
【摘要】目的建立一种快速、灵敏、特异的多重实时荧光RT-PCR方法,用于检测2019-nCoV。方法选取2019-nCoV ORF1ab和N基因的保守序列作为扩增靶区域,分别设计特异性引物及TaqMan探针,同时设计内标检测系统。通过条件优化建立多重实时荧光 RT-PCR检测体系,并进行准确性、重复性、特异性、和灵敏度检测;采用羧基荧光素(FAM)和绿色荧光蛋白(VIC)荧光基团标记目的探针,采用菁染料(Cy5)标记内标探针,实现多基因的同时检测。结果建立的多重实时荧光 RT-PCR方法具有较好的灵敏度和特异性,检测阳性样本的灵敏度为500 copies/mL,检测与感染部位相同或感染症状相似的其他病原体等无交叉反应。结论建立的多重实时荧光RT-PCR检测方法灵敏、特异、重复性好,可用于2019-nCov感染的快速检测。
2019新型冠状病毒(2019-novel Coronavirus, 2019-nCoV)因2019年武汉病毒性肺炎病例而被发现[1],2020年1月12日被世界卫生组织(World Health Organization, WHO)正式命名。此次中国武汉发现的2019-nCoV是一种先前尚未在人类中发现的新型冠状病毒,迄今为止共发现7种可感染人类的冠状病毒,包括:属于α属冠状病毒的HCoV-229E和HCoV-NL63和β属冠状病毒HCoV-OC43、SARS-CoV、HCoV-HKU1、MERS-CoV和2019-nCoV [2-8]。2019-nCoV具有较强的传染性,主要通过呼吸道飞沫传播,亦可通过接触传播,对公共卫生安全和人类的健康有很大的威胁。其感染的临床症状主要是发热,可合并干咳、乏力、呼吸不畅等症状,流涕、咳痰等其他症状少见[9],严重者快速进展为急性呼吸窘迫综合征(Acute Respiratory Distress Syndrome, ARDS)、脓毒症休克、难以纠正的代谢性酸中毒和出现凝血功能障碍[10]。随着疫情防控形势的变化,基于目前对新型冠状病毒感染的肺炎的病原、流行病学、临床特征等特点的认识,国家卫生健康委于2020年1月21日将新型冠状病毒感染的肺炎纳入法定传染病乙类管理,采取甲类传染病的预防、控制措施。截止2020年2月9日,中国已经累计报告确诊40171例2019新型冠状病毒病例,死亡病例为908例。日本、新加坡、泰国、韩国、澳大利亚、马来西亚、德国、越南、美国、法国、加拿大等国家也相继出现了疫情[11]。冠状病毒的实验室检测主要包括病毒核酸检测、抗原和抗体检测及作为病毒感染诊断“金标准”的病毒分离鉴定。其中核酸检测具有周期短、灵敏度高、易标准化等优势而广泛应用于病毒的病原学诊断工作[12]。在2019年的武汉疫情中,中国的研究人员采用高通量测序技术开展mNGS宏转录组测序分析,24小时内鉴定了2019-nCoV的相对丰度,并获取了全基因组序列及突变位点,为该病毒诊断工具及疫苗的研制奠定了重要基础[13]。基于TaqMan探针法的实时荧光PCR技术具有简单、快速、准确的优势,已广泛应用于临床病原体的检测,在2003年我国爆发的SRAS病毒的疫情控制方面做出重要贡献,是WHO推荐的MERS-CoV病毒检测方法之一,目前实时荧光RT-PCR技术已被列入我国2019-nCoV诊疗指南[14] ,国内已有数十家企业针对该病毒建立了体系进行检测,但仍然存在试剂供应不足,且检测结果准确性不佳,假阴性率严重等问题,市场亟需高质量的2019-nCoV核酸检测试剂以辅助临床医生对疑似患者进行快速确诊[15-18]。因此本研究针对ORF1ab和N基因,设计特异性引物及TaqMan探针对2019-nCoV进行双重检测,同时为提高试剂检测结果的准确性,采用人管家基因作为内参基因对样本质量进行监控,建立了新型冠状病毒2019-nCoV核酸特异的多重实时荧光RT-PCR检测方法。
一、 材料与方法
1.材料:
(1) 主要试剂引物、探针、热启动Taq酶、c-MMLV酶、阳性病毒样颗粒样本、核酸提取或纯化试剂盒(磁珠法)(粤穗械备20170583号和粤穗械备20150302号)均由中山大学达安基因股份有限公司提供,RNasin购自普洛麦格(Promega,北京)生物技术有限公司。
(2)主要仪器 Smart 32核酸提取仪由中山大学达安基因股份有限公司提供,ABI 7500实时荧光定量PCR仪购自美国Thermo Fisher Scientific公司。
2.方法:
(1)引物和探针的设计与合成根据全球流感序列数据库(GISAID)公布的2019-nCoV核酸序列,选取ORF1ab和N基因的保守区及人管家基因RNase P的保守区,采用Primer Premier 5.0软件分别设计3套引物对和3条特异性探针,并使用Primer blast (http://www.ncbi.nlm.nih.gov/tools/primer-blast/)进行评估。最终各基因分别确定1对引物和1条特异性探针,序列见表1(其余引物对和探针未列出)。

(2)阳性标准品的制备根据全球流感序列数据库(GISAID)公布的2019-nCoV核酸序列制备WH(2020)和WH-nCoV-N及HRP病毒样颗粒样本,并进行梯度稀释,采用数字PCR对标准品拷贝数进行标定和确认,将制备合格的标准品分装并于-20℃保存备用。
(3)实时荧光RT-PCR反应体系的建立采用105 U/L 热启动Taq酶、4×106 U/L c-MMLV酶、8×105 U/L RNasin、200 mM dNTPs、10 mM Tris-HCl (pH=8.3)、20 mM KCl、3.5 mM MgCl2配制预混反应液,与引物及探针进行荧光PCR体系配制,对引物和探针进行筛选,确认2019-nCoV各基因检测引物和探针,再将各基因检测引物和探针进行混合,配制成2019-nCoV多重实时荧光RT-PCR检测反应体系,混匀后于ABI 7500荧光定量PCR仪进行扩增,反应条件为:50℃,15 min;95℃,15 min;94℃,15 s;55℃,45s;共45个循环。
(4)准确性和重复性测试采用建立的多重实时荧光RT-PCR反应体系分别对5×106 copies/mL、5×104 copies/mL WH(2020)和WH-nCoV-N标准品进行批内和批间精密度试验,每个浓度重复测试3次,采用SPSS 16.0 软件计算Ct值的平均值、标准差和变异系数(CV),验证该检测体系的准确性和重复性。
(5)灵敏度测试分别将已定值的WH(2020)和WH-nCoV-N标准品以10倍差进行稀释,梯度浓度分别为1×106 copies/mL、1×105copies/mL、1×104 copies/mL、1×103 copies/mL、1×102 copies/mL,经优化好的多重实时荧光RT-PCR反应体系进行检测,初步测试该方法的灵敏度范围,再进一步测试确定灵敏度。
(6)特异性测试使用优化好的多重实时荧光RT-PCR反应体系对2019新型冠状病毒种属相近或引起症状相似的其他病原体样本进行检测,包括冠状病毒OC43、NL63、229E、HKU1、副流感病毒、甲型流感病毒、腺病毒、呼吸道合胞病毒、SARS冠状病毒、MERS冠状病毒,验证该检测体系的特异性。
二、结果及分析
1. 2019-nCoV检测体系的建立及优化:
(1)2019-nCoV单重实时荧光RT-PCR反应的建立:采用10倍差浓度梯度(1×108~1×104 copies/mL)的WH(2020)、WH-nCoV-N和HRP病毒样颗粒提取的核酸作为检测样本分别筛选两个目的基因及内参基因检测引物探针。分别确定一套相对较优的引物探针组合体系(表1),具有明显S形扩增曲线,且曲线光滑,Ct值较为合适,绝对荧光强度与背景荧光强度的差值(ΔRn)较大(图1)。因此确定ORF1ab、N及内参基因扩增单重体系。
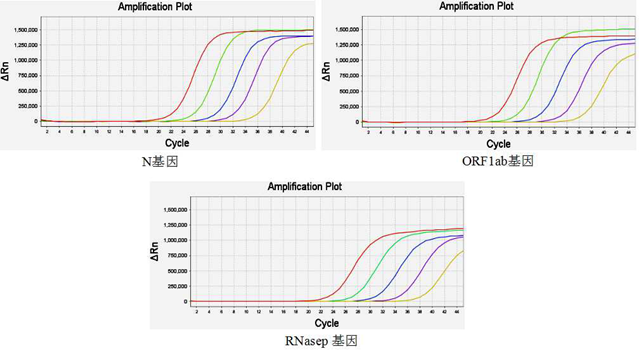
图1 单重实时荧光 RT-PCR反应扩增曲线
(2)2019-nCoV多重实时RT-PCR反应的建立:将通过单重实时荧光RT-PCR反应筛选出的2019-nCoV ORF1ab和N基因检测体系及内参基因检测体系进行混合,以1×106 copies/mLWH(2020)、WH-nCoV-N和HRP病毒样颗粒样本提取的核酸为模板进行检测,发现ORF1ab和N基因检测通道的荧光值明显高于内参基因检测通道(图2-A)。为了实现多重实时荧光RT-PCR中多个基因扩增效率的一致性,通过改变引物探针用量对多重实时荧光RT-PCR反应体系进行优化,最终获得较好的多重实时荧光RT-PCR扩增体系(图2-B)。
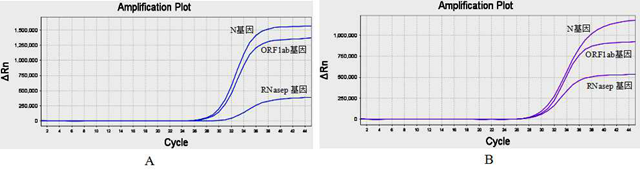
图2 多重实时荧光RT-PCR反应扩增曲线
2. 2019-nCoV多重实时荧光RT-PCR检测体系性能研究:
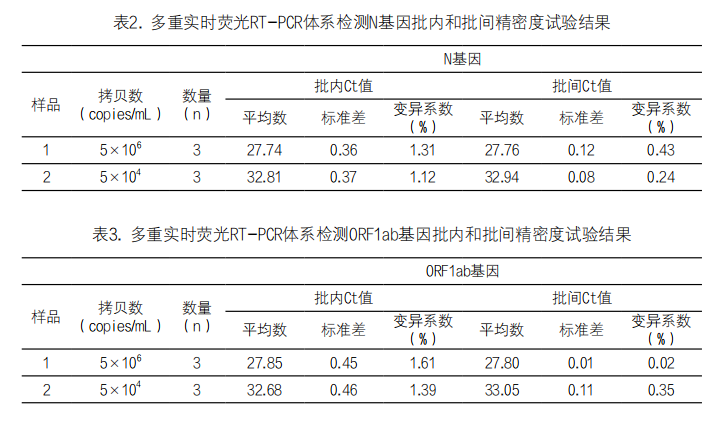
(1)多重实时荧光RT-PCR反应体系的准确性和重复性:使用2.1.2建立的多重实时荧光RT-PCR反应体系分别对不同浓度的WH(2020)和WH-nCoV-N标准品进行批内和批间精密度试验,每个浓度重复测试3次,统计计算批内和批间重复试验Ct值的变异系数CV均小于5%,见表2和表3。
(2)多重实时荧光RT-PCR反应体系的特异性:通过Blast进行同源性分析,多重体系的引物探针与其他冠状病毒和流感病毒等无同源性。使用2.1.2建立的多重实时荧光RT-PCR体系检测冠状病毒OC43、NL63、229E、HKU1、副流感病毒、甲型流感病毒、腺病毒、呼吸道合胞病毒、SARS冠状病毒及MERS冠状病毒核酸,同时以5×106 copies/mL的WH(2020)和WH-nCoV-N病毒样颗粒提取的核酸为阳性模板,并设置空白对照,结果只有阳性模板有正常扩增曲线,其它均无扩增,表明该检测体系具有较好的特异性(图3)。
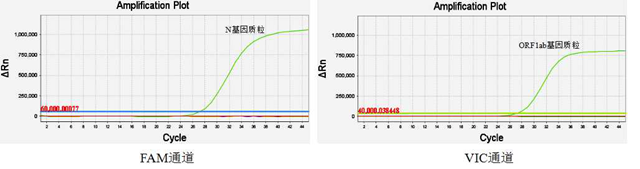
图3 多重实时荧光RT-PCR反应特异性扩增曲线
(3)多重实时荧光RT-PCR反应体系的灵敏度:使用10倍差梯度浓度(1×106~ 1×102 copies/mL)的病毒样颗粒提取的核酸作为模板进行多重实时荧光RT-PCR反应体系的灵敏度度测试,结果显示,体系检测1×102 copies/mL的样本ORF1ab基因检测通道(VIC)无荧光信号,N基因检测通道(FAM)虽然存在荧光信号和扩增曲线,但Ct值为40.5,检测1×103 copies/mL的样本,ORF1ab 检测通道(VIC)和N基因检测通道(FAM)均存在明显扩增曲线,且曲线形态良好(图4)。因此,采用1×104 copies/mL、1×103 copies/mL、500 copies/mL、200 copies/mL病毒样颗粒提取的核酸作为模板进一步测试,各进行20个重复测试,确定本研究建立的多重实时荧光RT-PCR体系的灵敏度为500 copies/mL(图5)。
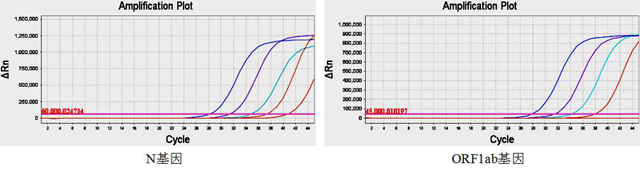
图4 多重实时荧光 RT-PCR体系灵敏度测试扩增曲线
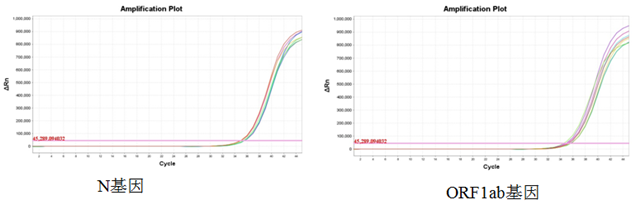
图5 多重实时荧光RT-PCR灵敏度确定扩增曲线
冠状病毒为不分节段的单股正链RNA病毒,易发生基因组的变异和重组,病毒颗粒呈圆形或椭圆形,直径50~200nm,具有包膜,包膜上有类似日冕的刺突[19]。人冠状病毒常引起呼吸道感染,该类病毒主要通过飞沫或直接接触分泌物传播,潜伏期一般为7天左右[19] ,因2003年出现的SARS病毒(SARS-CoV)导致感染者出现严重急性呼吸综合征而引起全球普遍关注。在SARS-CoV病毒发现后,又相继发现6种引起呼吸系统疾病的HCoV-229E、HCoV-NL63、HCoV-OC43、HCoV-HKU1、MERS-CoV冠状病毒及新发现的2019-nCoV[4-8],目前,β属新型冠状病毒2019-nCoV流行病学尚不完全清楚,针对该病毒建立核酸检测方法,对疾病诊断和后期防控具有重要意义。
病毒的实验室常用检测方法主要包括病毒分离培养[20-21]、血清学检测[22-23]及核酸检测[24-28]。病毒分离培养虽然为病毒检测的金标准,但存在检测周期较长,且部分病毒培养阳性率较低的问题。血清学检测存在灵敏度低、操作繁琐等缺陷,不利于疾病的早期诊断和治疗。核酸分子检测具有较高的特异性及灵敏度,已广泛应用于病毒包括冠状病毒的临床检测,主要包括PCR[24]、实时荧光PCR[25-26]、芯片[27]及高通量测序技术[28]等。PCR需结合电泳法对扩增产物进行分析,存在耗时较长、操作麻烦且容易污染等缺点。芯片技术虽然自动化程度高且检测通量高,但其价格昂贵,且重复性差难以广泛应用于临床检验。高通量测序技术在检测通量及挖掘病毒深层信息方面具有独特优势,然而,其数据分析难度高且价格昂贵等问题,大大限制了该技术在临床方面的推广及应用。而荧光PCR技术操作简单、结果直观、具备较高敏感度、特异性、准确性和重复性且检测成本较低等优势,已广泛应用于临床呼吸道病原体、肠道病原体及性病病原体等常见临床感染性疾病的诊断,是我国新型冠状病毒疑似感染者推荐的确诊方式之一。
本研究根据全球流感序列数据库(GISAID)公布的2019-nCoV核酸序列,选择ORF1ab和N基因两个基因作为检测靶标,对其保守区进行检测,而目前市场上部分企业已建立的方法仅对ORF1ab基因进行检测,相比较而言,本研究可对新型冠状病毒的检测进行双重把控,检测结果更为可信。同时,本研究建立的方法选择人管家基因RNase P的保守区作为内参基因进行检测,可对检测中样本的采集及核酸提取过程进行监控,可有效避免因样本质量问题导致的假阴性结果的出现。然而,市场上大部分新型冠状病毒相关核酸检测产品并未采用人源性的内参基因对样本质量进行监控,这可能是导致目前新型冠状病毒核酸检测阳性率低的重要原因之一。通过性能研究分析发现,本研究建立的2019-nCoV检测试剂,灵敏度可达500 copies/mL,而已报道的传统实时荧光PCR灵敏度为1000 copies/mL[29-30],此外,目前市场上已公布的2019-nCoV检测产品大部分产品检测灵敏度亦为1000 copies/mL,因此本研究建立的多重实时荧光RT-PCR体系在检测灵敏度方面具有显著优势。检测试剂的灵敏度是影响试剂盒检测准确性的重要因素之一,灵敏度越高,低浓度样本被正确检出的几率越高,则假阴性率越低。但灵敏度高的同时意味着特异性可能会受影响,如何在保证试剂具有较高灵敏度的同时,特异性能满足临床需求是衡量检测试剂成功与否的重要指标。本研究建立的2019-nCoV多重实时荧光RT-PCR体系,在具有较高灵敏度的同时,经特异性分析发现具备良好特异性,与其余6种冠状病毒及其他呼吸道常见病原体均无交叉反应,其中SARS病毒与本病毒序列同源性达80%,亦能准确区分。因此,本研究所建立的多重实时荧光RT-PCR检测方法具有特异性强、灵敏度高等优点,在检测2019-nCoV时,采用人源性内标对样本质量进行监控,是一种更为准确可靠的2019-nCoV病毒核酸检测方法,可用于2019-nCoV的快速诊断,为新型冠状病毒(2019-nCoV)的预防和流行病学研究奠定了基础。
参考文献
[1]A Novel Coronavirus from Patients with Pneumonia in China, 2019[J].N Engl J Med. 2020 Jan 24. doi: 10.1056/NEJMoa2001017. [Epub ahead of print]
[2]陈嘉源,施劲松,丘栋安等. 武汉2019冠状病毒基因组的生物信息学分析[J]. 中国生物信息学杂志, 2019. DOI:10.12113/202001007
[3]Zumla, A., Chan, J., Azhar, E. et al. Coronaviruses — drug discovery and therapeutic options. Nat Rev Drug Discov 15, 327–347 (2016) doi:10.1038/nrd.2015.37.
[4]Lu R J , Zhang L L , Tan W J , et al. Characterization of human coronavirus 229E infection among patients with respiratory symptom in Beijing, Oct-Dec, 2007[J]. Chinese journal of experimental and clinical virology, 2009, 23(5):367-370.
[5]Esper F , Weibel C , Ferguson D , et al. Coronavirus HKU1 Infection in the United States[J]. Emerging Infectious Diseases, 2006, 12(5):775-779.
[6]Guan, Y. Isolation and Characterization of Viruses Related to the SARS Coronavirus from Animals in Southern China[J]. Science, 2003, 302(5643):276-278.
[7]Chafekar A , Fielding B C . MERS-CoV: Understanding the Latest Human Coronavirus Threat[J]. Viruses, 2018, 10(2):93-.
[8]Kamau E, Luka MM, de Laurent ZR, Adema I, Agoti CN, Nokes DJ. Genome Sequences of Human Coronavirus OC43 and NL63, Associated with Respiratory Infections in Kilifi, Kenya[J]. Microbiol ResourAnnounc. 2019 Nov 14;8(46). DIO: 10.1128/MRA.00730-19.
[9]Qun Li, M.Med., Xuhua Guan, Ph.D., et al. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia. DOI: 10.1056/NEJMoa2001316.
[10]史河水,韩小雨,樊艳青等.新型冠状病毒(2019-nCoV)感染的肺炎临床特征及影像学表现[J/OL].临床放射学杂志:1-8[2020-02-06].https://doi.org/10.13437/j.cnki.jcr.20200206.002.
[11]Michelle L. Holshue, M.P.H., Chas DeBolt, M.P.H., Scott Lindquist, M.D., et al. First Case of 2019 Novel Coronavirus in the United States. DOI: 10.1056/NEJMoa2001191.
[12]王佶. 病原体核酸检测新技术的建立及应用[D].中国疾病预防控制中心,2018.
[13]Chen L. RNA based mNGS approach identifies a novel human coronavirus from two individual pneumonia cases in 2019 Wuhan outbreak. Emerg Microbes Infect. 2020 Dec;9(1):313-319. doi: 10.1080/22221751.2020.1725399.
[14] 国家卫生健康委办公厅.《新型冠状病毒肺炎诊疗方案》(试行第五版 修正版).
[15]Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR.Euro Surveill. 2020 Jan;25(3). doi:10.2807/1560-7917.ES.2020.25.3.2000045.
[16]郭杨,陈世界,郭万柱等.荧光定量PCR技术及其应用研究进展[J].动物医学进展,2009, 30 (2):78-82.
[17]CormanVM, Eckerlei, BleickerT, etal. Detection of a novel human corona virus by real-time reverse-transcription polymerase chain reaction[J].Euro Surveil, 2012, 17(39): 20285.
[18]CormanVM, MulerMA, CostabelU, etal. Assaysforlabora-toryconfirmation of novel human corona virus(h-CoV-EMC) infection[J].Euro Surveil, 2012, 17(49): 20334.
[19]Sun SF, Wang YY, Liu LS, et al. Summary of coronavirus[J]. China AnimalHealth Inspection, 2013, 30(6):68-71.
[20] 鲍琳琳,涂新明, 蒋虹,等.SARS冠状病毒分离培养和鉴定的实验研究[J]. 病毒学报, 2005, 1(21):31-34.
[21] 江丽芳,赵卫,方丹云,等.SARS冠状病毒的分离培养与鉴定[J]. 中国病毒学, 2003, 18(6):544-547.
[22]Okba NMA, Widjaja I, GeurtsvanKessel CH, etal. Sensitive and Specific Detection of Low-Level Antibody Responses in Mild Middle East Respiratory Syndrome Coronavirus Infections[J]. Emerg Infect Dis, 2019, 25(10):1868-1877.
[23]More GD, Dunowska M, Acke E, etal. A serological survey of canine respiratory coronavirus in New Zealand[J]. N Z Vet J, 2020, 68(1):54-59.
[24]Greninger AL, Pepper G,Shean RC, etal.Myeloablation-associated deletion of ORF4 in a human coronavirus 229E infection[J]. NPJ Genom Med, 2017, 2:30.
[25]Corman VM, Landt O, Kaiser M, etal. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR[J]. Euro Surveill, 2020, 25(3).
[26]Shirato K, Nao N, Matsuyama S, etal. An ultra-rapid real-time RT-PCR method for detecting Middle East respiratory syndrome coronavirus using a mobile PCR device, PCR1100[J]. Jpn J Infect Dis, 2019.
[27]Yan Y, Luo JY, Chen Y, etal. A multiplex liquid-chip assay based on Luminex xMAP technology for simultaneous detection of six common respiratory viruses [J]. Oncotarget, 2017, 8(57):96913-96923.
[28]Li B, Si HR, Zhu Y, etal. Discovery of Bat Coronaviruses through Surveillance and Probe Capture-Based Next-Generation Sequencing[J]. mSphere, 2020, 5(1):pii: e00807-19.
[29] Elpaeva E A , Komissarov A B , Pisareva M M , et al. NEW METHOD FOR DETERMINING HEPATITIS B VIRUS RESISTANCE MUTATIONS M204I/V TO NUCLEOS(T)IDE ANALOGUES IN PATIENTS WITH CHRONIC HEPATITIS B[J]. Infekciâ I Immunitet, 2015, 5(3):265-272.
[30] Dong-Ying X , Bing-Liang L , Qi-Huan X U , et al. The predictive value of bsaeline HBeAg levels to the efficacy of 52 weeks treatment of adefovir dipivoxil in patients with HBeAg-positive chronic hepatitis B[J]. Chinese Journal of Clinicians(Electronic Edition), 2010, 4(8).


收稿日期:2020-02-10