【摘要】成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)是一种广泛应用于基因编辑的新兴技术,拥有附属切割活性的Cas蛋白的发现,促进了快速便携式检测工具的开发,为即时检测提供了新的研究方向。本文阐述了CRISPR检测反应原理,CRISPR-Cas系统的不同反应模式,以及对不同应用对象及结果输出方式,并对比分析了国内外相关专利和产业化情况,最后对该技术前景进行了展望。
【关键词】CRISPR-Cas技术;Cas蛋白;即时检测;核酸
新冠疫情防控凸显了核酸检测的重要性,并加速了即时诊断技术的研发。逆转录聚合酶链反应(RT-PCR)的核酸检测方法被视为金标准,但也存在实验步骤繁琐、依赖昂贵的基础设施和训练有素的实验人员等,难以满足现场检测的需求。近年来,CRISPR-Cas系统的出现为即时检测的发展开辟了新的方向。
1987年Nakata教授[1]在研究大肠杆菌时首次发现含有29个碱基的重复序列,但当时的研究并未发掘其生物学意义。2002年由Jansen[2]正式将其命名为成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)。随后,经过多年研究,最终由Oost团队[3]和Charpentie团队[4]补充crRNA及tracrRNA,完善了该系统功能的机制成分。自2016年张锋团队[5]发现具有附属切割能力的Cas13a后,CRISPR Cas系统逐渐在基因编辑功能以外的即时检测方面也大放异彩[6]。
目前,对于CRISPR Cas系统检测的综述性文章主要报道了病毒等核酸检测进展,对于该系统的其他应用对象及产业化方面鲜有报道。因此,本文综述了CRISPR-Cas系统丰富的检测对象及多样的结果呈现方式,并总结了其产业化情况。
一、CRISPR检测原理和主要反应模式
1. 检测反应原理:CRISPR-Cas系统是微生物体内的适应性免疫系统,它协助微生物抵抗核酸入侵,主要成分包括成熟crRNA(CRISPR RNA)及Cas效应蛋白。其工作原理主要分为三个部分,即适应、crRNA成熟及干扰[7](图2)。适应,即该系统适应性免疫作用的过程,是指当Cas1-Cas2蛋白复合物识别PAM(protospacer adjacent motif)序列后,对外源DNA进行切割,并整合到CRISPR基序中[8]。crRNA成熟即是当记忆后的外源DNA再次进入机体后,CRISPR序列转录成前体CRISPR RNA(pre-crRNA),在体内RNA内切酶的作用下加工成为成熟crRNA。最后,成熟crRNA与Cas蛋白形成复合物,其中crRNA发挥向导作用,指导Cas蛋白发挥剪切能力,完成干扰作用。
根据不同Cas效应蛋白的差异,目前,CRISPR-Cas系统可分为两大类,第一类系统干扰作用由多个Cas蛋白复合物执行,包含I型、III型和IV型;第二类系统由单个效应器实行干扰作用,包括II型、V型和VI型[9]。由于第二类系统的简便性,多种途径的应用均取自第二类系统。其中,Cas12及Cas13蛋白具有附带切割性能[5, 10],两者均可作为信号放大器,因此广泛应用于核酸检测中。而两者的主要区别在于,Cas12的检测对象为DNA,而Cas13的检测对象为RNA[11]。
2. 依赖扩增的CRISPR检测:CRISPR Cas系统的直接检测灵敏度并不高,通常需要对目标序列扩增放大,多种扩增手段与CRISPR Cas系统联合使用,为即时检测提供了新的思路。目前CRISPR检测信号放大方式一般采用聚合酶链式反应或等温扩增技术,通过扩增使得目标核酸指数级增长从而提高检测灵敏度。
第一个CRISPR-Cas检测系统是由张锋[12]团队开发的SHERLOCK(specific high-sensitivity enzymatic reporter unlocking)检测平台(图1)。在此检测系统中,重组酶聚合酶扩增(recombinase polymerase amplification,RPA)被用作第一层放大过程;随后的T7体外转录可令RNA大量增多,为第二层放大;最后触发的Cas13a附属切割可剪切体系中大量的报告RNA,完成第三层放大。这些放大方法的组合叠加使其能够在1-2h内检测aM级别的核酸。另一种等温扩增方式为环介导等温扩增(loop-mediated isothermal amplification,LAMP),2020年,Chen等人[13]应用cas12a与LAMP相结合,40min即可达到20拷贝的检测灵敏度。Hou等人[14]通过滚环扩增(rolling circle amplification,RCA)与CRISPR Cas12a联合使用,检测PIK3CA H1047R的SNV基因,检测限为10aM,有助于乳腺癌的早期诊断。此外,还有基于聚合酶链式反应(PCR)[15]、链置换扩增(SDA)[16]及核酸序列的扩增(NASBA)[17]等的方法,研究表明这些扩增方法均可与CRISPR Cas系统串联,发挥高效扩增,进一步提高反应灵敏度。
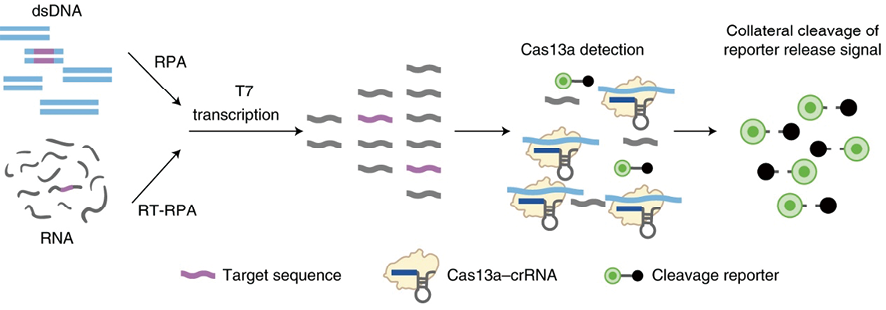
图1. SHERLOCK检测示意图[18]
3. 无需扩增的检测:然而,扩增也会带来一些负面影响,如扩增偏倚、交叉污染、仪器依赖、操作时间长等问题。Doudna等人[19]提出CRISPR核酸酶串联检测方法,通过化学激活剂将RNA引导的Cas13与Csm6结合,用于微流控芯片中检测鼻咽拭子中的新冠病毒RNA,20分钟内检测每微升RNA约30个分子。这种方法无需核酸扩增,简化操作,减少扩增污染,降低检测成本,同时串联核酸酶,提高酶识别效率。Fozouni等人[20]提出一种用于直接检测SARS-CoV-2鼻拭子RNA的无扩增CRISPR-Cas13a方法,该方法可以用手机显微镜读取,在30分钟内达到100copies/μL的灵敏度。
Choi等人[21]提出一种CRISPR介导的表面增强拉曼光谱技术,对病毒DNA进行无扩增检测。将修饰单链DNA拉曼探针的功能化金纳米颗粒集成在氧化石墨烯/三角形金纳米花阵列上(graphene oxide/triangle Au nanoflower,GO-TANF),当靶标基因存在时,Cas12a蛋白在crRNA引导下结合靶标并激活非特异性切割单链基因活性,即切割GO-TANF阵列上的单链DNA拉曼探针,导致阵列上的表面增强拉曼光谱(surface-enhanced Raman spectroscopy,SERS)信号降低。该法提高了乙型肝炎病毒、人乳头瘤病毒16和人乳头瘤病毒18等多病毒DNA的检测灵敏度,检测限在1aM至100pM范围内。Tian等人[22]通过使用一个简单的液滴微流控平台,在皮升体积的液滴中进行Cas13a分析,发现从单个RNA靶标激活的Cas13a反式切割中积累的荧光信号足以照亮1pL大小的液滴,从而实现无核酸扩增的数字化单分子水平的RNA定量。
二、CRISPR检测结果输出方式
2016年,Collins等人[17]结合等温RNA扩增,CRISPR/Cas9以及纸基传感器技术成功实现寨卡病毒的分型鉴定,其中Cas9蛋白优异的识别能力和切割活性,是精确区分寨卡病毒亚型的关键。这是CRISPR/Cas技术首次应用于核酸检测中,表现出高灵敏、高特异、简单快速的优点,近年来在病毒,细菌等生物检测领域发展势头迅猛。
1. 荧光分析法:荧光分析法通过检测荧光信号达到检测目的,是CRIPSR/Cas检测系统最常用的输出信号[23]。该法具有特异性高、受非荧光物质干扰小、对痕量分析物选择性高、反应快速可视化等优点[24, 25]。检测灵敏度可到微摩尔至纳摩尔级,但有设备依赖性且试剂成本较高[26]。2018年,Doudna等人[27]提出DNA内切酶靶向CRISPR反式报告基因(DNA endonuclease-targeted CRISPR trans reporter,DETECTR)的方法。并先后开发了基于CRISPR-Cas12a的DETECTR法检测人乳头瘤病毒诊断和基于CRISPR-Cas14的DETECTR法检测单核苷酸多态性基因分型,这些检测方法利用了Cas12和Cas14非特异性切割单链核酸分子特性,切割荧光报告分子,提供可检测的荧光信号。2021年,Curti等人[28]评估了一种基于逆转录、等温扩增和CRISPR-Cas12反应的冻干CRISPR-Cas12检测SARS-CoV-2试剂盒(Lyo-CRISPR SARS-CoV-2试剂盒)。在收集鼻咽拭子样本后先进行逆转录等温扩增,扩增产物再被投入到CRRISPR/Cas12反应体系中,Cas12蛋白在sgRNA引导下结合靶标核酸并激活Cas12非特异性切割荧光单链DNA报告分子。2021年,Liu等人[29]建立了一种高灵敏度检测非洲猪瘟病毒的荧光检测方法。他们将逆转录-重组酶聚合酶扩增技术耦合Cas12a系统与光学荧光淬灭报告基因,实现单管25min内等温可视化检测非洲猪瘟病毒,检测灵敏度可到单拷贝且与其他猪病毒无交叉反应。
2021年,Wang等人[30]开发了一种超敏碱性磷酸酶(ALP)检测方法,通过靶点诱导的转录扩增来触发Cas13a(TITAC-Cas)反式切割荧光报告分子,通过T7启动子/模板将ALP活性信号转化为RNA转录的扩增信号,省去制备激活Cas13a的化学分子探针,检测过程简单快速、高灵敏、高特异性,拓宽了CRISPR/Cas13a测量非核酸靶标应用范围。现有的CRISPR/Cas-荧光检测技术虽然具有超灵敏和高特异性,但比较依赖复杂的仪器及特殊的试剂,需要专业的人员和洁净的场地进行操作。
2. 电化学分析法:电化学分析法通过检测因生物或化学反应而产生的电信号而达到检测目的。电化学生物传感器具有选择性好、分析速度快、成本低、检测灵敏度高等优点,已广泛应用于医学诊断领域[31-33]。另外电化学生物传感器可制作成便携式检测仪器,可实现多靶标检测和多场景应用[34]。Dai等人[31]提出一种基于CRISPR-Cas12a(cpf1)的电化学生物传感器,在成本效益和便携性上均优于荧光生物传感器,用于人类乳头瘤病毒16和细小病毒B19检测,灵敏度达到皮摩尔级。如图2所示,在crRNA引导下,Cas12a结合靶标核酸同时激活非特异性切割单链DNA活性。在金电极上固定亚甲基蓝标记的单链DNA报告分子,在靶标存在的情况下,Cas12a-crRNA会剪切非特异性单链DNA报告基因,导致金电极上失去亚甲基蓝后产生电流变化。Xu等人[35]比较了Cas9和Cas12a增强的电化学DNA传感器的性能,并优化了CRISPR反应条件,在不进行酶放大的情况下实现了细小病毒B19飞摩尔级检测。
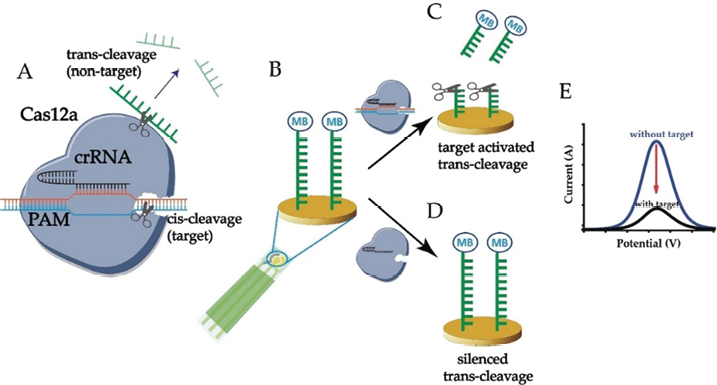
图2. 基于CRISPR-Cas12a(cpf1)的电化学生物传感器原理[31]
3. 比色分析法:比色分析通过敏感的颜色变化实现对目标物的识别,无需精密仪器,检测结果直观,非常适合现场检测与分析。Mukama等人[36]报道了一种基于CRISPR/Cas12的核酸检测横向流动生物传感器,可特异检测到单拷贝克隆铜绿假单胞菌(1cfu/ml)。首先样本经等温扩增和CRISPR/Cas12反应,反应后溶液进行侧流免疫层析检测。在有靶标DNA时,溶液中生物素修饰的报告探针被剪切,无法与层析条上链霉亲和素修饰的胶体金结合,进而在横向流动时报告探针与检测线(T线)互补探针结合后无显色;在无靶标DNA时,溶液中生物素修饰的报告探针不被剪切,与层析条上链霉亲和素修饰的胶体金结合,在流经T线时与互补探针结合显色。因此未裂解的ssDNA报告基因的数量可以用肉眼或便携式读取器检测。检测方法简单、廉价、超灵敏。Wang等人[37]提出了一种CRISPR/Cas9介导的侧流核酸测定分析,可以用肉眼或便携式读取器检测信号强度,检测限低至数百拷贝。由于AuNP-DNA探针与sgRNA茎环区的一个通用序列杂交,因此AuNP-DNA探针是通用的,与AuNP-DNA探针预组装的横向流动装置可以用于检测任何靶基因[38]。
4. 倏逝波分析法:光纤倏逝波传感器是依据倏逝波原理制作,生物分子可以吸附在纤芯表面,导致输出光的能量发生变化,输出光特性不同反映了被测物质的不同特性[39]。Koo等人[40]报告了一种CRISPR/dCas9介导的生物传感器,将核酸酶失活的Cas9(dCas9)和单微环谐振器生物传感器耦合,实现无标记实时检测致病DNA和RNA。Yang等人[41]报道了一种基于CRISPR的快速(<1h)、易于实现、准确检测SARS-CoV-2的倏逝波荧光生物传感平台。将Cas13a的间接效应与通用自主无酶杂交链反应(hybridization chain reaction,HCR)相结合,检测限可达nM或pM水平,其良好的识别能力显示出其在COVID-19即时诊断方面的潜力。倏逝波生物传感器具有操作简单,灵敏度高,实时监测的优点,在病原体检测方面有很大发展潜力。
三、CRISPR系统的主要检测对象
由于CRISPR Cas系统检测的强特异性与高灵敏度,随着检测技术及手段的提升,包括病毒、细菌、突变等多种检测对象都可应用CRISPR Cas系统进行分析。表1比较了为不同检测对象开发的CRISPR Cas系统。
表1. 基于CRISPR Cas系统的不同靶标检测
|
检测系统 |
Cas酶 |
靶标 |
参考文献 |
|
CREST |
Cas13a |
新冠病毒 |
[42] |
|
AIOD-CRISPR |
Cas12a |
新冠病毒 |
[43] |
|
SHERLOCK |
Cas13a |
寨卡、登革热病毒 |
[12] |
|
DETECTE |
Cas12a |
人乳头瘤病毒 |
[24] |
|
CRISPR-Cas-13a |
Cas13a |
miRNA |
[44] |
|
HOLMESv2 |
Cas12b |
SNP、甲基化、病毒 |
[45] |
|
APC-Cas |
Cas13a |
沙门氏菌 |
[46] |
|
MXene-CRISPR-Cas12a |
Cas12a |
脂多糖,革兰氏阴性菌 |
[47] |
|
CaT-SMelor |
Cas12a |
人血清,尿酸,对羟基苯甲酸 |
[48] |
1. 新冠病毒和其他病原体:2020年,Liu等人[43]提出了基于CRISPR Cas12a的超灵敏、视觉检测新冠技术。通过靶向SARS-CoV-2的核蛋白基因,该方法引入了两种没有PAM位点限制的CRISPR RNA,以高效驱动系统活性,开发双重CRISPR-Cas12a的检测。该方法的检测结果可以通过肉眼直接可视化,检测限为5个拷贝的病毒靶标,大大简化了检测过程。
除了核酸检测平台,为进一步扩展CRISPR检测的通用性,有研究将CRISPR-Cas系统与适配体联用,增加检测对象[49]。2021年,Zhang等[47]报道了一种基于MXene与CRISPR-Cas12a结合检测脂多糖(LPS)和细菌的高灵敏检测平台,该方法对LPS及革兰氏阴性菌的检测限可以到达11pg/mL和23CFU/mL。
2. 遗传病和肿瘤相关SNP,miRNA:由于CRISPR-Cas系统灵敏、高效的检测性能,多项研究开始着力于应用CRISPR-Cas系统检测作为癌症早期临床诊断的潜在生物标志物miRNA,实现该系统与临床检测的接轨。2019年,Xing等[44]基于Cas13a识别RNA并激活附属切割的特性,将其直接应用于miRNA的检测。当目标miRNA激活Cas13a后,附属切割特性将切割报告探针,释放大量荧光信号,具有单碱基分辨能力,可在30分钟内实现低至4.5amol的检测限(LOD)。Wang等[45]通过使用嗜热CRISPR-Cas12b,创建了HOLMES v2检测平台,可区分单核苷酸多态性(SNP)、简单检测病毒RNA、人细胞mRNA及环状RNA,同时结合Cas12b检测和亚硫酸氢盐处理,可以准确定量目标DNA甲基化程度。
3. 小分子检测:除了上述目标核酸,CRISPR-Cas系统也可用于多种小分子的检测。2019年,Zhang等[48]结合CRISPR-Cas12a的DNA切割能力与细菌变构转录因子(aTF)对小分子和DNA的竞争性结合活性,开发了简单、超灵敏及高通量的小分子检测平台CaT-Smelor,通过对多种嘌呤化合物和尿酸的测试证实了该检测平台的特异性。同时,通过与高效液相色谱(HPLC)相对比,确定该方法对分析样品尿酸的结果与生化分析仪器相同。
四、CRISPR检测技术的产业化
CRISPR/Cas检测系统在医疗卫生、生物科学、农业技术以及环境监测等各大领域中有着广泛的应用前景,这一情况可通过对国内外相关专利和产品的分析中得知。
1. 国内外代表性专利:使用“专利之星数据库”(https://cprs.patentstar.com.cn/Search/Index)对中国公开CRISPR技术专利的全部检索,发现自2013年以来该技术的应用次数持续增长,尤其在2015年之后上升的尤为明显。又使用“全球专利检索数据库”(https://www.iprdb.com/),将“CRISPR detect*”作为关键词,将时间(截止至2021年10月)和IPC(小分类)进行编辑,对全球不同国家的CRISPR专利分布进行探索,如表2所示。美国和中国是该技术的两个重要研发和应用国家,同时也是全球CRISPR技术竞争的两大主场。而欧盟、日本、韩国的技术手段虽然没有以上两个国家多,但是总体上也是呈现上升的趋势。此外我们对CRISPR的重点研究领域进行剖析,发现了该技术主要是集中于微生物、酶及其组合物方向。
表2. CRISPR技术全球分类
(检索关键词为CRISPR+detect*,WIPO为世界知识产权组织)
|
分类 |
WIPO |
美国 |
中国 |
欧盟 |
日本 |
韩国 |
|
微生物、酶,或其组合物 |
304 |
246 |
165 |
101 |
52 |
24 |
|
包含酶或微生物的测定或检验方法 |
188 |
171 |
129 |
81 |
44 |
28 |
|
医用、牙科用或梳妆用的配制品 |
121 |
83 |
19 |
30 |
21 |
5 |
|
化学或物理分析 |
70 |
60 |
23 |
25 |
17 |
9 |
|
肽 |
67 |
34 |
20 |
24 |
10 |
0 |
|
化合物或药物制剂的特定治疗 |
46 |
28 |
15 |
8 |
20 |
0 |
|
植物相关 |
24 |
33 |
21 |
7 |
6 |
4 |
|
畜牧业相关 |
15 |
19 |
20 |
3 |
5 |
2 |
|
微生物之C12C至C12Q小类相关 |
2 |
1 |
31 |
1 |
0 |
0 |
|
其他 |
8 |
21 |
1 |
4 |
1 |
0 |
2. 产业化情况:通过基因编辑和改造能够快速方便的检测和治疗临床上难以解决的疾病,成为新的研发热点。知名生物学家张峰博士和诺贝尔奖获得者Jennifer Doudna共同开创了Editas Medicine公司,主要发展方向是基于CRISPR技术来研究临床上治疗方法有限的一些重大疾病;该公司和艾尔建(Allergan)公司共同发明的CRISPR疗法AGN-151587(EDIT-101)实现了世界上治疗莱伯氏先天性黑蒙症的首例患者体内给药。从哈佛大学走出来的公司eGenesis主要致力于通过CRISPR技术生产可供人类移植的各种器官。而Caribou BioScience也是美国基于CRISPR方法在血液病和肿瘤治疗方面的公司。CRISPR Therapeutics是由Emmanuelle和其他同伴在瑞士创立的,主要致力于研发有关于CRISPR-Cas9的基因治疗药物,如:CTX001是一种基于CRISPR的造血干细胞疗法,产品CTX110则是应用于肿瘤免疫的治疗领域。国内,北京博雅基因生物科技有限公司(EdiGene)在2017年首次取得了中国CRISPR专利的使用权。军事医学研究院联合杭州众测公司获得国内第一个基于CRISPR的新冠病毒检测注册证书。上海的伯杰医疗科技公司创建了基于CRISPR的恒温新冠病毒核酸检测试剂盒和核酸扩增的恒温仪器,缩短了病毒检测的时间(30分钟内),于2021年9月份由国家药监局批准上市。
五、总结与展望
CRISPR-Cas系统可以通过与多种实验技术联用,在各个检测方向上发挥其简便、灵敏、高特异性的优点。该系统除了检测病原体外,还可检测遗传基因、小分子定量等目标对象。关于CRISPR的专利及产业化公司以病原检测或基因治疗药物为主,主要实验对象仍是核酸,对于非核酸的相关检测并未有深入进展。但相关实验研究已经证实,CRISPR-Cas系统还有更多的应用发展方向值得深入探索。
参考文献
[1]Ishino Y, Shinagawa H, Makino K et al: Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. Journal of bacteriology, 1987, 169(12): 5429-5433.
[2]Jansen R, van Embden JDA, Gaastra W et al: Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43(6): 1565-1575.
[3]Brouns SJJ, Jore MM, Lundgren M et al: Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891): 960-964.
[4]Deltcheva E, Chylinski K, Sharma CM et al: CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J]. Nature, 2011, 471(7340): 602-+.
[5]Abudayyeh OO, Gootenberg JS, Konermann S et al: C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299).
[6]Chen S, Yao Y, Zhang Y et al: CRISPR system: Discovery, development and off-target detection[J]. Cellular Signalling, 2020, 70.
[7]Wang Z, Cui W: CRISPR-Cas system for biomedical diagnostic platforms[J]. View, 2020, 1(3): 20200008.
[8]Wang H, La Russa M, Qi LS: CRISPR/Cas9 in Genome Editing and Beyond. In: Annual Review of Biochemistry, Vol 85. Volume 85, edn. Edited by Kornberg RD; 2016: 227-264.
[9]Makarova KS, Wolf YI, Iranzo J et al: Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants[J]. Nature Reviews Microbiology, 2020, 18(2): 67-83.
[10]Li S-Y, Cheng Q-X, Liu J-K et al: CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA[J]. Cell Research, 2018, 28(4): 491-493.
[11]Wang M, Zhang R, Li J: CRISPR/cas systems redefine nucleic acid detection: Principles and methods[J]. Biosensors & Bioelectronics, 2020, 165.
[12]Gootenberg JS, Abudayyeh OO, Lee JW et al: Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-+.
[13]Chen Y, Shi Y, Chen Y et al: Contamination-free visual detection of SARS-CoV-2 with CRISPR/Cas12a: A promising method in the point-of-care detection[J]. Biosensors & Bioelectronics, 2020, 169.
[14]Cao G, Chen X, Deng Y et al: Single-nucleotide variant of PIK3CA H1047R gene assay by CRISPR/ Cas12a combined with rolling circle amplification[J]. Analytica Chimica Acta, 2021, 1182.
[15]Zhang M, Liu C, Shi Y et al: Selective endpoint visualized detection of Vibrio parahaemolyticus with CRISPR/Cas12a assisted PCR using thermal cycler for on-site application[J]. Talanta, 2020, 214.
[16]Wang X, Wang X, Shi C et al: Highly sensitive visual detection of nucleic acid based on a universal strand exchange amplification coupled with lateral flow assay strip[J]. Talanta, 2020, 216.
[17]Pardee K, Green AA, Takahashi MK et al: Rapid, Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components[J]. Cell, 2016, 165(5): 1255-1266.
[18]Kellner MJ, Koob JG, Gootenberg JS et al: SHERLOCK: nucleic acid detection with CRISPR nucleases[J]. Nature Protocols, 2019, 14(10): 2986-3012.
[19]Liu TY, Knott GJ, Smock DCJ et al: Accelerated RNA detection using tandem CRISPR nucleases[J]. Nature chemical biology, 2021, 17(9): 982-988.
[20]Fozouni P, Son S, Diaz de Leon Derby M et al: Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy[J]. Cell, 2021, 184(2): 323-333 e329.
[21]Choi JH, Shin M, Yang L et al: Clustered Regularly Interspaced Short Palindromic Repeats-Mediated Amplification-Free Detection of Viral DNAs Using Surface-Enhanced Raman Spectroscopy-Active Nanoarray[J]. ACS nano, 2021.
[22]Tian T, Shu B, Jiang Y et al: An Ultralocalized Cas13a Assay Enables Universal and Nucleic Acid Amplification-Free Single-Molecule RNA Diagnostics[J]. ACS nano, 2021, 15(1): 1167-1178.
[23]Xie S, Ji Z, Suo T et al: Advancing sensing technology with CRISPR: From the detection of nucleic acids to a broad range of analytes-A review[J]. Analytica Chimica Acta, 2021: 338848.
[24]Medintz IL, Uyeda HT, Goldman ER et al: Quantum dot bioconjugates for imaging, labelling and sensing[J]. Nature materials, 2005, 4(6): 435-446.
[25]Zhu S, Meng Q, Wang L et al: Highly photoluminescent carbon dots for multicolor patterning, sensors, and bioimaging[J]. Angewandte Chemie (International ed in English), 2013, 52(14): 3953-3957.
[26]Spoelstra WK, Jacques JM, Gonzalez-Linares R et al: CRISPR-based DNA and RNA detection with liquid-liquid phase separation[J]. Biophys J, 2021, 120(7):1198-1209.
[27]Chen JS, Ma E, Harrington LB et al: CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity[J]. Science (New York, NY), 2018, 360(6387): 436-439.
[28]Curti LA, Primost I, Valla S et al: evaluation of a Lyophilized CRISPR-Cas12 Assay for a Sensitive, Specific, and Rapid Detection of SARS-CoV-2[J]. Viruses, 2021, 13(3).
[29]Liu S, Tao D, Liao Y et al: Highly Sensitive CRISPR/Cas12a-based Fluorescence Detection of Porcine Reproductive and Respiratory Syndrome Virus[J]. ACS Synth Biol, 2021.
[30]Wang X, Zhou S, Chu C et al: Target-induced transcription amplification to trigger the trans-cleavage activity of CRISPR/Cas13a (TITAC-Cas) for detection of alkaline phosphatase[J]. Biosens Bioelectron, 2021, 185: 113281.
[31]Dai Y, Somoza RA, Wang L et al: Exploring the Trans-Cleavage Activity of CRISPR-Cas12a (cpf1) for the Development of a Universal Electrochemical Biosensor[J]. Angewandte Chemie (International ed in English), 2019, 58(48): 17399-17405.
[32]Wang J: Electrochemical Glucose Biosensors[J]. Chemical Reviews, 2008, 108(2): 814-825.
[33]Wang Y, Shao Y, Matson DW et al: Nitrogen-Doped Graphene and Its Application in Electrochemical Biosensing[J]. ACS nano, 2010, 4(4): 1790-1798.
[34]Thévenot DR, Toth K, Durst RA et al: Electrochemical biosensors: recommended definitions and classification[J]. Biosensors & bioelectronics, 2001, 16(1-2): 121-131.
[35]Xu W, Jin T, Dai Y et al: Surpassing the detection limit and accuracy of the electrochemical DNA sensor through the application of CRISPR Cas systems[J]. Biosens Bioelectron, 2020, 155: 112100.
[36]Mukama O, Wu J, Li Z et al: An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids[J]. Biosens Bioelectron, 2020, 159: 112143.
[37]Wang X, Xiong E, Tian T et al: Clustered Regularly Interspaced Short Palindromic Repeats/Cas9-Mediated Lateral Flow Nucleic Acid Assay[J]. ACS nano, 2020, 14(2): 2497-2508.
[38]Yuan C, Tian T, Sun J et al: Universal and Naked-Eye Gene Detection Platform based on the Clustered Regularly Interspaced Short Palindromic Repeats/Cas12a/13a System[J]. Analytical Chemistry, 2020, 92(5): 4029-4037.
[39]张素真: 基于过渡金属硫化物的新型光纤倏逝波传感器的制备及性能研究. 硕士. 山东师范大学; 2019.
[40]Koo B, Kim DE, Kweon J et al: CRISPR/dCas9-mediated biosensor for detection of tick-borne diseases[J]. Sens Actuators B Chem, 2018, 273: 316-321.
[41]Yang Y, Liu J, Zhou X: A CRISPR-based and post-amplification coupled SARS-CoV-2 detection with a portable evanescent wave biosensor[J]. Biosens Bioelectron, 2021, 190: 113418.
[42]Rauch JN, Valois E, Solley SC et al: A Scalable, Easy-to-Deploy Protocol for Cas13-based Detection of SARS-CoV-2 Genetic Material[J]. Journal of Clinical Microbiology, 2021, 59(4).
[43]Ding X, Yin K, Li Z et al: Ultrasensitive and visual detection of SARS-CoV-2 using all-in-one dual CRISPR-Cas12a assay[J]. Nature Communications, 2020, 11(1).
[44]Shan Y, Zhou X, Huang R et al: High-Fidelity and Rapid Quantification of miRNA Combining crRNA Programmability and CRISPR/Cas13a trans-Cleavage Activity[J]. Analytical Chemistry, 2019, 91(8): 5278-5285.
[45]Li L, Li S, Wu N et al: HOLMESv2: A CRISPR-Cas12b-Assisted Platform for Nucleic Acid Detection and DNA Methylation Quantitation[J]. Acs Synthetic Biology, 2019, 8(10): 2228-2237.
[46]Shen J, Zhou X, Shan Y et al: Sensitive detection of a bacterial pathogen using allosteric probe-initiated catalysis and CRISPR-Cas13a amplification reaction[J]. Nature Communications, 2020, 11(1).
[47]Sheng A, Wang P, Yang J et al: MXene Coupled with CRISPR-Cas12a for Analysis of Endotoxin and Bacteria[J]. Analytical Chemistry, 2021, 93(10): 4676-4681.
[48]Liang M, Li Z, Wang W et al: A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules[J]. Nature Communications, 2019, 10.
[49]Zhao X, Li S, Liu G et al: A versatile biosensing platform coupling CRISPR-Cas12a and aptamers for detection of diverse analytes[J]. Science Bulletin, 2021, 66(1): 69-77.
[50]袁媛,王景林,孙岩松,王菁,辛文文,康琳,李岩伟. RPA联合CRISPR技术检测炭疽杆菌的方法及成套试剂[P]. 北京市:CN112522429A,2021-03-19.
[51]许腾, 曾伟奇, 杨敏玲, 陈文景, 徐学中, 李永军, 王小锐, 苏杭. 基于CRISPR技术的2019-nCoV快速检测引物组及其用途[P]. 广东省: CN111363847A, 2020-07-03.
[52]梁亚峰, 孙洁, 刘锐恒. 基于CRISPR技术进行靶核酸检测的方法[P]. 山东省: CN111996236A, 2020-11-27.
[53]余四斌, 田莉, 袁志阳, 凡凯. 一种水稻种子耐贮藏性基因sd1及其分子标记和应用[P]. 湖北省: CN110358773A, 2019-10-22.
[54]余四斌, 袁志阳, 蔡星星, 凡凯, 田莉, 邱先进. 一种水稻温敏核不育基因tms3突变体及其分子标记与应用[P]. 湖北省: CN110205327A, 2019-09-06.
[55]王功伟, 刘畅. OsLUT2基因在水稻光保护中的应用[P]. 湖北省: CN109112150A, 2019-01-01.
[56]熊立仲, 叶莹, 肖本泽. OsSEC3B基因在控制水稻抗旱性中的应用[P]. 湖北省: CN112321695A, 2021-02-05.
[57]赵云德, 张涛, 王荣臣. 一种调控水稻理想株型的基因OsVAS1[P]. 湖北省: CN109112138A, 2019-01-01.
[58]POLICE, Seshidhar, Reddy,NG, Robert, YANG, Yanfei. CRISPR/CAS ALL-IN-TWO VECTOR SYSTEMS FOR TREATMENT OF DMD[P]. WO2020225606, 2020-11-12.
[59]DE PUIG GUIXE, Helena,JOLLY, Pawan,COLLINS, James J., INGBER, Donald E.. ON-CHIP ASSAY STRATEGY FOR THE DEVELOPMENT OF ELECTROCHEMICAL READOUT FOR CRISPR-CAS DIAGNOSTICS[P]. WO2021087203, 2021-05-06.
作者单位:100069 北京,首都医科大学公共卫生学院
通讯作者:郝荣章,email:hao@ccmu.edu.cn