
陈威震,博士,助理研究员,就职于浙江大学医学院附属第一医院浙江省临床体外诊断技术研究重点实验室;主要研究领域:感染性疾病分子诊断,发表SCI论文10余篇,主持国家自然科学基金1项。
【摘要】基于规律成簇的间隔短回文重复序列及其相关蛋白(CRISPR/Cas)系统构建的基因编辑技术,凭借其简单、高效的特点,目前已广泛应用于生物、医学等多个研究领域。特别是近几年,CRISPR/Cas系统已被开发为一种快速、超敏、高特异性的分子检测工具,目前在病原体核酸检测、基因多样性如单核苷酸多态性与DNA甲基化、耐药性分析、疾病相关基因的筛查等领域展现了巨大的应用前景。本文就CRISPR/Cas系统在分子诊断的应用进行总结,并对其应用前景进行展望,以期为相关领域科研工作提供参考。
【关键词】间隔短回文重复序列及其相关蛋白(CRISPR/Cas);病原诊断;分子检测
长期以来,由病原体引起的感染性疾病,对人类生命安全和健康带来巨大威胁,是当前全球最为重要的公共健康卫生问题之一[1]。引发感染性疾病的病原体主要有细菌、病毒、真菌和寄生虫等。在感染性疾病的临床诊治中,一些不同的病原体感染可能会引发相似的临床症状,但是其发病机制和进程则不尽相同,需要采取的治疗方法也往往不同。因此如何及时、准确的确定感染原因,对于进一步控制患者病情、提高感染性疾病患者生存率,具有重要作用。特别是对于发病率和病死率较高、且传染性强的感染性疾病,快速、精准的对病原体进行筛查和鉴定,有利于及时采取疫情防控措施,从而控制疫情进一步蔓延。
近年来,随着分子生物学技术的飞速发展,基于核酸扩增技术的分子诊断方法,凭借其高度的特异性和灵敏性,目前已广泛应用于感染性疾病病原诊断领域,成为传染病防控和诊断的重要方法[2]。当前临床常用的分子检测技术主要以聚合酶链反应(polymerase chain reaction,PCR)、分子杂交技术和基因测序技术等为主,但是这几类诊断技术存在操作流程复杂,所需检测设备昂贵,人力成本和人员资质要求较高等不足。因此,在满足灵敏度高和特异性强的前提下,开发更为快速、廉价和操作简单的新一代病原诊断技术,是分子诊断领域亟需解决的问题。
近年来,基于规律成簇的间隔短回文重复序列及其相关蛋白(CRISPR/Cas)系统的基因编辑技术打破了传统分子诊断技术的局限性,已经被开发成为一种高效的分子诊断工具。基于CRISPR/Cas系统的分子诊断技术凭借其低成本、快速、便捷、高灵敏性、高特异性等优势,目前在分子诊断领域,尤其是感染性疾病病原体诊断应用中,展示出巨大的发展前景。本文旨在对CRISPR/Cas系统在病原诊断中的研究进展、优势和局限等方面进行介绍和探讨。
一、CRISPR/Cas系统概述
规律成簇间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)是一类广泛分布于细菌和古菌基因组中的重复结构。而Cas基因(CRISPR-associated genes)则是一类在CRISPR位点附近高度保守的基因家族,通过该基因家族编码的蛋白复合体具有多种RNA结合蛋白特性的结构域,以及核酸酶、解旋酶、整合酶和聚合酶等多种酶活性特征。近年来一系列的研究发现,Cas基因可以与CRISPR序列协同作用,Cas蛋白复合体在特定向导RNA(gRNA)介导下,赋予了细菌和古细菌对病毒的获得性免疫功能,而间隔序列在其中发挥编码引导Cas蛋白定位的向导RNA的功能。自2012年Jennifer Doudna等人首次发现利用RNA介导的CRISPR/Cas9系统可实现基因编辑之后,基于CRISPR/Cas系统的基因编辑工具已被广泛应用于生物、医学、农业等多个领域(图1)。
根据核糖核蛋白复合物特性及发挥功能方式,目前CRISPR/Cas基因编辑系统可以分为两大类别,6种型别和多种亚型:以多个Cas组成的效应复合体行使功能为特征的第1类CRISPR/Cas系统;以单个多结构域Cas为基础构成的第2类CRISPR/Cas系统。由于第2种类型CRISPR/Cas系统操作简单高效,是目前最为常用的基因编辑系统,以Cas9,Cas12a和Cas13a等为代表性。
注:在基因编辑过程中,Cas9内切酶会在gRNA分子的引导,识别在靶标DNA上与gRNA序列互补的PAM序列,并与之结合,对特定位点的DNA进行切割,形成双链DNA缺口。之后在同源重组机制或者非同源末端连接机制作用下对断裂的DNA进行修复
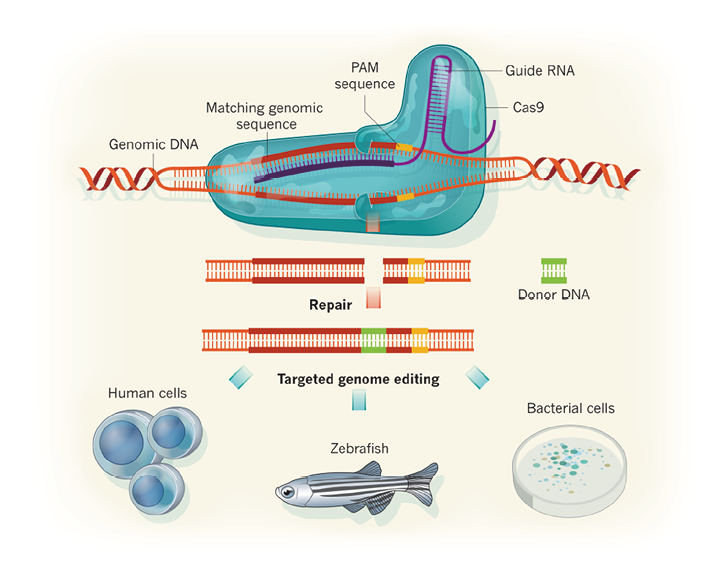
图1. 由gRNA介导的CRISPR-Cas9系统靶向基因编辑作用机制示意图[3]
二、基于CRISPR/Cas系统顺式切割功能的分子诊断系统
顺式切割(cis-cleavage)是指在gRNA引导下,CRISPR/Cas系统通过靶向特异性识别含间隔序列前体临近基序(PAM)的靶标核酸,并对其进行切割或编辑。序列特异性切割功能是CRISPR/Cas系统基因编辑技术的重要特性之一,使其不仅可以被用于基因编辑,同时在分子诊断领域也有巨大应用前景,以DNA为靶标核酸的CRISPR/Cas9系统就是典型代表之一。早在2016年,合成生物学家James J. Collins研究团队首次将CRISPR/Cas基因编辑技术用于病原微生物的核酸分子诊断(图2)。通过利用CRISPR/Cas9特异性识别和剪切双链DNA(dsDNA)的功能,Collins团队研发出一种用于寨卡病毒和登革病毒病原体核酸分子检测的NASBA-CRISPR裂解(NASBACC)方法[4]。该方法首先利用依赖核酸序列的等温扩增(NASBA)技术对病原体靶向RNA序列进行逆转录和扩增,产生大量双链DNA。之后CRISPR/Cas9在gRNA引导下,寻找双链DNA上的间隔序列前体临近基序(PAM)位点,对DNA进行切割,实现对靶标核酸序列识别。由于CRISPR/Cas9系统具有高度的通用性特征,在特定gRNA序列辅助下,可以将Cas9蛋白定向到任何具有相邻PAM的靶标核酸位点。此外,由于CRISPR/Cas9系统的高特异性识别特征,基于CRISPR/Cas9系统构建的分子诊断系统在单位点突变病原体分型,或其他单核苷酸多态性(single nucleotide polymorphism,SNP)等相关疾病的应用中显示出更为突出的优势。目前,CRISPR/Cas9系统已被开发用于人乳头状瘤病毒检测和基因分型,扩增子测序等分子诊断领域[5-7]。

图2. 基于CRISPR/Cas9顺式切割功能构建的NASBACC分子检测系统及其在寨卡病毒分型中的应用[4]
三、基于CRISPR/Cas反式切割功能的分子诊断系统
1. CRISPR/Cas反式切割RNA功能的分子诊断系统:随着CRISPR/Cas系统中Cas蛋白不断被挖掘,研究人员还发现一些Cas蛋白除了可通过“顺式切割”功能对靶标核酸进行切割外,还存在其他特性的核酸切割活性:反式切割(trans-cleavage)或附带切割(collateral effect)功能[8, 9],并将该功能用于研发分子诊断系统。2016年张锋团队首次发现了一种能够靶向结合和降解RNA的酶C2c2(即Cas13a),并发现了Cas13a还具有不依赖于核酸序列的反式切割(trans-cleavage)或附带切割(collateral effect)效应[10]。所谓反式切割活性,即Cas13a在与crRNA和靶标核酸形成切割三元复合体之后,能够对系统内存在的任意序列ssRNA进行切割。随后在2016年9月,Jennifer Doudna等人率先将CRISPR/Cas13a蛋白的附带切割活性用于检测RNA[11],但是该团队开发的检测方法灵敏度低,临床应用潜能有限。紧接着在2017年,James J. Collins团队和张锋团队合作,通过利用CRISPR/Cas13a的“反式切割”功能,开发了一种高效的体外核酸检测方法—SHERLOCK(Specific High Sensitivity Enzymatic Reporter UnLOCKing),已用于寨卡病毒、登革病毒等病原体核酸检测和DNA突变鉴定等[12]。之后,基于CRISPR/Cas基因编辑技术的分子诊断应用与研究立即驶入快车道,并取得系列重要成果。在该系统中,首先将靶标核酸片段进行核酸恒温扩增放大的同时添加上T7启动子,由此获得大量含有T7启动子的DNA模板,后经T7 RNA聚合酶转录获得Cas13a可识别的RNA靶标核酸片段。Cas13a在对靶标核酸进行识别切割后激活该系统的“反式切割”功能,对体系中存在的单链RNA进行切割,产生荧光信号。之后研究团队对SHERLOCK检测系统进行进一步优化,在系统中通过增加Cas蛋白的种类(LwaCas13a、PsmCas13b、CcaCas13b和AsCas12a)以达到可以同时检测四种病毒(或靶标)的目的,并利用Csm6酶放大其最终检测信号,进一步增强该方法的灵敏度[13]。在进一步优化的SHERLOCK检测系统(SHERLOCKv2)中,其性能方面有了更大提升,能够实现单管多靶标检测出,定量检测限可低至2Amol/L。由于该检测体系检测快速、价格低廉、灵敏度高,因此非常适用于POCT分子诊断产品研发与应用。
2. CRISPR/Cas反式切割DNA功能的分子诊断系统:2018年,Jennifer Doudna等人发现Cas12在gRNA引导下特异性结合和切割靶标双链DNA后,会产生类似于Cas13a的反式切割(附带切割)效应,但是其切割靶标为非特异性单链DNA(ssDNA)[14]。根据此特性,研究人员相继开发基于Cas12反式切割活性的DETECTR(DNA Endonuclease Targeted CRISPR Trans Reporter)[14]和HOLMES(an one-HOur Low-cost Multipurpose highly Efficient System)[15]等核酸检测系统。与SHERLOCK核酸检测系统不同,DETECTR或HOLMES检测系统中,Cas12a在gRNA引导下与双链DNA靶标序列结合并切割,由此激活Cas12a的附带切割活性,对反应体系中的非特异性ssDNA荧光报告序列进行切割,释放荧光信号。由于DETECTR或HOLMES检测系统中Cas12识别靶标为双链DNA,因此省去了体外将DNA转录成RNA这一繁琐步骤,诊断过程进一步简化。

图3. 基于不同CRISPR/Cas系统构建的NASBACC、SHERLOCK、DETECTR技术差异比较[16]
在左图NASBACC系统中,首先利用通过逆转录酶和RNase H生成cDNA单链,随后利用带有Toehold(立足点传感器)开关触发序列的反向引物与cDNA结合,通过酶促反应延伸形成含有触发序列(Trigger)的dsDNA。当dsDNA含有靶标特异性PAM序列,Cas9系统将靶标核酸切割,此时切割的dsDNA在T7 RNA聚合酶作用下,只能合成截断的RNA产物,无法激活传感器H的Toehold开关,显示阴性识别信号。当dsDNA无靶标特异性PAM序列时,dsDNA在T7 RNA聚合酶作用下,会合成完整的RNA产物,激活传感器H的Toehold开关,显示阳性识别信号;在右图基于CRISPR/Cas系统“反式切割”的分子诊断系统中,首先利用RPA或RT-RPA等恒温扩增方法对靶标核酸进行扩增,在靶向RNA的CRISPR/Cas分子诊断系统中,如基于Cas13a酶的SHERLOCK系统,会利用T7 RNA聚合酶将靶标DNA逆转录为RNA,激活Cas酶的反式切割功能;在靶向DNA的CRISPR/Cas分子诊断系统中,如基于Cas12a酶的DETECTR系统,恒温扩增产物dsDNA会直接激活Cas酶的反式切割功能。
四、小结与展望
短短五六年时间,基于CRISPR/Cas系统的基因编辑技术已经发展成一种功能强大的多用途生物学工具,特别是在分子诊断领域中,显示出其重要的临床应用价值与前景。特别是基于CRISPR/Cas技术研发的SHERLOCK和DETECTR核酸检测系统,由于其检测快速,成本低廉,且具有高灵敏度和特异性等优点,已证实在病原体核酸检测、SNP分型、耐药性分析以及基因突变相关疾病的检测等多个方面具有应用前景,被认为是重要的“下一代分子诊断技术”。
但是同时也要认识到,当前基于CRISPR/Cas系统的分子诊断技术仍处于起步阶段,面临诸多挑战。当前CRISPR/Cas分子诊断系统存在的最大不足就是大部分检测方法仍需核酸预扩增到足够量以达到可检测水平,而预扩增不仅大大增加反应时间,同时所需的特异性引物设计也会增加检测成本。另外,基于Cas13a的检测系统是以反式切割RNA为基础,这一过程极易受到环境中RNA酶的干扰,导致假阳性结果的产生。此外,通过“一锅法”反应和结果可视化等方式,可以进一步降低CRISPR/Cas分子诊断技术使用难度,但是目前该方法的样本热处理过程仍依赖一些加热设备,不适宜于居家或者其他特定场景检测。因此,通过利用材料学和工程学原理,如微流控技术等,研发一种集合样本制备、检测和结果报告于一体的CRISPR/Cas分子诊断一体机,有望解决这些难题。当前报道的CRISPR/Cas分子诊断技术尚处于实验室研发阶段,未来在应用于实际临床检测前,必须要进行大量临床试验评估,以确保其具有良好的临床适用性。总之,我们坚信随着研究的不断深入,基于CRISPR/Cas基因编辑系统的分子诊断技术必将重塑核酸检测的技术格局。
参考文献
Bajaj S, Banerjee M. Engineering Virus Capsids Into Biomedical Delivery Vehicles: Structural Engineering Problems in Nanoscale. J Biomed Nanotechnol. 2015; 11: 53-69.
Persing DH, Tenover FC, Hayden RT, Ieven M, Miller MB, Nolte FS, et al. Molecular microbiology: diagnostic principles and practice: John Wiley & Sons; 2020.
Charpentier E, Doudna JA. Rewriting a genome. Nature. 2013; 495: 50-1.
Pardee K, Green AA, Takahashi MK, Braff D, Lambert G, Lee JW, et al. Rapid, low-cost detection of Zika virus using programmable biomolecular components. Cell. 2016;165:1255-66.
Wang Q, Zhang B, Xu X, Long F, Wang J. CRISPR-typing PCR (ctPCR), a new Cas9-based DNA detection method. Scientific reports. 2018; 8: 1-13.
Zhang B, Wang Q, Xu X, Xia Q, Long F, Li W, et al. Detection of target DNA with a novel Cas9/sgRNAs-associated reverse PCR (CARP) technique. Anal Bioanal Chem. 2018; 410: 2889-900.
Quan J, Langelier C, Kuchta A, Batson J, Teyssier N, Lyden A, et al. FLASH: a next-generation CRISPR diagnostic for multiplexed detection of antimicrobial resistance sequences. Nucleic Acids Res. 2019; 47: e83-e.
李林显. CRISPR-Cas12b的反式切割活性研究及其介导的核酸检测技术开发 [硕士]: 河南大学; 2019.
王雪亮, 肖艳群, 王华梁. CRISPR/Cas系统在分子检测中的应用. 检验医学. 2020; 35: 181-5.
Abudayyeh OO, Gootenberg JS, Konermann S, Joung J, Slaymaker IM, Cox DB, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 2016;353.
East-Seletsky A, O’Connell MR, Knight SC, Burstein D, Cate JH, Tjian R, et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection. Nature. 2016; 538: 270-3.
Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 2017; 356: 438-42.
Gootenberg JS, Abudayyeh OO, Kellner MJ, Joung J, Collins JJ, Zhang F. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6. Science. 2018; 360: 439-44.
Chen JS, Ma E, Harrington LB, Da Costa M, Tian X, Palefsky JM, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 2018; 360: 436-9.
Li S-Y, Cheng Q-X, Wang J-M, Li X-Y, Zhang Z-L, Gao S, et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell discovery. 2018; 4: 1-4.
Kaminski MM, Abudayyeh OO, Gootenberg JS, Zhang F, Collins JJ. CRISPR-based diagnostics. Nature Biomedical Engineering. 2021; 5: 643-56.