免疫组库检测技术在衰老评估中的应用价值

作者:李晓宇 罗礼华 张伟 聂超
【摘要】T细胞受体(TCR)和B细胞受体(BCR)是获得性免疫系统的核心组成部分,直接与抗原进行结合产生免疫反应。TCR和BCR由于在淋巴细胞成熟过程中发生基因重排,产生了很大的多样性,而个体的TCR和BCR集合构成了免疫组库。随着高通量技术的普及,捕获和检测巨大数量的TCR和BCR成为可能。经过近十年的发展,已有多种免疫组库实验捕获技术,以及多个相应的信息分析软件。随着年龄的增长,TCR和BCR库也会发现相应的变化,影响着免疫系统的反应能力。在本文中,我们将从免疫组库重排原理、实验和信息技术进行介绍,免疫组库多样性与衰老的相关性,以及免疫组库在衰老疾病中的应用。
【关键词】免疫组库,高通量测序,TCR,BCR,衰老
一、免疫组库介绍
在人类的免疫系统中T细胞受体和B细胞受体的序列具有巨大的多样性,这种多样性的存在使人体的免疫系统能够针对多种病原进行免疫反应以起到广泛保护人体的作用。这些构成适应性免疫系统的T细胞受体和B细胞受体(也称为免疫球蛋白)的总和被定义为免疫组库。随着高通量测序技术的开发和发展,打破了之前低通量对免疫组库研究的限制,大规模的并行测序技术非常适合用于研究序列多样的免疫组库。适应性免疫的功能取决于T淋巴细胞表达的各种T细胞受体(TCR)和B淋巴细胞表达的B细胞受体(BCR,也称为免疫球蛋白)。多样化的TCR可以识别并结合抗原呈递细胞的主要组织相容性复合物(MHC)分子呈递的抗原的各种表位,并形成TCR-抗原-MHC样的结构复合物;另一方面,抗体可以直接结合并中和抗原。从研究历史上看,由于测序技术和工具的不足,针对TCR和BCR谱系的研究内容并不全面。高通量测序的发展使免疫组库的研究也随之得到了前所未有的发展,但是针对不同疾病的不同研究方法还有待开发。
二、TCR的重排和多样性
根据TCR序列的不同,T细胞可分为α/β型T细胞和γ/δ型T细胞。对于人类而言,大多数T细胞是α/β的TCR,而γ/δ型T细胞占总T细胞的0.5-16%(平均约4%)[1];对于α/β型T细胞来说TCR是由一条α和一条β链组成的异二聚体;对于γ/δ型T细胞,其TCR由一条γ和一条δ链组成。人类TCRα基因(TRA)和δ基因(TRD)基因座的位置在14号染色体上,β链基因(TRB)和γ链基因(TRG)基因座位于7号染色体上。在T细胞的发育中,TCR经历了与BCR基本相同的重组过程。TCR基因座重排始于双阴性祖细胞T细胞中的TCRβ链。成功表达TCRβ链的T细胞将开启T细胞膜表面CD4和CD8的表达以形成双阳性细胞,同时,α链被重新排列并与β链配对以形成αβTCR[2-4]。同样,大量的TCR基因片段可作为体细胞重组,连接多样性和α/β链配对的潜在候选者,从而有助于TCR多样化库识别MHC /抗原复合物。
三、BCR的重排和多样性
抗体,又称免疫球蛋白(IG),是由B细胞分泌的能特异性地免疫和中和多种抗原的蛋白质分子。抗体由一个非常独特的蛋白质编码系统编码,在编码基因中发生体细胞重排。抗体是由两个相同的大重链(由基因编码)和两个相同的小轻链(λ或κ)(由基因IGL或IGK编码)组成的Y形蛋白质。B细胞抗原受体(B-cell receptor, BCR)是一种位于B细胞表面的负责特异性识别及结合抗原的分子,BCR可变区的多样性决定了抗原识别的多样性和特异性。人类IGH、IGL和IGK基因分别位于14、22和2号染色体上。免疫球蛋白重链可变区由三个基因家族编码(免疫球蛋白重链基因重排见图1),包括可变V、D、J基因,它们包含许多具有高序列同源性的基因拷贝。对于轻链,只有V基因和 J基因家族参与重组。在BCR细胞发育成熟过程中,BCR的可变编码基因经历了一个称为V(D)J重组(重排)的体细胞重组过程。在这一过程中,随机选择的V,(D)和J基因片段,通过重组激活基因(RAG1/RAG2)和末端脱氧核苷酸转移酶(TdT)[5,6]等多种酶的协同作用,被导入到相邻的介质中,所排列的可变域包括4个框架区(FR)和三个互补决定区(CDR)。用于重组的V、D和J基因多样性为抗体的多样性提供了基础,V-D和D-J(轻链为V-J)连接区域中的随机非定位核苷酸的添加和删除也在很大程度上促成了BCR的巨大多样性[7,8]。随后体细胞超突变(Somatic Hypermutation,SHM)导致了进一步的多样性,主要是对重组的抗体编码基因进行单核苷酸替换。由于活化诱导(胞嘧啶核苷)脱氨酶(AID)和尿嘧啶DNA糖基化酶[9,10]的作用,体细胞超突变在B细胞中发生率极高,约为10-3每碱基对分裂[11],从而大大增加了BCR的多样性[7-12]。除此之外,基因转录和重/轻链配对也提高了功能性抗体的多样性,最终产生的人类免疫球蛋白理论上可以达到1013,超过了人体B细胞总数(1011)[13,14],正是这种多样性的潜在抗体使他们能够识别各种抗原。
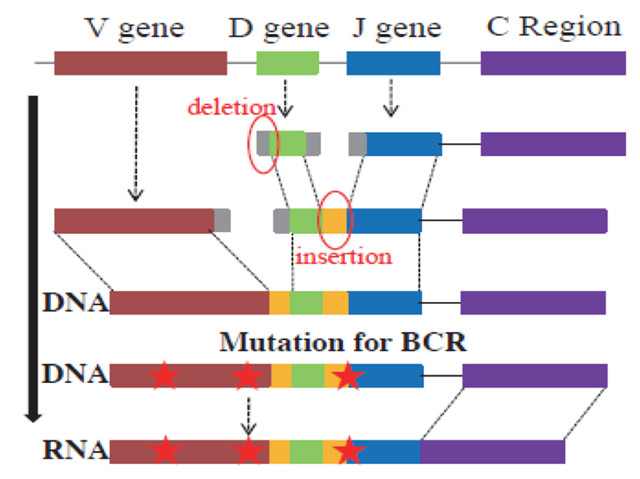
图1. 免疫球蛋白重链重排示意图
四、免疫组库的检测技术
高通量测序技术(High-throughput Sequencing,HTS)的发展使得从巨大的T/B细胞库里寻找抗原特异性T/B细胞成为可能。在该领域使用的最早的HTS平台是Roche 454系统,该系统平均提供500 bp的读取长度和每次运行适度的读取数量。它能够捕获TCR和BCR的整个VDJ区域,并且每个序列可以测序数百万个分子,这大大超过了Sanger测序技术。2009年,《科学》杂志发表了第一个使用454测序仪对斑马鱼抗体库进行测序的研究[15]。随后,又有两项研究采用454测序仪来测序人类抗体和TCR组成部分的整个VDJ重组区域[16,17]。Genome Research发表了第一项利用Illumina测序仪来完成可变性最大的CDR3区域的研究,以研究人类外周血中的TCRβ链的特征[18]。与454测序仪相比,Illumina Hiseq平台提供了较短的读取长度,但测序量显着提高,并且测序成本更低,这非常适合深度测序非常复杂的组库。从当前的应用现状来看,读取长度和测序量是研究者应该折衷和权衡的两个因素。
在各种研究中,选择DNA(gDNA)还是mRNA作为分析对象取决于研究的目的。TCR和BCR的gDNA以单拷贝形式存在于T和B细胞中,因此代表了T/B细胞群体的细胞比例和异质性。因此,gDNA适用于计算抗原特异性或目标T和B细胞(例如疫苗特异性克隆或白血病/淋巴瘤克隆)的比例,并适合研究特定TCR/BCR克隆型的功能/表型演变。另一方面,由于TCR/BCR的表达在T/B细胞的不同类型/状态之间(例如在原始细胞和浆母细胞上)有显着差异,因此TCR/BCR mRNA的定量与细胞功能/激活的相关性更高。从技术上讲,从T/B细胞群体中提取的gDNA通常包含重组前后的VDJ片段和非生产性VDJ重排,这将成为干扰TCR/BCR谱库正确扩增的背景因素。然而,gDNA比mRNA更容易储存和运输。
有多种方法可以从gDNA和mRNA样品制备TCR/BCR库的测序文库。由于特定VDJ分子的稀有性,尤其是低频率的拷贝,通常采用扩增的方法。多重PCR(多重PCR原理示意图见图2)是DNA样本最便捷,最直接的扩增方法[17,19],通常结合V基因和J基因(或mRNA的C基因)特异性多重引物来扩增完全重组的取决于引物靶向的V基因区域,该受体基因的可变区或CDR3区。这种单管反应很方便,但是各种引物的不同效率和交叉反应性通常会使扩增产物产生一定程度的偏差。目前还有几个团队仍在使用BIOMED-2引物或基于它们的改进版本。但是,其他研究团队正在尝试实施各种方法来最小化基因特异性偏倚,例如使用嵌套式PCR[17],使用合成模板设计无偏的PCR反应[20],或者在引物中掺入随机条形码以调节偏差和误差[21,22]。然而,通过所有这些努力,PCR偏倚只能被最小化,而几乎不能消除。
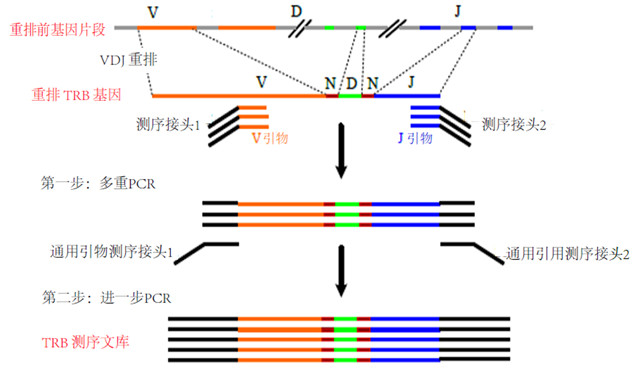
图2. 多重PCR原理示意图
富集重排抗体或TCR的另一种实验方法是快速扩增cDNA 5'末端(5'RACE,示意图见图3),理论上它可以避免V基因偏倚,因为它仅掺入C基因引物。5'RACE只能作用于mRNA,而不能作用于gDNA。
针对3'mRNA中的已知区域设计引物,该区域是TCR /抗体库分析中的恒定基因区域。将mRNA进行逆转录,并使用dNTP和TdT或仅通过逆转录酶的TdT特性通过模板转换过程将均聚物尾部合成添加到cDNA的3'端[23,24]。另一个5'RACE是连接锚定PCR,使用RNA连接酶逆转录后将连接子连接至cDNA的3'末端[25,26]。因此,可以通过恒定基因引物和通用接头引物扩增产物。为了缩短产品长度,可以通过Illumina Hiseq系统对产品进行测序,在此过程中进行了一些更改。例如,将生物素标记到恒定基因引物上,然后将cDNA产物随机片段化为合适的长度,以通过Hiseq机器的单次读数或双端读数进行测序。生物素标记的片段被磁珠特异性捕获,以富集含有CDR3和恒定基因区域的靶向片段。RACE方法已被当前的TCR/BCR研究广泛采用[27-29]。
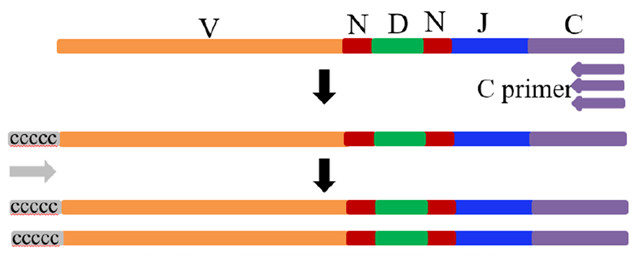
图3. 5’RACE示意图
多重PCR和5'RACE是目前研究中两种最流行的方法。然而,正如其他评论文章所分析的那样[30,31],两者都有其优缺点。关于VDJ重组的富集 还有其他方法,例如使用针对V/J种系基因序列设计的探针/诱饵来捕获重排的VDJ片段[32]。但是,这种方法需要在捕获步骤之前随机剪切gDNA/cDNA,并且会导致不完整的重排不完整,并在捕获和测序中丢失。因此,它仅适用于在淋巴瘤或白血病等样本中恢复大量的重排。
BCR的VH:VL和TCR的Vα:Vβ的链配对信息对于阐明T/B细胞的功能至关重要。先前的方法分别放大每个链,或在同一反应中同时富集两个链。但是,在总体水平上,这些方法无法分辨出每个VH:VL或Vα:Vβ对。直到最近,通过限制单个细胞上的稀释和Sanger测序,通过单细胞克隆对天然链对进行了研究。但是,该方法吞吐量低且扩展性强。与HTS合并后,已经开发出新的方法来提高吞吐量和发现本地链配对信息的可行性。开发了一种基于细胞的乳液RT-PCR方法,以允许TCRVα:Vβ基因的天然对的选择性融合,并且在单个乳液实验中可以回收700对以上[33]。DeKosky等人开发了一种VH:VL配对技术,该技术依赖于将单个B细胞隔离到纳升以下的容量孔中,裂解细胞,在poly-dT珠子上捕获RNA,并通过乳液重叠延伸PCR产生编码连接的VH:VL片段的扩增子[34]。实验在一天时间内,获得了成千上万对独特的内源性VH:VL对,验证了97%的配对精度。最近,Howie等报道了pairSEQ方法,其中将固定数量的T细胞随机分配到96孔板上的每个孔中,并将mRNA进行反转录,并附上特异的条形码,扩增,汇集在一起并测序。
共享唯一的一组孔(条形码)的任何对都被视为来自同一克隆,因为两个克隆共享同一组孔的可能性极小[35]结合乳液PCR和HTS的高通量配对方法正在开发中,我们可以预料在不久的将来该领域将常规采用TCR/BCR配对测序。
除了配对信息外,单细胞方法还可以将T/B细胞表型(例如基因表达特征)与受体配对序列联系起来,这可能有助于阐明T/B细胞的进化和功能异质性[36]。
五、免疫组库的信息分析技术
在高通量筛选测序出现之前,已经开发了可行的生物信息学工具来比对和分析TCR和BCR序列,包括iHMMune-align[37]、IMGT/V-QUEST[38]和NCBI的IgBLAST工具。这些工具有助于确定重组的V/D/J基因、可变区域注释和TCR/BCR序列的特征。高通量筛选测序为我们提供了前所未有的深度和多样性的免疫储备,更强大的工具和软件能够更准确、快速地处理这些庞大的数据。目前已经发表了用于免疫库数据分析和可视化的标准流程[39,40],并发布了一些新的计算工具来分析高通量筛选测序数据,如IMGT/HighV-QUEST[28]、new IgBLAST[41]、Decombinator[42],pRESTO[43]和MiTCR[44]其进一步更新为MiXCR,用于挖掘TCR和BCR数据[45]。这些工具可用于VDJ基因定位、CDR和FR注释、CDR3长度鉴定、插入和删除分析以及突变谱分析(BCR),Greiff等人对这些分析工具的特点进行了总结[39]。为了提高分析工具的质量,需要考虑几个技术方面的问题,包括从真实的生物变异中识别PCR和测序误差,以及调整PCR的偏差。可通过优化实验方法,也可以从数据分析方法上进行优化。考虑到这些因素,我们已经建立了自己的名为IMonitor的生物信息学工具,该工具是Immune Monitor的缩写,用于分析TCR和BCR的免疫组库数据[46]。我们在模拟数据和生物数据上都进行了验证,并与现有的一些工具进行了精度比较。
在基本分析工具的基础上,还开发了其他算法,以便能更详细地进行数据挖掘。免疫反应谱的指标可以反映克隆选择和扩增的程度,可以作为个体免疫状态的生物标志物。从生态学角度引入香农熵指数和辛普森多样性指数来估计物种多样性,但存在着灵敏度低、依赖测序深度等明显局限性。目前已经建立了新的生物信息框架,将免疫状态与多样性指数联系起来[47],用于评估物种丰富度和分布[48]。对于BCR,体细胞超突变使抗体亲和力成熟以及BCR更加多样化。因此,分子进化的概念被用来描述个体内BCR的动力学。B细胞进化树的构建可用于推断单个细胞之间的祖先关系,并且已经开发了一些计算方法来分析B细胞进化树[49,50]。
六、免疫组库多样性与衰老
衰老与适应性免疫反应的显著下降有关,这是因为衰老的T细胞[50,51]功能发生了改变以及幼稚T细胞丰度和TCR多样性总体下降引起的。免疫组库的多样性主要由TCR多样性产生,每106T细胞的TCR多样性随年龄的增长呈线性下降,在前10万个克隆中的丰度与TCR多样性相关,随着年龄的增长而降低,且随着年龄的增长幼稚CD45RAhigh/CD27highT细胞百分比呈线性下降, CD4+/CD8+子集中的幼稚T细胞百分比在70岁之前也呈直线下降[52],见图4。对1068名18至80岁的健康人进行分析发现[53],CD3+T细胞计数,CD3+CD8+T细胞计数,CD4+CD45RA+T细胞计数,CD4+CD45RA+CD45SRA+CD62L+T细胞计数,CD4+CD28+/CD4+比和CD8+CD28+/CD8+比随着年龄的增长而降低,记忆CD4+或CD8+T细胞随年龄增长而增加,但是在衰老个体中NK细胞和CD19+B细胞的绝对计数和百分比保持相对稳定。老年人的免疫系统下降,会影响他们对感染的反应能力以及对疫苗的有效反应能力。降低了来自老年人的反应中的有效和特异性抗体,有利于非特异性抗体的产生。使用IGHV互补决定区(CDR)3的光谱类型分析调查了86-94岁个体和19-54岁对照组外周血DNA样本中的B细胞组成。研究发现[54]一部分老年人的B细胞库多样性显着下降。从选择的样品中测序聚合酶链反应产物表明,这种多样性丧失的特征是体内B细胞的克隆扩增引起的;且B细胞多样性丧失的程度与老年人的健康状况相关。
TCR系统的多样性、结构和动态性在很大程度上决定了我们在免疫应答最小的错误目标下有效抵抗感染和恶性肿瘤的能力,为T细胞免疫的长期动态,对脐带血到百岁老人外周血的样本进行分析发现[55],免疫组库有着显著的稳定性,大约一半的外周血T细胞克隆在三年内持续存在;脐带血中无意义介导的mRNA衰变的效率降低,这可能反映了发育中的特定调节机制;人类TCR的组成在出生时在功能上更加相似,但在生活中却有所不同,并且我们追踪发现CMV和EBV特异性的T细胞克隆会终身存在。在瑞典的八十岁老人和非老年人队列研究中进行的两项纵向研究[56]显示,与死亡率增加相关的免疫生物标记物的识别被称为免疫风险表型(IRP)。在具有IRP的健康老年人中发现高死亡率,其特征在于CD4/CD8比值倒数(小于1),幼稚T细胞丢失,对促分裂原的T细胞增殖反应差,CD8+CD28-CD57+细胞增加,B细胞数量少以及CMV或EBV特异性CD8 T细胞的克隆扩增。
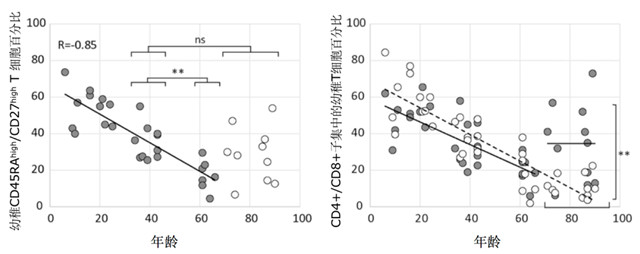
图4. 年龄和幼稚T细胞的关系[52]
七、免疫组库在衰老相关疾病中的应用
免疫系统的衰老(通常称为免疫衰老)显示在疫苗接种,病毒感染和神经系统疾病[57-60]的情况下会阻碍适应性免疫反应。老年人在免疫挑战期间有时会增加宿主介导的免疫病理学,这进一步凸显了在衰老过程中研究适应性免疫的重要性。例如,神经炎症在衰老和各种神经系统疾病(包括阿尔茨海默氏病,多发性硬化症和感染)中起着至关重要的作用。然而,技术局限性阻止了对淋巴细胞免疫受体组成及其伴随的转录状态如何随中枢神经系统(CNS)年龄的变化而进行的综合分析。我们可以利用单细胞测序来同时分析样本B细胞受体(BCR)和T细胞受体(TCR)组成以及相关的基因表达谱。以发现在衰老相关疾病中免疫组库的变化规律。
阿尔兹海默症是当前研究免疫与衰老性疾病的热点之一,这是一种不可治愈的神经退行性疾病,其相关的神经炎症在该病的研究重点;但针对神经炎症的适应性免疫反应在阿尔兹海默症中的研究比较少,有团队通过对阿尔兹海默症病人的多个队列进行研究,发现该病的免疫特征:阿尔兹海默症病人的CD8+T效应记忆CD45RA+(TEMRA)细胞数量增加,而CD8+TEMRA细胞与病人的认知呈负相关[58]。这些结果揭示了阿尔茨海默氏病在血液和脑脊液中的适应性免疫反应,后续的研究这可以以此为基础探究阿尔兹海默症的病理机制。
老年人体内保护性B细胞的反应也会因衰老被限制,所以老年人在应对新的病原体存在问题[59],这使得老年人接种疫苗的效果异于年轻人,因此探究改善老年人的疫苗接种策略也是当下研究的热点。针对老年人,提高疫苗接种效力的方法主要包括开发抗原传递系统和免疫调节剂,以增加天然B细胞的募集和产生,并诱导B细胞活化及分化成浆细胞。针对这些问题可以增加抗原剂量,提高抗原保留率等。针对婴儿和老人两个年龄极端的疫苗接种人群,这类研究也是很有必要的。
参考文献
Chien, Y.; Meyer, C.; Bonneville, M. γδ T Cells: First Line of Defense and Beyond . Annu. Rev. Immunol. 2014, 32, 121-155, doi:10.1146/annurev-immunol-032713-120216.
Germain, R.N. t-cell development and the CD4-CD8 lineage decision. Nat. Rev. Immunol. 2002, 2, 309-322.
Koch, U.; Radtke, F. Mechanisms of T Cell Development and Transformation. Annu. Rev. Cell Dev. Biol. 2011, 27, 539-562, doi:10.1146/annurev-cellbio-092910-154008.
Rothenberg, E. V.; Moore, J.E.; Yui, M.A. Launching the T-cell-lineage developmental programme. Nat. Rev. Immunol. 2008, 8, 9-21.
Gauss, G.H.; Lieber, M.R. Mechanistic constraints on diversity in human V(D)J recombination. Mol. Cell. Biol. 1996, 16, 258-269, doi:10.1128/mcb.16.1.258.
Schatz, D.G.; Swanson, P.C. V(D)J Recombination: Mechanisms of Initiation. Annu. Rev. Genet. 2011, 45, 167-202, doi:10.1146/annurev-genet-110410-132552.
Nezlin, R. Combinatorial Events in Generation of Antibody Diversity. Comb. Chem. High Throughput Screen. 2012, 4, 377-383, doi:10.2174/1386207013330977.
Rose, D.R. The generation of antibody diversity. Am. J. Hematol. 1982, 13, 91-99.
Larson, E.D.; Maizels, N. Transcription-couple mutagenesis by the DNA deaminase AID. Genome Biol. 2004, 5.
Teng, G.; Papavasiliou, F.N. Immunoglobulin Somatic Hypermutation. Annu. Rev. Genet. 2007, 41, 107-120, doi:10.1146/annurev.genet.41.110306.130340.
Kleinstein, S.H.; Louzoun, Y.; Shlomchik, M.J. Estimating Hypermutation Rates from Clonal Tree Data. J. Immunol. 2003, 171, 4639-4649, doi:10.4049/jimmunol.171.9.4639.
Li, Z.; Woo, C.J.; Iglesias-Ussel, M.D.; Ronai, D.; Scharff, M.D. The generation of antibody diversity through somatic hypermutation and class switch recombination. Genes Dev. 2004, 18, 1-11.
Schroeder, H.W. Similarity and divergence in the development and expression of the mouse and human antibody repertoires. Dev. Comp. Immunol. 2006, 30, 119-135, doi:10.1016/j.dci.2005.06.006.
Trepel, F. Number and distribution of lymphocytes in man. A critical analysis. Klin. Wochenschr. 1974, 52, 511-515.
Weinstein, J.A.; Jiang, N.; White, R.A.; Fisher, D.S.; Quake, S.R. High-throughput sequencing of the zebrafish antibody repertoire. Science (80-. ). 2009, 324, 807-810, doi:10.1126/science.1170020.
Boyd, S.D.; Marshall, E.L.; Merker, J.D.; Maniar, J.M.; Zhang, L.N.; Sahaf, B.; Jones, C.D.; Simen, B.B.; Hanczaruk, B.; Nguyen, K.D.; et al. Measurement and clinical monitoring of human lymphocyte clonality by massively parallel V-D-J pyrosequencing. Sci. Transl. Med. 2009, 1, doi:10.1126/scitranslmed.3000540.
Wang, C.; Sanders, C.M.; Yang, Q.; Schroeder, H.W.; Wang, E.; Babrzadeh, F.; Gharizadeh, B.; Myers, R.M.; Hudson, J.R.; Davis, R.W.; et al. High throughput sequencing reveals a complex pattern of dynamic interrelationships among human T cell subsets. Proc. Natl. Acad. Sci. U. S. A. 2010, 107, 1518-1523, doi:10.1073/pnas.0913939107.
Freeman, J.D.; Warren, R.L.; Webb, J.R.; Nelson, B.H.; Holt, R.A. Profiling the T-cell receptor beta-chain repertoire by massively parallel sequencing. Genome Res. 2009, 19, 1817-1824, doi:10.1101/gr.092924.109.
Klarenbeek, P.L.; Tak, P.P.; van Schaik, B.D.C.; Zwinderman, A.H.; Jakobs, M.E.; Zhang, Z.; van Kampen, A.H.C.; van Lier, R.A.W.; Baas, F.; de Vries, N. Human T-cell memory consists mainly of unexpanded clones. Immunol. Lett. 2010, 133, 42-48, doi:10.1016/j.imlet.2010.06.011.
Carlson, C.S.; Emerson, R.O.; Sherwood, A.M.; Desmarais, C.; Chung, M.W.; Parsons, J.M.; Steen, M.S.; LaMadrid-Herrmannsfeldt, M.A.; Williamson, D.W.; Livingston, R.J.; et al. Using synthetic templates to design an unbiased multiplex PCR assay. Nat. Commun. 2013, 4, doi:10.1038/ncomms3680.
Shugay, M.; Britanova, O. V.; Merzlyak, E.M.; Turchaninova, M.A.; Mamedov, I.Z.; Tuganbaev, T.R.; Bolotin, D.A.; Staroverov, D.B.; Putintseva, E. V.; Plevova, K.; et al. Towards error-free profiling of immune repertoires. Nat. Methods 2014, 11, 653-655, doi:10.1038/nmeth.2960.
Vollmers, C.; Sit, R. V.; Weinstein, J.A.; Dekker, C.L.; Quake, S.R. Genetic measurement of memory B-cell recall using antibody repertoire sequencing. Proc. Natl. Acad. Sci. U. S. A. 2013, 110, 13463-13468, doi:10.1073/pnas.1312146110.
Mamedov, I.Z.; Britanova, O. V.; Zvyagin, I. V.; Turchaninova, M.A.; Bolotin, D.A.; Putintseva, E. V.; Lebedev, Y.B.; Chudakov, D.M. Preparing unbiased T-cell receptor and antibody cDNA libraries for the deep next generation sequencing profiling. Front. Immunol. 2013, 4, doi:10.3389/fimmu.2013.00456.
Comprehensive Transcript Analysis in Small Quantities of mRNA by SAGE-lite-PubMed Available online: https://pubmed.ncbi.nlm.nih.gov/10572191/?from_single_lite.+Nucleic+Acids+Res.+1999%3B27%3Ae39. (accessed on Jun 15, 2020).
Gao, F.; Wang, K. Ligation-anchored PCR unveils immune repertoire of TCR-beta from whole blood. BMC Biotechnol. 2015, 15, doi:10.1186/s12896-015-0153-9.
Heather, J.M.; Best, K.; Oakes, T.; Gray, E.R.; Roe, J.K.; Thomas, N.; Friedman, N.; Noursadeghi, M.; Chain, B. Dynamic perturbations of the T-Cell receptor repertoire in chronic HIV infection and following antiretroviral therapy. Front. Immunol. 2016, 6, doi:10.3389/fimmu.2015.00644.
Bashford-Rogers, R.J.M.; Palser, A.L.; Idris, S.F.; Carter, L.; Epstein, M.; Callard, R.E.; Douek, D.C.; Vassiliou, G.S.; Follows, G.A.; Hubank, M.; et al. Capturing needles in haystacks: A comparison of B-cell receptor sequencing methods. BMC Immunol. 2014, 15, doi:10.1186/s12865-014-0029-0.
Li, S.; Lefranc, M.P.; Miles, J.J.; Alamyar, E.; Giudicelli, V.; Duroux, P.; Freeman, J.D.; Corbin, V.D.A.; Scheerlinck, J.P.; Frohman, M.A.; et al. IMGT/HighV QUEST paradigm for T cell receptor IMGT clonotype diversity and next generation repertoire immunoprofiling. Nat. Commun. 2013, 4, doi:10.1038/ncomms3333.
Warren, R.L.; Freeman, J.D.; Zeng, T.; Choe, G.; Munro, S.; Moore, R.; Webb, J.R.; Holt, R.A. Exhaustive T-cell repertoire sequencing of human peripheral blood samples reveals signatures of antigen selection and a directly measured repertoire size of at least 1 million clonotypes. Genome Res. 2011, 21, 790-797, doi:10.1101/gr.115428.110.
Georgiou, G.; Ippolito, G.C.; Beausang, J.; Busse, C.E.; Wardemann, H.; Quake, S.R. The promise and challenge of high-throughput sequencing of the antibody repertoire. Nat. Biotechnol. 2014, 32, 158-168.
Robins, H. Immunosequencing: applications of immune repertoire deep sequencing. Curr. Opin. Immunol. 2013, 25, 646-652.
He, J.; Wu, J.; Jiao, Y.; Wagner-Johnston, N.; Ambinder, R.F.; Diaz, L.A.; Kinzler, K.W.; Vogelstein, B.; Papadopoulos, N. IgH gene rearrangements as plasma biomarkers in non-Hodgkin’s Lymphoma patients. Oncotarget 2011, 2, 178-185, doi:10.18632/oncotarget.235.
Turchaninova, M.A.; Britanova, O. V.; Bolotin, D.A.; Shugay, M.; Putintseva, E. V.; Staroverov, D.B.; Sharonov, G.; Shcherbo, D.; Zvyagin, I. V.; Mamedov, I.Z.; et al. Pairing of T-cell receptor chains via emulsion PCR. Eur. J. Immunol. 2013, 43, 2507-2515, doi:10.1002/eji.201343453.
Dekosky, B.J.; Ippolito, G.C.; Deschner, R.P.; Lavinder, J.J.; Wine, Y.; Rawlings, B.M.; Varadarajan, N.; Giesecke, C.; Dörner, T.; Andrews, S.F.; et al. High-throughput sequencing of the paired human immunoglobulin heavy and light chain repertoire. Nat. Biotechnol. 2013, 31, 166-169, doi:10.1038/nbt.2492.
Howie, B.; Sherwood, A.M.; Berkebile, A.D.; Berka, J.; Emerson, R.O.; Williamson, D.W.; Kirsch, I.; Vignali, M.; Rieder, M.J.; Carlson, C.S.; et al. High-throughput pairing of T cell receptor α and β sequences. Sci. Transl. Med. 2015, 7, doi:10.1126/scitranslmed.aac5624.
Han, A.; Glanville, J.; Hansmann, L.; Davis, M.M. Linking T-cell receptor sequence to functional phenotype at the single-cell level. Nat. Biotechnol. 2014, 32, 684-692, doi:10.1038/nbt.2938.
IMGT/V-QUEST: The Highly Customized and Integrated System for IG and TR Standardized V-J and V-D-J Sequence Analysis-PubMed Available online: https://pubmed.ncbi.nlm.nih.gov/18503082/?from_single_result=Brochet+X%2C+Lefranc+MP%2C+Giudicelli+V.+IMGT%2FV-QUEST%3A+the+highly+customized+and+integrated+system+for+IG+and+TR+standard-+ized+V-J+and+V-D-J+sequence+analysis.+Nucleic+Acids+Res.+2008%3B36%3AW503-8. (accessed on Jun 15, 2020).
Greiff, V.; Miho, E.; Menzel, U.; Reddy, S.T. Bioinformatic and Statistical Analysis of Adaptive Immune Repertoires. Trends Immunol. 2015, 36, 738-749.
Yaari, G.; Kleinstein, S.H. Practical guidelines for B-cell receptor repertoire sequencing analysis. Genome Med. 2015, 7.
IgBLAST: An Immunoglobulin Variable Domain Sequence Analysis Tool-PubMed Available online: https://pubmed.ncbi.nlm.nih.gov/23671333/?from_single_result=Ye+J%2C+Ma+N%2C+Madden+TL%2C+Ostell+JM.+IgBLAST%3A+an+immunoglob-+ulin+variable+domain+sequence+analysis+tool.+Nucleic+Acids+Res.+2013%3B41%3AW34-40. (accessed on Jun 15, 2020).
Decombinator: A Tool for Fast, Efficient Gene Assignment in T-cell Receptor Sequences Using a Finite State Machine-PubMed Available online: https://pubmed.ncbi.nlm.nih.gov/23303508/?from_single_result=Thomas+N%2C+Heather+J%2C+Ndifon+W%2C+Shawe-Taylor+J%2C+Chain+B.+Decombinator%3A+a+tool+for+fast%2C+efficient+gene+assignment+in+T-cell+receptor+sequences+using+a+finite+state+machine.
+Bioinformatics.+2013%3B29%3A542-50. (accessed on Jun 15,
2020).cell+receptor+sequences+using+a+finite+state+
machine.+Bioinformatics.+2013%3B29%3A542-50. (accessed on Jun 15, 2020).
pRESTO: A Toolkit for Processing High-Throughput Sequencing Raw Reads of Lymphocyte Receptor Repertoires-PubMed Available online: https://pubmed.ncbi.nlm.nih.gov/24618469/?from_single_result=Vander+Heiden+JA%2C+Yaari+G%2C+Uduman+M%2C+
Stern+JN%2C+O%27Connor+KC%2C+Hafler+DA%2C+et+al. +pRESTO%3A+a+toolkit+for+processing+high-+throughput+sequencing+raw+reads+of+lymphocyte+receptor+repertoires. +Bioinformatics.+2014%3B30%3A1930-2. (accessed on Jun 15, 2020).
Bolotin, D.A.; Shugay, M.; Mamedov, I.Z.; Putintseva, E. V.; Turchaninova, M.A.; Zvyagin, I. V.; Britanova, O. V.; Chudakov, D.M. MiTCR: Software for T-cell receptor sequencing data analysis. Nat. Methods 2013, 10, 813-814.
Bolotin, D.A.; Poslavsky, S.; Mitrophanov, I.; Shugay, M.; Mamedov, I.Z.; Putintseva, E. V.; Chudakov, D.M. MiXCR: Software for comprehensive adaptive immunity profiling. Nat. Methods 2015, 12, 380-381.
Zhang, W.; Du, Y.; Su, Z.; Wang, C.; Zeng, X.; Zhang, R.; Hong, X.; Nie, C.; Wu, J.; Cao, H.; et al. IMonitor: A Robust Pipeline for TCR and BCR Repertoire Analysis. Genetics 2015, 201, 459-72, doi:10.1534/genetics.115.176735.
Greiff, V.; Bhat, P.; Cook, S.C.; Menzel, U.; Kang, W.; Reddy, S.T. A bioinformatic framework for immune repertoire diversity profiling enables detection of immunological status. Genome Med. 2015, 7, doi:10.1186/s13073-015-0169-8.
Laydon, D.J.; Melamed, A.; Sim, A.; Gillet, N.A.; Sim, K.; Darko, S.; Kroll, J.S.; Douek, D.C.; Price, D.A.; Bangham, C.R.M.; et al. Quantification of HTLV-1 Clonality and TCR Diversity. PLoS Comput. Biol. 2014, 10, doi:10.1371/journal.pcbi.1003646.
Bashford-Rogers, R.J.M.; Palser, A.L.; Huntly, B.J.; Rance, R.; Vassiliou, G.S.; Follows, G.A.; Kellam, P. Network properties derived from deep sequencing of human b-cell receptor repertoires delineate b-cell populations. Genome Res. 2013, 23, 1874-1884, doi:10.1101/gr.154815.113.
Larbi, A.; Pawelec, G.; Wong, S.C.; Goldeck, D.; Tai, J.J.Y.; Fulop, T. Impact of age on T cell signaling: A general defect or specific alterations? Ageing Res. Rev. 2011, 10, 370-378.
Li, G.; Yu, M.; Lee, W.W.; Tsang, M.; Krishnan, E.; Weyand, C.M.; Goronzy, J.J. Decline in miR-181a expression with age impairs T cell receptor sensitivity by increasing DUSP6 activity. Nat. Med. 2012, 18, 1518-1524, doi:10.1038/nm.2963.
Britanova, O. V.; Putintseva, E. V.; Shugay, M.; Merzlyak, E.M.; Turchaninova, M.A.; Staroverov, D.B.; Bolotin, D.A.; Lukyanov, S.; Bogdanova, E.A.; Mamedov, I.Z.; et al. Age-Related Decrease in TCR Repertoire Diversity Measured with Deep and Normalized Sequence Profiling. J. Immunol. 2014, 192, 2689-2698, doi:10.4049/jimmunol.1302064.
Qin, L.; Jing, X.; Qiu, Z.; Cao, W.; Jiao, Y.; Routy, J.P.; Li, T. Aging of immune system: Immune signature from peripheral blood lymphocyte subsets in 1068 healthy adults. Aging (Albany. NY). 2016, 8, 848-859, doi:10.18632/aging.100894.
Gibson, K.L.; Wu, Y.C.; Barnett, Y.; Duggan, O.; Vaughan, R.; Kondeatis, E.; Nilsson, B.O.; Wikby, A.; Kipling, D.; Dunn-Walters, D.K. B-cell diversity decreases in old age and is correlated with poor health status. Aging Cell 2009, 8, 18-25, doi:10.1111/j.1474-9726.2008.00443.x.
Britanova, O. V.; Shugay, M.; Merzlyak, E.M.; Staroverov, D.B.; Putintseva, E. V.; Turchaninova, M.A.; Mamedov, I.Z.; Pogorelyy, M. V.; Bolotin, D.A.; Izraelson, M.; et al. Dynamics of Individual T Cell Repertoires: From Cord Blood to Centenarians. J. Immunol. 2016, 196, 5005-5013, doi:10.4049/jimmunol.1600005.
An Immune Risk Phenotype, Cognitive Impairment, and Survival in Very Late Life: Impact of Allostatic Load in Swedish Octogenarian and Nonagenarian Humans-PubMed Available online: https://pubmed.ncbi. nlm.nih.gov/15972602/?from_single result=Wikby+A%2C+Ferguson+F%2C+Forsey+R%2C+Thompson+
J%2C+Strindhall+J%2C+Lofgren+S%2C+Nilsson+BO%2C+
Ernerudh+J%2C+Pawelec+G+and+Johansson+B.+An+immune+
risk+phenotype%2C+cognitive+impairment%2C+and+survival+
n+very+late+life%3A+impact+of+allostatic+load+in+
Swedish+octogenarian+and+nonagenarian+humans.+J+
Gerontol+A+Biol+Sci+Med+Sci.+2005%3B+5%3A556-565.
(accessed on Jun 15, 2020).
Doyle, K.P.; Quach, L.N.; Solé, M.; Axtell, R.C.; Nguyen, T.V. V.; Soler-Llavina, G.J.; Jurado, S.; Han, J.; Steinman, L.; Longo, F.M.; et al. B-lymphocyte-mediated delayed cognitive impairment following stroke. J. Neurosci. 2015, 35, 2133-2145, doi:10.1523/JNEUROSCI.4098-14.2015.
Gate, D.; Saligrama, N.; Leventhal, O.; Yang, A.C.; Unger, M.S.; Middeldorp, J.; Chen, K.; Lehallier, B.; Channappa, D.; De Los Santos, M.B.; et al. Clonally expanded CD8 T cells patrol the cerebrospinal fluid in Alzheimer’s disease. Nature 2020, 577, 399-404, doi:10.1038/s41586-019-1895-7.
Leng, J.; Goldstein, D.R. Impact of aging on viral infections. Microbes Infect. 2010, 12, 1120-1124.
Siegrist, C.A.; Aspinall, R. B-cell responses to vaccination at the extremes of age. Nat. Rev. Immunol. 2009, 9, 185-194.





