重视细菌耐药监测数据分析, 促进临床抗生素合理用药
作者:喻华 蒋黎
抗菌药物耐药(AMR)被认为是对于全球公共卫生的主要威胁,据估计,目前每年全球范围内耐药菌(包括结核菌)至少造成700,000人死亡[1, 2]。到2050年,全球范围每年将有1,000,000人死于耐药菌(包括结核菌)感染、疟疾和HIV感染,且亚洲和非洲将会是重灾区,90%的死亡会发生在亚非区域。据世界银行估计,到2050年,由于抗菌药物耐药,我们将会损失世界总GDP的3.8%[3]。因此,2015年,世界卫生大会批准了WHO的全球行动计划,以应对AMR,此全球行动计划的关键组成部分之一就是提高耐药监测能力[4]。
在这一大背景下,我国已经建立全国性的细菌耐药监测网络,也有各个省级的以及区域性、针对特殊人群(如儿科)的耐药监测网。但是耐药监测过程中仍然存在一些问题:药敏方法不统一、信息化程度不完备,以及临床沟通不足等等[5]。如果不对耐药监测数据进行准确的分析,并加强与临床的沟通,就无法为临床抗感染治疗提供及时有效的指导。同时通过耐药监测,可以对多重耐药菌(MDR),泛耐药菌(PDR)和全耐药菌(XDR)在医院环境内的流行播散进行预警,从而进行有效的感染预防[6, 7]。在2020年7 月,国家卫健委办公厅发布了“关于持续做好抗菌药物临床应用管理工作的通知”[8],通知中提出了六个方面的具体要求,包括:“依托信息化建设,提升细菌真菌药敏试验结果报告,提升耐药监测数据质量,做好细菌和真菌耐药情况的分析,为及时干预措施提供科学依据”。
在与临床的沟通中发现,耐药监测数据的有效传达对于促进抗菌药物的合理应用非常重要。通过深入分析耐药监测数据,可以发现不同地区、不同医院,以及同一医院内不同科室间的差异,如病原菌分布、MRDO发生率、重要抗菌药物敏感性差异等。通过对于不同感染部位,不同感染类型的深入分析和检测,可以为抗菌药物的管理和临床经验治疗提供更好的依据。
在耐药数据的解读上,CLSI专门提供了相关的规范性文件M39-A4[10],笔者就CLSI规范中的相关建议,以及在实际耐药监测数据分析中发现的一些问题进行说明。
一、数据的完整性
在进行耐药分析时,首先需要确保数据的完整性,目前国家细菌耐药监测网已经制定了专门的技术方案[13],提出了上报数据的基本需求。但是想要做好耐药监测,给临床提供更有价值的信息,还需要整合更多信息。CLSI M39文件中[10],对于耐药监测的数据完整性提出了更高的要求。
1. 患者:必备信息:唯一的患者编号;来源的医疗保健机构(当实验室同时为多个医疗保健机构提供服务时必须)推荐信息:年龄或出生日期(推荐);性别;标本采集时病人的所属科室和病区(如:外科,急诊等);给与患者的临床服务(如:外科,产科等);患者的入院日期。
2. 标本:必备信息:唯一的标本编号;标本类型*(中段尿、血液、脑脊液等);标本采集日期。
* 在统计时需要将不同目的送检的标本进行区分,并分别进行统计分析。如出于感控目的送检的肛周拭子,就需要标记为感控目的,同一般的感染病原菌区分开。推荐信息:标本来源的原始部位(如来自右腿伤口)。
3. 菌株:必备信息:鉴定结果(理想情况下需要鉴定到种水平,如果不能鉴定到种水平,需要区分到属或生物群的水平,如凝固酶阴性葡萄球菌,草绿色链球菌等)。
推荐信息:分离编号(特别是当在同一标本的培养中,分离到一个以上的分离株时);菌株鉴定结果完整,可以考虑将因菌株命名不一致造成的差异进行统一(如对于产气肠杆菌Enterobacter aerogenes,最近重新命名为产气克雷伯菌Klebsiella aerogenes,需要避免因细菌命名变更造成的错误);来自感控和临床的补充信息——是感染病原还是定植菌,是社区获得性还是医院获得性。
4. 药敏信息:必备信息:定量测试结果(抑菌圈直径或MIC值)以及最终药敏结果解释,如果使用专家系统,则需要保存专家系统规则(如对于肺炎链球菌,可能根据红霉素诱导克林霉素耐药的结果,将克林霉素的药敏结果从S更改为R,则需要保存此条药敏结果);完整的药敏测试结果,应尽量将所有抗菌药物敏感性结果导入分析数据包中;药敏方法(KB法或MIC法)
推荐信息:使用的药敏检测系统(如商品化药敏系统…),是否可以根据折点变化来重新分析数据。
二、数据准确性
为了确保最后的耐药数据分析的准确,必须参考一定的标准,如何验证数据的准确性,如何处理重复数据等:
1. 数据准确性验证:最终统计分析药敏数据必须经过验证。许多LIS系统,微生物系统中间件和药敏专家系统都具有此功能,可以自动检测数据,对异常结果进行警告。实验室人员也需要加深对于细菌耐药机制和罕见的药敏结果的认识,这样可以迅速发现不合理药敏结果。如在CLSI M100[11]文件中,附录A中就列举了罕见的耐药表型,同时我们在日常工作中也了解的目前国内的基本耐药情况,可据此对数据进行审核。如目前国内尚无万古霉素耐药的金黄色葡萄球菌的分离报告,如果分离到万古霉素中介或耐药的金黄色葡萄球菌需要复核确认;又如对于大肠埃希菌,阿米卡星的体外抗菌活性优于妥布霉素和庆大霉素,在耐药分析中如果大肠埃希菌对于阿米卡星的耐药率高于庆大霉素和妥布霉素,这种情况是不常见的。
2. 建议统计频率:建议每年至少进行一次耐药数据分析,当分离到足够的病原菌、新药物上市并提供药敏测试,或在院感以及临床上存在重要变化时,可以增加统计分析的频率。但需要注意的是,感染病原菌存在季节性差异(如:下呼吸道的感染病原菌)。在做病原菌分析时,必须需要考虑到病原菌季节变化的影响。(如分析病原菌组成时,四季度数据直接和三季度比较,有可能会有季节因素影响,和去年四季度同期进行比较,可以排除季节性因素的干扰。)
3. 菌株分析原则:菌株统计和分析的原则:从同一患者的连续培养物中多次培养到同一种菌的多个分离株,这些分离株有可能是同源的一株菌,也有可能是不同源的不同株。出于感控、质量保证、评估稀有表型,以及检测患者在治疗过程中耐药性的变化等目的而言,必须要将所有分离株纳入整体的分析数据库,一旦省略这些分离株,可能会导致与细菌种群相关信息的大量丢失。但是在计算耐药率时,来自单个患者的多个分离株可能会造成耐药率的不准确,特别送检较为频繁的患者中的分离株比例较高,可能会高估耐药率。
因此CLSI M39建议在每一次计算分析耐药率时,特别是此耐药分析报告用于协助临床进行抗感染经验治疗时,对于每位患者,无论分离自何部位、药敏结果和其他表型特征(如生物型等等),仅统计第一次分离到的分离株。同时M39也强调这个仅仅是一种耐药率统计计算的分析方法,此外还有一些其他的耐药率计算方法可供参考:如“最耐药的分析”——对于某种细菌,在进行统计分析时仅纳入针对单个药物耐药性最高的分离株。这种统计方法常用于MDRO的统计分析,如对于MRSA的分离率进行统计分析。此外还有“加权平均法”;将单个患者的所有耐药率进行平均计算,而后将累计耐药率中单个患者的耐药率进行平均统计分析。此外可以设定一定的间隔,如7天、30天,将经过这个间隔的分离株分别进行统计。
还有非常重要的一点,需要将处于感控目的送检的标本以及分离株和处于诊断目的检测送检的分离株的耐药率分开进行统计。如下图1中,某科室送检的标本17.6%为拭子标本,主要出于感控目的送检的肛周拭子、鼻拭子和咽拭子,这些标本需要与出于诊断目的送检的病原菌分开进行统计。
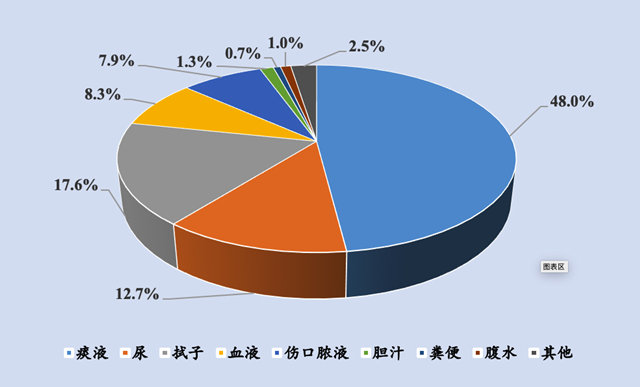
图1. 某科室标本类型分析
4. 药敏分析:在进行药敏分析时,首先需要避免选择性药敏的测试问题,这样会造成耐药率极大的偏差,胡付品教授[12]曾经指出过这种情况:某院连续进行了6年头孢哌酮/舒巴坦的耐药监测,其中2010、2011和2014年,是所有菌株进行头孢哌酮/舒巴坦的敏感性测试,而2012、2013和2015年是对于碳青霉烯耐药的不动杆菌加做头孢哌酮/舒巴坦的药敏结果,这就会造成耐药率极大的偏差,导致无法正常分析数据。
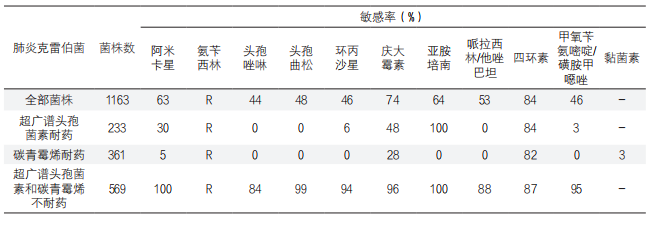
此外,CLSI中有一些替代药物,在进行耐药统计时,规范统计的药物和报告的方式,如使用头孢西丁纸片检测金黄色葡萄球菌对于苯唑西林的耐药率,在统计时需要计算MRSA发生率,而不是头孢西丁耐药率。而在实际工作中,实验室确实有可能根据临床需求对于部分分离株进行了选择性的药敏测试,如对于碳青霉烯耐药的肺炎克雷伯菌,额外增加黏菌素的敏感性测试,这种情况有可能造成耐药性增高。对于这种情况,建议可以将细菌按重要的耐药机制进行分类,再统计其耐药率。如将肺炎克雷伯菌区分为三代头孢敏感株;三代头孢耐药株和碳青霉烯耐药株,仅碳青霉烯耐药株统计耐药性。如下表中对于肺炎克雷伯菌耐药率进行了统计,仅CRKP补充了黏菌素,所以单独进行了耐药率统计分析,但是切记不能把这个耐药率等同于所有肺炎克雷伯的耐药率。
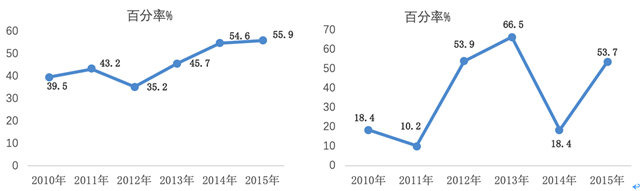
图2. 某院2010-2015年鲍曼不动杆菌耐药率变迁
注:图2左某院鲍曼不动杆菌对于亚胺培南耐药率的变迁;
图2右某院鲍曼不动杆菌对于头孢哌酮/舒巴坦耐药率变迁。
表1. 肺炎克雷伯菌敏感率分析示意图
5. 统计敏感率:在统计敏感率时,特别是对历史耐药数据进行分析时,需要考虑折点更新的因素并进行注明,或分别统计,这样可以更明显地展示细菌耐药率的变迁趋势,如在下图中[10],对于历年来肺炎链球菌的对于青霉素的敏感性进行了分析,但是由于期间肺炎链球菌青霉素的折点进行过变更,所以特地根据新、老折点分别绘制了曲线,这样可以排除折点变更这一因素,更加客观的反映细菌耐药性的变迁趋势(图3)。
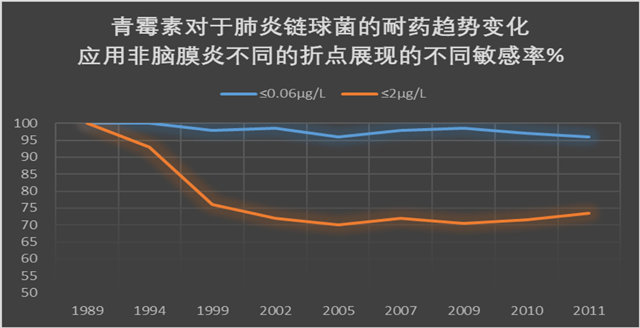
图3. 青霉素对于肺炎链球菌的耐药趋势变化-应用非脑膜炎不同的折点展现的不同敏感率
对于在使用商品化的药敏系统时,必须要考虑到MIC的检测范围是否能覆盖新、老折点,如美罗培南在2010年调整了折点,敏感折点从4μg/ml下降到1μg/ml;当使用商品化系统美罗培南检测下限为2μg/ml时,就无法直接使用新折点,对于所有标记为≤2μg/ml的药敏结果无法区分中介还是敏感。当使用软件进行分析时,可直接标记为“当前标准无法解释”。在调整折点时必须注意此类问题,同时尽量选择MIC测试浓度更广的药敏检测系统,可以减少此类情况的发生。
在进行数据统计时,需要注意MIC值的表述方式,当表述方式不同,可以造成统计的偏差,特别是当折点以及MIC<1μg/ml时,如敏感折点为≤0.12μg/ml,中介折点为0.25-2μg/ml;当药敏系统报告某菌MIC=0.125μg/ml,在使用软件进行分析时,实验室有可能错误的将这些结果解释为耐药。对于这个问题,CLSI M100文件[11]中已经有了专门的说明,当MIC<1μg/ml时,仅保留2位有效数字,需要按规范报告和记录MIC结果。
使用ETEST或类似产品时,可能会报告非2的整数次幂的 MIC结果。如出现报告MIC=1.5,3,6μg/ml情况。在统计MIC分布时,可以直接按实际报告MIC值进行统计分析。但是在记录药敏分类解释结果时主要将此MIC值上调至2的整数次幂后,再给出药敏分类解释结果。如当敏感折点为≤2μg/ml;中介折点=4μg/ml;耐药折点为≥8μg/ml;当MIC检测为6μg/ml,需要将其视为8μg/ml,报告为耐药,而不能将其归类为中介。
三、统计分析报告
1. 数据验证:目前大部分实验室的LIS、药敏检测系统及其中间件均可以进行累计耐药率分析,因此在分析耐药数据时,可以使用多个系统同时分析统计,如WHONET、LIS和药敏系统中间件。确认其耐药率是否一致,如果不一致,差异在哪里,造成差异的原因是什么。排除差异后,可以确保耐药数据在统计过程中的准确性。
2. 在统计分析报告中,还有一些事项需要注意,如报告的抗菌药物种类和范围;抗菌药物的缩写和表达方式,天然耐药的统计等。同时也需要注意结合临床实际需求,因为此类报告的作用是支持临床抗感染经验治疗,协助抗菌药物合理应用,需要有针对性的进行分析。
(1)报告抗菌药物种类和范围:CLSI M100[11]文件中的表格1推荐了药敏实验应该报告的药物种类;是在耐药率分析时报告抗菌药物种类的一个重要参考;也可以参考每年更新全国细菌耐药监测网技术方案[13]。同时需要考虑到临床需求,特别是耐药分析报告是否能覆盖临床经验治疗中常用的抗菌药物。如下图中某院对呼吸道分离的流感嗜血杆菌的耐药情况进行了分析,但是目前对于社区获得性肺炎,临床最常用的经验治疗方案口服喹诺酮、阿莫西林/克拉维酸等[12],此耐药分析中缺少此类药物的敏感性分析,不能给与临床经验治疗以支持。
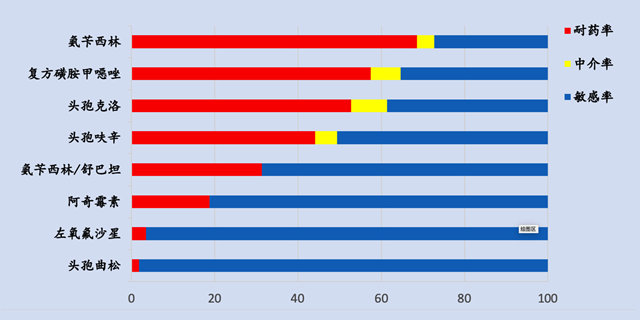
图4. 某院对于呼吸道分离的流感嗜血杆菌耐药率分析
(2)对于天然耐药和耐药机制监测规范报告:对于天然耐药,在耐药分析报告中可以直接标记为R,不进行耐药率的统计分析,具体的天然耐药列表,可以参考CLSI M100文件中的附录B[11]。对于一些重要的耐药机制也需要规范报告,如对于肠球菌的高浓度庆大霉素实验(HLAR),不能直接记录统计为庆大霉素耐药率,需要特别注明为高浓度庆大霉素耐药性检测实验。
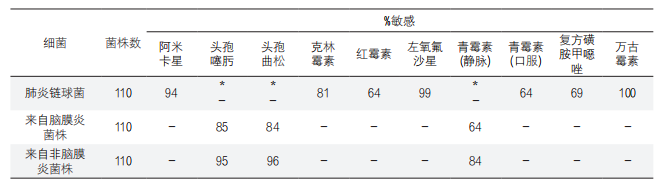
(3)复杂折点的分析:部分细菌对于特定的抗菌药物可能有多个折点,如肺炎链球菌对于青霉素具有脑脊液、非脑脊液和口服三个折点,在进行耐药率分析时,需要根据三个不同折点计算耐药率。如下表示例:
表2. 肺炎链球菌敏感率分析示例
(4)重要的耐药菌分析:需要对于常见的 MRDO的发生率进行统计和分析,包括MRSA,MRCoNS,VRE,CRE等等,同时需要分类进行统计和分析,在上文中的表1示例,将肺炎克雷伯菌分为了三代头孢耐药,碳青霉烯耐药,以及对于三代头孢和碳青霉烯均敏感的三大类,并分别统计耐药率。3.2.5进阶分析:
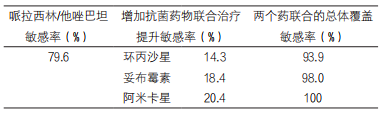
在实际的经验治疗中,对于重症患者,临床常用广覆盖的经验治疗策略,联合1种以上的抗菌药物进行治疗。分析经验治疗的常用抗菌药物组合,有助于临床选择覆盖面更广,成功率更高的治疗方案。但是在沟通前需要强调,此数据仅仅是多种抗生素耐药数据的简单统计分析,并未考虑到在实际治疗过程中药物间潜在的协同或拮抗作用,同时也并不意味在实际治疗过程中,多种药物的组合必然优于1种药物。如下表3示例中分析了血流感染中分离到的977株革兰阴性菌的耐药率分布,使用哌拉西林/他唑巴坦对于79.6%的病原菌为敏感,加上环丙沙星后,可以覆盖93.9%的病原菌;加上阿米卡星可以覆盖全部的革兰阴性菌。
表3. 某院对于血流感染革兰阴性菌分析(2015年,n=977)示例
小结:在抗感染治疗过程中,早期经验治疗是很重要的一环。耐药监测数据的总结和分析,可以协助临床优化早期经验治疗方案。特别是对于本院特定科室的耐药监测数据的分析,更具针对性,对临床的帮助更大。但是在数据整理和分析的过程中,需要注意核对数据,避免传递有偏差的信息。注重临床反馈和沟通,才能利用好数据协助临床,为抗菌药物的合理应用服务。
参考文献
O'neill J. Antimicrobial resistance[J]. Tackling a Crisis for the Health and Wealth of Nations, 2014.
Limmathurotsakul D, Dunachie S, Fukuda K, et al. Improving the estimation of the global burden of antimicrobial resistant infections[J]. The Lancet Infectious Diseases, 2019, 19(11): e392-e398.
Adeyi O O, Baris E, Jonas O B, et al. Drug-resistant infections: a threat to our economic future[J]. World Bank Group, Washington, DC, 2017.
World Health Organization. Global action plan on antimicrobial resistance[J]. 2016.
胡付品, 朱德妹. 我国细菌耐药监测工作需要进入2.0时代[J]. 中国感染与化疗杂志, 2018,18(02):129-131.
汪复. 应加强细菌耐药性和耐药菌感染的研究力度[J]. 中华医学杂志, 2006,86(9):579-580.
朱德妹. 进一步加强细菌耐药监测 [J]. 中华检验医学杂志, 2006,29(10):865-866.
国家卫生健康委办公厅. 国家卫生健康委办公厅关于持续做好抗菌药物临床应用管理工作的通知. 国卫办医发〔2020〕8号,2020.
医政医管局. 《国家卫生健康委办公厅关于持续做好抗菌药物临床应用管理工作的通知》解读
Guideline A, Edition F. CLSI document M39-A4[J]. Wayne, PA: Clinical and Laboratory Standards Institute, 1995.
Clinical and Laboratory Standards Institute. 2020. Performance standards for antimicrobial susceptibility testing, 30th ed. CLSI supplement M100. Clinical and Laboratory Standards Institute, Wayne, PA.
瞿介明, 曹彬. 中国成人社区获得性肺炎诊断和治疗指南 (2016 年版) [J]. 中华结核和呼吸杂志, 2016 (2016 年 04): 241-242.
http://www.carss.cn/





