西格玛度量应用于HBV DNA 定量分子检测
慢性乙型肝炎病毒(hepatitis B virus,HBV)感染对患者构成重大健康风险。患者HBV DNA水平已被证明与肝细胞坏死程度相关,增加了发展为肝细胞癌及死亡率的风险。因此,HBV DNA检测在患者的诊断和管理中有着重要的作用[1]。
对于HBeAg阳性和阴性患者的HBV DNA的医学决定水平分别是20000IU/ml和2000IU/ml。优化慢性乙肝的管理需要由定量PCR设定的HBV DNA基线,后者随后可用于监测与病毒抵抗力相关的响应和病毒反弹[2]。HBV DNA检测通常需要延续几个月至几年的时间,因此一个稳定的分析系统对于得到可重复的和可比较的结果是至关重要的。虽然达到这一目标最直接的方法是使用已批准的试剂盒而不是自建的方法,但是美国临床和实验室标准化研究院(CLSI)要求对于整个系统来说至少需要一个长期的独立的分析质量控制,即使是对于已批准的检测方法。在最近几十年,诊断实验室已经研究了质量控制(quality control,QC)方法的不同方面,如QC方法选择、误差检出概率和假失控概率。在QC系统中,误差检出概率和假失控概率取决于QC材料的选择、靶值、质控品检测频率和QC规则[3]。
与常规生化方法相比,定量PCR的统计QC还有待进行综合的研究[4]。本研究的目的是确定HBV DNA检测的性能特征,并进一步为实时定量PCR设计统计QC系统。
1 方法
1.1 研究设计
HBV感染患者治疗的临床最小阈值是在HBV DNA水平1log(IU/ml)的改变。换句话说,抗病毒治疗的疗效定义为一种抗病毒药物导致HBV DNA水平从基线至少减少1log(IU/ml)。因此总误差如果小于1 log(IU/ml)将不会影响临床决策。由于其他参数包括血清的微小改变也可能会影响HBV DNA水平,所以随意值0.5log(IU/ml)作为本研究中试验的最大允许总误差(total allowable error,TEa)。根据这个假设,在4.2log(IU/ml)和3.2log(IU/ml)处测量系统误差和随机误差的值,这两个数值均在临床决定水平附近。
统计计算中,对血浆中HBV DNA值进行对数转换。这是有效的换算,因为转换的数值可能会服从正态分布。在标准化的操作过程规范(operational process specifications,OPSpecs)图上可以确定操作点。最后,可以评价分析方法的性能和选择候选QC程序。
1.2 系统误差测量
冻干的二级WHO标准品是从英国国家生物标准和控制研究所(赫特福德郡,英国)购买,其浓度水平为106IU/ml。按照制造商说明书,使用A级移液管加入0.5ml的蒸馏水复溶该标准物质。然后使用A级移液管加入100μL复溶标准物质到5ml HBsAg阴性血浆制备稀释液,以获得HBV DNA浓度为19608IU/ml。接下来,将500μL稀释标准物质加入5ml HBsHg阴性血浆中以获得HBV DNA浓度为1783IU/ml。最后对每个浓度水平制备成200μL等份样品并冰冻在-20℃条件下。
在五天时间内每天分别取出每个水平标准物质一式两份的样品,用QIAGEN Artus HBV RG PCR检测试剂盒进行定量分析。计算每个水平的均值与靶值的差值。差值转换为对数后作为系统误差的估计值。
我们应该注意到病毒载量试验趋向于没有偏倚。也就是说一种方法的偏倚并不是那么重要,因为患者是用一种方法检测的,所以检测结果随着时间的改变比某个特定的截断值更重要。因此对于大多数临床情况来说,我们可以考虑使用有效偏倚为零的σ度量。
1.3 随机误差测量
将HBV DNA阳性和阴性人血浆进行混合来制备两种血浆样品,其HBV DNA水平为2log IU/ml和4log IU/ml。对每个水平制备200μL等份样品,并冷冻在-20℃下。超过15个月,分别取出以上描述的每个水平20份样品,用QIAGEN Artus HBV RG PCR 检测试剂盒进行定量分析。以对数表示的每个浓度水平的检测结果的总标准差(total standard deviation,st)作为随机误差的估计值。
为了确定PCR阶段的标准差,每个水平14份纯化的样品混合后再分为几等份,然后在5个不同的PCR批中进行检测。因为通过混合可以忽略纯化的变异,这样测量只反映PCR阶段的变异。
1.4 统计计算
采用单样本Kolmogorov–Smirnov 检验来证实HBV DNA水平对数转换后(log HBV)是服从正态分布。通过将离群值定义为偏离均值±3.5s的值,来剔除离群值后,我们可以获得每个控制水平log HBV DNA的均值、标准差(s)和变异系数(CV)。采用自助抽样(bootstrapping)方法计算均值、s和CV的95%置信区间。方法的总不精密度主要来自DNA纯化和定量PCR过程。因此,纯化阶段的不精密度计算公式为:,其中st指的是总标准差,se和sp分别指的是纯化阶段和PCR阶段的标准差。HBV DNA高低水平的纯化阶段的不精密度比较采用F检验。考虑到本研究中的样本量,将犯Ⅰ型错误概率定为0.05,F值大于2.46为有统计学意义。采用SPSS 18.0(2009)进行统计分析,P小于0.05为统计学意义。
1.5 标准化操作过程规范图
标准化的OPSpec图可以按照Westgard的描述进行绘制。简单的过程为:计算每个水平的观察系统误差(%)和变异系数(%),并通过除以TEa即0.5log(IU/ml)进行标准化。然后将每个水平的操作点绘制到OPSpec图中(见图1)。
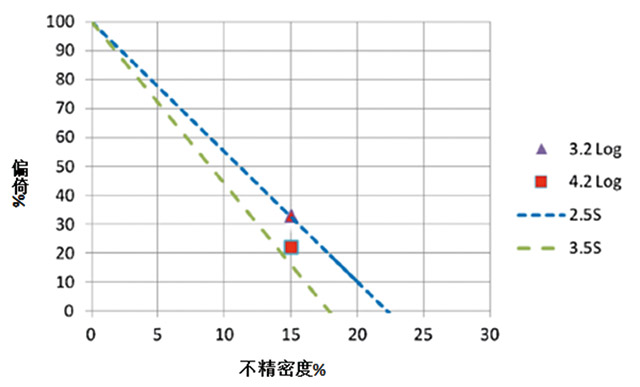
图1 90%分析质量保证的标准化OPSpecs图;3.2log,3.2log(IU/ml); 4.2log,4.2log(IU/ml);2.5s,12.5s;3.5s,13.5s;两个质控结果
2 结果
批间随机误差测量的结果是通过2011年1月12日到2012年4月12日的质控数据获取(见图2)。系统误差测量结果来源于2012年4月和5月的数据。HBV DNA值对数转换后显示服从正态分布(Kolmogorov–Smirnov 检验, P>0.050。
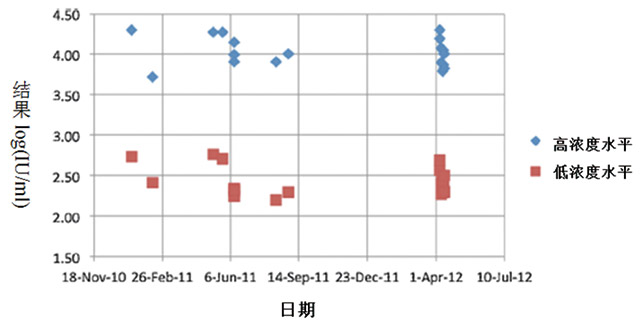
图2 散点图显示了4.2和3.2log(IU/ml)两个水平结果的随机变异
在低浓度质控水平测量的系统误差和随机误差分别为:0.33log(IU/mL)或11%(0.33/2.9);0.17log(IU/mL)或7%(0.17/2.43)。在高浓度质控水平测量的系统误差和随机误差分别为:0.22log(IU/mL)或5.4% (0.22/4.07);0.17log(IU/mL)或4.2%(0.17/4.00) (见表1)。超过16个月结果的统计变异显示在图2中,定量PCR阶段的QC参数如表1所示。对于HBV DNA水平为4.07log(IU/mL)和2.9log(IU/mL),在纯化阶段的标准差分别为0.16和0.13,差异没有统计学意义(F=1.56)。对于所描述的DNA病毒载量试验方法,本研究中使用的OPSpecs图显示采用单个12.5S QC规则且两个水平的质控品,其误差检出率是可接受的(见图1)。
表1 批间、PCR和纯化阶段的质量控制参数
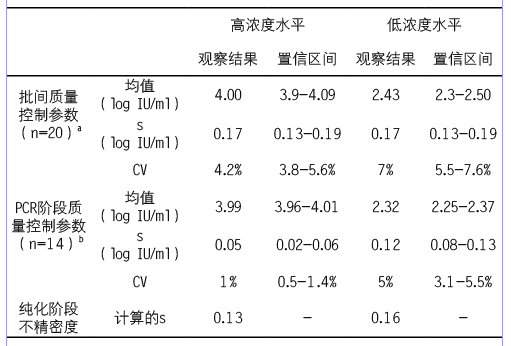
a 批间随机误差测量结果是通过从2011年1月12日到2012年4月12日质控结果中获取。HBV DNA高浓度水平(4log)和低浓度水平(2log)血浆样品是用HBV DNA阳性和阴性人血浆混合制备。
b 14份每个浓度水平纯化的样品单独混合,分为几个等份,并在5个不同的PCR批中检测。
2.1 西格玛度量应用于HBV DNA定量检测的性能评价
2.1.1 单位换算
0.5log(IU/ml)作为本研究中试验的最大允许总误差,换算之后其目标为216.23%。log决定点与常规数值的转换为:低浓度水平:2.43 log=269;2.93log=851.1;2.43log与2.93log的差异是0.5log,但也是常规数值582。常规数值为269水平的582的TEa=216.23%;高浓度水平:4log=10,000;4.5log=31,622.8;4.5log与4.0log的差异是0.5log,但也是常规数值21622.78;常规数值为1000水平的21622.78的TEa=216.23%。TEa值应该与%表示的常规数值单位相匹配,因为TEa是以log单位表示的。
2.1.2 σ度量的计算
σ度量计算公式中同时考虑不精密度和偏倚。我们将同时使用”Ricos目标”和CLIA目标来计算σ度量。记住σ度量的计算公式为(TEa-bias%)/CV
低浓度水平的HBV:TEa=216.23%,不精密度为47.9%,偏倚为39.7%,其σ度量=(216.23-39.7)/47.9=176.53/47.9=3.68σ;高浓度水平的HBV:TEa=216.23%,不精密度为47.9%,偏倚为53.2%,σ度量=(216.23-53.2)/47.9=163.03/47.9=3.40σ。如果将偏倚设为0,σ度量变为:低浓度水平的HBV:σ度量=216.23/47.9=4.5σ;高浓度水平的HBV:σ度量=216.23/47.9=4.5σ。
2.1.3 西格玛度量方法性能决策图和OPSpecs图
我们可以使用标准化的西格玛度量方法决策图来对性能进行直观的评价,如图3所示。
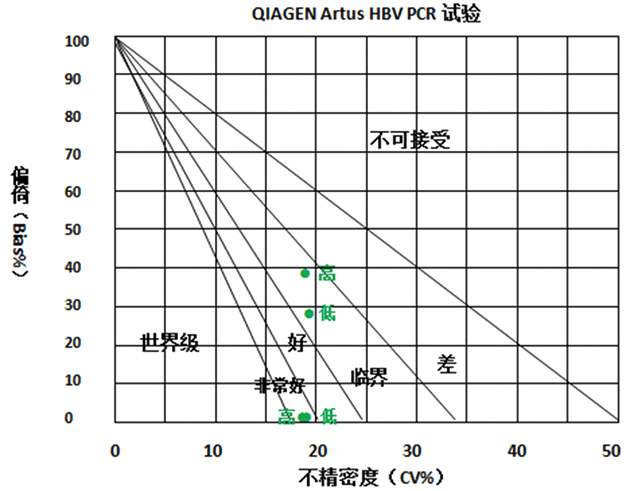
图3 标准化的西格玛度量方法决策图评价QIAGEN Artus HBV PCR试验性能
为了对检测方法进行控制和监测,我们需要一个标准化的OPSpecs图(如图4)
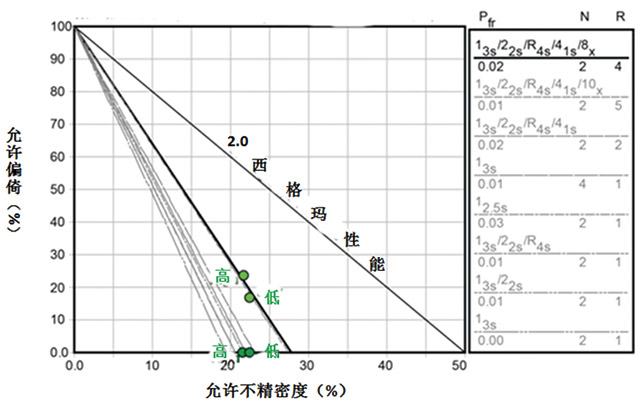
图4 TEa=100.0%具有90%分析质量保证(SE)的标准化OPSpecs 图
如果我们将偏倚考虑进去,我们需要引入大量的由8到10个检测结果建立的”Westgard 规则”。如果每个批只检测2个质控品,这意味着往回看3到4个批。如果我们期望是单独使用病毒载量结果(没有其他检测方法比对),我只需要不包含8x或10x的一套“Westgard 规则”,并且只需要往回看一个批次。
3 讨论
对于慢性乙肝,临床上根据HBV DNA的绝对数量或相对数量改变来做出临床决策。对于前者,系统的总误差,包括随机和系统误差,要控制在可接受范围内。为了实现这个目标,可通过使用质量控制材料和对测量程序进行统计评价来监测分析系统。另一方面,当临床决策是根据HBV DNA水平的改变来确定时,重要的是要把患者HBV DNA水平的真实改变从由试验变异导致的改变中区分开来。
实施统计过程控制包括:QC材料的选择,确定标准差(s)和均值及QC规则的选择。如图2所示,HBV DNA含量在-20℃可以稳定好几个月,因此将血浆等份分装可用作QC材料。虽然每个实验室必须建立自己的s和均值,在本实验室,在HBV DNA水平为3.2log(IU/ml)和4.3log(IU/ml)确定s,结果如图2所示。另外,分析系统的性能由操作点在OPSpecs图中的位置决定。如图1所示,单个12.5S规则并具有两个水平的质控品,具有90%的可信度检出误差。
假设系统误差是恒定的,当HBV DNA水平改变明显超过了试验变异可认为是真实的变化。为了区别HIV-RNA拷贝数真实改变和试验变异,Yen-Lieberman和他同事根据以两个数值(Z1-α/2+Z1-β)= log(X1/X2)/(1.4×SD)表示的样本量计算最大的标准差。使用同样的表达式,发现两次结果1log(IU/ml)(也就是10倍)HBV DNA水平的改变的最大标准差是0.22log(IU/ml),获得第I类型误差大小为0.05,第II类型误差大小为0.10。换句话说,如果试验报告的结果的SD超过0.22 log(IU/ml)可能导致试验中不应该带来的实验室误差造成的临床错误决策。同时,即使统计QC方法中s可接受,s也是越小,误差检出概率也就越高。
本研究中,人工纯化是高低HBV DNA水平变异的主要来源,这与其他研究结果也是一致的。另外,人工纯化造成HBV DNA的低估,是系统误差的来源。因此,本研究中恒定的负偏倚部分也归因于人工纯化过程。改成自动化纯化将不仅提高方法的整体性能,还可以降低样品间的携带污染。
但是定量PCR中的变异给整体不精密度造成较小影响,其中较低水平的HBV DNA的统计变异较大。当样品中目标分子较少时,引物二聚体和非特异性产物将与靶分子竞争从而导致效率降低。因此获得更高PCR性能的方法是通过增大提纯过程中目标DNA浓度来降低PCR反应的CT值。而提高DNA浓度可能需要增加最初血浆提取的体积。
总的来说,使用长期的质控品和简单的QC规则来监测检测系统的关键变化和防止错误的实验结果,否则会导致误诊和处置失当。另外,本文中描述的手工纯化方法的性能较好。然而,改成自动纯化可能降低携带污染和提高系统整体性能。
参考文献
[1] Gish, R.G., Locarnini, S.A. Chronic hepatitis B: current testing strategies. Clinical Gastroenterology and Hepatology, 2006(4), 666-676.
[2] Espy, M., Uhl, J., Sloan, L., Buckwalter, S., et al. Real-time PCR in clinical microbiology: applications for routine laboratory testing. Clinical Microbiology Reviews 2006(19), 165-256.
[3] Kubista, M., Andrade, J.M., Bengtsson, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine 2006(27), 95-125.
[4] Caliendo, A.M., Valsamakis, A., Bremer, J.W., et al. Multilaboratory evaluation of real-time PCR tests for hepatitis B virus DNA quantification. Journal of Clinical Microbiology, 2011(49), 2854-2858.




