真菌持留菌发生机制与耐药性检测分析
作者:侯欣 陈新飞 徐英春 李永哲
【摘要】侵袭性真菌感染的发病率和病死率高,但治疗选择十分有限。耐药真菌感染的发生率逐渐升高,给临床治疗带来巨大挑战。更重要的是,有相当一部分患者即使感染了对药物敏感的真菌,仍对治疗无反应,可能与菌株对抗真菌药物的耐受性有关。真菌持留菌是一小群对高浓度抗真菌药物有高度耐受性的细胞,是非增长的、野生型细胞的表型变异,与耐药性的发生发展和临床顽固性感染相关。持留菌具有高度异质性和可塑性,能够影响真菌生物膜的形成。真菌持留菌的研究是关注真菌在形成稳定耐药之前的表型和分子机制,为控制真菌耐受发生发展、遏制真菌耐药和研发新药提供新的思路。
真菌感染的发病率和病死率高,每年导致约150万人的死亡,与结核、HIV数量相当,高于疟疾、乳腺癌和前列腺癌[1]。而抗真菌药物种类十分有限,主要有四大类:唑类、多烯类、棘白菌素类和嘧啶类。因此,病原真菌对任何一种药物耐药,都会给临床治疗带来巨大挑战。临床治疗选择有限,并且抗真菌药物的耐药性正在增加。耐药真菌感染已成为一个全球关注的问题,特别是“超级真菌”——耳念珠菌感染的爆发和非白念珠菌感染比例的不断升高,引起临床医生、流行病学家和感控专家的重视。抗真菌药物预防治疗及长期使用诱导耐药的相关问题已引起国内专家学者们的关注。此外,尽管有相当一部分患者感染了对药物敏感的真菌,但治疗效果欠佳,可能与宿主免疫状况和/或药理等非真菌学因素相关[2],药物与真菌之间的特异性相互作用也可能影响治疗反应。总体治疗结果欠佳与临床低耐药率之间的偏差可能归因于抗真菌药物的耐受性。因此,探索真菌耐药发展的过程和相关机制,对于寻找有效的措施和手段,遏制耐药的发生与发展和快速筛查耐药靶点具有非常重要的意义,是针对性开发新型抗真菌感染治疗策略的前期基础和重要保障。
一、真菌持留菌的发现
持留菌(persister)现象发现于1944年,Joseph Bigger等发现金黄色葡萄球菌在用青霉素处理时会产生低代谢活性的持留菌细胞[3]。60年后,人们才逐渐确定,这些非生长状态的持留菌细胞正是抗生素不能完全清除细菌的主要原因[4]。细菌持留菌形成的诱发因素较多,包括抗生素、营养缺乏、氧气不足等逆境压力胁迫,相关研究发现持留菌的形成与药物胁迫有直接联系[5]。细菌持留菌在抗生素胁迫下,通过一些通路的开启与关闭,发生表型转换,并以一种特殊状态逃避抗生素的杀伤作用,从而有利于菌株获得耐药基因,在铜绿假单胞菌[6]、大肠埃希菌[7]和结核分枝杆菌[8]中均有所证实。药物耐受(tolerance)不仅能够增加细胞存活和突变数量,而且提高突变率和转变为耐药菌的概率[9],耐受与耐药(resistance)的发展密切相关[10]。然而,真菌持留菌方面的研究还有待开展。
真菌持留菌是一小群对高浓度抗真菌药物有高度耐受性的野生型细胞,是具有休眠特点的表型异质体,可能参与了真菌感染的耐药与复发[11]。国际上针对真菌持留菌的研究甚少。与耐药不同,真菌持留菌的耐药特点不是基因突变所赋予的,因而不具有遗传性。研究显示:真菌持留菌的形成受营养感知和代谢控制协同调控。白念珠菌持留菌的蛋白组学研究发现持留菌能够依靠替代碳源生存,有利于适应葡萄糖不足的环境[12]。此外,持留菌细胞能够适应氧化应激压力,使细胞免受药物诱导的活性氧簇(reactive oxygen species,ROS)产生的损害[13]。压力应激保护剂如糖原和海藻糖能够通过稳定蛋白质来保护细胞,起到化学分子伴侣的作用。而持留菌细胞中积累了大量的压力应激保护剂,使其在ROS诱导的细胞凋亡中存活。
二、抗真菌药物耐受性的检测
抗真菌药物耐药性的检测有标准的参照方法,包括CLSI和EUCAST相关文件,主要包括:纸片扩散法、E-test法和微量肉汤稀释法。抗真菌药物耐受性的检测采用相似的方法,通常在菌株生长48h后进行判读。在纸片扩散法中,利用专业软件diskImageR计算特定平均半径(radius,RAD)抑制区域内(通常为20%或50%抑制)的生长分数(fraction of growth,FoG),是相对于最大可能区域(在选定的RAD水平,对于RAD20图示为虚线矩形,图1a)曲线下区域的像素强度(图1a的粉色区域)。具有相似敏感性水平的菌株可表现出不同的耐受水平。在距纸片一定距离的范围内,耐受增长与密度相关,可用相似的像素强度来衡量。微量肉汤稀释法中,48h分析结果也提供了一种耐受性指标,称为超MIC生长(supra-MIC growth,SMG)[11]。SMG以阳性生长对照孔作标准化,用高于MIC的每孔平均生长(以光密度衡量)进行量化(图1b)。E-test法可用抑制区内的生长相对于抑制区外的生长所占的比例,比较不同菌株的耐受性(图1c)。与液体培养基方法相比,固体培养基测定法(图1a和1c)可较直观显示持留菌生长的情况,肉眼观察到单菌落以及药物浓度的影响。
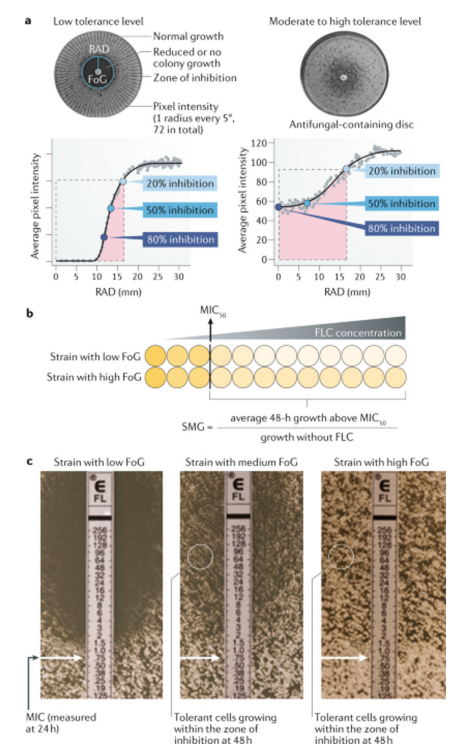
图1. 抗真菌药物耐受性的检测[16]
三、真菌持留菌的表型特点
真菌持留菌是药物敏感菌株的特征,在抑制药物浓度下具有缓慢生长的能力。通常,给定群体中只有一小部分细胞能够进行这种缓慢生长。持留菌与耐药菌的区别:持留菌的生长比耐药菌的生长慢且时间依赖性强,并且对药物浓度的依赖性较小[14]。不同的临床分离株可表现出不同的耐受水平,在纸片扩散法或微量肉汤稀释法中,可表现为48h时,高于MIC生长的细胞比例不同(图1)。耐受程度与超过MIC的持留菌的菌落大小以及细胞形成可见菌落的速率有关[14],表明持留菌是由于种群中某些细胞的缓慢生长所致。
耐受程度在不同分离株之间有所差异,可能是分离株固有等位基因的多样性所致。临床分离株之间的表型差异涉及成千上万的单核苷酸多态性,并且在生物膜形成以及对特定转录因子的依赖性等表型上也有明显差异[15]。从该意义上说,以分离株之间FoG或SMG(见图1)衡量的耐受水平差异与遗传相关。但对于单克隆分离株而言,驱动某些细胞缓慢生长而阻止其他细胞生长的表型异质性归因于群体中同基因细胞之间的生理或代谢变化。
四、真菌持留菌产生的机制
真菌持留菌产生机制的相关研究较少。除了不同菌株之间的异质性外,种群中也存在异质性。在氟康唑胁迫下,白念珠菌在生长停滞前,可进行1-3次分裂生长[14],群体中的持留菌可在数小时后恢复生长,而非持留菌停滞时间更长,并且仅可在不使用药物的情况下恢复分裂。因此,耐受性可能是细胞群体中异质出现的生理反应,与分离株的遗传性相关。由于在杂合二倍体中筛选隐性突变较为困难,并且非相关临床分离株有数万个不同的基因位点,因此影响该遗传性的点突变仍待确定。
非整倍性以及杂合性的丢失可快速改变基因组信息,导致菌株耐受性增强,并可能改变影响耐受性水平的一个或多个基因。已有研究发现,白念珠菌暴露于唑类时,能够促进有丝分裂缺陷,产生四倍体和非整倍体细胞[17]。非整倍体可改变白念珠菌对氟康唑的耐受性[18]。此外,佐剂(包括热休克蛋白90,heat shock protein 90,Hsp90)的抑制剂(格尔德霉素和雷地可尔),钙调神经磷酸酶抑制剂(环孢菌素A和FK506等)可降低菌株对抗真菌药物的耐受性,且与不同分离株的耐受水平无关[14],表明不同分离株的耐受性由共同的关键通路调控,干扰耐受性的药物可与现有抗真菌药物联合治疗发挥协同作用。除了耐受性水平的遗传决定因素外,生长条件也会影响菌株的耐受性。培养基的pH,温度和营养成分均会影响白念珠菌氟康唑持留菌的形成[14]。因此,不同菌株持留菌的形成差异受遗传因素和生长条件、细胞生理学等非遗传因素的影响。
对于同一菌株,细胞之间的表型差异可能是由于基因表达状态(如染色质修饰和/或拷贝数变化)、新陈代谢状态(如细胞间代谢物交换的相互作用)和/或生物物理状态的不同[16]。研究影响耐受性的一种或多种状态,有助于了解在药物胁迫下,特定细胞及其子代维持持留菌和非持留菌细胞状态的条件。此外,白念珠菌可通过调节液泡运输途径关键成分的转录能力影响耐受性[19]。持留菌形成可能是应对细胞压力,提高适应能力的细胞内过程。对持留菌形成的分子和细胞机制及其临床意义的研究,有助于提供治疗真菌感染的新策略。
五、真菌持留菌的临床意义
真菌持留菌可能是耐药性产生的“温床”,有利于提高菌株在抗真菌药物暴露初期的存活率,是发展为稳定耐药的重要前提。白念珠菌在氟康唑的胁迫下,高耐受性菌株比低耐受性菌株更频繁地分裂,因此它将具有更大的种群规模和更高获得新突变的可能性[9]。此外,药物压力继续对适应性强的突变体进行阳性选择。因此,持留菌种群大小、突变率和应对抗真菌药物的压力适应性对耐药的发生发展十分重要。
生物膜可影响药物与真菌的作用,降低抗真菌药物敏感性,影响临床治疗效果。念珠菌在两性霉素B胁迫下形成的持留菌,通常在生物膜中检测到,生物膜不同的粘附表面类型会影响持留菌的比例[11]。抗真菌药物能够在体外彻底清除快速生长的液体培养菌体,却不能完全清除临床环境中的真菌感染,可能与生物膜中的真菌持留菌有关[20]。真菌持留菌的临床意义有待进一步研究,目前缺乏长期临床预后(如感染复发)的相关研究。真菌在宿主体内存在抗真菌药物的情况下,具有继续分裂的能力,可能是导致反复感染以及获得耐药性的重要原因。一项小规模的回顾性研究中,分别从氟康唑治疗成功和无法清除感染的顽固性念珠菌血症患者中分离白念珠菌,比较不同菌株的耐受水平,研究发现治疗成功患者与持续性感染患者分离的初始菌株相比,具有较低的耐受性,表明耐受性可能会影响氟康唑治疗的疗效[21]。后续仍需要更多大规模的回顾性和前瞻性研究,以明确患者预后与感染菌株初始耐受水平之间的关系。
六、结论与展望
关于真菌持留菌依然有许多问题尚待解决,但随着单细胞技术和抗真菌动力学研究技术的发展,有助于我们明确持留菌的形成原理以及对临床感染患者的影响。细菌持留菌研究者已可通过微流控技术鉴定和分离持留菌,类似的研究策略也可用于真菌持留菌。真菌持留菌的研究可推动对真菌耐药机制研究的进一步探索,在菌株发展为耐药菌之前就采取相应措施,协助临床更加合理地使用抗真菌药物以预防耐药发生,有利于加强临床真菌治疗规范化,为控制真菌的耐药率和新型抗真菌药物的研发提供新思路。
参考文献
[1]Brown GD, Denning DW, and Levitz SM. Tackling human fungal infections[J]. Science. 2012, 336(6082): 647.
Lepak AJ, and Andes DR. Antifungal pharmacokinetics and pharmacodynamics[J]. Cold Spring Harb Perspect Med. 2014, 5(5): a019653.
Balaban Nq MJ, Chait R, Kowalik L, Leibler S. Treatment of staphylococcal infections with penicillin by intermittent sterilisation[J]. Lancet. 1944, 244:497-500.
Balaban NQ, Merrin J, Chait R, Kowalik L, and Leibler S. Bacterial persistence as a phenotypic switch[J]. Science. 2004, 305(5690): 1622-1625.
Fisher RA, Gollan B, and Helaine S. Persistent bacterial infections and persister cells[J]. Nat Rev Microbiol. 2017, 15(8): 453-464.
Vogwill T, Comfort AC, Furio V, and Maclean RC. Persistence and resistance as complementary bacterial adaptations to antibiotics[J]. Journal of Evolutionary Biology. 2016, 29(6): 1223-1233.
Levin-Reisman I, Ronin I, Gefen O, Braniss I, Shoresh N, and Balaban NQ. Antibiotic tolerance facilitates the evolution of resistance[J]. Science. 2017, 355(6327): 826-830.
Sebastian J, Swaminath S, Nair RR, Jakkala K, Pradhan A, and Ajitkumar P. De Novo emergence of genetically resistant mutants of Mycobacterium tuberculosis from the persistence phase cells formed against antituberculosis drugs in vitro[J]. Antimicrob Agents Chemother. 2017,61(2).
Cowen LE, and Lindquist S. Hsp90 potentiates the rapid evolution of new traits: drug resistance in diverse fungi[J]. Science. 2005, 309(5744): 2185-2189.
Windels EM, Michiels JE, Fauvart M, Wenseleers T, Van Den Bergh B, and Michiels J. Bacterial persistence promotes the evolution of antibiotic resistance by increasing survival and mutation rates[J]. The ISME Journal. 2019.
Wuyts J, Van Dijck P, and Holtappels M. Fungal persister cells: The basis for recalcitrant infections?[J]. PLoS Pathog. 2018, 14(10): e1007301.
Li P, Seneviratne CJ, Alpi E, Vizcaino JA, and Jin L. Delicate metabolic control and coordinated stress response critically determine antifungal tolerance of Candida albicans biofilm persisters[J]. Antimicrob Agents Chemother. 2015, 59(10): 6101-6112.
Bink A, Vandenbosch D, Coenye T, Nelis H, Cammue BP, and Thevissen K. Superoxide dismutases are involved in Candida albicans biofilm persistence against miconazole[J]. Antimicrob Agents Chemother. 2011, 55(9): 4033-4037.
Rosenberg A, Ene IV, Bibi M, Zakin S, Segal ES, Ziv N, Dahan AM, Colombo AL, Bennett RJ, and Berman J. Antifungal tolerance is a subpopulation effect distinct from resistance and is associated with persistent candidemia[J]. Nat Commun. 2018, 9(1): 2470.
Ropars J, Maufrais C, Diogo D, Marcet-Houben M, Perin A, Sertour N, Mosca K, Permal E, Laval G, Bouchier C, Ma L, Schwartz K, Voelz K, May RC, Poulain J, Battail C, Wincker P, Borman AM, Chowdhary A, Fan S, Kim SH, Le Pape P, Romeo O, Shin JH, Gabaldon T, Sherlock G, Bougnoux ME, and D'enfert C. Gene flow contributes to diversification of the major fungal pathogen Candida albicans[J]. Nat Commun. 2018, 9(1): 2253.
Berman J, and Krysan DJ. Drug resistance and tolerance in fungi[J]. Nat Rev Microbiol. 2020, 18(6): 319-331.
Harrison BD, Hashemi J, Bibi M, Pulver R, Bavli D, Nahmias Y, Wellington M, Sapiro G, and Berman J. A tetraploid intermediate precedes aneuploid formation in yeasts exposed to fluconazole[J]. PLoS Biol. 2014,12(3): e1001815.
Mount HO, Revie NM, Todd RT, Anstett K, Collins C, Costanzo M, Boone C, Robbins N, Selmecki A, and Cowen LE. Global analysis of genetic circuitry and adaptive mechanisms enabling resistance to the azole antifungal drugs[J]. PLoS Genet. 2018, 14(4): e1007319.
Cornet M, Bidard F, Schwarz P, Da Costa G, Blanchin-Roland S, Dromer F, and Gaillardin C. Deletions of endocytic components VPS28 and VPS32 affect growth at alkaline pH and virulence through both RIM101-dependent and RIM101-independent pathways in Candida albicans[J]. Infect Immun. 2005, 73(12): 7977-7987.
Muzny CA, and Schwebke JR. Biofilms: an underappreciated mechanism of treatment failure and recurrence in vaginal infections[J]. Clin Infect Dis. 2015, 61(4): 601-606.
Colombo AL, Guimaraes T, Sukienik T, Pasqualotto AC, Andreotti R, Queiroz-Telles F, Nouer SA, and Nucci M. Prognostic factors and historical trends in the epidemiology of candidemia in critically ill patients: an analysis of five multicenter studies sequentially conducted over a 9-year period[J]. Intensive Care Med. 2014, 40(10): 1489-1498.





