基于方法学的流行病学临界值用于Sensititre YeastOne显色肉汤法 与Etest法检测念珠菌属和曲霉菌属对三唑类的耐药研究
编译:聂晶晶 艾效曼
【摘要】尽管商品化Sensititre Yeast-One显色药敏板(SYO)和Etest方法已被广泛使用,但念珠菌属或曲霉菌属对三唑类药物敏感性检测结果仍没有解释标准。我们从39家代表全球的实验室中(鉴于方法/药物)收集菌株对氟康唑、伊曲康唑、泊沙康唑及伏立康唑的SYO和Etest的 MIC检测结果,包括白色念珠菌11,171株、都柏林念珠菌215株、光滑念珠菌复合群4,418株、季也蒙念珠菌(季也蒙毕赤酵母)157株、克柔念珠菌(库德里阿兹威毕赤酵母)676 株、葡萄牙念珠菌(葡萄牙棒孢酵母)298株、近平滑念珠菌911株、近平滑念珠菌复合群3,691株、似平滑念珠菌36株、拟平滑念珠菌110株、热带念珠菌1,854株、酿酒酵母244株、烟曲霉1,409株、黄曲霉389株、构巢曲霉130株、黑曲霉233株和土曲霉复合群302株。用于测定SYO/Etest MICs值的282株非野生型(non-WT)菌株包括:ERG11(白色念珠菌)、ERG11 和MRR1(近平滑念珠菌)、cyp51A(烟曲霉)、CDR2和CDR1过表达(分别为白色念珠菌和光滑念珠菌)。用SYO检测酵母菌和用Etest检测曲霉菌属,均呈现出良好的实验室间一致性。对满足CLSI流行病学临界值(ECV)定义的分布进行汇总后,我们提出了酿酒酵母和9种酵母以及3种曲霉属菌种的 SYO ECVs,及5种酵母菌和4种曲霉属菌种的Etest ECVs。白色念珠菌的泊沙康唑SYO ECV为0.06µg/ml,烟曲霉的伊曲康唑Etest ECV为2µg/ml,它们是非野生型菌株的最佳预测指标。这些发现支持对基于方法学ECVs(method-dependent ECVs,MD-ECVs)的需求,因此总体来看,SYO更适用于酵母菌的药敏试验,Etest可能更适用于曲霉属的药敏试验。进一步研究可以对更多念珠菌突变株进行深入评估。
【关键词】曲霉属、光滑念珠菌、白色念珠菌、真菌突变株Etest MICs、Etest ECVs、真菌突变株SYO MICs
三唑类(氟康唑、艾沙康唑、伊曲康唑、泊沙康唑和伏立康唑)是目前治疗严重念珠菌病和曲霉病的药物(例如作为第一线或预防性、辅助性、经验性药物,或用于从另一种药物过渡,或作为补救疗法) [1-3]。这些真菌感染可能导致免疫功能低下患者的发病率和死亡率升高[3-5]。唑类药物耐药的影响及流行情况已得到广泛认识,在四种最常见的念珠菌特别是白色念珠菌,和烟曲霉中,已经阐明了各种突变耐药机制[6-10]。在大多数念珠菌中,对唑类耐药性(异常高或升高的MIC值)大多与两种主要的分子机制有关:ERG11基因(在真菌麦角甾醇生物合成途径中编码的酶)或MRR1转录调节因子的唑靶唑甾醇去甲基化酶增加(过表达)或改变(氨基酸取代)[6, 8, 9]。然而,就光滑念珠菌而言,对唑类耐药性常与调节外排泵的PDR1基因过表达或改变有关[7]。另外,烟曲霉对唑类药物的主要耐药性机制是cyp51A基因改变[10]。
建议对所有血流感染和其他临床相关念珠菌分离株进行唑类敏感性检测(检测MICs)[1]。尽管通常不建议在初次曲霉病治疗期间对曲霉属菌株进行常规MIC测定,MICs在识别潜在耐药菌株(例如从治疗失败患者中分离的菌株)方面有重要作用[2]。有几种可以测定念珠菌属和曲霉菌属MICs的药敏方法,包括美国临床和实验室标准协会(CLSI)[11, 12]及欧洲抗菌药物敏感性试验委员会(EUCAST)[13](http://www.eucast.org/ast_of_fungi/)抗真菌小组委员会提出的微量肉汤稀释法M27和M38参考方法。此外,比色肉汤微量稀释显色药敏板Sensititre Yeast-One(SYO;Trek Diagnostic System,Cleveland,OH)方法以及琼脂扩散 Etest(bioMérieux,Marcy l’Etoile,France)方法等商业化方法也广泛应用于临床实验室进行真菌药敏检测。这些方法在日常使用中更为实用且省时[14-16]。
评估SYO和Etest方法性能的早期研究,其目的是将这些方法获得的唑类MICs与参考方法获得的MICs进行比较,所用菌株为常见念珠菌和曲霉菌[17-19]。其中一些早期研究还评估了有分界点菌株间的排序一致性,但很少关注实验室间重复性这一关键问题。最近,用这些商品化方法获得的烟曲霉和光滑念珠菌突变株的三唑类MIC数据已被报道[20-24]。然而,由于缺乏合适的临床数据,无法通过这两种方法建立折点(BPs)来对念珠菌属或曲霉菌属的三唑类 MICs进行分类解释。因此,这两种检测方法均依赖于念珠菌属的CLSI折点用于分类解释和质量控制(QC)[14,16]。念珠菌属或曲霉菌属的两性霉素B或棘白菌素药敏试验的SYO/Etest流行病学临界值(ECVs)的提议显示在一些数据中存在较大的与方法相关的差异,尽管监管要求在上市前必须证明该方法与参考方法相当
。这些结果强调需要为这两种广泛使用的商品化检测方法建立三唑类的基于方法学ECVs(method-dependent ECVs,MD-ECVs),以便临床实验室判断念珠菌属和曲霉菌属对三唑类药物的敏感性。
在过去两年中,我们收集了三唑类的MICs,这些MICs值是使用SYO和Etest方法检测流行和非流行酵母菌获得的(包括白色念珠菌、都柏林念珠菌、光滑念珠菌复合群、季也蒙念珠菌[季也蒙毕赤酵母]、克柔念珠菌[库德里阿兹威毕赤酵母]、葡萄牙念珠菌[葡萄牙棒孢酵母]和近平滑念珠菌复合群[包括近平滑念珠菌、拟平滑念珠菌和似平滑念珠菌])、酿酒酵母和曲霉复合群(烟曲霉[包括烟曲霉sensu stricto]、黄曲霉、构巢曲霉、黑曲霉和土曲霉)。另外还报道了较少见酵母菌如无名念珠菌(汉逊德巴利酵母)、乳酒念珠菌(马克斯克鲁维酵母)和似平滑念珠菌的SYO MIC分布,这些菌株来自至少三家实验室并且具有可比性。从此处开始,我们将使用最常见的临床名称。这些三唑类MICs是由全球39家独立实验室(依赖于方法/药物/菌种)提交的,目的是(i)通过商品化药敏试验方法/药物和菌种获得 MIC分布;(ii)评估这些分布是否适用于设置ECV,包括评估实验室间模式的一致性;和(iii)评估每个菌种/药物/方法的ECVs是否满足CLSI标准中对ECV的定义(实验室间模式的一致性,每种菌种/方法/药物至少100个MICs,来源于≥3个独立实验室),使用迭代统计方法在97.5%的临界值下估算ECV[27-29],或当推定的野生型(WT)处于分布中的最低浓度时使用二阶数值导数方法估算ECV[30]。
尽管未评估大多数菌株的耐药机制,但我们还是收集了282株含已知或确定突变菌株(非野生型[non-WT])的MIC数据,如下所示:白色念珠菌(ERG11)的SYO MICs和Etest MICs、近平滑念珠菌(ERG11、MRR1)突变株和/或CDR2基因过表达菌株的SYO MICs、CDR1基因过表达的光滑念珠菌的SYO MICs,以及带有cyp51A突变的烟曲霉的SYO MICs和Etest MICs。这些数据主要来自欧洲、阿根廷、泰国和南非的实验室,以及一份已发表的Etest研究[20]。提交的58株含PDR基因突变的光滑念珠菌的SYO数据未列入本研究,因为与总体模式相比,非突变株模式存在较大变异。
一、材料与方法
1. 分离株:用于评估的念珠菌和其他酵母菌大多收集自念珠菌血症或其他深部感染(>90%)以及浅表、口腔和阴道感染和鹅口疮患者的血液和其他无菌部位。曲霉属菌种也是从深部感染、无菌及其他部位(主要是[>90%]支气管肺泡灌洗液和痰)收集的,来源于以下医疗中心:美国弗吉尼亚州里士满弗吉尼亚大学医疗中心;西班牙马德里,马亚达洪达卡洛斯三世健康研究所国家微生物学中心真菌学参考实验室;法国克雷泰伊细菌学病毒学卫生真菌学寄生虫学系;西班牙巴伦西亚,圣拉菲研究所严重感染组;西班牙塞维利亚,德瓦尔梅医院传染病和微生物学临床管理科;中国台湾台北市国立台湾大学附属医院和医学院内科;加拿大安大略省公共卫生局;瑞典斯德哥尔摩,卡罗林斯卡大学医院,卡罗林斯卡大学实验室,临床微生物学;法国巴黎-笛卡尔大学医学院,APHP,欧洲乔治·蓬皮杜医院,微生物学寄生虫-真菌学科;阿根廷国家科学技术研究理事会(CONICET)全国生命科学大学生物化学与生物科学学院,真菌学和分子诊断实验室,寄生虫学和真菌学教研室;墨西哥新莱昂自治大学;南非约翰内斯堡威特沃特斯兰德大学,国家传染病研究所(医疗保健相关感染、耐药和真菌中心);西班牙马德里,格雷奥里奥·马拉尼翁大学综合医院;澳大利亚阿德莱德,国家真菌学参考中心南澳病理学研究所;奥地利,因斯布鲁克医科大学卫生与医学微生物学系;西班牙科尔多瓦,科尔多瓦大学医学与护理学院微生物学系;西班牙巴拉卡多市,克鲁塞斯大学医院微生物学系;西班牙阿斯图里亚斯,阿斯图里亚斯中央大学医院(HUCA),阿斯图里亚斯公国生物医学研究和生物卫生创新基金会(FINBA);西班牙毕尔巴鄂UPV/EHU大学医学和护理学院免疫学、微生物学和寄生虫学;西班牙加的斯大学生物医学生物技术与公共卫生系;西班牙阿尔卡尼兹医院;西班牙巴塞罗那,圣克鲁医院和圣保罗医院;意大利罗马,天主教圣心大学微生物研究所;美国宾夕法尼亚州匹兹堡大学;波兰格但斯克工业大学药物技术与生物化学系;意大利米兰大学健康生物医学科学系;美国明尼苏达州,罗切斯特市梅奥诊所;意大利蒙扎,圣杰拉多医院微生物学实验室;意大利帕维亚,IRCCS Policlinico圣马特奥微生物学和病毒学研究室;意大利米兰人道研究医院微生物科;意大利布雷西亚,A.O. Spedali Civili,微生物学实验室;意大利米兰,IRCCS CàGranda O. Maggiore Policlinico基金会微生物学实验室;意大利米兰,尼瓜达医院微生物学实验室;希腊雅典,雅典国立卡波季斯特里安大学,Attikon医院医学院临床微生物学实验室;巴西圣保罗联邦大学保利斯塔医学院,传染病学科,真菌学特别实验室;意大利贝加莫,ASST Papa Giovanni XXIII微生物研究所;西班牙瓦伦西亚,拉费奥大学医院微生物学部真菌科;意大利科莫,微生物学-ASST Lariana;意大利米兰,IRCCS Policlinico San Donato医学检验室。
表1. 通过三种药敏试验方法a,检测念珠菌属、糖酵母属和曲霉菌属的四种三唑类药物的MD-ECVs
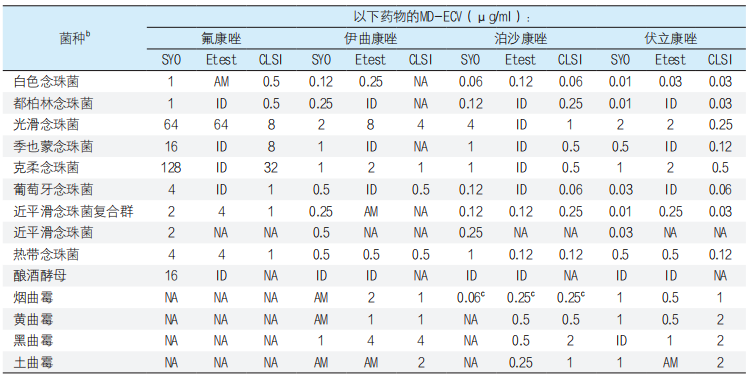
a 本研究中提出的SYO和Etest ECVs以及CLSI M27和M38方法[11,12]。AM,异常模式,模式差异;ID,进入ECV定义池的实验室数或菌株数不足;NA,不可用或不适用。
b 新分类学名称或重新分类如下:季也蒙念珠菌(季也蒙毕赤酵母)、克柔念珠菌(库德里阿兹威毕赤酵母)和葡萄牙念珠菌(葡萄牙棒孢酵母)。
c 烟曲霉的泊沙康唑 ECVs另有报道[23,26];拟平滑念珠菌的SYO ECV为4µg/ml。
对11,171株白色念珠菌、215株都柏林念珠菌、4,418株光滑念珠菌复合群(包括349株光滑念珠菌)、157株季也蒙念珠菌、676株克柔念珠菌、298株葡萄牙念珠菌、3,691株近平滑念珠菌复合群、922株近平滑念珠菌和1,854株热带念珠菌(表1和表2),使用SYO和/或Etest方法检测并根据ECV定义进行评估,3-30家实验室提交了酵母菌的4种三唑类药物的MIC值(取决于方法/药物)(表1和表2)。对25株无名念珠菌、55株乳酒念珠菌、36株似平滑念珠菌和110株拟平滑念珠菌,收集了至少3家实验室的少见念珠菌和酵母菌的SYO MICs,还收集了244株酿酒酵母的SYO数据。另外,我们汇总了来自3-11家独立实验室的5种最常见曲霉复合群的SYO和大部分Etest数据,菌种如下所示(基于方法/药物):1409株烟曲霉、389株黄曲霉、103株构巢曲霉、233株黑曲霉和302株土曲霉。
我们还获得了总共282个突变株的MICs:59个SYO MICs和10个Etest MICs,分别来自白色念珠菌突变株(Erg11基因突变)、2株白色念珠菌和13株光滑念珠菌突变株(分别过表达CDR2或CDR1外排泵)和78株近平滑念珠菌突变株(Erg11和MRR1)。SYO和Etest MICs分别收集自39株和81株烟曲霉突变株,它们具有cyp51A基因耐药机制(TR34/L98H、G54、M220等)[20](表1、表2和表5),数据来自5-7家参与实验室和一项已发表Etest研究。在每家医学中心,使用常规方法和分子方法对菌株进行鉴定,包括宏观和微观形态学观察、耐热性(在50℃下孵育)、MALDI-TOF质谱法以及β微管蛋白和钙调蛋白测序[31,32]。由于并未对本研究中评估的所有菌株进行分子鉴定,我们在相应表格中列出非突变菌株,如光滑念珠菌或近平滑念珠菌或曲霉属的复合群。提交的含有突变的烟曲霉、白色念珠菌和光滑念珠菌是在参加实验室中使用已发表的实验方案筛选的[31,36-38]。
在每个参加实验室中,通过两种方法评估质控菌株(QC)和/或参考菌株中的至少一种:质控菌株近平滑念珠菌ATCC22019、克柔念珠菌ATCC6258和宛氏拟青霉ATCC MYA-3630,参考菌株烟曲霉ATCC MYA-3626和黄曲霉ATCC MYA-204304[14,16]。参加实验室报告其使用的质控菌株的MIC均应在预期的范围内,否则MIC数据不被采纳在本研究中。
2. 真菌药敏试验:三唑类药物SYO MICs和Etest MICs是按照制造商的指导原则通过两种商业化真菌药敏检测方法获得的[14-16]。SYO MIC是在培养24小时(念珠菌属),或大多数情况下48小时(曲霉菌属)后,首先出现的蓝色或紫色孔的数值。Etest MIC是孵育24-48小时后,形成的被抑制生长的椭圆形边界对应的抗菌试纸条的刻度,为最低药物浓度。只有念珠菌的Etest MICs允许存在拖尾生长。
3. 定义:在以往研究和上文中已对ECV作为分类终点的定义进行了大量描述[27,28]。简而言之,ECV是野生型菌株中最高的MIC/MEC分布,它是使用至少3家实验室的可靠MIC/MEC分布建立的。与野生型(无表型耐药)菌株相比,非野生型菌株通常表现出对被评估药物的敏感性降低。除MIC分布外,ECV的计算还考虑到每家实验室的分布模式、检测方法的固有差异性(通常为1倍稀释),事实上ECV可能包含95%-97%的菌株。我们提议的Etest和SYO MD-ECVs是使用相同的标准和要求建立的。如我们的研究所示(表1),大多数发布的ECVs都基于参考方法的MIC分布,基于其他方法的ECVs可能有所不同。
4. 数据整理和分析:三唑类药物MICs是由全球39家独立实验室(基于方法/药物/菌种)提交的,以便(i)通过商品化药敏试验方法/药物和菌种获得MIC分布;(ii)在ECV设置之前检查这些分布是否适合合并,包括评估多家实验室间模式的一致性;和(iii)合并后,评估每个菌种/药物/方法的ECV是否满足CLSI标准中对ECV定义(每种菌种/方法/药物至少包括100个MICs,来源于≥3个独立实验室)[27,28]。使用迭代统计方法在97.5%的临界值下估算ECVs[29],或当推定的野生型模式处于分布中的最低浓度时,使用二阶数值导数方法估算ECVs[30](表2表4)。对于较少见的酵母菌(无名念珠菌和乳酒念珠菌)和似平滑念珠菌,当它们来自至少三家实验室并且模式具有可比性时,也报告SYO MIC分布。
二、结果与讨论
当方法和菌种相关的折点或ECVs可用于评估菌株和药物时,临床菌株的药敏实验最有用。根据折点将菌株分为敏感或耐药,根据ECV可将菌株分为野生型(WT;无可检测的耐药表型)或非野生型(很可能有耐药机制)[27]。由于ECV仅源于体外数据(MIC或最低有效浓度[MEC]结果),将菌株归类为推定的野生型不能直接预测治疗结果是否成功。将菌株归类为非野生型则表明它对被测药物可能具有隐匿的获得性耐药机制,并且使用被测药物治疗很可能无效[27]。但是,将菌株归类为非野生型不一定需要推测其耐药机制。CLSI折点是基于体外和临床数据、耐药基因机制以及药代动力学/药效学(PK/PD)参数[27,28]。EUCAST ECVs和折点是基于MIC分布和PK/PD参数(http://www.eucast.org/ast_of_fungi/)。因此,当折点可用于评估菌株和药物时,该值也应当被使用。据我们所知,基于SYO或Etest检测法对四种三唑类药物得到的ECV或折点尚未用于念珠菌属或曲霉菌属的分类。我们的ECVs是根据CLSI最新发布的标准定义的[27]。它们基于SYO或Etest三唑类MIC分布,这些MIC值来自3-30家(SYO)或3-11家(Etest)实验室(依赖于菌种和药物)(表1至表4)[27]。正如前文所述,SYO MICs是由多家实验室根据以下突变株提供的:59株白色念珠菌ERG11突变株(4家实验室)和39株烟曲霉cyp51A突变株(5家实验室)。Etest MICs是由多家实验室根据81株烟曲霉cyp51A突变株(7家实验室和一项已发表的研究[20])提供的(表2、表3和表5)。SYO MICs分别来自1家实验室:含过表达CDR1和CDR2基因的13株光滑念珠菌和2株白色念珠菌,以及78株近平滑念珠菌(49株含ERG11突变,29株含MRR1突变)。Etest MICs来自单家实验室收集的10株白色念珠菌(ERG11)突变株(未列入表2、表3或表5)。这些已确认的突变株MICs可用于初步评估我们用ECVs来识别非野生型菌株的提议的效果。因此,虽然折点无法用于商品化检测方法,但本研究提出的ECVs 可以帮助临床医生和实验室人员判定菌株是否存在获得性耐药机制,或用于监测或流行病学研究。
尽管所评估菌株的SYO MICs来自39家参与单位中的30家,我们仍根据CLSI标准中ECV的定义进行了数据排除(表1)[27]。在数据整合期间,以下个别念珠菌和曲霉菌的SYO MIC分布不纳入ECV分析中,如当MIC分布为双峰、特定模式的分布比总体模式多1-2个稀释度、分布模式中菌株少于5株,或者模式异常或未定义时。当质控菌株的MIC值超出规定范围时,此MIC分布也被排除[14,16]。
表2. 使用SYO方法汇总的念珠菌属、糖酵母属和曲霉菌属的三唑类MIC分布a
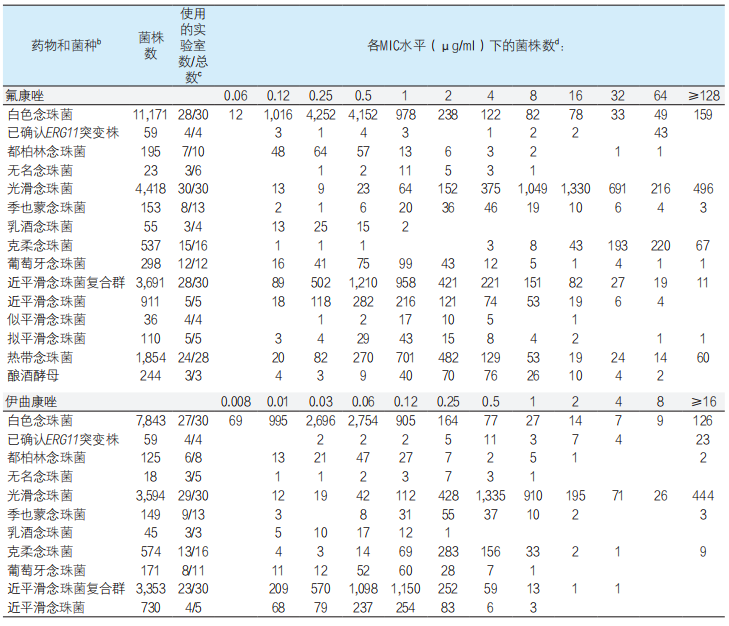
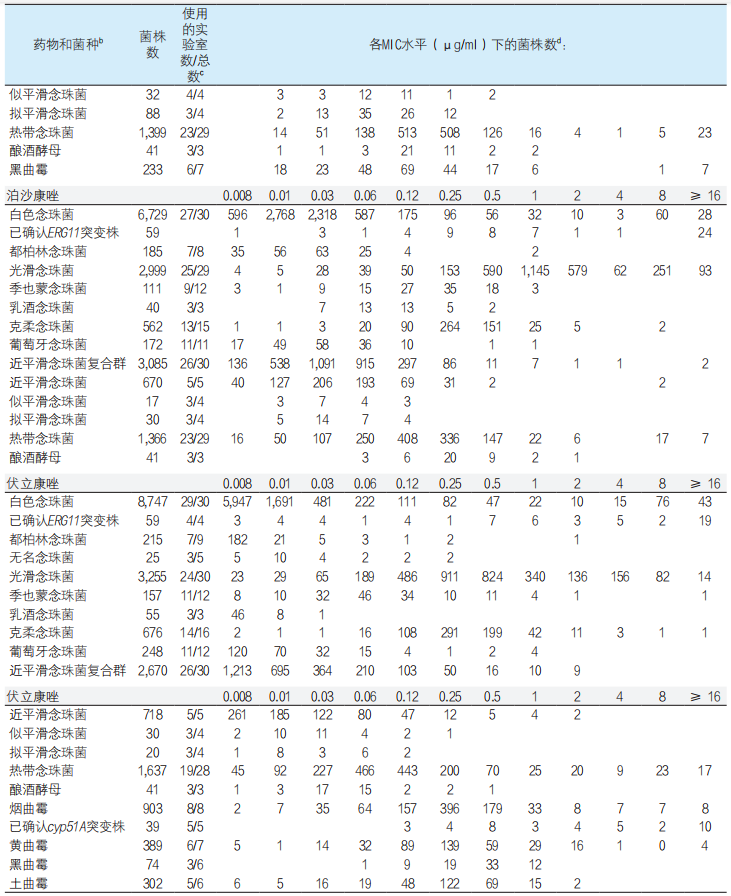
a 包括光滑念珠菌、近平滑念珠菌和曲霉菌属的复合群。(cyp51At突变株为烟曲霉)。
b 新分类学名称或重新分类如下:无名念珠菌(汉逊德巴利酵母)、季也蒙念珠菌(季也蒙毕赤酵母)、乳酒念珠菌(马克斯克鲁维酵母)、克柔念珠菌(库德里阿兹威毕赤酵母)和葡萄牙念珠菌(葡萄牙棒孢酵母)。
c ECV定义池中包含的实验室数/提交数据的实验室总数。
d 数据来自3-30家实验室,并通过比色微量肉汤稀释SYO法测定[14];每行中最高的数字(显示最多或众数MIC)用粗体表示。
12种念珠菌属菌株(17株至11,171株)来自3-30家独立实验室,这些菌株对4种三唑类药物的SYO MICs汇总后用于定义ECV,包含近平滑念珠菌、近平滑念珠菌复合群、似平滑念珠菌、拟平滑念珠菌、无名念珠菌、乳酒念珠菌和酿酒酵母的数据。来自多家实验室的59株白色念珠菌突变株和39株烟曲霉突变株的SYO MIC分布也列在表1中。就曲霉菌属的SYO数据而言,实验室间模式一致性是一个总体问题,对于提交的5个菌种的数据,仅对伏立康唑(4株菌中的3株)和伊曲康唑(黑曲霉)提出了ECV(表1和表3)。在表1列出的903株烟曲霉中,71%(640个数据点)被鉴定为烟曲霉,而29%(263个数据点)被鉴定为烟曲霉复合群(通过形态学方法、基质辅助激光解吸电离飞行时间[MALDI-TOF]质谱法或分子方法[如β微管蛋白和钙调蛋白测序]进行鉴定)[31]。在实验室提交的数据中,还通过生化实验、MALDI-TOF质谱法和/或分子方法将念珠菌属鉴定到种水平[31,32];近平滑念珠菌和光滑念珠菌主要以菌种复合群提交(表1)。表1还描述了念珠菌属和曲霉菌属SYO检测值的众数。白色念珠菌、都柏林念珠菌和乳酒念珠菌SYO氟康唑众数最低(0.25µg/ml),克柔念珠菌的SYO氟康唑众数(64µg/ml)最高。在泊沙康唑MICs中,可观察到类似的众数多样性(白色念珠菌为0.01µg/ml,光滑念珠菌为1µg/ml)。然而,伊曲康唑众数大多分布在0.06-0.12µg/ml,伏立康唑众数分布在0.008-0.03µg/ml。例外的有:伊曲康唑对光滑念珠菌众数(0.5µg/ml)、对季也蒙念珠菌及克柔念珠菌(0.25µg/ml)的众数,以及伏立康唑对季也蒙念珠菌及热带念珠菌(0.06µg/ml)、对光滑念株菌及克柔念珠菌(0.25µg/ml)的众数。近平滑念珠菌复合群的SYO众数大多为±1倍稀释度,但近平滑念珠菌复合群中四个菌种的泊沙康唑众数均为0.03µg/ml。曲霉属的SYO伏立康唑众数和黑曲霉的伊曲康唑众数范围为0.12-0.5µg/ml。与期望相同,白色念珠菌和烟曲霉突变株的SYO众数比非突变株的SYO众数高得多,在较低药物浓度可观察到两组MICs之间存在重叠(表2)。因此,念珠菌属的SYO数据显示出极好的模式一致性,而曲霉菌属的大多数SYO数据点不适用于ECV定义,如以往报道的烟曲霉的SYO泊沙康唑数据[23]。
表3. 使用Etest方法汇总的念珠菌属和曲霉菌属的三唑类药物的MIC分布a
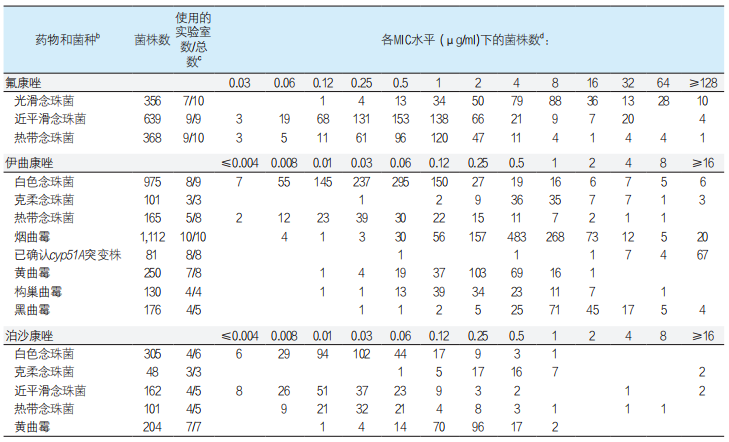
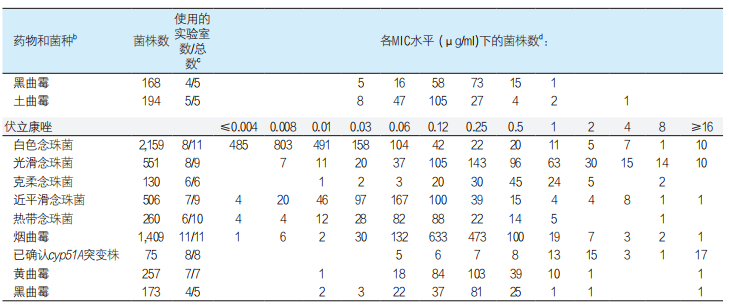
a 包括近平滑念珠菌、光滑念珠菌和曲霉菌属的复合群(cyp51A 突变株为烟曲霉)。
b 新分类学名称或重新分类如下:克柔念珠菌(库德里阿兹威毕赤酵母)。
c ECV定义池中包含的实验室数/提交数据的实验室总数和一份已发表的研究[20]。
d 数据来自3-11家实验室,并通过琼脂扩散Etest法确认[15],每行中最高的数字(显示最多或众数MIC)以粗体显示。
39家实验室中有11家实验室为念珠菌属中较流行的4个菌种、克柔念珠菌和曲霉菌属提供了Etest MICs。8家实验室和已发表的一项研究[20]分别提供了75个和81个烟曲霉突变株的伏立康唑和伊曲康唑Etest数据(表2和表5)。64%烟曲霉菌株(在1,112个伊曲康唑MICs中占712)和大多数念珠菌属菌株[31,32]鉴定到种水平,但是光滑念珠菌和近平滑念珠菌主要鉴定到种复合群水平。因此使用Etest方法,我们无法像SYO方法那样提供近平滑念珠菌复合群中各菌种之间抗真菌药敏感性的潜在差异(表1)。由于进入ECV定义数据池中的Etest MIC分布模式间存在差异,我们也排除了白色念珠菌/氟康唑、光滑念珠菌/伊曲康唑和泊沙康唑、近平滑念珠菌/伊曲康唑、克柔念珠菌/氟康唑的ECV定义。然而,大多数曲霉菌属/药物组合的Etest数据点都适合ECV定义池,虽然我们也观察到伊曲康唑和伏立康唑对土曲霉的抑制存在模式差异。因此,我们收集了更适合曲霉菌属的Etest数据,而念珠菌属的总体SYO数据非常好。最低的Etest众数是近平滑念珠菌和氟康唑组合(0.5µg/ml)、近平滑念珠菌和泊沙康唑组合(0.01µg/ml)、热带念珠菌和伊曲康唑组合(0.03µg/ml)以及白色念珠菌和伏立康唑组合(0.008µg/ml)。曲霉菌属的所有Etest众数值范围为0.12-0.25µg/ml,除烟曲霉和黑曲霉对伊曲康唑的Etest众数分别为0.5µg/ml和1µg/ml。烟曲霉突变株的Etest众数数值也较非突变株高得多。
表4和5显示了ECOFFinder程序中三唑类药物分别使用SYO和Etest方法检测的ECVs,97.5% MIC(菌种和三唑类药物组合)满足CLSI的ECV计算标准[27]。由于没有一个单独的分布在总体中的贡献≥50%,因此无需对数据进行加权。除了较流行的念珠菌属的SYO ECVs外,还提出了拟平滑念珠菌和酿酒酵母的氟康唑ECVs(分别为4µg/ml和16µg/ml)(表4)。尽管近平滑念珠菌及其菌种复合群的氟康唑ECVs相同(2µg/ml),但近平滑念珠菌的其他ECVs会高1倍稀释度。据我们所知,近平滑念珠菌及其复合群以及酿酒酵母的ECVs尚未用于参考方法[26,28](http://www.eucast.org/ast_of_fungi/)。由于Etest的异常结果,我们只定义了5种念珠菌属的伏立康唑 Etest ECVs(0.03-2µg/ml),对于其他三种药物和3-4个菌种的组合,将ECVs定义为0.12-64µg/ml(表4)。但是,我们提出了3-4个与曲霉菌属相关的ECVs[2,4,5]。4家实验室提供的数据中,土曲霉的伊曲康唑众数和伏立康唑众数不一致,而且提供的构巢曲霉的泊沙康唑MICs和伏立康唑MICs数量不足(只有两家实验室提交了数据),因此无法定义这两种菌株/药物组合的ECV[27](表2中未显示数据)。在表6,将我们的SYO ECVs和Etest ECVs与CLSI M59文件中列出的ECVs进行了比较[26]。通常,SYO ECVs比CLSI或Etest的ECVs高1-2个稀释度。在某些情况下,例如氟康唑和伏立康唑/光滑念珠菌的SYO ECVs和CLSI ECVs分别被确定为64和8µg/ml以及2和0.25µg/ml[26]。所有这些观察结果都强调MD-ECVs,以便对被评估为野生型或非野生型的感染菌株的MIC进行正确分类。该观察结果还表明,尽管商品化系统可以根据FDA标准成功建立等效性,来自多家实验室的数据汇总可以更轻松地检测出这些方法与参考方法之间的差异,至少在野生型菌株中如此。
正如上文所述,ECV的主要作用是鉴定可能具有天然耐药或获得性耐药机制的菌株(非野生型或突变株)[27,28]。在文献中可找到念珠菌属和曲霉菌属突变株的CLSI MICs[6-10,33-36],但是商品化检测方法的MICs很少[20-24]。本研究共收集到162株白色念珠菌,光滑念珠菌和近平滑念珠菌突变株的SYO和Etest MICs。对于59株白色念珠菌ERG11突变株,SYO MICs高于4种三唑类药物ECVs的菌株数量取决于所选药物。利用泊沙康唑的ECV 0.06µg/ml,识别出最高的突变株百分比(55/59;93%),其次是伊曲康唑的ECV 0.12µg/ml(53/59;90%)、伏立康唑的ECV 0.01µg/ml(52/59;88%)和氟康唑的SYO ECV 1µg/ml(48/59;81%)。这些白色念珠菌突变株在ERG11基因有如下突变:F145L、Y132H、S442F、S405F、G464S、A114S、G464S、F145T、T22OL和P98A(单独或联合)。尽管已报道大多数突变株对三唑类药物的CLSI MICs[6,8,33-35]较高,尚未报道T22OL和P98A(单独或与E266D、G448R、V437I、V488I、K143R和Y132H/X同时存在)的MICs。考虑到它们的MICs较高(>8µg/ml)(表1),这些菌株可能存在多种耐药机制(例如,最常见的外排泵过表达联合erg11过表达和/或erg11突变)。这些分子层面的组合是由非整倍体引起的(5号染色体重复或其长臂增殖)。然而,我们没有收到59株白色念珠菌突变株的外排泵过表达数据。另一方面,在表5中我们列出了白色念珠菌和烟曲霉突变株,根据MD-ECV可将其分为野生型(其MICs小于或等于ECV)和/或非野生型(其MICs大于ECV)。
表4. 念珠菌属、糖酵母属、曲霉菌属对四种三唑类药物的基于SYO方法的ECOFFinder ECVs a
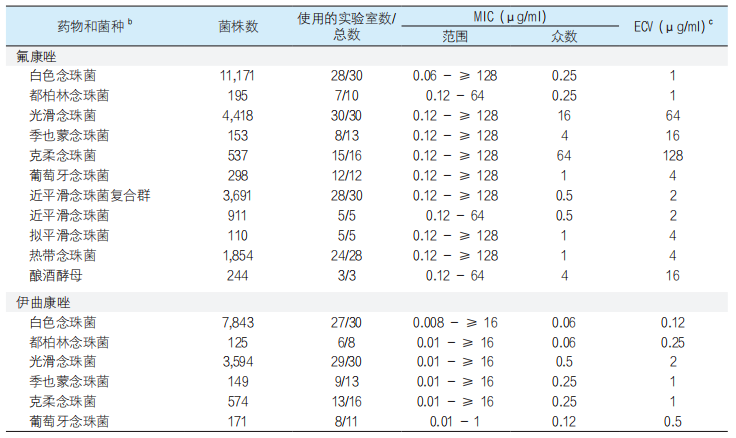
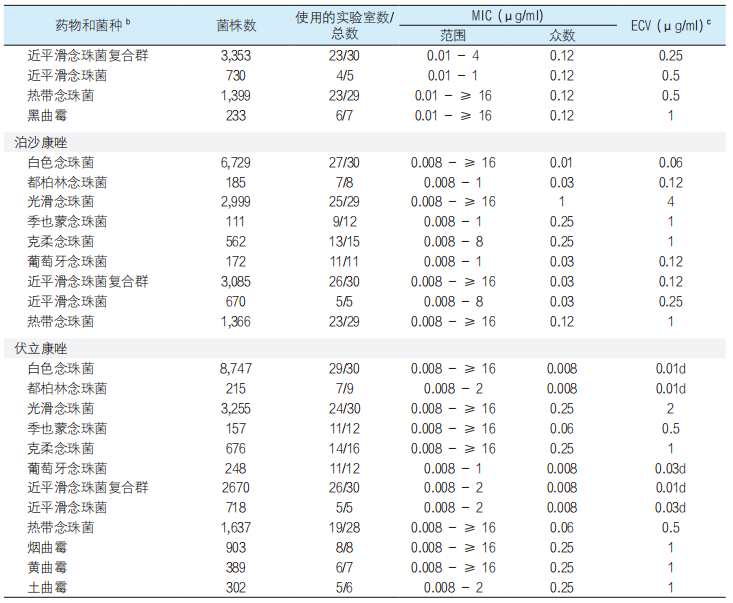
a 包括近平滑念珠菌、光滑念珠菌和曲霉属的复合群。由于模式变异性或数据不足,未提出某些念珠菌和曲霉菌的ECV。
b 新分类学名称或重新分类如下:季也蒙念珠菌(季也蒙毕赤酵母)、克柔念珠菌(库德里阿兹威毕赤酵母)和葡萄牙念珠菌(葡萄牙棒孢酵母)。
c 97.5%的统计建模菌种的ECOFFinderECVs[14,29]是基于比色微量肉汤稀释SYO法所得MICs得出的,上标c所指的除外,参考脚注c。根据其他报道,烟曲霉和泊沙康唑的SYO ECV为0.06µg/ml[23]。无论MIC如何,克柔念珠菌都对氟康唑天然耐药。
d 使用二阶导数法估算的ECV[30]。
根据CLSI方法和折点,含这些突变的菌株中,敏感株及耐药株均被报道过[8,33-35]。单家实验室的数据显示,2株白色念珠菌和13株光滑念珠菌中的11株分别过表达CDR2和CDR1基因外排泵,其对四种药物的SYO MICs均高于ECVs(表5中未显示数据)。然而只有氟康唑和伏立康唑的ECVs(分别为2和0.03µg/ml)可在78株近平滑念珠菌突变株中鉴定出96%以上的突变株。因此,在最流行的念珠菌中(白色念珠菌、光滑念珠菌),我们的SYO ECVs能够识别至少90%的耐药株,为其临床价值提供了初步证据。如果能收集更多其他念珠菌突变株的数据,将有助于评估SYO方法在临床实验室检测酵母菌的效果。
表5. 念珠菌属和曲霉菌属的四种三唑类药物基于Etest方法的 ECOFFinder ECVs a
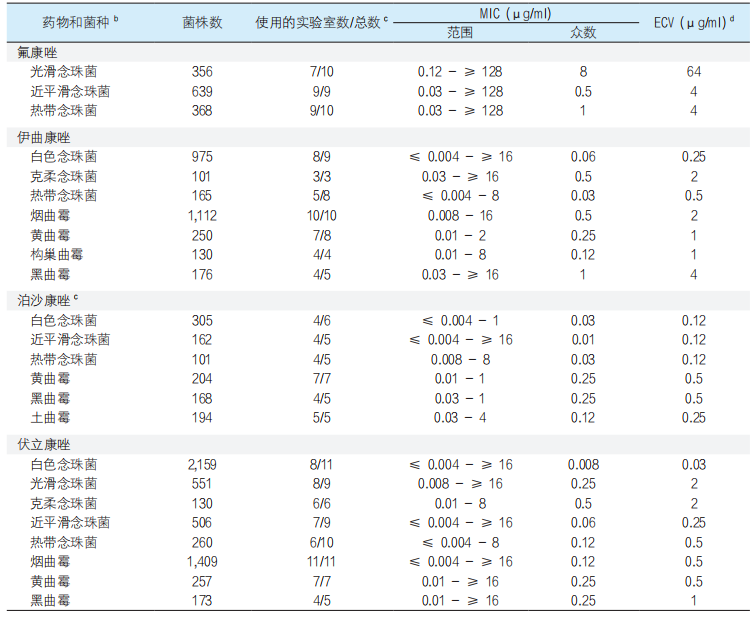
a 包括近平滑念珠菌、光滑念珠菌和曲霉属的复合群。由于模式变异性或数据不足,未提出某些念珠菌和曲霉菌的ECVs。
b 新分类学名称或重新分类如下:克柔念珠菌(库德里阿兹威毕赤酵母)。
c ECV定义池中包含的实验室数/提交数据的实验室总数(包括一项已发表研究的数据[20])。
d 97.5%的统计建模菌种的ECOFFinder ECVs[15,29]是基于商品化琼脂扩散Etest法所得MICs得出的。根据其他报道,烟曲霉和泊沙康唑的Etest ECV为0.25µg/ml[23]。
在本研究中,总共评估了烟曲霉cyp51A突变株的75个伏立康唑Etest MICs和81个伊曲康唑Etest MICs以及39个伏立康唑SYO MICs。我们提出的伊曲康唑Etest检测烟曲霉的ECV为2µg/ml,在识别cyp51A突变株(78/81;96%)方面优于0.5µg/ml的伏立康唑Etest ECV(50/75;67%)和1µg/ml的SYO ECV(26/39;67%)(表6)。在含有以下突变的菌株中,伊曲康唑的Etest MICs高于ECVs:48株具有TR34/L98(59%)突变、12株具有G54(15%)突变、9株具有M220(11%)突变、5株具有G448S(6%)突变, 7株具有混合突变(9%),包括2株具有TR46/Y121F突变的菌株(表6中未列出相应数据)。然而,文献中报道cyp51A G54突变与伊曲康唑和泊沙康唑的交叉耐药有关,M220突变与三唑类药物的MICs值高低有关[36]。
表6. 确定存在ERG11突变的白色念珠菌和cyp51突变的烟曲霉的三唑类药物SYO MICs和Etest MICs a
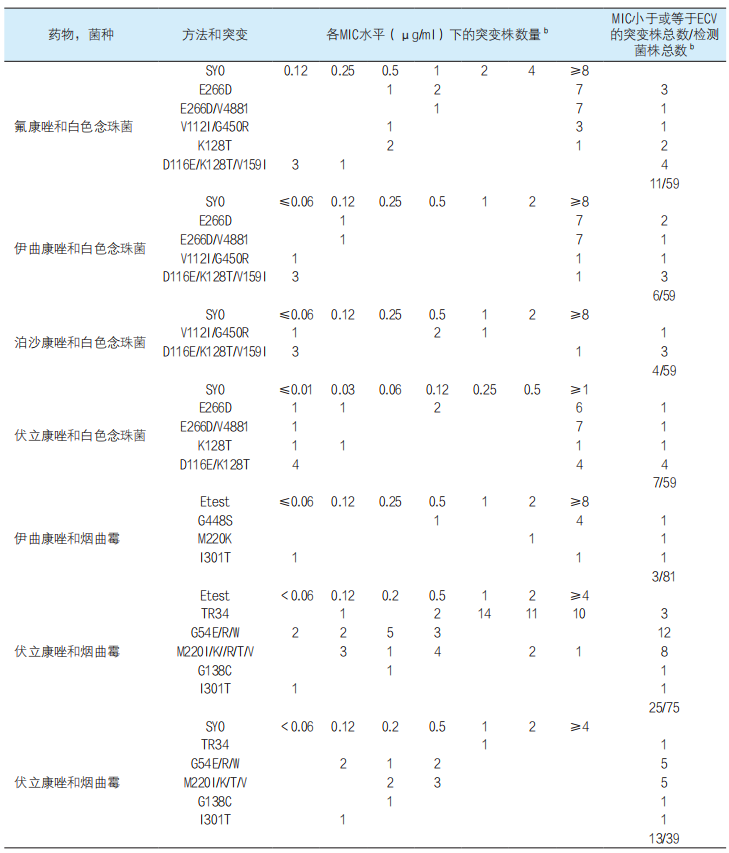
a 列出的是白色念珠菌和烟曲霉突变株的SYO和Etest MICs,在总数据点中,这些MICs或低于或高于(分别以阴影和非阴影表示)相应的ECV,包括59株白色念珠菌突变株和75、81或39株烟曲霉突变株。数据来自多个参与实验室(4至8个)提交的数据和一项已发表的研究[20]。
b 提出的SYO ECVs如下:白色念珠菌/氟康唑,1µg/ml;白色念珠菌/伊曲康唑,0.12µg/ml;白色念珠菌/泊沙康唑,0.06µg/ml;白色念珠菌/伏立康唑,0.01µg/ml;烟曲霉/伏立康唑,1µg/ml。Etest ECVs如下:烟曲霉/伊曲康唑,2µg/ml;烟曲霉/伏立康唑,0.5µg/ml。
据报道使用三种真菌药敏检测方法(CLSI、EUCAST和Etest),烟曲霉非突变株的泊沙康唑MICs与大量突变株的MICs存在重叠[23]。曲霉菌属的初步结果显示,Etest方法在检测烟曲霉突变以及其他曲霉属突变时可能更适用。这些结果再次强调需要MD-ECVs。对于曲霉属的SYO数据,需进一步的合作研究来评估判定终点;已有文献报道了颜色变化和生长抑制。
总之,我们对各种菌种/三唑类药物组合,提出了基于方法学的SYO ECVs和Etest ECVs,数据合理且来自多家实验室(3-30个)。对于念珠菌和其他酵母菌,包括近平滑念珠菌复合群(近平滑念珠菌、似平滑念珠菌和拟平滑念珠菌)和酿酒酵母的MIC分布,使用SYO方法评估具有良好实验室间一致性的大量数据。因此,我们提出了四种三唑类药物对8-10种酵母菌的SYO ECVs,包括氟康唑作用于拟平滑念珠菌和酿酒酵母。我们还提供了MIC范围,更重要的是,提供了其他少见酵母菌的众数。另外,曲霉菌属的实验室间Etest众数一致性比酵母菌好。于是我们为曲霉属3-4种菌提供了伊曲康唑、泊沙康唑和伏立康唑的Etest ECVs,为念珠菌属中4种最流行的菌种和克柔念珠菌提供了伏立康唑ECVs。最终,白色念珠菌的SYO泊沙康唑ECV为0.06µg/ml,烟曲霉的Etest伊曲康唑ECV为2µg/ml,它们在预测非野生株或突变株时效果最好(对于高于ECV的突变株,其MICs的百分比最高)。尽管氟康唑和伏立康唑的ECVs可以识别出近平滑念珠菌中96%以上的非野生株,泊沙康唑和伊曲康唑的ECVs结果却不令人满意。其他念珠菌属突变株的数据可以更好地评估MD-ECVs。SYO方法似乎更适用于检测念珠菌属并获得MIC数据,Etest方法则更适用于检测曲霉菌属。
鸣谢:我们特此感谢阿根廷国立利特瑞尔大学真菌学和分子诊断实验室的S. Gamarra、F. Leonardelli、C. Dudiuk和D. Macedo,以及澳大利亚阿德莱德国家真菌学参考中心、南澳病理学研究所的H. Alexiou、S. R. Davis和D. H. Ellis。
参考文献
Pappas PG, Kauffman CA, Andes DR, Clancy CJ, Marr KA, Ostrosky- Zeichner L, Reboli AC, Schuster MG, Vazquez JA, Walsh TJ, Zaoutis TE, Sobel JD. 2016. Clinical practice guidelines for the management of candidiasis: 2016 update by the Infectious Diseases Society of America. Clin Infect Dis 62:1-50. https://doi.org/10.1093/cid/civ747.
Patterson TF, Thompson GR, III, Denning DW, Fishman JA, Hadley S, Herbrecht R, Kontoyiannis DP, Marr KA, Morrison VA, Nguyen MH, Segal BH, Steinbach WJ, Stevens DA, Walsh TJ, Wingard JR, Young JA, Bennett JE. 2016. Practice guidelines for the diagnosis and management of aspergillosis: 2016 update by the Infectious Diseases Society of America. Clin Infect Dis 63:e1-e60. https://doi.org/10.1093/cid/ciw326.
Cornely OA, Bassetti M, Calandra T, Garbino J, Kullberg BJ, Lortholary O, Meersseman W, Akova M, Arendrup MC, Arikan-Akdagli S, Bille J, Cast- agnola E, Cuenca-Estrella M, Donnelly JP, Groll AH, Herbrecht R, Hope WW, Jensen HE, Lass-Flörl C, Petrikkos G, Richardson MD, Roilides E, Verweij PE, Viscoli C, Ullmann AJ, ESCMID Fungal Infection Study Group. 2012. ESCMID guidelines for the diagnosis and management of Candida diseases 2012: non-neutropenic adult patients. Clin Microbiol Infect 18:19-37. https://doi.org/10.1111/1469-0691.12039.
Klingspor L, Saaedi B, Ljungman P, Szakos A. 2015. Epidemiology and outcomes of patients with invasive mould infections: a retrospective observational study from a single centre (2005-2009). Mycoses 58: 470-477. https://doi.org/10.1111/myc.12344.
Kosmidis C, Denning DW. 2015. The clinical spectrum of pulmonary aspergillosis. Thorax 70:270-277. https://doi.org/10.1136/thoraxjnl-2014-206291.
Berkow EL, Lockhart SR. 2017. Fluconazole resistance in Candida species: a current perspective. Infect Drug Resist 10:237-245. https://doi.org/10.2147/IDR.S118892.
Vale-Silva LA, Moeckli B, Torelli R, Posteraro B, Sanguinetti M, Sanglard D. 2016. Upregulation of the adhesin gene EPA1 mediated by PDR1 in Candida glabrata leads to enhanced host colonization. mSphere 1:e00065-15. https://doi.org/10.1128/mSphere.00065-15.
Sanglard D, Coste AT. 2016. Activity of isavuconazole and other azoles against Candida clinical isolates and yeast model systems with known azole resistance mechanisms. Antimicrob Agents Chemother 60: 229-238. https://doi.org/10.1128/AAC.02157-15.
Forastiero A, Mesa-Arango AC, Alastruey-Izquierdo A, Alcazar-Fuoli L, Bernal-Martinez L, Pelaez T, Lopez JF, Grimalt JO, Gomez-Lopez A, Cuesta I, Zaragoza O, Mellado E. 2013. Candida tropicalis antifungal cross- resistance is related to different azole target (Erg11p) modifications. Antimicrob Agents Chemother 57:4769-4781. https://doi.org/10.1128/ AAC.00477-13.
Bernal-Martínez L, Gil H, Rivero-Menéndez O, Gago S, Cuenca-Estrella M, Mellado E, Alastruey-Izquierdo A. 2017. Development and validation of a high resolution melting assay to detect azole resistance in Aspergillus fumigatus. Antimicrob Agents Chemother 61:e01083-17. https://doi.org/ 10.1128/AAC.01083-17.
Clinical and Laboratory Standards Institute. 2017. Reference method for broth dilution antifungal susceptibility testing of yeasts, 4th ed. CLSI standard M27. Clinical and Laboratory Standards Institute, Wayne, PA.
Clinical and Laboratory Standards Institute. 2017. Reference method for broth dilution antifungal susceptibility testing of filamentous fungi, 3rd ed. CLSI standard M38. Clinical and Laboratory Standards Institute, Wayne, PA.
Arendrup MC, Meletiadis J, Mouton JW, Guinea J, Cuenca-Estrella M, Lagrou K, Howard SJ, Arendrup MC, Meletiadis J, Howard SJ, Mouton J, Guinea J, Lagrou K, Arikan-Akdagli S, Barchiesi F, Hamal P, Järv H, Lass-Flörl C, Mares M, Matos T, Muehlethaler K, Rogers TR, Torp Andersen C, Verweij P, Subcommittee on Antifungal Susceptibility Testing (AFST) of the ESCMID European Committee for Antimicrobial Susceptibility Testing. 2016. EUCAST technical note on isavuconazole breakpoints for Aspergillus, itraconazole breakpoints for Candida and updates for the antifungal susceptibility testing method documents. Clin Infect Dis 22: 571.e1-571.e4. https://doi.org/10.1016/j.cmi.2016.01.017.
Trek Diagnostic Systems. 2012. Sensititre Yeast One: Yeast One suscep- tibility, v1.8. Trek Diagnostic Systems, Cleveland, OH.
bioMérieux SA. 2013. Etest antifungal susceptibility testing package insert. bioMérieux SA, Marcy-l’Etoile, France.
bioMérieux SA. 2013. Etest performance, interpretative criteria and qual- ity control ranges table. bioMérieux SA, Marcy-l’Etoile, France.
Pfaller MA, Espinel-Ingroff A, Jones RN. 2004. Clinical evaluation of Sensititre YeastOne colorimetric antifungal plate for antifungal suscep- tibility testing of the new triazoles voriconazole, posaconazole, and ravuconazole. J Clin Microbiol 42:4577-4580. https://doi.org/10.1128/ JCM.42.10.4577-4580.2004.
Espinel-Ingroff A, Pfaller M, Erwin ME, Jones RN. 1996. Interlaboratory evaluation of Etest method for testing antifungal susceptibilities of pathogenic yeasts to five antifungal agents using a Casitone agar and solidified RPMI 1640 medium with 2% glucose. J Clin Microbiol 34: 848-885. https://doi.org/10.1128/JCM.40.6.2101-2107.2002.
Espinel-Ingroff A, Rezuzta A. 2002. E-test method for testing suscepti- bilities of Aspergillus spp. to the new triazoles voriconazole and po- saconazole and to established antifungal agents: comparison with NCCLS broth microdilution method. J Clin Microbiol 40:2001-2107.
Burgel P-R, Baixench M-T, Amsellem M, Audureau E, Chapron J, Kanaan R, Honoré I, Dupouy-Camet J, Dusser D, Klaassen CH, Meis JF, Hubert D, Paugam A. 2012. High prevalence of azole-resistant Aspergillus fumigatus in adults with cystic fibrosis exposed to itraconazole. Antimicrob Agents Chemother 56:869-874. https://doi.org/10.1128/AAC.05077-11.
Wu C-J, Wang H-C, Lee J-C, Lo H-J, Dai C-T, Chou P-H, Ko W-C, Chen Y-C. 2015. Azole-resistant Aspergillus fumigatus isolates carrying TR34/L98H mutations in Taiwan. Mycoses 58:544-549. https://doi.org/10.1111/myc.12354.
Mello E, Posteraro B, Vella A, De Carolis E, Torelli R, D’Inzeo T, Verweij PE, Sanguinetti M. 2017. Susceptibility testing of common and uncommon Aspergillus species against posaconazole and other mold-active antifun- gal azoles using the Sensititre method. Antimicrob Agents Chemother 61:e00168-17. https://doi.org/10.1128/AAC.00168-17.
Espinel-Ingroff A, Turnidge J, Alastruey-Izquierdo A, Dannaoui E, Garcia- Effron G, Guinea J, Kidd S, Pelaez T, Sanguinetti M, Meletiadis J, Botterel F, Bustamante B, Chen Y-C, Chakrabarti A, Chowdhary A, Chryssanthou E, Córdoba S, Gonzalez GM, Guarro J, Johnson EM, Kus JV, Lass-Flörl C, Linares-Sicilia MJ, Martín-Mazuelos E, Negri CE, Pfaller MA, Tortorano AM. 2018. Posaconazole MIC distributions for Aspergillus fumigatus species complex by four methods: impact of cyp51A mutations on estimation of epidemiological cutoff values. Antimicrob Agents Chemother 62:e01916-17. https://doi.org/10.1128/AAC.01916-17.
Szweda P, Gucwa K, Romanowska E, Dzierz˙anowska-Fangrat K, Naumiuk Ł, Brillowska-Da˛ browska A, Wojciechowska-Koszko I, Milewski S. 2015. Mechanisms of azole resistance among clinical isolates of Candida glabrata in Poland. J Med Microbiol 64:610-619. https://doi.org/10.1099/jmm.0.000062.
Espinel-Ingroff A, Arendrup M, Cantón E, Cordoba S, Dannaoui E, García- Rodríguez J, Gonzalez GM, Govender NP, Martin-Mazuelos E, Lackner M, Lass-Flörl C, Linares Sicilia MJ, Rodriguez-Iglesias MA, Pelaez T, Shields RK, Garcia-Effron G, Guinea J, Sanguinetti M, Turnidge J. 2017. Multi- center study of method-dependent epidemiological cutoff values for detection of resistance in Candida spp. and Aspergillus spp. to ampho- tericin B and echinocandins for the Etest agar diffusion method. Anti-microb Agents Chemother 61:e01792-16. https://doi.org/10.1128/AAC.01792-16.
Clinical and Laboratory Standards Institute. 2018. Epidemiological cutoff values for antifungal susceptibility testing. CLSI supplement M59, 2nd ed. Clinical and Laboratory Standards Institute, Wayne, PA.
27. Clinical and Laboratory Standards Institute. 2016. Principles and proce- dures for the development of epidemiological cutoff values for antifun- gal susceptibility testing. CLSI M57 document, 1st ed. Clinical and Lab- oratory Standards Institute, Wayne, PA.
Espinel-Ingroff A, Turnidge J. 2016. The role of epidemiological cutoff values (ECVs/ECOFFs) in antifungal susceptibility testing and interpreta-tion for uncommon yeasts and moulds. Rev Iberoam Micol 33:63-75. https://doi.org/10.1016/j.riam.2016.04.001.
Turnidge J, Kahlmeter G, Kronvall G. 2006. Statistical characterization of bacterial wild-type MIC value distributions and the determination of epidemiological cut-off values. Clin Microbiol Infect 12:418-425. https:// doi.org/10.1111/j.1469-0691.2006.01377.x.
Meletiadis J, Curfs-Breuker I, Meis JF, Mouton JW. 2017. In vitro antifun- gal susceptibility testing of Candida isolates with the EUCAST method- ology, a new method for ECOFF determination. Antimicrob Agents Chemother 61:e02372-16. https://doi.org/10.1128/AAC.02372-16.
Alastruey-Izquierdo A, Alcazar-Fuoli L, Cuenca-Estrella M. 2014. Antifun- gal susceptibility profile of cryptic species of Aspergillus. Mycopathologia 178:427-433. https://doi.org/10.1007/s11046-014-9775-z.
Howell S, Hazen KC. 2011. Candida, Cryptococcus and other yeasts of medical importance. In Versalovic J, Carroll KC, Funke G, Jorgensen JH, Landry ML, Warnock DW (ed), Manual of clinical microbiology, 10th ed. ASM Press, Washington, DC.
Chau AS, Mendrick CA, Sabatelli FL, Loebenberg D, McNicholas PM. 2004. Application of real-time quantitative PCR to molecular analysis of Candida albicans strains exhibiting reduced susceptibility to azoles. Antimicrob Agents Chemother 48:2124-2131. https://doi.org/10.1128/ AAC.48.6.2124-2131.2004.
Morio F, Loge C, Besse B, Hennequin C, Le Pape P. 2010. Screening for amino acid substitutions in the Candida albicans Erg11 protein of azole- susceptible and azole-resistant clinical isolates: new substitutions and a review of the literature. Diagn Microbiol Infect Dis 66:373-384. https:// doi.org/10.1016/j.diagmicrobio.2009.11.006.
Flowers SA, Brendan CB, Whaley SG, Schuler MA, Rogers D. 2015. Con- tribution of clinically derived mutations in ERG11 to azole resistance in Candida albicans. Antimicrob Agents Chemother 59:450-460. https:// doi.org/10.1128/AAC.03470-14.
Rivero-Menendez O, Alastruey-Izquierdo A, Mellado E, Cuenca-Estrella M. 2016. Triazole resistance in Aspergillus spp.: a worldwide problem? J Fungi 2:21. https://doi.org/10.3390/jof2030021.
Garcia-Effron G, Dilger A, Alcazar-Fuoli L, Park S, Mellado E, Perlin DS. 2008. Rapid detection of triazole antifungal resistance in Aspergillus fumigatus. J Clin Microbiol 46:1200-1206. https://doi.org/10.1128/JCM.02330-07.
Sanguinetti M, Posteraro B, Fiori B, Ranno S, Torelli R, Fadda G. 2005. Mechanisms of azole resistance in clinical isolates of Candida glabrata collected during a hospital survey of antifungal resistance. Antimicrob Agents Chemother 49:668-679. https://doi.org/10.1128/AAC.49.2.668-679.2005.
a 美国弗吉尼亚州里士满弗吉尼亚大学医疗中心
b 澳大利亚阿德莱德大学
c 西班牙马德里,马亚达洪达卡洛斯三世健康研究所国家微生物学中心真菌学参考实验室
d 法国克雷泰伊,细菌学病毒学卫生真菌学寄生虫学系
e 西班牙巴伦西亚,圣拉菲研究所严重感染组
f 西班牙塞维利亚,德瓦尔梅医院传染病和微生物学临床管理科
g 中国台湾台北市国立台湾大学附属医院和医学院内科
h加拿大安大略省多伦多,安大略省公共卫生局
i 瑞典斯德哥尔摩,卡罗林斯卡大学医院,卡罗林斯卡大学实验室,临床微生物学
j 法国巴黎-笛卡尔大学医学院,APHP,欧洲乔治·蓬皮杜医院,微生物学寄生虫-真菌学科
k 阿根廷国家科学技术研究理事会 (CONICET) 全国生命科学大学生物化学与生物科学学院,真菌学和分子诊断实验室,寄生虫学和真菌学教研室
l 墨西哥新莱昂州,新莱昂自治大学
m 南非约翰内斯堡威特沃特斯兰德大学,国家传染病研究所(医疗保健相关感染、耐药和真菌中心)
n 西班牙马德里,格雷奥里奥·马拉尼翁大学综合医院
o 澳大利亚阿德莱德,国家真菌学参考中心南澳病理学研究所
p 奥地利因斯布鲁克,因斯布鲁克医科大学卫生与医学微生物学系
q 西班牙科尔多瓦,科尔多瓦大学医学与护理学院微生物学系
r 西班牙巴拉卡多市,克鲁塞斯大学医院
s 西班牙阿斯图里亚斯,阿斯图里亚斯中央大学医院 (HUCA),阿斯图里亚斯公国生物医学研究和生物卫生创新基金会 (FINBA)
t 西班牙毕尔巴鄂,巴斯克地区大学/巴斯克大学 (UPV/EHU)
u 西班牙加的斯,加的斯大学生物医学生物技术与公共卫生系
v 西班牙萨拉戈萨,阿拉贡健康研究所
w 西班牙巴塞罗那,圣克鲁医院和圣保罗医院
x 意大利罗马,天主教圣心大学微生物研究所
y 美国宾夕法尼亚州匹兹堡,匹兹堡大学
z 波兰格但斯克,格但斯克工业大学药物技术与生物化学系
aa 意大利米兰,米兰大学健康生物医学科学系
bb 美国明尼苏达州,罗切斯特市梅奥诊所
cc 意大利蒙扎,圣杰拉多医院微生物学实验室
dd 意大利帕维亚,IRCCS Policlinico圣马特奥微生物学和病毒学研究室
ee 意大利米兰人道研究医院微生物科
ff 意大利布雷西亚,A.O. Spedali Civili,微生物学实验室
gg 意大利米兰,IRCCS Cà Granda O. Maggiore Policlinico 基金会微生物学实验室
hh 意大利米兰,尼瓜达医院微生物学实验室
ii 希腊雅典,雅典国立卡波季斯特里安大学,Attikon 医院医学院临床微生物学实验室
jj 巴西圣保罗,圣保罗联邦大学保利斯塔医学院,传染病学科,真菌学特别实验室,
kk意大利贝加莫,ASST Papa Giovanni XXIII 微生物研究所
ll 西班牙瓦伦西亚,拉费奥大学医院微生物学部真菌科
mm 意大利科莫,微生物学-ASST Lariana
nn 意大利米兰,IRCCS Policlinico San Donato医学检验室




