微生物多样性分析系统及其临床应用
微生物是人体内的各种微小生物的总称,包含细菌、真菌、病毒等等。它们中的大部分为与人体共生的有益生物,其形成的微生态系统(也叫微生物多样性)在维持机体健康状态、调节免疫功能、防止代谢功能障碍或病原体入侵引起的疾病等方面扮演重要角色。统计分析表明[1],菌群多样性与约三分之一的人类微生物相关性疾病间存在显著联系。由于物种组成随疾病状态的变化而变化,一些微生物群落构成和丰度的变化可能作为疾病诊断和监测的潜在指标。
微生物多样性分析通常采用宏基因组测序方法,以特定环境中整个微生物群落作为研究对象,无需分离培养,直接提取样本中全部微生物的DNA进行研究[2]。目前对微生物多样性的研究主要聚焦于细菌多样性的分析,微生物多样性分析系统也主要以使用16S rRNA基因测序结合生物信息学分析对样本中菌群的多样性进行识别和鉴定为主。
一、16S rRNA是主要的检测靶标
16S rRNA即16S ribosomal RNA,是原核生物核糖体的重要组成部分,参与形成细菌生命活动所需物质提供相互结合、相互作用的重要场所,对细菌蛋白质的合成至关重要。所有细菌基因组中都至少有一个16S rRNA编码基因,由于它高度保守且进化缓慢,因此它是细菌和古细菌系统发育生物学研究中最广泛应用的基础指标[3]。如图1所示,16S rRNA基因全长约1500个核苷酸(有一些细菌生物体可能具有更短或更长的16SrRNA序列),包括9个可变区(Variable region)和10个保守区(Constant region),呈交替排列。保守区在物种之间高度相似,而可变区则体现物种间的差异。不同细菌编码的16s rRNA基因均具有独特的可变区序列,由于其长度适中,序列较为保守,因此可以设计特异性的引物,通过检测16S rRNA基因可变区的序列变异和丰度,了解不同微生态菌落的多样性信息[4,5]。迄今为止,细菌16S rRNA 基因仍是临床实验室中细菌属或种级别鉴定的首选分子靶标[6]。
最近发现,人类胃肠道还含有大量真菌,它们与肠道菌群的结构及人体免疫发育也是存在内在联系的,主要表现为白色念珠菌的丰度和异质性[7]。这使得ITS和18S rRNA,这些指示真菌和真核生物(藻类等)的高度保守序列成为了测序检测的靶标。但是目前临床应用较少。
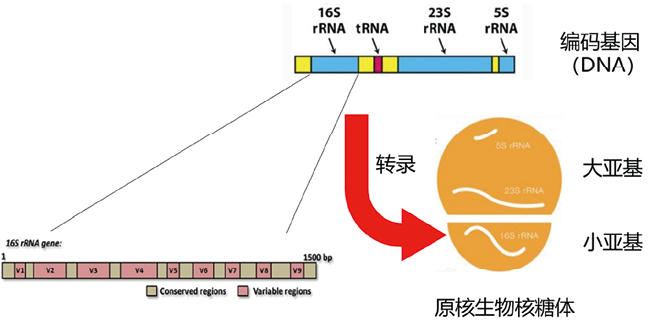
图1. 16S rRNA基因
二、分析系统
早在20世纪70年代测序技术就已经产生,但最初Sanger测序(一代测序技术)方法存在劳动强度大、耗时、实验条件苛刻、技术复杂及成本昂贵等特点,限制了其在临床实验室中的应用[8]。随着技术的革新和进步,下一代高通量测序技术(next generation sequencing,NGS)逐渐替代了传统的Sanger测序方法,使得基于16S rRNA基因测序的微生物多样性分析系统进入临床实验室成为可能。目前,已有基于16S rRNA基因测序的微生物多样性分析系统从基础研究实验室过渡到公共卫生领域和微生物临床检测的先例[9,10]。同时,自新型冠状病毒疫情爆发以来,出于对人类病原体明确诊断和快速鉴定的迫切需要,临床医学实验室对NGS的采用大大加快,相关的微生物多样性分析系统得到了政府和权威机构重视和支持,将会具有更加广泛的应用前景。
Sanger测序能够提供接近全长16S rDNA片段的测序信息,仍是检测准确度最高的测序方法。但是,其检测成本高且通量较低,难以满足现有临床实验室的需求。NGS的出现虽然迅速替代了一代测序的方法,三代测序技术检测16S rRNA基因的方法也很快被应用于微生态分析当中。然而,使用什么样的手段对16S rRNA进行测序,以及对不同区域的16S rRNA编码基因进行测序,是个有争议的领域。不同研究中感兴趣的检测区域可能因实验目标、设计和样本类型等因素而异。
1. 二代测序检测方法:目前使用的短读长大规模平行测序技术,通常也称为第二代测序技术[11]。现有的16S rRNA基因测序技术大部分是基于二代测序技术。这种测序技术通常在单台机器运行中生成和检测数千到数百万个短测序读数(50-600bp),且无需克隆,极大降低了测序成本,缩短了测序时间。在每次运行期间,测序会产生高达数十亿个核苷酸序列,其中每个基因组在小的随机片段中被多次测序读取以生成非常大的数据集。尽管每个微生物多样性分析平台可能使用不同的生化反应和阵列排布,但工作流程基本相似,具体包括以下步骤:(1)基因组DNA提取;(2)文库制备(包括DNA模板制备);(3)自动测序;(4)数据分析。
对于细菌的16S rRNA基因序列分析,Illumina公司的MiSeq平台是目前使用最为普遍的测序平台。IlluminaMiSeq平台(图2)使用的是边合成边测序(sequencingbysynthesis,SBS)的技术。具体来说,就是以单分子阵列的方式在小型芯片流动池(Flowcell)上进行桥式PCR反应,通过可逆阻断技术实现每次只合成一个碱基。在清除未反应的碱基和荧光试剂后,可利用相应的激发光激发荧光基团,读取荧光信号并获得碱基信息。而后,使用DNA酶切除荧光基团和阻断基团,便可重复进行下一个碱基的检测。

图2. Illumina公司的MiSeq分析平台
使用MiSeq2×300平台进行16S rRNA基因序列分析的具体流程如下(图3)[12]:(1)文库制备(约90分钟):使用两对特异性引物进行两步法扩增构建文库,第一步以16S rDNA部分可变区的序列为目标进行扩增,第二步通过扩增添加索引序列。MiSeq平台用于制备16S rRNA宏基因组测序文库的引物主要针对16S rRNA基因的V3和V4可变区序列设计的,可创建约460bp的单个扩增子。同时,也可以自行设计引物针对不同的可变区序列进行研究。(2)测序(约56小时):使用MiSeqv3试剂盒进行双端测序(所需测序引物已包含在 MiSeq试剂盒中),片段长度最长延伸至2×300碱基对,最大片段读数(reads)可达2500万个,数据产出量为13.2-15Gb(600个循环)。整个运行中,大于70%的数据碱基质量分值(Q值)高于Q30(碱基检出中错误发生几率小于0.1%)。(3)数据分析(约3小时):片段读数经过合并、去噪及质检后,利用Illumina审核的GreenGenes数据库对16S rRNA靶向扩增读数进行分类,进而得到α多样性、β多样性等微生物多样性分析结果。
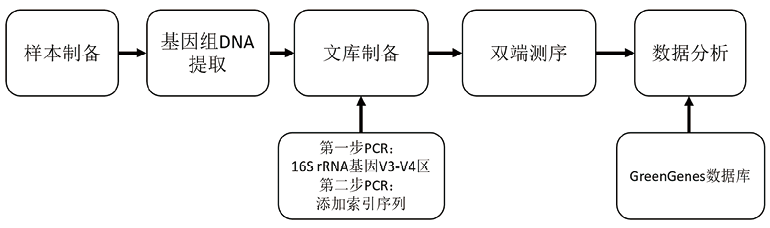
图3. MiSeq平台进行16S rRNA基因分析的流程
Illumina公司的MiSeq平台是目前唯一一台在单仪器上整合了扩增、测序和数据分析的微生物多样性分析平台,具有占用空间小、质量轻,便于组装和运输的特点。同时,一次检测产生高达十几个Gb的数据,也使MiSeq有着其他二代测序平台无可比拟的测序深度和准确性[13]。在2016年,使用MiSeq分析系统开展16S rRNA基因测序,每个样本的估算成本为18美元(约合人民币120元人民币),表明其是一种经济可行的微生物多样性分析方法,非常适合临床实验室的应用和推广。
虽然有众多优势,然而,以Illumina MiSeq为代表的二代测序也存在缺点。由于其受读取片段长度短的限制,一般只能选择1-2个可变区作为扩增片段,如V3-V4、V4-V5等,无法对测得物种进行更加精确的分类鉴定(仅能到属水平),从而影响了检测结果的准确性和精度。因此,对16S rRNA基因的多个可变区进行扩增及测序可能成为解决引物偏差和分类分辨率问题的其中一种方案。同时,随着测序数据量的增大,测序时间也在成倍延长,这样也会给未来应用带来较大局限。
三、三代测序检测方法
第三代测序技术是一种长读长单分子测序技术,是指在DNA测序时,不需要经过PCR扩增,而对每一条DNA分子进行单独测序。目前使用第三代测序技术进行16S rRNA基因分析的平台有Pacific Biosciences(简称 PacBio)公司的PacBio RSII平台(图4A)和Oxford公司的Nanopore平台,分别代表了单分子荧光测序和纳米孔测序两种主流的三代测序技术。单分子荧光测序的基本原理与Illumina MiSeq平台相似,均为使用荧光基团标记脱氧核苷酸(dNTP),实时记录荧光的强度改变。纳米孔测序技术则是利用纳米孔直径非常小的特点,在电场力和孔控制蛋白的联合作用下一次仅允许单个核苷酸分子从纳米孔通过,而由于不同碱基带电性质同,通过位移过程中电信号的波动就能识别通过每个纳米孔的碱基类别,从而实现DNA的精准测序。


A B
注:A.PacBio RSII分析平台 B. PacBio Sequel分析平台
图4. Pacific Biosciences公司基于SMRT技术的PacBio分析平台
PacBio公司的PacBio Sequel平台(图4B)是2015年推出的基于单分子实时(single molecule real time,SMRT)测序技术和纳米孔技术的新一代分析系统。其以SMRT Cell为载体,每个SMRT Cell上布满了一百万个零模波导孔(zero-mode waveguides,ZMW)。测序时DNA聚合酶捕获DNA序列锚定在ZMW孔底部,不同荧光标记的dNTP进入ZMW孔底部参与反应。位于小孔底部的激发光能够激发核苷酸上标记的荧光基团,通过监测系统记录荧光信号存在时间的长短可以把匹配的碱基与游离的碱基区别开来,进而获得序列信息。整个测序过程,一方面使链延伸,另一方面进行荧光检测,不需要经过PCR扩增便实现了对每一条DNA分子的实时测序。
使用PacBio Sequel平台进行16S rRNA基因序列分析的具体流程如下(图5):(1)文库制备(约90分钟):同样采用两步PCR法进行SMRT Cell文库构建[14]。与Illumina MiSeq不同的是,第一步是针对16S rRNA全长基因的扩增。具体来说就是,使用特异性引物对跨越可变区V1-V9的全长16S rRNA基因序列(>1400bp)进行扩增。推荐使用的通用16S rRNA基因全长引物如下:
前向引物(27F):
5′-GCAGTCGAACATGTAGCTGACTCAGGTCACAGRGTTYGATYMTGGCTCAG-3′
后向引物(1492R):
5′-TGGATCACTTGTGCAAGCATCACATCGTAGRGYTACCTTGTTACGACTT-3′
(R=A或G,Y=C或T,M=A或C)
由于三代测序所需DNA量较大,PCR扩增体系推荐为30-50μL。PCR扩增程序为:95℃ 5min,95℃ 30s,57℃ 30s,72℃ 60s,72℃ 10min,其中2-4步为20个循环。PCR产物使用微量分光光度计和1%琼脂糖凝胶电泳检测。然后,在使用PacBio标签引物进行二次扩增添加索引序列。每个样品至少设置3个重复,并将其等量混合,以减少PCR偏差。同时,每批次PCR都应设置空白对照。(2)测序(约20小时):目前PacBio Sequel平台使用的SMRT测序技术的平均读取长度为8000-12000bp,运行时间为0.5~20小时(时间越长得到的读取长度越长),可以轻松覆盖16S rRNA基因所有可变区域,获得16S rRNA基因全长序列。在该平台下每个SMRT Cell可以产生超过25万个reads和高达20 Gb的数据,用于解析16S rRNA基因。SMRT测序可以实现超过99.999%(QV50)的高精确度。(3)数据分析(约3小时):对多个16srRNA基因的全长测序读数进行一致性校正,得到一个唯一读数,也称为CCS(Circular Consensus Sequencing)读数,然后通过删除特定长度范围(小于1300bp和大于1700bp)之外序列的方式进行质量控制,最后通过数据库获得α多样性、β多样性等微生物多样性分析结果。
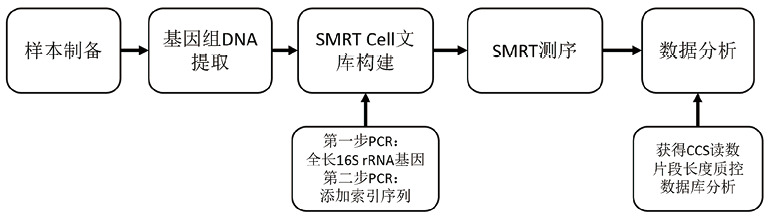
图5. PacBio Sequel平台进行16S rRNA基因分析的流程
16S rRNA基因测序区域和测序深度对微生物多样性分析具有明显影响,增加测序深度将显著提升微生物多样性组成谱解析的精准性和全面性。有研究[15]系统比较了以PacBio RS II为代表的三代测序平台和以Illumina MiSeq为代表的二代测序平台对模拟细菌菌落的分辨能力。根据两种平台的测序结果,两者菌群整体结构虽然较为相似,但当菌群复杂程度增加时,两者的检测结果便会存在差异,而三代测序的结果则更加精确,不确定性更低。最近的研究[16, 17]分别使用了PacBio Sequel平台对年轻人牙菌斑中最初定植的细菌及早产儿粪便中的微生物组进行了全长16S rRNA基因测序分析,结果均准确、详细地获得了菌群多样性的报告结果,为临床提供了帮助。因此,三代测序集合了Sanger测序的读长优势和二代测序的高通量优势,可在较短的时间内用较低的成本获得16S rRNA基因全长序列,有望从根本上解决二代测序的瓶颈问题,实现对16S rRNA基因的“高分辨率”检测。
然而,三代测序技术距离真正实现临床应用还有很长一段路要走。首先,平台运行通量低且每个样本检测成本高,加上仪器价格昂贵和占用空间大,使得三代技术更适合于对微生物多样性研究有更高质量要求的科研机构。其次,与其他长读长测序技术一样,PacBio Sequel平台比Illumina MiSeq平台具有更高的原始数据错误率(11%~15%)。因此,需要增加读取数量和纠错算法来降低错误率。目前,通量更高、测序成本更低的PacBio SequelⅡ平台已经面世,三代平台存在的问题也在逐步解决。
四、微生物多样性分析结果的解读
在微生物多样性分析过程中,首先需要引入聚类单元(Operational Taxonomic Units,OTU)这个概念。对序列信息按照一定的相似程度进行聚类,每形成一个聚类单元称为一个OTU。一个OTU中序列的差异程度不能大于规定的相似程度,在细菌16S多样性研究中,目前主要按照序列97%的相似性进行OTU聚类。微生物多样性分析平台使用OTU进行物种注释,即从OTU中选择一条代表序列与现有数据库进行比对,从而获得该OUT的物种信息。
临床实验室可以根据OTUs进行微生物多样性分析和个性化分析,其中微生物多样性分析包括物种组成分析、单样本多样性分析(Alpha多样性分析)和组间多样性分析(Beta多样性分析)等,可以帮助我们了解样本中微生物的种类和构成情况,以及寻找不同来源样本间具有显著性差异的微生物群落;个性化分析可以通过高级统计分析方法提供微生物群落结构和丰度变化与感兴趣的某种疾病或某种影响环境因素间的相关性分析,以及通过物种进化树、基因预测等手段提供特定物种的耐药性监测和临床用药指导。因此,根据微生物多样性分析平台提供的结果,临床实验室可以进行更加快速、更加准确的物种分类鉴定,了解微生态变化情况,发现传统微生物检测流程中难以发现的潜在致病菌种,为临床工作提供更有价值的指导。
五、面临的问题和临床应用前景
对技术技能的高要求、设备成本的高投入以及对用户友好的比较测序分析软件和经过验证的数据库的需求,是目前基于测序的微生物多样性分析平台所面临的主要问题[18]。其中,最重要的是数据分析方法的标准化。当前各家公司微生物多样性分析系统数据对比分析使用的一个或多个参考数据库通常不同。对于同一个测序结果,可能会出现不同平台提供的微生物多样性分析报告不一致的问题。在临床微生物学实验室推广微生物多样性分析的另一个障碍是缺乏掌握生物信息分析能力的医学专业人员。结合临床信息和数据分析结果,报告才能被正确解读。因此,在这方面的比较、统一和规范,非常必要,具有重要的临床意义。
测序技术和生物信息学的迅速发展,预计在未来将会给临床实验室带来巨大变化,这种变化将极大地影响微生物多样性分析在常规临床和公共卫生中的应用。微生物多样性分析系统及其临床应用,有可能为临床医生和公共卫生专家提供新的健康指标和诊断、监测依据。此项工作将极大地提高临床微生物学实验室对疾病发生和发展的诊断和预测能力,让实验室更加深入地参与到临床疾病的诊治当中。
参考文献
Ma ZS, Li L, Gotelli NJ. Diversity-disease relationships and shared species analyses for human microbiome-associated diseases. ISME J. 2019; 13(8): 1911-1919.
国家标准化管理委员会. 环境微生物宏基因组检测高通量测序法. 中华人民共和国国家标准. 2021. GB/T 40226-2021.
Fox GE, Magrum LJ, Balch WE, Wolfe RS, Woese CR. Classification of methanogenic bacteria by 16S ribosomal RNA characterization. Proc Natl Acad Sci U S A. 1977. 74: 4537-4541.
Sun DL, Jiang X, Wu QL, Zhou NY. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity. Appl Environ Microbiol. 2013. 79: 5962-5969.
Marchandin H, Teyssier C, Simeon De Buochberg M, Jean-Pierre H, Carriere C, Jumas-Bilak E. Intra-chromosomal heterogeneity between the four 16S rRNA gene copies in the genus Veillonella: implications for phylogeny and taxonomy. Microbiology. 2003. 149: 1493-1501.
Clinical Laboratory Standards Institute (CLSI). Interpretive criteria for identification of bacteria and fungi by DNA target sequencing; MM18-A2 approved guideline. 2018. CLSI, Wayne, PA.
Leonardi I, Li X, Semon A, Li D, Doron I, Putzel G, Bar A, Prieto D, Rescigno M, McGovern DPB, Pla J, Iliev ID. CX3CR1(+) mononuclear phagocytes control immunity to intestinal fungi. Science. 2018 Jan 12; 359(6372): 232-236.
Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. 1977; 74(12): 5463-7.
Besser J, Carleton HA, Gerner-Smidt P, Lindsey RL, Trees E. Next-generation sequencing technologies and their application to the study and control of bacterial infections. Clin Microbiol Infect. 2018; 24(4): 335-341.
Church DL, Cerutti L, Gürtler A, Griener T, Zelazny A, Emler S. Performance and Application of 16S rRNA Gene Cycle Sequencing for Routine Identification of Bacteria in the Clinical Microbiology Laboratory. Clin Microbiol Rev. 2020; 33(4): e00053-19.
Schadt EE, Turner S, Kasarskis A. A window into third-generation sequencing. Hum Mol Gen. 2010; 19(R2): R227-R40.
Illumina. Illumina 16S metagenomics SequencingWorkflow. 2017.
Loman NJ, Misra RV, Dallman TJ, Constantinidou C, Gharbia SE, Wain J, Pallen MJ. Performance comparison of benchtop high-throughput sequencing platforms. Nat Biotechnol. 2012; 30(5): 434-9.
PacBio. Procedure & Checklist-Full-Length 16S Amplification, SMRTbell® Library Preparation and Sequencing. 2018. Part Number 101-599-700 Version 01.
Singer E, Bushnell B, Coleman-Derr D, Bowman B, Bowers RM, Levy A, Gies EA, Cheng JF, Copeland A, Klenk HP, Hallam SJ, Hugenholtz P, Tringe SG, Woyke T. High-resolution phylogenetic microbial community profiling. ISME J. 2016; 10(8): 2020-32.
Ihara Y, Takeshita T, Kageyama S, et al. Identification of Initial Colonizing Bacteria in Dental Plaques from Young Adults Using Full-Length 16S rRNA Gene Sequencing. mSystems. 2019; 4(5): e00360-19.
Graf J, Ledala N, Caimano MJ, Jackson E, Gratalo D, Fasulo D, Driscoll MD, Coleman S, Matson AP. High-Resolution Differentiation of Enteric Bacteria in Premature Infant Fecal Microbiomes Using a Novel rRNA Amplicon. mBio. 2021; 12(1) pii: mBio.03656-20.
中华医学会检验医学分会.高通量宏基因组测序技术检测病原微生物的临床应用规范化专家共识. 2020; 43(12): 1181-1195.




