宏基因组测序技术流程面临 的挑战与技术操作规范难点
宏基因组测序(metagenomics next generation sequencing,mNGS)技术是对样本中所有核酸进行无偏倚测序,结合病原微生物数据库及特定算法,检测样本中含有的病原微生物序列,在病原微生物的鉴定、分型、耐药基因突变检测及新型病原体鉴定等方面具有广泛的应用。相较与传统病原微生物检测方法,mNGS可一次性检测多种类型病原体,无需预先假设,具有灵敏度高和检测范围广等优势。
mNGS检测流程主要包括样本采集和运输、核酸获得、文库构建、上机测序、生物信息分析、报告发送和结果解读,每一个环节的操作不规范都可能对实验产生影响,对结果造成偏倚。本文将简述mNGS检测流程中的样本采集运输、核酸提取富集、生信分析和报告解读这四个步骤的注意事项和技术难点,以及可行的解决方案。
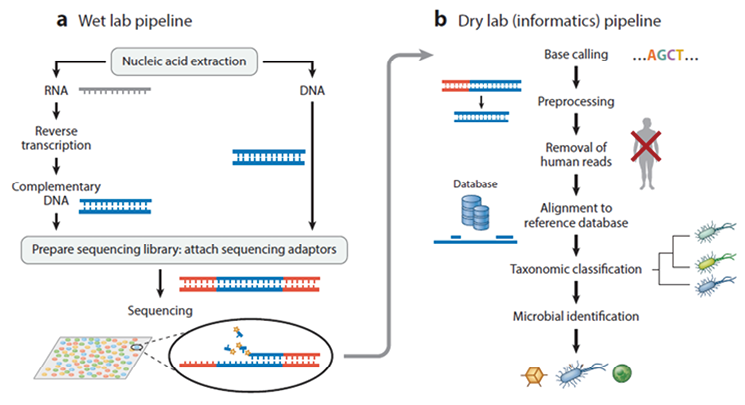
图1. 宏基因测序技术流程示意图
一、样本采集和运输
mNGS适用标本类型包括脑脊液、静脉血、肺泡灌洗液、痰液、咽拭子和组织等。为保证标本质量,减少标本污染,采集过程中需做好消毒和清洁工作。无菌部位的标本,应严格遵守无菌原则;有菌定植或污染部位的标本采集时应采取必要措施,尽量减少污染(如采集支气管肺泡灌洗液时弃去前段可能污染的部分等)。原则上,样本直接从患者感染部位的体液或组织中进行采集,如果感染部位的不易采集或标本不理想,可以选择血液标本,并及时送检。
采集后的标本转运装置的类型、储存时间和温度影响核酸的数量和完整性。欧蒙未一针对脑脊液和肺泡灌洗液标本开发了常温运输方式,专用常温保存管中的保护剂和稳定剂,不仅能够避免微生物游离核酸的降解,同时防止人源细胞破裂带来人源核酸干扰,从而提高病原微生物检出的敏感度。
二、核酸提取与富集
由于细菌、真菌和寄生虫等微生物细胞结构差异大,核酸提取的方法需要兼容不同微生物的特征,使得“破壁”成为难点。目前常用的破壁方法包括物理破壁法(如超声波破壁)、化学法(如二硫代苏糖醇、氢氧化钠)和酶法(如溶菌酶、胰蛋白酶)等,然而,常规的破壁方法对细胞壁较厚的病原体(如胞内菌和真菌)细胞壁裂解效率低,从而影响胞内菌和真菌的检出。因此,欧蒙未一采用珠磨(玻璃珠匀质破碎)和酶解的双重破壁方式,通过三维高速振动,辅助不同粒径大小研磨玻璃珠的敲打,再经过生物酶将细胞壁和细胞膜消化溶解,不仅大大提高了真菌、结核分枝杆菌等厚壁及胞内微生物的核酸提取效率,同时保证病毒的提取效率不受影响。
mNGS的另一个关键限制因素是,如果样本中含有高水平宿主DNA,将影响检测病原微生物的灵敏度,因此,在测序前进行去人源处理尤为重要。目前常用的去人源技术包括提取DNA前去除法(如物理法、生物法、化学法),以及提取DNA后去除法(主要通过抗甲基化DNA磁珠来实现)。需要指出的是,去人源处理虽然可以提高病原体检测的敏感性,但同时也会显著增加试剂来源的背景微生物的干扰,以及降低对没有细胞壁的微生物(如支原体)的检出敏感性,因此不建议对所有临床样本都进行去人源处理。欧蒙未一根据不同样本类型人源细胞量不同的特点,采用选择性去人源方式,对于高人源背景的样本(如胸腔积液、呼吸道样本、严重血性脑脊液等)进行去人源处理,同时优化了去人源的方法,达到有效去除背景噪音,提高病原体检出效率的效果。
三、生物信息学分析
生物信息学分析是mNGS检测中的重要部分,是对测序产生的原始数据进行处理和分析,最终鉴别出检测样本中所有临床相关的病原微生物物种,为微生物解读人员进一步解读与出具报告提供参考。mNGS应用于临床时的生物信息分析一般包括:数据质控、参考基因组数据库构建、序列比对与物种鉴定。目前,宏基因组测序技术应用于临床病原微生物检测主要以二代测序平台为代表被广泛使用,但mNGS数据分析流程同实验流程一样,较为复杂,尚无该行业内标准化数据分析程序及软件。
mNGS数据分析流程由多种分析软件配套完成。其中,数据质控是对原始测序数据进行质量控制,去除低质量序列、剪切掉模糊碱基(如“N”序列)、剪切掉接头序列、过滤掉过短序列等,常用的软件有Fastp、Trimmomatic、FastQC等。参考基因组数据库构建包括两类,一类是构建人源参考基因组数据库,用于对质控后的测序数据,进一步的去除宿主序列;另一类是构建微生物参考基因组数据库,用于对非人源测序数据进行物种鉴定。微生物参考基因组数据来源较多(如NCBI、PATRIC、EuPathDB、ViPR、Wormbase、RVDB、FDA-ARGOS等),数据质量良莠不齐。
进行构建本地基因组数据库时,需要建立数据质控标准进行筛选,充分考虑微生物基因组的组装水平、基因组版本和基因组组装质量等,并需要进行性能验证,评估物种鉴定的性能。物种鉴定是将非人源序列与微生物参考基因组数据库进行序列比对,常用的软件较多有Kraken2、metaPhlAn2、MegaBlast、PhymmBL等,每个软件都有自己的适用范围和性能特点。
四、报告解读
mNGS结果解读是另一个重大挑战,报告结果需要紧密结合临床。实验室应按照不同临床标本类型和微生物类型,确定各自的阳性阈值和判读标准。例如:无菌部位(血和无菌体液等)与有微生物定植的部位(呼吸道和开放性伤口等)要区别对待。传统微生物学中的正常无菌部位(血液和脑脊液等)在mNGS检测时也可以测到一些低浓度病原体的核酸序列,但是没有微生物活体存在。对于有微生物定植部位的mNGS报告的解读需要紧密联系临床表现、实验室检查和影像学特征等一系列临床指标进行综合解读,并与临床专家密切沟通。mNGS检测的目标是核酸,并非存活的病原体,DNA在体内需要一定时间降解,可能存在病原微生物已死亡但mNGS检测结果是阳性这种看似不相符的情况,应该从方法学角度进行解释与甄别。同时注意mNGS报告解读要与传统培养结果紧密结合,从而更好的指导临床,提高临床诊断率。
另一方面,阳性阈值一般采用受试者工作特征(Receiver Operator Characteristic,ROC)曲线等统计方法确认,然而针对某些特殊传染病病原微生物,即使检出1条序列也应视为阳性(在排除污染的前提下),如MTB、鼠疫耶尔森菌、霍乱弧菌、流行性出血热病毒等。对于某些破壁困难微生物(分枝杆菌属、诺卡菌属、曲霉菌、隐球菌属等)要考虑由于提取效率存在差异,应降低报出阈值,以免漏检。同时这类病原体应采用多方法学(PCR、GM实验、隐球菌荚膜多糖抗原实验等)进行验证,从而进一步提高报告的准确性。
小结:虽然mNGS技术的发展和兴起为感染性疾病的诊断提供了新的途径和思路,但其本身的技术仍需优化和规范。在目前的技术能力条件下,为实现感染性疾病的精确诊断,建议将mNGS与传统培养、其他分子生物学以及血清学检测等方法联合使用,使mNGS测序技术真正的受益于更多医生和患者。
参考文献
Gu W, Miller S, Chiu CY. Clinical metagenomic Next-Generation Sequencing for Pathogen Detection. Annu Rev Pathol. 2019; 14: 319-338.
Li N, Cai Q, Miao Q, et al. High-Throughput metagenomics for Identification of Pathogens in the Clinical Settings[J]. 2021; 5(1): 2000792.
王辉. 宏基因组高通量测序技术应用于感染性疾病病原检测中国专家共识[J]. 中华检验医学杂志, 2021, 44(02): 107-120.
Chiu C Y, Miller S A. Clinical metagenomics[J]. Nature Reviews Genetics, 2019.

欧蒙未一医学检验实验室(以下简称“未一医学”)成立于2014年,是国内最早一批开展病原微生物mNGS检测的第三方实验室,拥有自主研发的mNGS检测技术平台,一次实验可检测目前已知的16000余种微生物、2600多种耐药基因以及600多种毒力因子,创新核提取和微生物富集技术,与中科院生信分析团队合作,建立以“PAI-CRP临床病原体知识库”为核心的专属四级数据库智能分析系统。拥有多领域临床医生组建的临床专家团队,提供专业报告解读支持。未一医学已在北京、杭州和广州分别设有独立医学检验实验室,致力于为风湿免疫科、呼吸科、神经科、儿科等领域提供“多平台”、“一体化”疾病诊断解决方案,覆盖疾病类别逾两千种,合作的三甲医院超千家。质量是立足的根本,创新是前进的动力。未一医学秉承认真、严谨的精神,不断创新,积极推动先进诊断技术在临床和科研领域的应用与发展。

欧蒙医学诊断(中国)有限公司 供稿




