应用机器学习结合全光谱MALDI-TOF检测产碳青霉烯酶肺炎克雷伯菌的研究
【摘要】基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)被认为是未来快速微生物技术发展的重要工具,我们建议将MALDI-TOF MS用于细菌鉴定和耐药性检测的双重技术而无需额外的手工操作。我们开发了一种机器学习(ML)方法,根据完整细胞的光谱,使用随机森林(RF)算法直接预测产碳青霉烯酶肺炎克雷伯菌(CPKP)分离株。我们使用了一个包含4,547个质谱图谱的数据库,其中包括715株不重复的临床分离株,这些分离株由324株具有37种不同STs的CPKP代表。在CPKP预测中,培养基的影响是决定因素,因为与用于构建模型的分离株(血琼脂)相比,这些分离株在相同的培养基中进行检测和培养。该方法预测CPKP的准确率为97.83%,预测OXA-48或KPC携带的准确率为95.24%。对于CPKP预测,RF算法的受试者工作特征曲线下面积(AU ROC)和精确率-召回率曲线下面积(AU PRC)均为1.00。使用Shapley值确定单个质量峰对CPKP预测的贡献,该值揭示了完整的蛋白质组而不是一系列质量峰或潜在的生物标志物,负责基于算法的分类。因此,如本文所述,将全光谱与模式匹配分析算法结合使用可产生最佳结果。使用MALDI-TOF MS与机器学习算法处理相结合,可以在几分钟内识别出CPKP分离株,从而缩短检测耐药性的时间。
产碳青霉烯酶肺炎克雷伯菌(CPKP)的日益增多被欧洲疾病控制与预防中心(ECDC)、美国疾病控制与预防中心(CDC)和世界卫生组织(WHO)等不同组织认为是一个全球卫生问题,因为这些细菌产生的感染与较高的发病率、死亡率和医疗费用相关。碳青霉烯酶可使几乎所有可用的β-内酰胺类抗生素产生耐药性,而β-内酰胺类抗生素是治疗肠杆菌感染最常用的抗生素,因此,早期识别可以改进治疗方案的选择。抗微生物药物耐药性(AMR)的检测通常基于广泛认可的分子技术,然而,这些技术比基质辅助激光解吸电离飞行时间技术(MALDI-TOF)更耗时、更昂贵,此外,分子技术一般都是单基因靶标的窄谱检测,需要全基因组测序(WGS)技术来鉴定细菌基因组。目前基于表型和培养的AMR方法,从样本采集到耐药性报告可能需要48-72h,因为必须先分离细菌,而使用MALDI-TOF作为双重技术,细菌鉴定和耐药性检测可以同时进行。
基于MALDI-TOF MS蛋白组学的生物分型技术可通过分析来自全细胞核糖体蛋白(2-20kDa不等)来识别微生物,这表明这些蛋白在不同种类的细菌中具有高度的多样性,MALDI-TOF MS可在几分钟内鉴别单个细菌物种的蛋白质组成,被认为是未来快速微生物技术发展的重要手段。与WGS等分子技术不同,该方法已经在许多临床微生物实验室中应用。直接从MALDI-TOF MS中提取更多信息还能检测AMR。由于需要加快程序并提高结果的可追溯性,临床实验室对双重技术(细菌鉴定和AMR检测)的需求日益增加。在抗生素耐药性检测领域,MALDI-TOF MS最初用于检测与耐药性相关的标记物。然而,由于缺乏针对所有潜在病原体和药物组合的全面、可靠的标记物目录,人们开始转向更复杂的方法。神经网络、支持向量机和随机森林(RF)等机器学习工具是强大的分类系统,已被用于癌症基因组学等健康科学领域。随着高通量技术的发展,产生大量的数据,这些分类特征适合应用于基于蛋白质组学的临床微生物诊断。已经开发了几种基于MALDI-TOF MS的程序和数据分析程序,然而,这些技术的生物学和技术可重复性方面仍然存在一些不一致之处,此外,缺乏通用的参考质谱数据库限制了MALDI-TOF MS作为一线临床工具的整体适用性。
在之前发表的一篇论文中,我们介绍了用于检测CPKP的MALDI-TOF MS数据分析流程,该流程使用在线Clover MS数据分析软件实现ML分析。此后,我们收集了更大的肺炎克雷伯菌(KP)分离株,以最大限度地减少不同收集地点的影响,并通过使用标记数据集调整模型来改进预测。本研究的目的是验证该程序并证明在样本采集后24h内在临床环境中使用MALDI-TOF MS可以直接追踪CPKP分离株。
一、材料与方法
1. 细菌分离株:该研究收集了具有代表性的715株不重复的临床分离株,其中324株CPKP,在研究的324株CPKP中有307株是在西班牙全国15家医院开展的产碳青霉烯酶肠杆菌(CPE)调查中收集的,该调查在2018年为期两个月的时间内由西班牙传染病和临床微生物学会(SEIMC)与西班牙传染病研究网络(REIPI)推动进行,其他17株是我们自己收集的,391株非产碳青霉烯酶肺炎克雷伯菌(NCPKP)来自西班牙不同的医院。根据欧洲抗微生物药物敏感性试验委员会(EUCAST)推荐的筛选临界值(cut off值,即通过自动微量稀释法获得的美罗培南或厄他培南MIC高于0.125mg/L的菌株)对分离株进行碳青霉烯酶产生的筛选。采用全基因组测序(WGS)对全国调查的307株分离株进行特征分析。使用Genomic-Tip 20/G(Qiagen)的基因组DNA缓冲液套装获得总基因组DNA。使用短读长(Illumina MiSeq benchtop)和长读长(MinION)方法对所有分离株的纯化基因组DNA进行平行测序,用Unicycler v0.4.6组装每个分离株的长读长和短读长,使用Bandage软件包对重叠群(contigs)进行可视化。使用Prokka v1.13对获得的组装结果进行注释。使用Resfinder v3.2软件包和综合抗生素耐药性数据库(CARD)对分离菌株的总抗生素耐药性基因含量进行了计算机模拟分析。多位点序列分型(MLSTs)是利用现有的在线数据库,从组装的全基因组测序数据中进行计算机模拟。
我们采用常规基因组技术对实验室收集的17株分离菌株进行了鉴定。采用PCR法检测碳青霉烯酶编码基因OXA-48和KPC。采用煮沸法提取DNA。使用特异性寡核苷酸扩增不同基因(OXA-48 Fw:GCGTGGTTAAGGATGAACAC;OXA-48 RV:CATCAAGTTCAACCCAACCG;KPC Fw:CGTCTAGTTCTGCTGTCTTG;KPC Rv:CTTGTCATCCTTGTTAGGCG)。通过对PCR产物进行测序,确认了不同碳青霉烯酶基因的存在。按照参考方案,在以下条件下进行肺炎克雷伯菌的MLST分析:94℃初始变性2min;94℃ 20s、50℃ 30s、72℃ 30s共35个循环;72℃下延伸5min。将核苷酸序列与MLST数据库中的现有条目进行比较,以生成等位基因编号和确定STs。不符合碳青霉烯酶筛查EUCAST标准的分离株被归类为非产碳青霉烯酶的分离株,未对这些分离株进行耐药基因含量或克隆复合体分析,将细菌分离株随机分为两个集:训练集和验证集。
2. 获取MALDI-TOF MS光谱:MALDI-TOF MS和数据处理工作流程总结如图1。将训练集中的细菌分离株保存在-80℃的加入玻璃冻存珠小瓶中。需要时,将分离株在血琼脂平板上解冻,方法是用无菌环从管中取出一颗冻存珠,然后将其在琼脂表面滚动。培养18h后,在平板上继续传代培养18h,平板在37℃的有氧环境中孵育。为了控制与衰老相关的质谱变化,分析的所有分离株的菌龄相同。对分离株进行改良Hodge试验,以检查是否存在碳青霉烯酶。表型和基因型注释方面未发现不一致的结果。同一操作者分析了训练集中的所有分离株,以减少相关的变异。
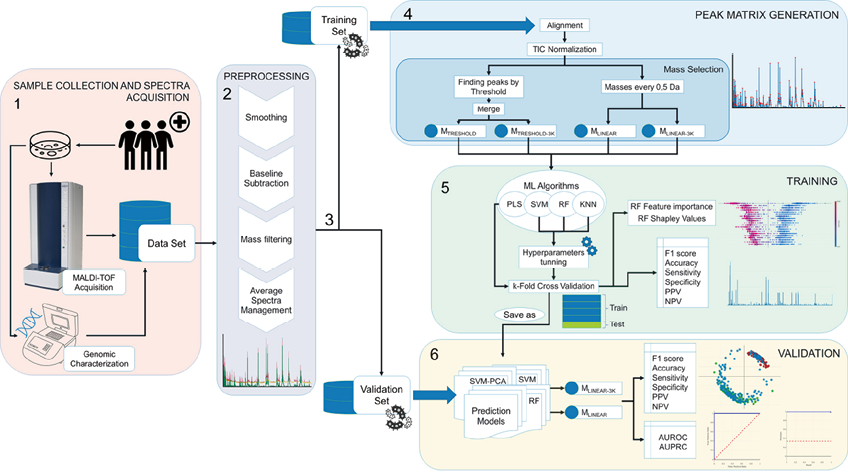
注:1. 样本采集和光谱采集;2. 预处理;3. 数据分割,分为训练集和验证集;4. 生成峰值矩阵;5. 训练;6. 验证
图1. 基于MALDI-TOF MS的CPKP预测工作流程
在没有标准化培养条件的情况下分析了验证集中的细菌分离株。为了改进现有方法,并设计一种使用通用样本培养基的程序,验证阶段首先使用了不同的培养基。质谱图谱会受到培养基相关的变化和差异,这是由于培养基中所含的营养物质不同,导致细菌生长的生物学差异。我们想说明基于质谱的AMR预测所面临的挑战和局限性,因此研究了是否可以从不同的细菌培养基(血琼脂、巧克力琼脂和麦康凯琼脂)开始进行CPKP分类,最多有三名操作者随机对验证集进行分析。通过“靶向”蛋白质提取法分析细菌蛋白质组,简而言之,该方法包括从MALDI靶标中直接提取蛋白质,将1μL甲酸滴在干燥样本上,然后添加IVD HCCA基质。在训练集中处理每个分离株的九个重复(三个点,每个点有三个光谱),在验证集中处理一个重复(一个点,一个光谱)。采用Microflex LT/SH SMART质谱仪,FlexControl 3.4软件在线性正离子模式下进行MALDI-TOF光谱采集,质量范围为2-20k Da。每次运行前使用细菌检测标准(BTS)进行外部校准,采用MALDI Biotyper Compass软件包(v.4.1.100)与质谱文库比对确认菌种,碳青霉烯酶耐药分析要求>2.0分。
使用Clover MS数据分析软件(Clover Biosoft)对光谱进行预处理。第一步是用Savitzky-Golay滤波器对所有光谱进行降噪预处理,然后用Top-Hat滤波器减去基线。在构建模型时,通过连续的对齐和合并过程,获得训练集中每个分离株的平均光谱,这样可以最大限度地减少同一分离株重复样本之间的差异。这一过程完成后,每个分离株只剩下一个平均光谱,所有光谱都会相互对齐。所有对齐过程都是通过考虑对齐集合中包含的每个样本最具代表性的峰值进行的,这些峰随后被用来创建参考峰列表。每个光谱峰都在2,000ppm的线性容差范围内移动,以与该列表相对应。在对训练集中的每个分离株进行预处理并形成平均光谱后,在两个不同范围内应用两种不同的质量选择方法:2,000-20,000 m/z(MTHRESHOLD和MLINEAR)和3,000-20,000 m/z(MTHRESHOLD-3K和MLINEAR-3K)。由此,共得到4个峰矩阵,MTHRESHOLD和MTHRESHOLD-3K方法的峰值矩阵是通过应用阈值算法(0.01)生成的,因此在两个范围内都考虑了至少为光谱最大强度1%的峰值。在600ppm的线性容差和3Da的恒定容差内,所有得到的峰合并为一个共同列表。MLINEAR和MLINEAR- 3k方法的矩阵是通过将每个分离株的整个谱合并在一个质量列表中获得的,每个范围值为0.5Da。随后用总离子电流法(TIC)对峰矩阵进行归一化处理。
3. 用于识别CPKP分离株的ML算法:通过对每种方法使用两个训练集得到4个不同的矩阵:MTHRESHOLD、MTHRESHOLD-3K、MLINEAR和MLINEAR-3K。这些峰值矩阵被用作四种监督机器学习算法的输入数据,这些算法随后被应用于这些矩阵:偏最小二乘判别分析(PLSDA)、应用和不应用主成分分析(PCA)的支持向量机(SVM)、应用和不应用近邻成分分析(NCA)的k最近邻(KNN)和随机森林(RF)。训练算法并优化其超参数(适用时)。然后,通过计算 K折交叉验证(k-fold cross-validation)得出的指标对训练步骤进行评估,对算法进行训练和评估后就可以为每种方法和峰值矩阵建立预测模型。使用外部验证集对这些预测模型进行验证。验证集中的样本按照训练集的相同程序进行预处理。验证样本被用作预测模型的输入数据,应用各自的质量选择方法进行分类。这些样本先前已被分类,以便比较(并由此评估)预测模型获得的结果与实际分类结果。本研究分两步进行,第一步鉴别CPKP菌株,第二步鉴别不同碳青霉烯酶型别:blaOXA-48-like,blaKPC,blaNDM和blaVIM。
4. 评估指标和统计分析:对于24种分析组合中的每一种,我们报告了用于评估性能的主要指标。对于训练中的内部评价,使用10折交叉验证(k=10)进行CPKP鉴别。在第二步中,可用于鉴别碳青霉烯酶类型的两个少数类别样本数量有限,因此实施了7折交叉验证(k=7)。报告的性能指标如下:准确度,用正确分类的百分比表示;准确度和灵敏度的调和平均值,用F1分数表示;灵敏度;特异性。还报告了RF分析算法在鉴别CPKP分离株方面的重要特征。分析它们的贡献有两种方法:直接获得作为RF函数输出之一的特征重要性值以及计算Shapley值。在第一种方法中,特征重要性值越高,表明对分类树中样本的分割贡献越大。特征重要性计算为该特征所带来的标准的(归一化的)总减少,这也称为基尼系数的重要性。Shapley值以多元的方式衡量特征的重要性。因此,特征在列表中的位置越高,对区分阳性类别就越重要。预测器使用高强度值或没有测量到的强度判定阳性类别。
为了验证,我们还报告了受试者工作特征曲线下面积(AU ROC)作为主要性能指标之一,平均精度(AP)和精准率-召回率曲线下的面积(AU PRC)用来描述精准率-召回率曲线下的值。AU ROC表示真阳性率(即真阳性率与假阳性率的比值),用于评估模型的鉴别能力,该值直接表示模型在成对类别之间的判别能力(即值越高表示能力越强)。另一方面,AU PRC或AP-PRC表示召回率与精确度(即,正确检测出阳性样本,同时最小化假阳性百分比的能力)的比率。在不平衡数据集的情况下,AU PRC得到了较高评价,因为AU ROC并不反映相对于精确度(或阳性预测值)的性能,因此,AU ROC值可以很高,而精度却很低。在本例中(324株CPK/391株NCPK),两组数据几乎是平衡的,但是,精确度在临床环境中的重要性使得该工具对分析很有价值。
二、结果
1. 细菌分离株:715株分离株代表来自西班牙各地15家不同医院的样本。CPKP分离株共检出37种不同的序列型(STs)。324株CPKP分离株中,228株携带blaOXA-48基因,82株携带blaKPC基因,7株携带blaNDM基因,7株携带blaVIM基因。在715株代表性分离株中,作为训练集的479株由246株CPKP分离株和233株NCPKP分离株组成。CPKP菌株包括170株产OXA-48菌株、62株产KPC菌株、7株产NDM菌株和7株产VIM菌株。我们从整个数据库中随机选择了分离株,但不包括产NDM和产VIM的分离株,因为两组的分离株数量都很少,因此有意选择这两种分离株作为训练集的一部分。我们在3个不同的点对这些分离株进行了3次重复检查,从而产生了构成数据库的4,311个光谱。剩余236株作为验证集,包括78株CPKP和158株NCPKP。其中58株产OXA-48菌株,20株产KPC菌株。进一步分析了获得的236个光谱,用于该程序的临床验证。分离菌株来自不同类型的样本:活检样本、导管样本、胆汁、腹腔液、脓肿、呼吸道样本、伤口样本、血培养、尿液样本和不明来源的样本。
2. 基于MALDI-TOF MS的CPKP预测的ML:(1)训练:在检测CPKP和随后区分碳青霉烯酶类型这两个步骤中,所有24种方法和分析算法组合都获得了较高的kappa值(>80)。对MLINEAR和MTHRESHOLD方法的评价显示,在CPKP检测步骤中,MLINEAR方法的准确率平均较高,正确鉴定出97.95%的CPKP菌株(MTHRESHOLD方法正确鉴定出96.44%的CPK菌株)。对算法(PLS、SVM、PCA-SVM、KNN、NCA-KNN和RF)的预测性能进行评估后,对所有方法(MLINEAR和MTHRESHOLD)进行平均,其结果表明,RF算法的性能最佳(准确率为99.27%)。对分析中质量范围影响的评估显示,考虑到从2-20k Da光谱中所有质量峰会产生出色的度量性能(即MLINEAR方法与RF算法相结合可提供最佳指标[准确率为99.79%,灵敏度为100%,特异性为99.57%])。在第二步中,MLINEAR方法也找到了最佳指标,其正确识别率为87.85%(MTHRESHOLD方法为86.65%)。对5种分析算法(PLS、SVM、KNN、NCA-KNN和RF)的预测性能进行评估,并对所有方法(MLINEAR和MTHRESHOLD)进行平均,结果表明RF算法的性能最佳(准确率为90.39%)。在这一步中,由于之前的PCA分析无法进行聚类,因此省略了SVM和PCA的组合。根据我们的结果,能够区分肺炎克雷伯菌中碳青霉烯酶的分类器是采用MLINEAR方法的RF算法,它的性能指标非常出色,无论是分析全部质量范围还是仅使用3kDa以上的质量峰,准确率都达到了90.91%。
然后,确定特征重要性和Shapley值,用于评估RF算法的CPKP预测性能。图2显示了平均贡献最高的30个特征的平均Shapley值和每个数据点的Shapley值,对于每个特征(每行),每个样本都有一个点(Shapley值),点向右移动表示对选定为阳性类别(CPKP)的贡献更大。图2中,每个特征的分布图尾部的颜色表示最高(红色)或最低(蓝色)特征值。因此,我们可以看到RF算法使用高强度值(红色)或无任何测量值或低强度值(蓝色)来预测CPKP。对于Shapley分析来说,大多数质量(特别是在MLINEL方法中)是那些在CPKP预测中以高强度出现的质量。位于3,514Da的质量峰对CPKP预测的贡献很大,在两种方法中都有体现。由于没有在很大程度上通过MS/MS分析蛋白质序列,蛋白质的理论鉴定与碳青霉烯耐药性无关,而是与糖苷酶(Uniprot:A0A2V1LFY6)有关,因此模式匹配变得更加重要。该质量峰的分布与分离株的ST无关,因为在所有被分析的CPKP中它都是一个高强度的质量峰区。
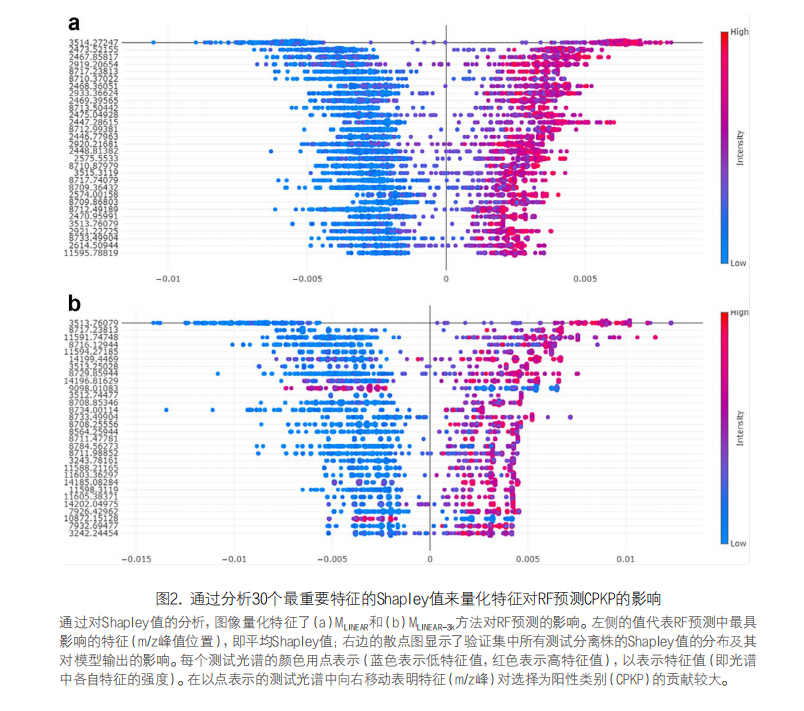
此外,分析表明任何特征的存在与否本身与CPKP的分类无关,任何特定特征的重要性在两种方法之间是否为CPKP进行平衡,这一发现证明了分析一系列质量峰而不仅仅是特定质量峰的重要性。此外,在MLINEAR方法中,观察到几乎所有(29/30)具有最高平均影响的特征箱(bins)都是质荷比(m/z)值小于10,000 Da的特征箱。在MLINEAR-3K的情况下,在预测影响最大的30个特征箱中,该比例下降到19个。这可以归因于MLINEAR方法中使用的30个特征箱中有15个质量低于3,000 Da,这意味着MLINEAR-3k方法可以探索其他质量范围进行分类,因为结果在两个质量范围之间非常接近。这一发现再次证明了分析一系列质量峰的重要性,而不仅仅是分析可以充当生物标志物的特定质量峰,从而突出了ML工具的重要性,因为它们能够分析大数据,而不是关注质量峰的存在与耐药表型之间的纯粹相关性。特征重要性的扩展条形图(图3)表明,在这两种方法中,分类器都使用了整个质量范围内的某些区域,能够适应不同的情况,在这种情况下,MLINEAR中2k-3k Da的特征重要性与MLINEAR-3k方法中3k Da的其他特征的重要性相抵消。
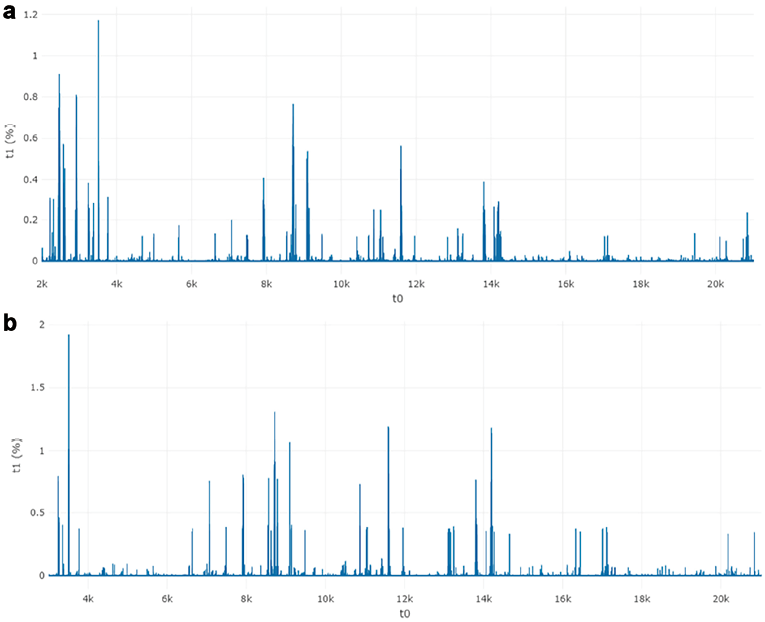
注:图表示在(a)MLINEAR和(b)MLINEAR-3k方法的情况下,每个质量峰对于RF分析结果的重要性,x轴表示个体样本质量,y轴表示在将每个分离株分配到一个分析类别(CPK或NCPK)的决策中,每个质量峰的重要性百分比。
图3. CPKP预测中RF算法的特征重要性条形图
(2)验证:首先试图确定使用不同样本培养基是否会影响AMR的预测。为此,我们使用在血琼脂、巧克力琼脂和麦康基琼脂上培养的临床样本测试了数据集分类器。观察到在巧克力琼脂和麦康基琼脂中收集的分离株分类均较差,这凸显了培养基在算法预测性能中的重要性。我们将这种性能下降的原因归结为营养物质对细菌生长的影响决定了某些肽和蛋白质表达的差异。如图4所示,首次应用PCA 的所有分离株(n=114)的分层聚类(HC)显示出三个明确的聚类,每个聚类都与特定的培养基相对应。该图像还包括一个热力图来说明差异,所有分离株均为NCPK,分类的差异完全取决于培养基。
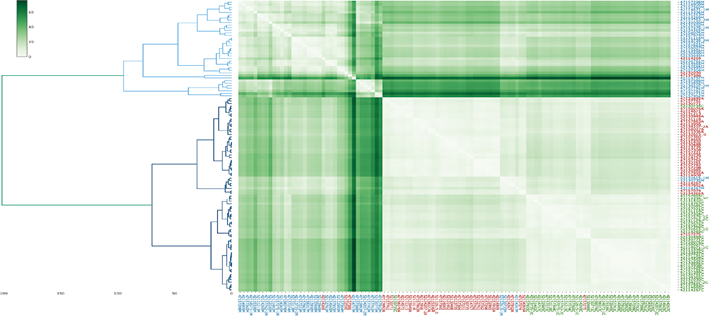
图4. 生长培养基对肺炎克雷伯菌细菌蛋白质组的影响
在0.1阈值峰值矩阵中,在114株NCPK中应用带有热力图的PCA的分层聚类。数据被缩放,样本间的欧氏距离用绿色刻度和Ward’s度量表示. 在麦康凯琼脂中培养之前获得质谱的分离株用蓝色表示,血琼脂上培养的分离株用红色表示,巧克力琼脂上培养的分离株用绿色表示,绿色刻度表示成对分离株之间的距离。
验证阶段的kappa值略低于训练阶段,尤其是采用MTHRESHOLD方法时。在CPKP检测步骤中,MLINEAR方法和MTHRESHOLD方法正确识别CPKP的平均准确度分别为93.60%和84.71%。因此,与分析方法无关,MLINEAR方法的一致性较高,相对于数据集,准确度仅下降4.35%,代表了优秀的模型性能。在MTHRESHOLD方法中,准确性下降了11.73%(即较MLINEAR方法下降了2倍以上)。对6种分析算法(PLS、SVM、PCA-SVM、KNN、NCA-KNN和RF)在方法(MLINEAL和MTHRESHOLD)和质量范围的预测性能评估表明,最能提供精确CPKP预测的分类器是RF算法和MLINEAL方法。也就是说,考虑到2-20 kDa谱图中的所有质量峰,得出了出色的性能指标,准确度为97.83%,灵敏度为100%,特异性为96.73%,F1分数为96.85%。在区分碳青霉烯酶类型的第二阶段,算法的表现甚至比训练数据集更好,在20种方法-算法组合中的8种几乎分类完美。因此,根据我们的结果,当应用MLINEAR方法时,最能区分产KPC和OXA-48肺炎克雷伯菌的分类器是RF算法,该算法产生了出色的性能指标,在分析整个质量范围时准确率为98.70%。
我们还将AU ROC和AU PRC作为性能指标进行报告。AU ROC可以理解为正确分类一对样本(即CPKP或NCPKP)的概率,而AU PRC则量化了从不平衡的二元分类(CPKP/NCPKP)中正确检测样本的能力,同时最大限度地减少错误结果。我们决定从进一步研究的分析中排除MTHRESHOLD方法和KNN、NCA-KNN算法,因为它们的性能低于其他方法-算法组合,且不能提供任何相关信息。我们还决定排除只考虑3k Da以上的质量峰的质量范围分析,因为它们提供的结果与之前度量中使用完整光谱的结果相似,而且略差。我们分析了最佳模型(RF)预测分离株是否为CPKP的能力,观察到AU ROC和AU PRC均具有较高的总体性能。PLS和RF是唯一对CPKP分离株具有显著区分能力的算法,其AU ROC和AUPRC均高于0.90,更具体地说,RF算法对CPKP分离株进行了完美的分类,其AU ROC和AU PRC均为1.00。在区分碳青霉烯酶类型的第二阶段,由于训练集中只使用了产NDM和VIM的分离株,而这两类分离株的数量较少,因此只对预测产OXA-48样和产KPC分离株的算法进行了验证。在第二阶段,SVM和RF算法在鉴别CPKP分离株方面表现突出,RF算法再次表现最佳,对产OXA-48样分离株的AU ROC值为0.99,MLINEAR方法的AU ROC值为0.978,对于产KPC分离株,AU PRC为0.97,AU ROC为0.975。
三、讨论
操作程序分析和后续数据管理对于从MALDI-TOF MS质谱图中恢复信息至关重要。在前期的研究中,我们分析了操作过程的生物学和技术可重复性,评估了这种差异对最终结果的影响,并提出了一种改进的用于鉴定CPKP的MALDI-TOF MS数据分析流程。在本研究中,我们使用了更大样本的肺炎克雷伯菌分离株来验证该方法,以便该方法能够完全融入临床微生物实验室的正常工作流程,我们已经证明,基于MALDI-TOF MS的常规临床诊断样本的CPKP预测可以在样本采集后的24h内提供准确的预测。
Weis等人报告称,在一个地点质谱上训练的分类器的性能不能推广到在其他地点质谱测量,因为分析可能受到许多因素的影响,包括不同的系统发育菌株、不同的抗性流行率、技术变异性以及不同的机器特定参数和设置。上述研究和本研究均局限于特定国家(分别为瑞士和西班牙)。我们没有通过基于不同地点构建不同的数据库来比较分类器的结果,因为我们的研究中包含的光谱数量要少得多,然而,使用我们的数据库获得的预测准确率为97.83(即优秀分类),该数据库由具有不同系统发育起源的分离株构建(研究中检测到37种STs)。请注意,未正确分类的分离株(n=5)都是来自同一地点(西班牙拉科鲁尼亚大学医院综合楼)的NCPKP分离株,并被鉴定为CPKP。因此,数据库的完善是一个重要因素。探索为较小区域构建特定分类器的可能性是一个可行的解决方案,使用最新和相似的分离株进行训练可以提供更好的预测性能,因为数据中的差异当然有可能降低算法的性能。然而,一般来说,大型数据集可以提高训练算法的性能。不能保证我们构建的MALDI-TOF模型适合细菌的所有生物学和流行病学变异,因为我们没有在不同中心进行随时间推移的前瞻性验证。因此,我们建议使用已知的、有特征的分离株对该方法进行单独的初步验证,以便在需要时,数据库可以适应个体的、区域特定的特征,并定期用新的分离株重新评估数据集。因此,该方法将被训练以识别进化细菌的变化和适应性。
培养基类型和培养条件会产生一些影响,但不会影响MALDI-TOF MS鉴别细菌种类的整体能力,这一点在之前的研究中已经得到了证实。不过,我们观察到培养基会影响CPKP的预测性能。必须考虑到被评估的分离株应与数据库中的分离株生长在相同的培养基(在本例中为血琼脂)上,当然,这也是该方法目前存在的局限性。因此,应进一步改进算法,尽量减少培养基产生的差异,以提高该方法的临床适用性。
很少有研究考虑将全质谱而不是单峰用于抗微生物药物预测,然而,耐药表型峰值预测的负面影响已有报道。该方法的一个缺点是依赖可能与耐药表型相关但不能与分离株的功能特征直接相关的单一生物标志物,此外,与单一生物标志物相关的变异性更高,特别是在蛋白质缺乏恒定的特异性表达的情况下。许多引起耐药性的蛋白质超出了MALDI-TOF质谱的有效质量范围,例如,肺炎克雷伯菌中的β-内酰胺酶重约30 kDa。因此,我们提出了一种基于模式匹配的算法,通过使用完整的蛋白质组而不是特定的质量峰来与细菌产生的耐药相关变化进行匹配。还评估了预测性能是否主要由部分峰值驱动或者全谱。Shapley值表明,多个质量峰(而不是一个特定的质量峰)有助于CPKP的鉴定。蛋白质组分析从3 kDa开始,以尽量减少2-3 kDa质量范围内噪声的影响,因为该部分光谱的噪声通常较大。不过,使用MLINEAR和MLINEAR-3K方法得到的结果很相似,这表明该算法消除了噪声的潜在影响,并突出了其对不同光谱的适应性。此外,Shapley值表明,极高或极低的特征值(对应于MALDI-TOF质量峰的存在或不存在)对预测结果有贡献,而不是特征幅度的较小变化,这与我们对MLINEAR和MLINEAR-3K方法的结果一致,证实了蛋白质检测是预测能力的决定因素,而不是将信号与噪声混为一谈。因此,研究结果支持提出的一系列质量峰的分析,而不仅仅是可以作为生物标志物的特定肽或蛋白质。将重点放在模式匹配上,而不是核心峰上,通过使用构建良好的数据库,而不是物种特异性峰恒定表达(已经被证明不存在),将提高细菌耐药性鉴定的可靠性。
对碳青霉烯酶类型的预测准确率为95.24%。纳入其他VIM和NDM型碳青霉烯酶将使该方法能够在临床环境中对这些组碳青霉烯酶进行验证。对于OXA-48和KPC型碳青霉烯酶,模型均表现出良好的性能,AU ROC和AU PRC值均大于0.95,这有利于OXA-48型的鉴定,因为这组菌株的数量较多(228株OXA-48,82株KPC)。在全球范围内,ST11、ST14、ST101、ST147和ST258/512是产碳青霉烯酶的主要肺炎克雷伯菌克隆。在我们的研究中共检测到37种STs。CPKP分离株的克隆结构结果显示,ST-15、ST-147和ST-392只表达OXA-48碳青霉烯酶,而ST-512在所有分离株中都与KPC有关。其他克隆(如ST-307)在两种碳青霉烯酶类型中的分布相似,ST-11 虽然在两组中都有表达,但在OXA-48分离株中占主导地位。两组中克隆的巨大变异性以及某些克隆同时携带两种碳青霉烯酶类型的事实证明了MALDI-TOF MS卓越的分类性能。在特定流行病学环境中,例如,在产OXA-48或产KPC的肺炎克雷伯菌分离株流行率高的地区,在获得分子结果之前确定碳青霉烯酶的类型将有所帮助。因此,MALDI-TOF技术有助于快速制定针对碳青霉烯酶型菌株的靶向治疗方案,如新型β-内酰胺/β-内酰胺酶抑制剂,头孢他啶-阿维巴坦和亚胺培南-瑞巴坦,这些抑制剂专门设计用于对抗碳青霉烯酶型分离株。
总之,研究结果表明,基于MALDI-TOF MS的机器学习可以提供新的方法来预测高度相关的临床场景中抗微生物药物耐药性,例如CPKP鉴定。我们认为,这种基于MALDI-TOF技术的理想应用是作为临床实验室中CPKP分离株鉴定的初步筛选测试,因为可以在细菌鉴定的同时,使用相同的操作程序和相同光谱进行直接跟踪,唯一的区别是机器学习算法的后处理,这只需几分钟即可完成,唯一需要的额外步骤是将光谱上传到配置了上述算法的分析平台中,生物信息学处理是完全自动化的。因此,除了MALDI-TOF MS的基本使用之外,不需要任何特殊技能,这使得该技术能够完全融入日常实验室工作中。由于菌血症和/或脓毒对临床影响很大,因此该方法未来的一个潜在应用是直接从菌血症和/或脓毒的阳性血液培养物中鉴定CPKP。虽然不能单独使用耐药性的预测,但缩短血培养中报告CPKP的时间可能有助于临床决策以及抗菌药物管理规划,从而显著减少治疗降级的时间。该方法还具有扩展到细菌鉴定/耐药检测的其他组合的巨大潜力,在潜在的暴发疫情中,基于MALDI-TOF MS的CPKP分离株鉴定可能为启动重症医疗和感染控制措施提供第一手证据,从而改变这些感染的临床结局。
编译节选自:《J Clin Microbiol》.v.61(6); 2023 Jun.




