胃肠微生物群和结肠癌
结肠直肠癌(CRC)是世界第三大常见癌症,每年产生超过140万新诊断病例,并造成近一成的癌症总诊断人数死亡。在全球范围内,CRC的发病率因地域的变化而不同,并且与称之为西方生活方式的关键要素关系密切。男性CRC的发病率比女性高,而且发病率随着年龄上升。发达国家的中值诊断年龄大约为70岁。尽管已知许多癌症如CRC和遗传以及环境因素有关,但是病毒/细菌/寄生虫感染是此种癌症发展的强大风险因素。国际癌症研究机构发现几乎20%的新发生癌症病例是由归类为人体致癌物质的特定的致病因子导致。幽门螺旋杆菌、乙肝病毒、丙肝病毒、人乳头瘤病毒是大部分癌症病例产生的原因。特定病原体引起癌症的范例如幽门螺旋杆菌引发胃癌、乙肝病毒引发肝癌,详见表1。与此相反,大量的研究将肠道微生物群与结肠癌症的发展联系起来,指出其具有和特定病原体一样的肿瘤促进作用,而且集体微生物群也有这样的作用。这强调了实际上与现有的报告相比,更多的癌症的发生都至少有部分微生物因素。
表1、和癌症相关的微生物
| 微生物 | 癌 症 |
| 艾普斯登-巴尔病毒 | 淋巴瘤、鼻咽癌、卡波西肉瘤 |
| 人类疱疹病毒8 | 卡波西肉瘤 |
| 幽门螺旋杆菌 | 胃腺癌、粘膜相关淋巴瘤 |
| 人乳头瘤病毒 | 口咽癌、宫颈癌、肛门与生殖器癌 |
| 乙肝病毒 | 肝细胞癌 |
| 丙肝病毒 | 肝细胞癌、淋巴瘤 |
| 肝吸虫 | 胆管癌 |
| 梅克尔细胞多瘤病毒 | 皮肤癌 |
| 血吸虫 | 膀胱癌 |
| 伤寒沙门菌 | 胆囊癌 |
| 溶血性链球菌 | 结肠癌 |
| 肺炎衣原体 | 肺癌 |
大量的微生物存在于人体肠道内,比人体细胞至少多10个量级。尽管个体间存在自然变化,但人体内肠道微生物的复杂程度相对恒定,发生变化部分受宿主遗传学的影响还受到饮食、年龄和健康状况的驱动。胃肠道内,大肠的微生物数量最密集,代谢最为活跃(每克含量>1011个细胞),并受2个主要门类控制—厚壁菌门和拟杆菌门。大部分的厚壁菌门术语2个主要类型—梭菌群XIVa和IV。大部分的拟杆菌门术语拟杆菌属。其他的门类包括变形菌门、放线菌门、疣微菌门和梭杆菌门,但是数量较少。据估计预测的基因组大小/胃肠道微生物群含量比人类基因组多2个数量级。这强调了肠道微生物的潜在新陈代谢能力等同于肝脏。因此,将其称之为‘器官’并不奇怪,这些微生物群显著促进了一些宿主进程,包括关键生长因子的合成、饮食成分的解毒、防范病原生物体、改变宿主细胞增殖还能影响免疫功能。肠道微生物群不是在一旁凑热闹的旁观者,它们对人类的健康产生着巨大的影响。
尽管存在强大的遗传因素,但是大部分CRC病例(>95%)是不定时发生的且在开始的几年内发展缓慢。患有炎症性肠病的个体,尤其是那些患病时间长病情较严重的疾病,增加了CRC的风险,如结肠炎相关癌症(CAC)。由于在疾病出现症状/最初发生之前,没有关于患者肠道微生物群的详细研究,因此确定是CRC或CAC引起了肠道微生物群变化,还是肠道微生物群变化引起了CRC或CAC是一个极大的挑战。尽管世界范围内对潜在微生物出发了疾病的发生进行了深入的理解,但是这需要超过数十年的时间对大量的参与者进行筛查获得足够的有价值的分析数量。
CRC通过一系列的遗传变异发展,这些变异被认为能驱使正常黏膜向癌前病变如腺瘤息肉发生恶性进展,向恶性肿瘤进展通常需要多年—很可能数十年。这一致病进程称之为腺瘤—癌变续发事件。有趣的是,尽管所有的腺瘤息肉都有恶化的可能,但是不是所有的腺瘤息肉都会癌变,这就证明了仅仅因为遗传变异不足以驱动其癌变为恶性肿瘤。但是很明显,腺瘤息肉的病理学特点(大小、组织学等级和中的异常增生组织)能够预测腺瘤息肉恶化的可能。腺瘤息肉沿着肠道黏膜发展并不断受到物理损伤和氧化损伤。这样的创伤必然会影响腺瘤的生理学,与健康结肠组织相比,这样的腺瘤生理学导致了慢性发炎,庇护了更多数量的炎症细胞。有证据表明,尽管炎症并不是恶性的,变异的微生物群落仍与腺瘤息肉有关,这表明微生物群的变化是恶性肿瘤的前奏。
大量的动物研究已经证明肠道微生物群参与CRC的进展。无菌动物研究(无微生物的动物)已经连续证明与常规饲养的对照组相比,无菌动物的肿瘤负荷较低。同样,已经证明通过使用抗生素消耗肠道微生物群载量能够减缓CRC的发展。鼠科致瘤性研究已经显示了肠道微生物群成分的不同点(化学诱导肿瘤发生)。已经记录了拟杆菌属、Akkermansia菌属和odoribacter菌属的数量增加,同时普氏菌属、卟啉单胞菌属数量下降。人类研究也观测到了类似的变化如拟杆菌属数量增加,包括拟杆菌脆弱类杆菌,以及一些变性菌门数量增加包括肠杆菌属弯曲菌属,以及梭杆菌门成员数量增加包括具核梭杆菌和纤毛属菌。这些潜在病原生物体数量的增加与有益菌种包括多种毛螺旋杆菌科数量的下降有关。很明显,目前潜在致癌细菌的名单仅仅初步确立,这一事实表明不仅微生物新陈代谢功能和/或不针对特定个体病原体的途径驱使癌症进展,微生物群更是促使癌症进展的主体。似乎如果发生肠道微生物群失衡,就会潜在导致非共生生物体增殖。但是似乎非共生生物体的整合没有规则,本地环境容易发生生物群失衡。因此,一些个体中可能包括驱使CRC 发展的潜在致病生物体。
特殊微生物和CRC风险
大量的微生物微生态失衡研究得到的尤其强大的证据表明特殊生物体是CRC易感性增加的原因。与健康对照组相比,CRC患者的肠道微生物群中核梭杆菌更为常见(腺瘤性息肉阶段以及恶性肿瘤阶段)。研究还证明核梭杆菌通过黏膜骨髓细胞群补充在具有腺瘤性结肠息肉病的(Apc)Min/+小鼠模型中驱使肠道肿瘤发生。核梭杆菌表达FadA—一种结合到宿主钙粘蛋白细胞表面毒性因子,导致直接影响上皮细胞增生和增殖的β连环蛋白途径的活化。FadA的其他影响包括肠道通透性变异、提供其他菌群渗透上皮的机会并影响宿主免疫系统。
其他涉及CRC的细菌包括产肠毒素拟杆菌属脆弱拟杆菌(ETBF)和促使CAC而非不定时发生CRC结肠肿瘤发生的大肠杆菌,溶血性链球菌亚种和肠球菌。
根据毒性因子脆弱拟杆菌的活动机制,ETBF并不是CRC的微生物起始因子。和拟杆菌的活动模式类似,脆弱拟杆菌诱导E钙粘蛋白(一种肿瘤抑制基因)分裂,导致Wnt/β连环蛋白信号增加,细胞增殖增强,原癌基因c-MYC表达增加。拟杆菌还上调NF-kB信号,因此增加促炎细胞因子分泌,并且直接诱导DNA损伤,进一步促进慢性炎症级联反应和CRC发展。(Apc)Min/+小鼠模型定殖研究证明ETBF同时加快时间进程并改变结肠肿瘤的分布。后续的机制研究发现黏膜IL-17反应诱导Stat3活化。尽管一项侧重于ETBF患病率的研究发现C与匹配的对照组相比,检测出ETBF的人群CRC人群更容易出现CRC病例,但是目前Stat3活化参与人体CRC的确认仍然很有限。
连续的研究表明与无炎症的组织相比,发炎的结肠粘膜的肠杆菌科(尤其是大肠杆菌)数量增加。IBD(CRC的一种已知风险因素)患者的肠道微生物群中肠杆菌科(包括大肠杆菌)的水平上升。尽管变形菌们激增的机制仍然不明,但是它们有可能利用宿主产生的炎症产物(如硝酸盐)作为能源,是他们在肠道中与其他菌群相比获得竞争优势。IBD和CRC患者体内分离的大肠杆菌比较性基因组分析显示存在一定数量的能潜在提供选择性优势的毒性因子。包括多种多样的毒性,例如细胞膨胀致死毒素和colibactin都能诱导直接的DNA损伤,导致染色体不稳定。
肠道微生物和肠道炎症
结肠粘膜不间断地暴露于肠道微生物群,处于‘持续不断的或生理学炎症’状态。黏膜是一种单细胞厚度的上皮,执行着过多的功能包括营养吸收/分泌,物理障碍功能以及免疫调节。上皮和肠道微生物群的交互通过对肠道免疫内环境稳定有深远影响的微生物群形成肠道环境。肠道微生物群的构成能产生炎症并影响免疫细胞的补充/调节。上皮必须与微生物细胞以及主要通过细菌产生的微生物代谢物相抗衡。代谢物组学暴露可以通过防护净平衡抑制癌症发生,还可以通过有致癌潜力的有害代谢物促使癌症发生。微生物产生的短链脂肪酸(SCFA)包括来自饮食纤维素发酵的醋酸盐、丙酸盐、丁酸盐,通常认为富含纤维的食物能降低CRC风险。SCFA能从肠道快速吸收,但是对不同的细胞类型产生一系列的影响,包括对骨髓细胞和表达转录因子FOXP3的结肠调控T细胞产生抗炎效果,对肿瘤相关炎症产生影响。丁酸盐是结肠细胞的首选能源。SCFA包括丁酸盐能被出现在结肠细胞和选定的免疫细胞表面G蛋白结合受体(GPR41、GPR43和GPR109A)识别。结肠细胞和免疫细胞内丁酸盐和丙酸盐抑制组蛋白去乙酰化酶的活性。这两个功能都能导致结肠调控细胞和抗炎细胞因子的总量增加。组织蛋白去乙酰化酶抑制还被认为能促进CRC细胞凋亡,减少细胞增殖。尽管可以认识到SCFA对宿主细胞进程的影响随着你的和宿主基因型的不同而变化,但是高水平的丁酸盐还可能影响/限制对微生物群的免疫反应,反而低丁酸盐浓度可能触发促炎状态,导致肠道微生物群重组,通过抑制潜在的致病生物体,提高能产生有益丁酸盐菌群的水平。但是一些关于丁酸盐对CRC发展的影响的研究仍存在争议,这些研究证明在不同的动物模型中存在促癌作用。
一般认为高量摄入红色肉类蛋白、动物脂肪和精制糖的饮食模式具有促癌和促炎作用。高蛋白摄入导致支链脂肪酸和苯乙酸水平升高,苯乙酸是肠道微生物群的成分,包括一些拟杆菌属和厚壁菌门。新陈代谢产生一定数量的生物活性产物,包括酚类化合物吲哚和对甲苯酚。富含饱和脂肪的食物也会促进胆汁酸水平的升高,已知肠道细菌有促进胆汁酸生成的机制。高脂肪摄入饮食模式的研究发现与健康对照组相比,CRC患者的去氧胆酸和次级胆汁酸石胆酸的水平相对较高。这些刺激胆汁酸通过7α去羟基作用生成,已经确定这一作用至少部分受细菌促使。已知胆汁酸有能力通过损伤细菌细胞膜发挥强大的抗菌活性。石胆酸和脱氧胆酸都能促炎,诱发活性氧和活性氮形态生成并诱导肠道上皮细胞内的NF-kB活化。活性氧和活性氮都能导致DNA损伤,包括点突变、DNA断裂和蛋白DNA交联,蛋白DNA交联能潜在导致染色体不稳定,增加CRC风险。
微生物生物膜和CRC
目前癌症的微生物研究仍然处于初级阶段,但毫无疑问随着组学技术的发展,如下一代测序和代谢组学的发展,将有助于加快我们的理解过程。其他复杂的微生物群落包括海洋和土壤环境,但是为使用较旧的概念/技术进一步阐明肠道微生物群对结肠疾病包括CRC的作用提供了绝佳机会。一个需要考虑的概念是生物膜形成和对肠道环境的影响。微生物生物膜十分普遍且成分中通常包括多种微生物。生物膜通常显示出协调的群内和群间多细胞状态,依靠局部细胞密度具有多种功能。现在我们对肠道内微生物生物膜的功能和结构仍然知之甚少。人体大肠的菌落主要为厌氧菌,形成了成百上千的不同菌群,这些菌群都在争夺有限的营养盐供应。生物膜形成的驱动机制以及特定菌群对生物膜形成敏感,而其他菌群却产生抵抗的原因尚不明了。由于和健康组织以及肿瘤相关的菌群存在大量重叠,因此生物膜功能以及菌群结构部分受宿主和宿主环境影响的理论似乎可信。目前已经提出了细菌生物膜和CRC之间的联系。90%的右侧结肠肿瘤患者检出了侵袭性多种微生物生物膜,但是仅有10%的左侧结肠肿瘤患者检出了这样的生物膜。生物膜阳性正常粘膜存在于具有生物膜阳性肿瘤的个体体内,这表明全球检测由于本地特定肿瘤检测。这表示特定的个体更有可能发展出生物膜。传统主要的CRC牵涉机制包括ETBF和核梭杆菌,这二者都出现在肠道生物膜内,这强调了生物膜‘易感’个体内的致病微生物存在貌似可信的补充系统。生物膜阳性组织显示出较强的上皮渗透性和较高的炎症活性,这表明生物膜的形成增加了发展为CRC的风险。细菌生物膜复杂的结构和功能导致了一种类推,即生物膜功能在似乎来自于更高级的生物体组织。尽管这仅仅是初步论断,但是生物膜能修改宿主细胞生物学的证据已经存在,这进一步表明微生物实际上真的具有未知的能力影响人体健康。
微生物驱动的癌症免疫疗法
本文中呈现的只要证据强调了微生物在癌症中的负面影响。但是,在微生物与癌症发病机理的联系出现之前,历史上有微生物对抗癌症的证据。从19世纪开始,微生物就开始用于免疫疗法。科利毒素(一种热灭活链球菌和粘质沙雷氏菌的组合)曾被用于治疗近1000名肉瘤患者,其中80%的患者延长了至多5年的存活期。科利假设在患者体内科利诱导了刷持久的免疫反应。无论其准确与否,操控患者的免疫系统杀死肿瘤细胞仍是癌症免疫疗法的关键焦点。
癌症靶向治疗
在过去的30年中出现了一些细菌免疫疗法。合成生物学家设计了能区分患病和健康细胞的细菌来鉴别、侵入和摧毁癌症细胞。鉴于有明确的证据表明使用微生物与特定肿瘤细胞结合能区分与健康和肿瘤组织环境相关的微生物群落,这种可能有效的理论增加了我们的治疗方法。利用这种理论的研究包括通过设计源于紧密结合到哺乳动物β1整合素的假结核耶尔森菌侵袭粘着蛋白表达,使大肠杆菌内具有细胞侵袭性。在厌氧可控诱导启动子前提下,进行侵袭素表达,确保细菌仅仅侵袭缺氧环境中的细胞。第二个研究设计了瞄准特定肿瘤发生途径的癌症侵袭细菌。这种经过设计的细菌生成关于β连环蛋白的短发卡RNA片段干扰癌症发展。细菌的静脉内管理显示进行本地管理时可以瞄准远端癌症位点。这对在体内瞄准CRC具有巨大的潜在影响。将这些概念验证研究推动到临床前模型中得到了广泛的关注。这一挑战仍能保证受益大于并发症。需要该模型确保最大的效果在癌症环境中发挥杠杆作用,同时最小化毒性和其他副作用。
微生物组处理
在过去的十年中,微生物组设计已经作为有前景的研究领域出现。总体上,肠道微生物组耐受良好;因此,利用同质性产生特殊治疗分子也具有前景。已经可以设计共生菌株生成糖尿病促胰岛素蛋白、HIV融合抑制剂和细胞白介素-2。尽管目前的研究工作还未侧重于CRC,但是通过控制对特定致病条件产生反应的细胞传感器研究治疗分子具有巨大的潜力(图1)。
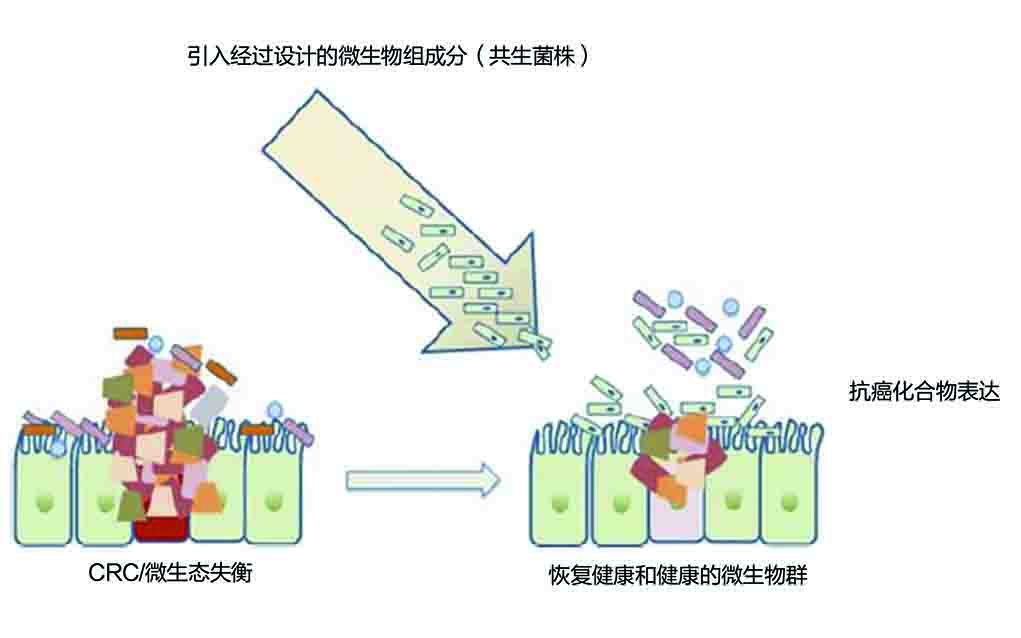
图1. 癌症免疫疗法的微生物组处理
前景和未来导向
理解肠道微生物群以及肠道微生物群促进健康和诱发疾病的机制是一个连续不断的研发领域。在CRC方面,更加完整地理解肠道微生物群对CRC发病机理的促进机制将为我们开启CRC防治的新道路。但是一些重要的问题仍然没有解决。阐明CRC发展是否源于肠道微生物群致癌活动或变异的微生物群是否源于肿瘤环境仍然是一个主要的障碍。现实很有可能是这两种现象的组合。需要在肠道炎症和CRC的情况下进一步了解生物膜群落及其影响微生物代谢的机制。进一步描述微生物及其代谢物影响CRC进展的机制将在未来影响CRC治疗方法,以及预防这一全球癌症杀手方面发挥重要作用。
(参考文献略)
(摘自《Digestive Diseases》,
版权归其所有,仅供内部参考)
编译:张凯
审校:王小茜




