循环肿瘤细胞浓缩后的简要工作流程: 从系统计数到突变分析
摘 要
目的:此次研究的目的是建立一种具有高度适应性的微流体工作流程促进系统性CTC计算和遗传研究。
方法:为了促进CTC的计算,我们利用一种开源图像分析仪将CK/EPCAM免疫染色策略和自动化CTC分析流水线相结合。利用这种工作流程的优势,用一种高通量螺旋微流体芯片系统进行了一项包括56名癌症患者和21个健康个体的试点研究。为了促进CTC体细胞突变的研究,我们将CTC计算和下一代测序结合,建立了一个包括多元化随机序列和经过测序的CTC样品的简单扩增子文库。
结果:经过ROC分析,CTC染色和计算工作流程达到了80.4%的灵敏度和85.7%的特异性(AUC=0.87,P=0.004,power=0.985)。单变量和多变量分析证明CTC(CK/EPCAM+CD45-),但没有验证其他细胞群,验证工作证明其是癌症患者的重要而独立的生物标志物(P<0.01)。癌症患者的连续CTC监测揭示治疗后CTC数量减少,药效学研究表明其具有临床实用性。CTC样品的深度测序揭示TP53和ESR1存在体细胞突变。
结论:该报告的意义是证明一个系统性的和具有适应性的工作流程能消除微流体浓缩和CTC分析之间的鸿沟,使得CTC在临床和理论研究中得到更加广泛的应用。
前 言
循环肿瘤细胞(CTC)已经成为癌症无创监测的焦点。在精准医疗的时代,它能够指导患者选择有效的疗法。毫无疑问,生物工程的迅速发展业已推动CTC领域的进步。引人注目的微流体技术的数量正在迅速增长,极大地促进了从上百亿红细胞中富集极端罕见的CTC群体。更重要的是,后续工作流程中对系统性和无偏性CTC的分析对CTC的计算和遗传研究都有极大的帮助。对于CTC的计算和鉴别,CTC上抗原的免疫检测是目前最常见的方法。由于在上皮癌细胞中的表达及其临床相关性,细胞角蛋白和EPCAM是广泛用于该领域的替代CTC标志物。但是,通常在107WBC中仅有一个CTC,在白细胞背景下对罕见CTC进行鉴别和计数仍然是一项挑战。因此,急需一种详细的且经过确认的方案及进行后续CTC的鉴别、计数和可视化。在一些报告中,通常都是通过人工进行CTC鉴别和计数。人工细胞计数复杂难做、费时费力且存在偏倚,显著减弱了CTC分析在临床使用中的效果。因此,系统的和可靠的CTC计数工作流程应该能获得高质量的结果并促进CTC的应用。此次研究的目的是证明并确认一种具有高度可接受性的系统CTC计数方法。
除CTC计数之外,CTC的遗传分析能够帮助医生和患者了解具有潜在转移可能性的癌细胞群体。在精准医疗时代,大部分现有治疗方法依靠原发肿瘤的遗传信息,而不是转移的癌细胞亚群体信息。CTC被认为是转移的罪魁祸首,是大约90%癌症死亡病例出现的原因。CTC下一代测序分析可能是快速了解这些转移的罪魁祸首突变情况的有效工具。该方法是一种治疗基因标靶和药物耐药性突变的无创实时监测工具。随着多种可靠的CTC分离技术的出现,我们需要直接的靶向CTC测序流程来扩展CTC技术的应用。
为了填补上述微流体浓缩和CTC应用之间的技术鸿沟,此次研究的目的是:
(1)证明一种系统性CTC计数方法;
(2)建立CTC样品的直接测序流程;
(3)使用临床样品确认计数和测序工作流程。
材料和方法
患者和血液采集
2015年7月至2016年10月之间,所有此研究的参与者均为诊断为患有结直肠、肺部、乳腺、胃部、肝脏和前列腺六种原发癌症之一的患者。在患者签署知情同意书之后进行血样采集。该研究由香港大学机构审查委员会批准。共招募56名癌症患者(22名肝癌患者、11名结直肠癌患者、7名肺癌患者、6名胃癌患者、5名乳腺癌患者和5名前列腺癌患者)和21名健康对照献血者。在BCT无细胞DNA试管中采集8毫升外周血。出于进行连续癌症监测的目的,血样的采集时间为治疗前一周和治疗后三个月(治疗方法为放疗或靶向索拉非尼疗法)。所有样品均在采集后72小时内进行处理。
样品制备和CTC分离
每次操作使用8毫升的血液样品进行微流体浓缩。在使用无标记微流体设备进行分离之前,血样进行RBC溶解处理。基于细胞大小从血液中分离CTC。WBC背景中分离的CTC在带正电荷的显微镜载玻片上进行固化,随后采用免疫荧光染色进行鉴别。在添加了2nM谷氨酸盐和10%胎牛血清的RPMI1640培养基中培养KYSE30和KYSE270食道癌细胞。
免疫荧光染色
使用DAPI、Alexa Fluor 555-conjugated pan-CK、Alexa Fluor 555-conjugated EpCAM 和 APC-conjugated CD45鉴别CTC。将与荧光染料结合的抗体添加到细胞涂片上并在室温下培养2小时。染色后,将玻片置于添加了DAPI的抗褪色固定试剂中固定并使用Cytation 5细胞成像多模式阅读器进行扫描。在相同的条件下捕获所有图像。我们使用DAPI染色来标记DNA从而鉴别有核细胞,使用CK/EPACM染色来标记CTC,使用CD45染色来标记WBC。
通过细胞分析仪(CellProfiler)和细胞分析仪分析(CellProfiler Analyst)进行的CTC计算
通过细胞分析仪和细胞分析仪分析来进行CTC的计算和可视化过程如下:1)主要目标鉴别:在细胞分析仪上采用全局阈值策略在DNA染色图片上首先鉴别有核细胞(主要目标)。该策略在具有一般背景的荧光图片上检测细胞目标的性能稳定;2)主要的过滤目标:测量胞核大小。过滤直径为9μm到36μm的胞核进行进一步分析;3)形状测量:“离心率”,离心率是焦点距离和长轴长度的比率,需要测量该比率。离心率的值为0到1(0值表示圆形目标)。离心率表示核的圆度,用于排除有小叶和外形不规则的WBC(例如中性粒细胞、嗜碱粒细胞和单核细胞);4)次要目标鉴别:基于主要目标定量特性测量的坐标系确定次要目标;5)平均密度测量:“平均密度”是目标内的平均像素密度,基于CK/EPCAM和CD45图片建立的次要目标进行测量;6)数据报告:测量结果报告的格式为SQL轻量数据库文件,细胞分析仪分析可以打开此类文件;7)细胞计数:在显示平均密度和目标外形的交互式散点图上利用内置过滤器和图片浏览工具完成细胞分析仪分析、细胞控制、计数和可视化。为CTC计数和可视化设定CK/EPCAM和CD45阳性和阴性控制值。使用2个染色对照组进行参考:使用同种型抗体对CK、EPCAM和CD45抗体染色的癌症细胞系(KYSE-30)和同一患者的血沉棕黄层碎片进行染色。
文库制备和下一代测序
利用QIAamp DNA微型试剂盒将来自微流体芯片的输出进行DNA提取。同时,计数500个WBC并进行DNA提取。在使用Qubit dsDNA HS试验试剂盒进行DNA定量后,使用Q5高保真DNA聚合酶执行一个2步的PCR文库制备。
第一次PCR用于扩增目标外显子区域,引入1个10bp的多元化序列和1份测序适配蛋白(即P5和P7适配蛋白)到扩增子文库中。该多元化序列以10个随机核苷酸中的1个序列,设计用于增加文库序列的多样性,进而通过Illumina测序仪进行准确的簇鉴别。第二次PCR用于扩展Illumina适配蛋白序列的剩余部分。使用磁性SeqCap EZ 纯化微粒纯化文库,接着使用NEBNext文库定量试剂盒进行定量。文库被多重化为等摩尔量并使用MiSeq 测序试剂盒V2根据Illumina指南进行测序。总体上,载入12pM的多重文库和5%phiX跟踪标定。
生物信息学
为了进行生物信息学分析,使用Trimmomatic处理MiSeq的测序数据配平P5和P7适配蛋白和多元化序列。使用Burrows—Wheeler调整器将测序读数调整到人类参考基因组(hg19)。随后,使用GATK插入缺失标记重排工具计算调整的文件。使用VarScan2执行体细胞变异调用,随后使用ANNOVAR进行变异注释。为了提高变异鉴别的准确度,输入CTC技术评估的肿瘤纯度(即CTC纯度)作为VarScan2的参数来调整变异频率预支,优化费舍尔精密检验。该检验将肿瘤样品(即CTC输出样品)中参考和变异支持的读数数量与正常样品(即WBC样品)进行比较。
为了对临床样品进行测序,每名患者采集2试管血液(每管8mL)同时分配给测序流程和计数流程。计数流程评估的肿瘤纯度输入VarScan2进行体细胞突变分析。2次计数一致的p值低于0.05的体细胞突变称作一致的体细胞突变。
统计分析和数据可视化
使用SPSS19进行曲线下面积、灵敏度和特异性的受试者操作特征曲线(ROC)分析。在R环境(3.3.1版)中执行相关系数评估、单变量和多变量分析、双尾t检验和95%置信区间评估。使用R内的pwr数据包完成统计功效计算。通过2个平均值除以合并标准差的差异评估双尾t检验的效应量。基于让步比评估单变量和多变量分析的效应量。对于Kaplan—Meier(KM)存活分析,从cBioPortal上下载TCCGA和METABRIC数据,接着在R内进行分析和可视化。癌症患者NRAS、ESR1、EGFR、KRAS、BRAF或TP53的隐匿突变被分为“有突变”组,上述基因无突变的患者被分为“无突变”组。在R内使用ggplot2、plot3D、plotROC、OmicCircos、survminer和venneuler软件完成此次研究的数据可视化工作。
结果和讨论
CTC工作流程的概况
螺旋微流体芯片系统是一种具有较高满意度的CTC浓缩方法,该方法利用螺旋微通道内的内在离心力去除WBC并根据细胞大小富集CTC。螺旋微流体芯片通过一种分流CTC富集出口富集CTC。这样可以连续富集CTC,具有较高的工作能力。与免疫介导CTC捕获系统相比,微流体平台的CTC回收率较高,去除WBC的灵敏度也较高。因此,我们采用该技术富集CTC并开发了后续计算和测序流程(图1)。
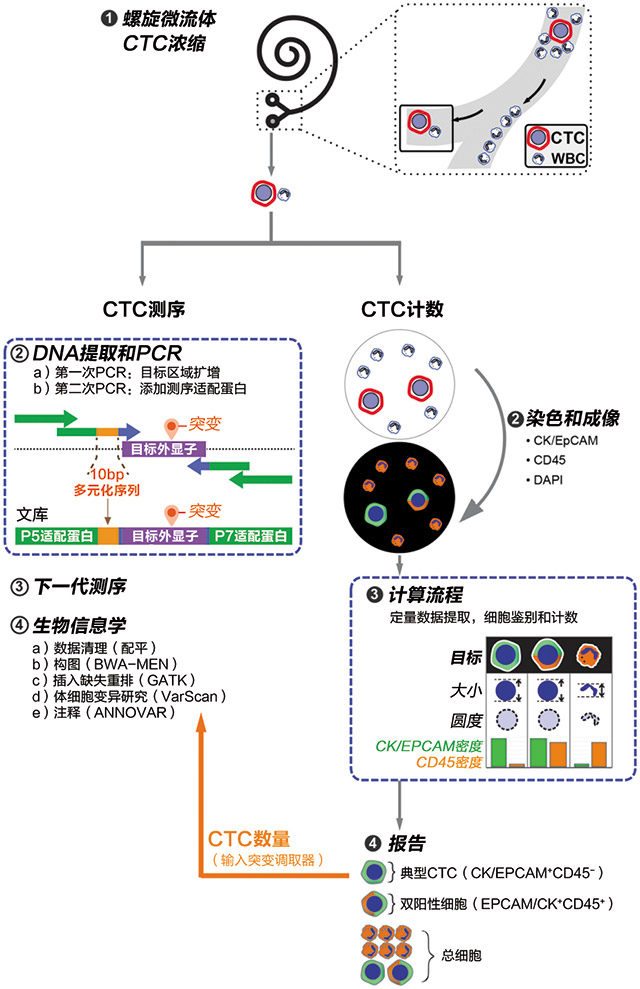
在随后的章节中,我们将展示CTC计数和测序分析的平行工作流程(图1)。对于CTC计数流程,我们将展示CK/EPCAM结合的染色策略和定量图像分析,随后将在包括77个个体的试点研究中对该流程进行流程确认。最后我们将展示NGS文库设计、文库制备方法以及模拟CTC样品的分析确认以及临床CTC样品的测序。
图1. CTC浓缩、计数和测序流程概述
CTC的CK/EPCAM结合染色策略
为了确保CTC免疫染色的质量,应该避免多种荧光光谱的重叠来最小化荧光干扰。因此,明智地采用有限数量的标记通道十分重要。传统CTC的鉴别需要对4种标志物进行荧光染色,包括CK、EPCAM、CD45和DAPI。将具有相同功能的不同标志物结合到1个通道内是建立简单CTC计数流程加速CTC计数过程的最佳选择。其余的染色通道通过对额外标志物进行染色加深对CTC生物学的认识。例如,癌症干细胞标志物(例如CD44)、EMT标志物(例如波形蛋白)、增殖标志物(例如Ki—67)是执行进一步研究的良好标志物。出于上述原因,我们通过将CK和EPCAM染色结合到1个Alexa Fluor 555荧光通道内完成CTC免疫染色(图2A)。这样做的目的是建立简单的染色工作流程加速染色过程和计数过程,并让剩余的荧光通道用于其他染色研究。
CTC计算和可视化
为了建立系统的CTC计算和可视化流程,我们利用细胞分析仪和细胞分析仪分析,免费通用图像分析程序。利用荧光图像建立自动化的分析流程测量定量特征(即平均染色密度、细胞大小和细胞外形;见流程的建立方法)。如图2B所述,CTC微流体输出中的主要的细胞群体表达CK/EPCAM蛋白。尽管这些细胞的CK/EPCAM表达具有多样性,但是表达的水平可以和阴性对照组进行明显的区分,因此能够在控制后进行准确的CTC计数。随后利用细胞分析仪分析进行CTC控制、计算和可视化(图2B和2C)。利用该流程,我们从微流体输出的免疫染色中规定了不同的细胞群体用于进一步确认,即典型CTC(EPCAM/CK+CD45-),双阳性细胞(EPCAM/CK+CD45+)和总细胞。
CTC染色和计算流程的临床确认
为了确认我们的CTC染色和计算流程,我们利用螺旋微流体芯片系统进行了一项包括77个临床样品(56个癌症患者和21个健康个体)的试点研究。癌症患者的典型CTC数量显著高于健康个体(p=0.004,power=0.985,双尾t检验;图3A)。图3B描述的是诊断为患有6种常见癌症类型个体患者的CTC计数。除典型CTC外,癌症患者的血样中观测到了非典型双阳性细胞,但是该方法的临床显著性不确定。我们计算了双阳性细胞并将数量与典型CTC细胞比较。ROC曲线分析确认了典型CTC计数是区分癌症患者和正常个体的显著生物标志物(AUC=0.87,95%CI=0.79-0.95,p=0.000,图3C)。相反,双阳性细胞核总细胞的数量没有区分价值(AUC<0.5,P>0.05)。CTC试验的灵敏度达到80.4%,特异性达到85.7%(临界值=3.5)。此外,如图3D所示,单变量和多变量分析鉴别的CTC数量和癌症的出现具有明确的相关性(单变量分析:让步比=1.41,p=0.004,power=1;多变量分析:让步比=1.51,p=0.007,power=1)。与之相反,双阳性细胞或总细胞与癌症的出现之间没有关系(p>0.05;图3D)。对单一癌症类型也执行了多变量分析,CTC数量的让步比连续大于1。总之,CTC染色与计算流程相结合的方法得到了确认,在临床环境中对CTC的系统分析具有很高的适用性。
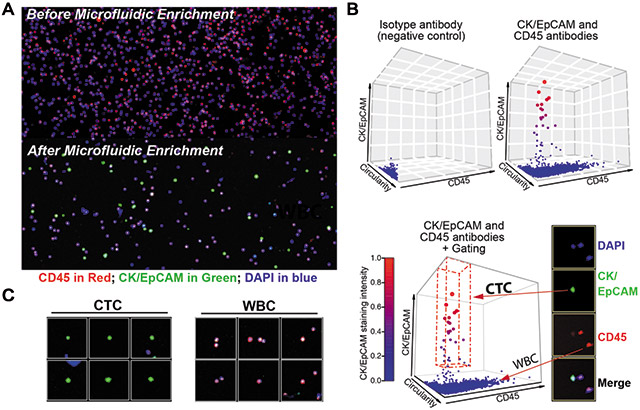
图2. CTC染色、控制和可视化。
(A)微流体浓缩前后的血样免疫荧光染色。样品使用DAPI(蓝色)、CK/EPCAM(绿色)和CD45
(红色)抗体染色。
(B)3D点显示细胞分析仪提取的数据。血沉棕黄层中的细胞使用同种型对照组抗体染色并作为
阴性对照。红点线框标示CTC控制。
(C)计算流程生成的个体细胞图集。显示了CTC(DAPI+CK/EPCAM+CD45-)和WBC(DAPI+CK/
EPCAM-CD45+)。
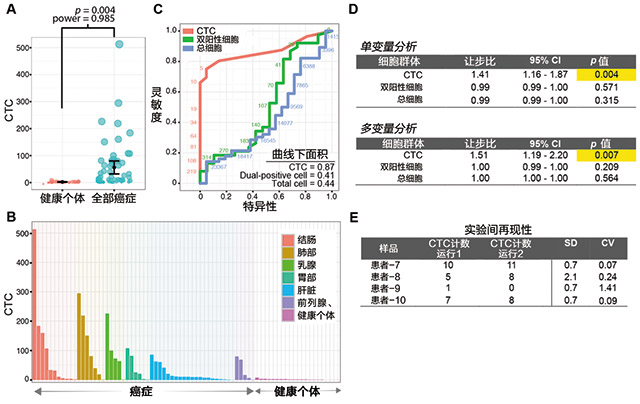
图3. CTC计算流程的确认分析。
(A)癌症患者(n=56)的CTC计数显著高于健康个体(n=21),p=0.004。
(B)长条图显示的是每个癌症患者和健康献血者的CTC计数。
(C)受试者操作特征(ROC)点表示癌症患者和健康个体不同样品CTC计数的效果。
(D)单变量和多变量分析实验验证了CTC、双阳性细胞和总细胞与临床癌症出现的相关性(即
癌症出现率)。
(E)CTC试验的实验间再现性。SD标准差;CV,变异系数。
通过CTC计数进行的癌症监测
为了检验我们的CTC试验的再现性,从4名患者中采集2试管血液(每管8mL)。在两台微流体机器上单独运行血样。如图3E所述,CTC计算结果的再现性很高且变异性低。而且我们寻求在连续癌症监测中将我们的CTC计算流程进行临床应用。采集肝细胞癌患者的治疗前和治疗后血样(n=4)进行CTC计数。我们观测到治疗后CTC的数量显著下降 [即CRC数量减少:患者1:82.5%;患者2:66.7%;患者3:47.7%;患者4:100%]。治疗后CTC数量的下降趋势表明药效学研究的潜在临床实用性。在HCC中,表达EPCAM的CTC报告为肿瘤抑制细胞,与肿瘤切除后患者的不良预后显著有关。尽管该证据充分支持了表达EPCAM的CTC的重要性,但是需要样品量更大的试点研究证明CTC在癌症监测中的作用。总之,我们的CTC分析方法能实现可靠而系统的CTC评估,可以扩大CTC分析在后续工作流程中的应用。
CTC计算对CTC测序的重要性
除了药效学研究,上述CTC计算流程还在NGS研究的体细胞突变精确检测中发挥着关键作用。由于微流体输出包含造血细胞背景,CTC中的体细胞突变的变异频率(VF)很低。为了促进体细胞突变的准确评估,微流体输出中的CTC纯度用于调整频率阈值,减少统计分析期间的假阴性错误。为了达到这一目的,CTC计算流程中获得的CTC数量用于评估CTC纯度(即CTC数量/总细胞数量)。CTC纯度输入到VarScan2中调整费舍尔精密检验中体细胞突变的调取(详见方法)。
NGS文库设计和制备
NGS文库的制备很直接;文库制备包括2个简单的PCR步骤(图4A)。生成的扩增子文库包括:1)结合到流动细胞的P5和P7蛋白;2)1个10bp多元化序列;3)目标外显子区域。10bp多元化序列由随机的核苷酸组成,是强化文库多样性达到测序期间精密DNA簇鉴别所必须的。在Illumina测序系统的流动细胞中,每个DNA簇是一个单一的测序读数,需要进行精密的检测得到高质量测序输出。在Illumina MiSeq、NextSeq和HiSeq平台中,成功地进行DNA鉴别和构图很大程度上取决于前5步测序中文库序列的多样性。但是,扩增子文库的序列多样性通常很低,导致模糊的簇检测。因此导致读数质量低和序列过滤的数据丢失。为了提高准确簇鉴别的序列多样性,我们在文库中引入由随机核苷酸构成的序列。
在我们的测序流程中,我们寻求对具有隐匿病原突变和/或临床可用突变的临床相关癌症基因进行测序。我们文库设计中的目标基因包括NRAS、ESR1、EGFR、KRAS、BRAF和TP53。我们利用TGGA和METABRIC数据库探索这类基因的临床相关性。KM存活分析显示突变的存在与乳腺癌患者(n=1865)、肝癌患者(n=319)、前列腺癌患者(n=491)、甲状腺癌患者(n=388)、肾癌患者(n=64)和胰腺癌患者(n=115)的存活之间存在显著的相关性(时序p值<0.05)(图4B)。基因存在突变的癌症患者与较差的总无病生存率有关。我们设想CTC中该基因的测序可能能够提供重要的理论和临床信息。
为了证明该基因中此测序流程的作用,我们设计了目标外显子NRAS、ESR1、EGFR、KRAS、BRAF和TP53基因的侧面参数,如体细胞突变目录(COSMIC)和ClinVar数据库的记述,这些基因隐匿了临床相关体细胞突变。图5A中的框1-3描述的是COSMIC和ClinVar数据库中测序基因目标和相关突变(突变病原学和药物反应)。
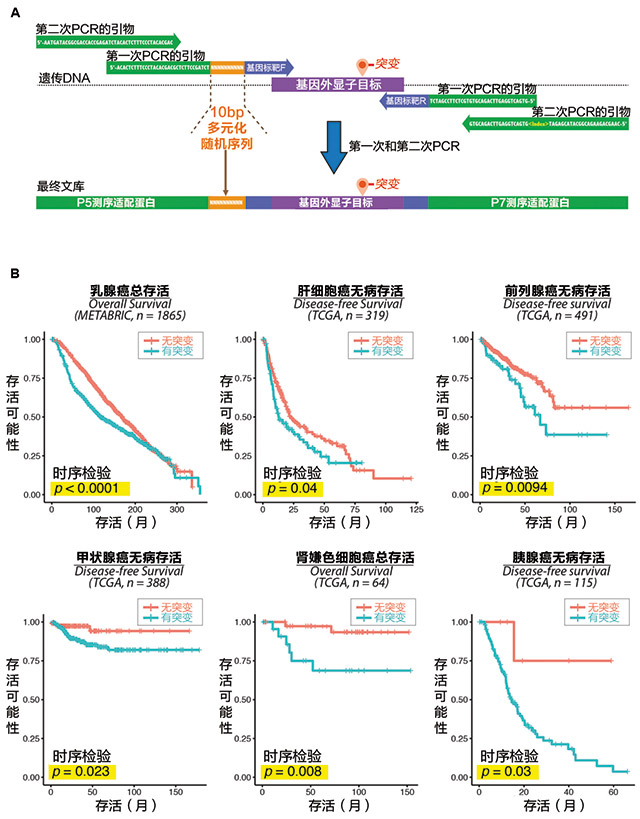
图4. 2步文库制备的原理图。
(A)采用2步PCR制备文库。第一步PCR的引物对包括1)靶向杂交序列
(蓝色),结合并扩增外显子目标;2)一个由随机核苷酸组成的10bp
多元化序列(橙色),测序运行期间提高DNA簇检测的准确度;3)1份
P5和P7适配蛋白序列(绿色)。第二次PCR的通用引物对增加全P5和P7
测序适配蛋白(绿色)到文库中。
(B)根据肿瘤组织的突变存在性对癌症患者的KM存活曲线进行分层。
根据NRAS、ESR1、EGFR、KRAS、BRAF和TP53基因的突变状态经过分
析,癌症患者分为“无突变”和“有突变”。每个点显示比较存活曲线
的时序检验p值。
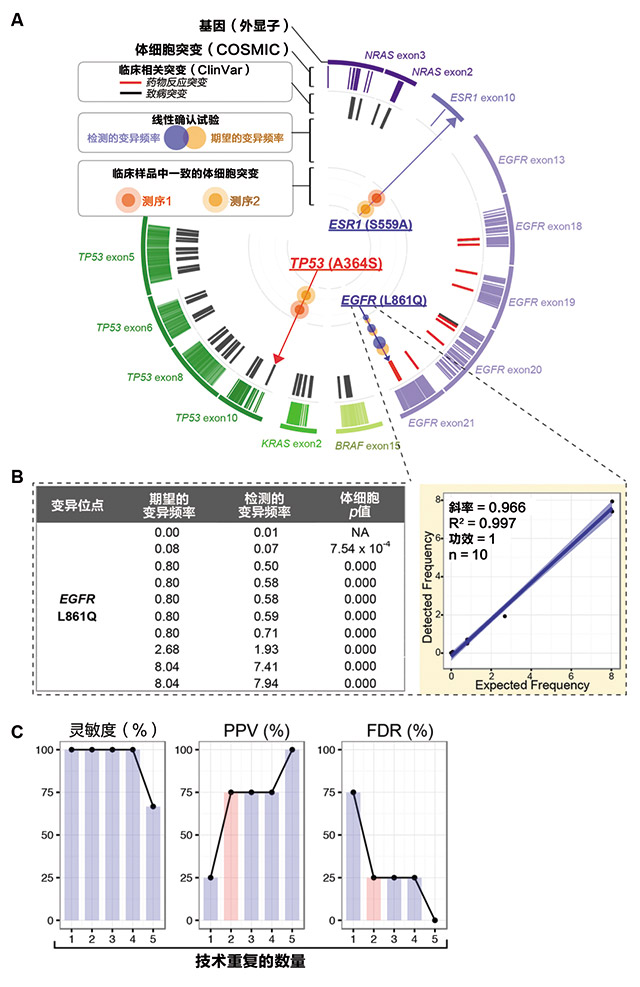
图5. 测序目标区域和测序结果。
(A)从外到内:1、测序的目标基因和外显子;2、体细胞突变;长
条表明COSMIC数据库报告的体细胞突变,3、临床相关突变:长条表示
ClinVar数据库报告的临床相关体细胞突变;4、确认试验:交错的点表
示3次模拟CTC样品典型的测序结果。点的大小与频率成比例。频率相
关性见B。5、临床样品中检测的一致的体细胞突变:在技术重复中执行
的CTC微流体输出的文库制备和测序。用点表示一致的突变。
(B)线性确认稀释测序试验的结果。显示期望频率和检测的频率之间
的关系。
(C)长条图显示增加技术重复数量后灵敏度、阳性预测值(PPV)和错
误发现率(FDR)的变化。粉色条表示明显的PPV和FDR的改进。
测序性能的确认
下面通过对一系列的模拟微流体输出进行测序,来对我们的靶向测序流程进行标准检查。为了模拟造血细胞背景,我们采用被广泛应用的参考DNA对照组(NA12878),该对照组是NIST基因组确认的检查金标准。为了模拟CTC,我们使用食道癌细胞系的DNA(KYSE270),该细胞系包括COSMIC数据库记录的与临床相关的EGFRL861Q的突变。
为了评估测序工作流程检测的变异频率是否与期望的VF呈线性相关,我们通过在背景NA12878DNA(模拟造血细胞背景)中添加不同比例的KYSE270 DNA(模拟CTC)模拟CTC微流体输出执行线性确认。EGFR L861Q突变的CTC样品期望的VF为0%、0.08%、0.8%、2.68%和8%。我们的测序工作流程显示期望的VF和检测的VF之间显著较高的线性和相关性(n=10,R2=0.997,斜率=0.966,power=1;图5B)。此外,即使期望的VF下降到0.08%,还是可以检测到EGFR L861突变(体细胞p值=7.54×10-4),表明该测序工作流程具有很高的检测灵敏度。不仅如此,通过分析模拟的CTC样品(n=5,0.8% VF),我们观测到通过提高技术复制的数量能提高突变鉴别的性能(图5C)。尤其是,在技术副本中,调用测序结果中的一致突变时,阳性预测值(PPV)显著升高(从25%提高到75%),相应的错误发现率(FDR)显著下降(图5C),但是,灵敏度仍然很高(100%)。此外,通过试验多种生物信息学工具能改进测序性能,但这超出了目前研究的范围。
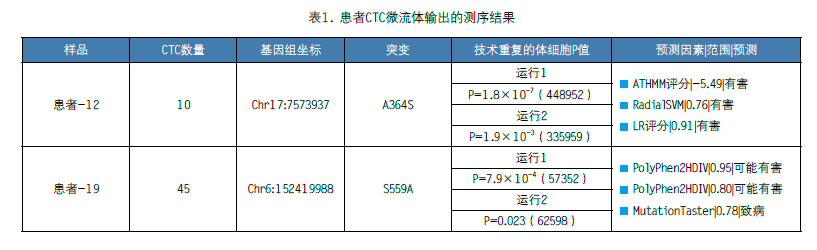
对临床CTC样品进行测序
利用测序流程的优势,我们对2个诊断为肝细胞癌的患者的微流体输出进行了测序。患者的WBC用作生物信息学分析的参考对照。在技术副本中执行CTC微流体输出的文库制备和测序。具有显著体细胞p值的技术重复的一致突变被调用于进一步分析。我们在CTC样品(图5A和表1)中发现了TP53体细胞错义突变(A364S)和ESR1错义突变(S559A)。肿瘤预测表明TP53 A364S突变(表1)存在损伤效应。值得注意的是COSMIC数据库(突变ID:COSM46361)中卵巢癌中报告了A364位点的错义突变。另一方面,ESR1与HCC的敏感性有关,可能是HCC的肿瘤抑制基因。TCGA 临时HCC数据库的ESR1的突变频率为1.6%(6/366个经过测序的案例)。总之,我们证明直接的、基于扩增子的靶向测序有助于对CTC的遗传研究。
结 论
总之,该报告证明我们的CTC染色、计算和靶向测序工作流程具有较高的适用性。CTC计算和遗传分析的系统流程能填补CTC微流体浓缩和CTC系统研究的技术鸿沟。结合微流体优势的工作流程能为理解CTC的生物学和扩展其在癌症监测和个体医疗中的临床应用铺平道路。
摘自《PLOS ONE》,版权归其所有,仅供内部参考
编译:张凯
审校:方研




