实施游离DNA检测的策略
直到最近,产前筛查唐氏综合征和其他常见的非整倍体都是基于孕妇血清标志物的测定和超声检查。基于标记组合的检测被用来评估个人受影响怀孕的风险。那些具有足够高风险(指定为阳性)的患者可以通过早孕期的绒毛膜绒毛取样(chorionic villus sampling,CVS)或中孕期的羊膜穿刺术进行侵入性产前诊断,两者都与胎儿丢失有关。根据当地或国家的指导方针,选择的截止风险有所不同。在大多数医疗系统中,不向结果为阴性的人提供侵入性检测,尽管在一些地方这样的检测是个人选择的问题。
这种已经发展了几十年的方法即将发生根本性的变化,因为一种单一的标记物,即母体血浆游离(cf)DNA,比任何传统的筛选试验都有更好的性能。然而,新的标志物具有几个局限性;传统检测具有非整倍性以外的作用。这些并发症意味着在临床实践中实施cfDNA检测时,没有单一的策略可以推荐。在不久的将来,世界各地可能会采用不同的策略。
一、筛查试验
非整倍性产前筛查的结果由两种因素进行评估:检出率(detection rate,DR),筛查结果为阳性的受影响的妊娠比例;假阳性率(false-positive rate,FPR),筛查结果为阳性的整倍体妊娠比例。另外,可以进一步评估的两个因素,它们是试验性能和被检测人群的函数:阳性预测值(positive predictive value,PPV),阳性中的非整倍风险,以及阴性预测值(negative predictive value,NPV),阴性中的风险。这些因素可以在筛查时或出生时计算;由于常见的非整倍体具有较高的宫内病死率,出生时的死亡率要低得多。
二、传统的唐氏综合征筛查
最广泛使用的标记物是孕妇血清妊娠相关血浆蛋白(PAPP)-A、人绒毛膜促性腺激素(hCG)、hCG游离β亚基、甲胎蛋白(AFP)、游离雌三醇(uE3)和抑制素-A、超声颈部透明度(NT)。每个标志物的鉴别能力因胎龄不同而不同,这限制了标志物的可能并发组合。此外,现在对于早孕期的超声标志物有更多的经验:胎儿鼻骨缺失(NB)、静脉导管血流异常、三尖瓣反流以及额上颌面角。还有一种孕早期母体血清标志物,胎盘生长因子(placental growth factor,PlGF),被认为是孕中期超声面部轮廓标记物、鼻骨皮质(NF)、鼻骨长度(NBL)和鼻前厚度(PT)。
由于前瞻性筛查研究存在生存偏差,这必然会高估非整倍性检出率,因此传统筛查试验的性能最好使用统计建模进行评估。建模显示,结合孕早期标志物的检测比孕中期标志物的检测性能更高,使用早中孕期标志物的检测性能最高。
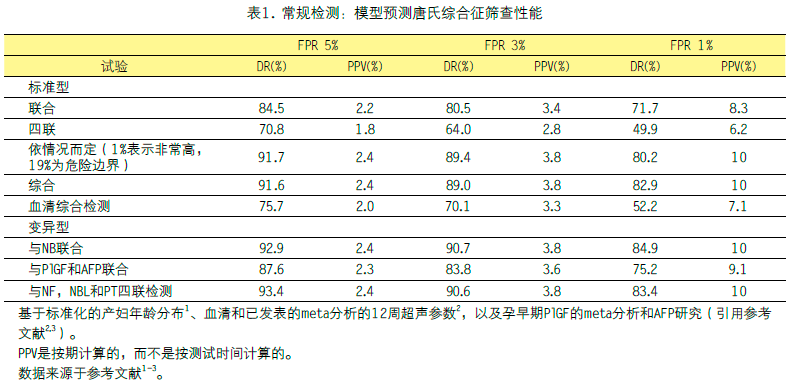
表1显示了5种最佳方案的模型预测性能:NT、PAPP-A和游离β-hCG(联合试验);孕中期游离β-hCG、AFP、uE3、抑制素-A(四联试验);联合试验标志物,但将侵入性检测限制在风险最高的1%和初始边缘危险的19%,孕中期四联标志物随后风险修正(依情况而定测试);NT和PAPP-A在12周时与所有妇女的孕中期四联标记物(综合检测);相同但无NT(血清综合试验)。表1还显示了一些带有额外标志物的变体。包含超声标志物的检测效果最好。
三、游离DNA检测的类型
目前,美国和中国的大型实验室有三种应用广泛的方法,分别是鸟枪(全基因组)大规模平行测序(s-MPS)、靶向MPS(t-MPS)和单核苷酸多态性(SNP)方法。在这些国家之外,这些检测要么通过经销商代表美国和中国实验室,或者在某些情况下通过当地实验室授权。在不久的将来,cfDNA的新方法将会变得更简单、更便宜,并且具有高通量。

表2总结了s-MPS、t-MPS和SNPs之间的技术差异。用这两种计数方法,数以百万计的全基因组胎儿和母体片段被测序,信息序列被映射到离散的染色体特异性位点;对于t-MPS,对感兴趣的染色体有富集作用。在非整倍体中,染色体的分布会因一个染色体的过量或不足而发生偏差染色体。另一种方法不使用计数,而是放大了大约20000个SNPs在淡黄色皮毛(母体)和血浆(母体和胎儿),然后进行测序。在非整倍体中,两种来源的产物模式不同。这三种方法都对胎儿胎盘单位产生的cfDNA比例,即胎儿分数(fetal fraction,FF)敏感。
最近,商业提供者正在考虑采用无需对大量片段进行测序的方法,这是该检验程序中一个昂贵且耗时的步骤。
四、游离DNA检测唐氏综合征的鉴别能力
在有创产前诊断(所谓的高风险研究)之前,已经有13项已发表的研究在血浆样品中进行4-16。此外,有7项已发表的研究,其中样品是作为现有筛查项目的一部分抽取的17-23,还有一项是混合了这两项研究设计的24。对高风险研究的meta分析得出的DR为99.3%,FPR为0.11%。其他研究的性能甚至更好,但由于随访不完整和生存偏倚,这些研究的可靠性较低。
五、游离DNA筛查策略
目前,正在讨论三种策略,cfDNA检测仅针对常规筛查结果为阳性的女性(二级cfDNA检测);对所有妇女,取代传统的筛查(一级cfDNA检测);根据常规筛查结果,有10%到30%的女性被选中(依情况而定cfDNA检测)。最后一种策略有4种变体:选择的组具有最高的常规测试风险;有创性产前诊断的风险最高的人群非常少,其余的人都有cfDNA;使用额外的常规测试标志物;高龄产妇(advanced maternal age,AMA)自动提供cfDNA以及根据常规检测风险选择较年轻的产妇。在所有的策略中,对那些cfDNA结果为阳性的患者都提供了有创的产前诊断。
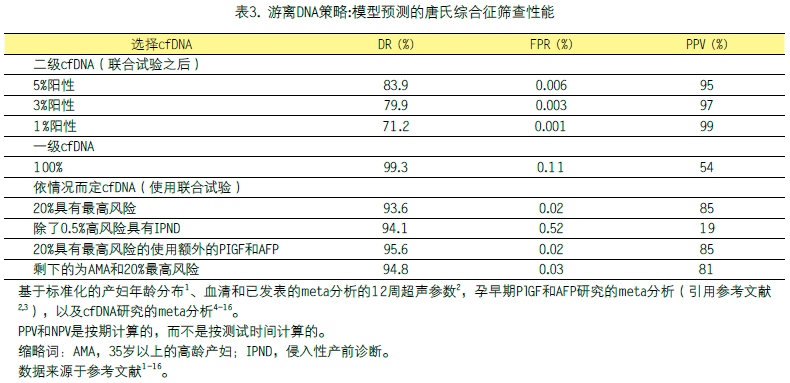
表3显示了每种策略的模型预测性能。二级cfDNA将大大减少常规筛查中羊膜腔或CVS绒毛活检的数量,从而减少医源性胎儿损失;但这也会导致检出的减少。一级cfDNA能最大限度提高检出,同时又能确保相对较少的产妇有侵入性的产前诊断。依情况而定cfDNA大大减少了需要进行昂贵的cfDNA检测的妇女人数(见后面的讨论),同时保持检出率比联合检测高而更接近于一级cfDNA。尽管假阳性率要高得多,但有侵入性检测的风险较高的孕妇检出率略高,这被认为是值得的,因为大约有三分之二的受影响怀孕属于极高危人群,因此可以避免等待cfDNA结果。所有其他策略的PPV都比常规测试高得多,而且所有策略的NPV都非常低。
六、成本效益
在公共卫生非整倍筛查项目中,最有效的成本效益衡量指标是边际成本或增量成本比(incremental cost ratio,ICR)。ICR是新检测避免的每一个额外的受影响出生的成本,超过现有检测所避免的成本。这种费用可以与受影响个人的终生费用进行比较,限于直接医疗、教育和社会服务费用或包括间接社会费用,例如收入损失。在一些成本效益研究,将终生成本包括在ICR中,然后将其与一些一般的负担能力指标进行比较,后者是国家可用资源数量的函数,如人均国内生产总值(gross domestic product,GDP)。例如,根据世界卫生组织(World Health Organization)的指导方针25,如果每项残疾调整后的生命年避免的成本低于人均国内生产总值(GDP),那么一项新的干预措施就具有很高的成本效益。除了边际成本外,公共卫生规划人员还需要考虑改变新战略的总成本,或每位女性筛查的平均成本。
二级cfDNA策略通常是成本中性的,或者能够节约成本,因为cfDNA检测的单位成本与侵入性产前诊断的单位成本相同或更低。需要更详细的成本效益分析来得出关于一级cfDNA和依情况而定cfDNA策略的结论。最简单的输入形式是常规筛选试验、cfDNA、侵入性产前诊断和摄取率的单位成本。这些费用在各国之间会有相当大的差别,而终生费用也会有相当大的差别,但各地的费用可能是相同的相对比例。表4为一个国家的单位成本分配示意图,例如美国,唐氏综合征出生的直接终生成本约为90万美元26。该例子显示,当cfDNA检测的单位成本降低到低于联合检测单位成本的1.5-2.5倍时,转换为一级cfDNA的边际成本只会小于唐氏综合征出生的终生成本。如果保留NT扫描,此范围的高端将适用(请参阅后面的讨论)。依情况而定cfDNA比一级cfDNA便宜得多,而且以cfDNA检测的单位成本4到5倍于联合测试的费用是可以负担得起的。应该理解的是,这些检测的单位成本不仅仅是试剂的价格,还包括实验室管理费用、技术人员时间,以及在公共卫生环境之外的利润。
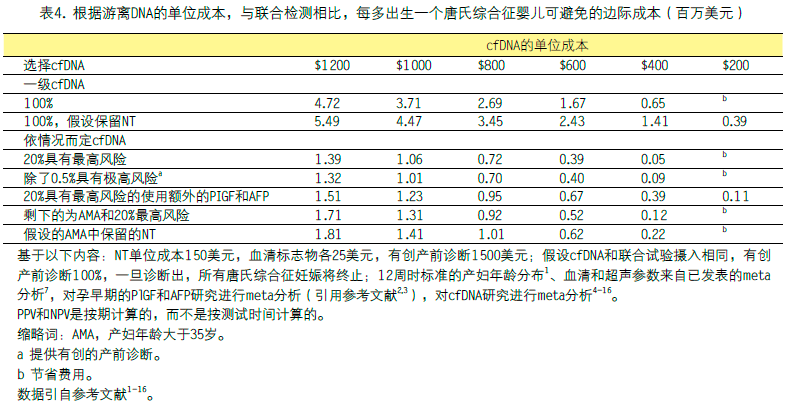
表4简单假设cfDNA和传统策略将有类似的应用。如果接受取决于对最终结果的看法需要有创的产前诊断,前者可能有较高的摄取,边际成本会较低。一项研究的初步结果表明,这可能是事实,但当cfDNA结果为阳性时,有创产前诊断的摄取减少弥补了这一点。
七、检测无反应
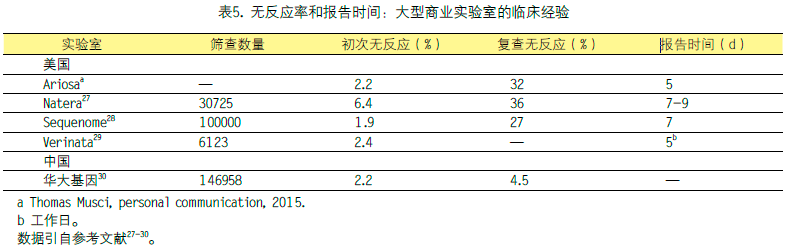
从表5可以看出,美国和中国大型实验室进行的cfDNA检测中,有很大一部分没有得到阳性或阴性的结果。这些数字是根据他们的常规临床经验报告的,而不是发表的高危或一般人群筛查研究,因此产生结果的标准可能更严格。有3种情况下可能会发生无反应:不合格的样品或质量评估失败、低的FF,或者实验室认为过于接近临界值的结果无法解释。最后一种情况只发生在一个实验室中,而且它们不测量FF。
那些测量FF的实验室设定了4%的下限。选择这个限值,是考虑到FF的测量误差,以及唐氏综合征21号染色体每个基因组中基于公式3×FF+12×(1-FF)给出的FF的平均拷贝数之间的必要关联。随着FF的减少,拷贝数接近2,cfDNA的鉴别能力降低。然而,可以认为,如果cfDNA测量的精度很高(例如,使用更深度测序),那么即使在非常低的水平上,鉴别也足够了,因为cfDNA是一种筛查性试验,并不意为着是诊断性的。
FF主要取决于胎盘的大小,在怀孕早期检测的样品中,胎盘越小,无反应的几率就越高。另一个重要的因素是孕妇的体重,因为在一个较大的女性体内,一定数量的胎儿DNA会被更大的血容量稀释,导致FF降低。表5显示,第二次无反应新样品的概率甚至高于第一次。这种可能性部分是由于胎盘大小在测试和体重稳定之间没有足够的变化。
表5还显示,所有实验室的周转时间都很长。因此,对于无反应的样品,有一个主要的实际问题,尤其是从国外寄出的样品。有些人会考虑不再重复这种检测,从而降低检出率或进行有创的产前诊断(因为无反应时非唐氏综合征非整倍体的风险增加;见后面的讨论),从而增加假阳性率。对于一级cfDNA来说,这个问题更加值得考虑,因为有了依情况而定cfDNA,联合检测的风险可以被自己用来告知选择。
八、对于其他非整倍体的游离DNA性能
除了唐氏综合征,传统的筛查方法还能检测出大量的非整倍体。一些实验室有单独的算法来计算Edwards和Patau综合征的风险,但联合检测只使用唐氏综合征风险的临界值,对这些三体有很高的附带诊断。模型预测,当唐氏综合征假阳性率为5%时,Edwards综合征和Patau综合征的检出率分别为85.0%和96.2%(见参考文献2)。常规的筛查也会导致对其他非整倍体的附带诊断。在一项meta分析中,唐氏综合征仅占被检出的非整倍体的58%,甚至所有3个常见的三倍体加在一起也仅占76%31。
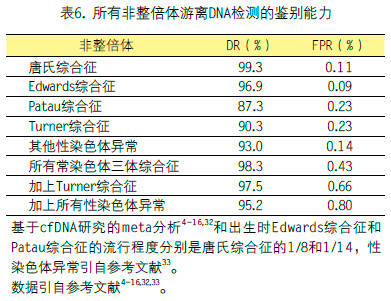
表6显示了cfDNA对Edwards和Patau综合征的区别能力,基于之前提到的唐氏综合征和Palomaki及其同事的高风险研究的meta分析32。与常规筛查相似,但所有商业机构将这些非整倍体纳入cfDNA检测中,与单纯性唐氏综合征相比,假阳性率将显著提高。表6还显示了基于所有已发表研究的meta分析,cfDNA在检测Turner综合征时的区分能力34。这些比率不如Edwards综合征和Patau综合征那么安全,因为它们大多只包括单染色体X病例,而Turner综合征的嵌合病例比出生时的单染色体X更为常见。因此,很大一部分被研究的病例可能是注定要自然流产的孕妇,没有反映出Turner综合征更重要的临床存活病例35。cfDNA还可检测到35例其他性染色体异常(sex chromosome abnormalities,SCAs);表6显示了对所有已发表的研究的meta分析中47,XXX;47,XXY和47,XYY的鉴别能力34。
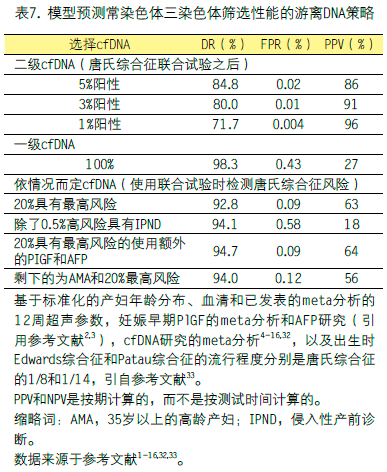
表7显示了3种常见常染色体三体检测的模型预测性能。对于二级、一级和依情况而定cfDNA策略的相对效率,可以得出同样的一般结论。对于一级和二级cfDNA来说,假阳性率越高,表现为PPV降低,而不是依情况而定cfDNA,即0.5%的高风险患者具有侵袭性产前诊断。由于在没有筛查的情况下,所有3个三体的出生流行率仅比唐氏综合征高约五分之一,婴儿死亡率极高,因此,纳入额外的三体对成本效益分析几乎没有影响。
九、假阳性和假阴性结果的原因
传统的非整倍筛查试验会产生较高的假阳性率,根据选择的临界值,其范围在1%到5%之间。假阴性率甚至更高,最好的标志物组合的假阴性率至少为7%左右。这一比率预计将用于筛查试验,这些试验的性质只是为了确定人群的一个亚组,以便进行明确的诊断试验。由于cfDNA检测的区别能力远远优于传统测试,一些人认为它几乎是诊断性的。从这个角度来看,少量的假阳性和假阴性被认为是不一致的。虽然这是有误导性的,但是值得记录这种结果的已知原因,如表8所示。
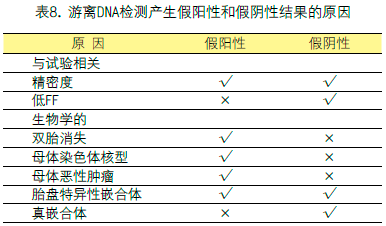
有两种与试验有关的原因。与生化和超声标志物一样,由于测量不精密度,cfDNA结果在受影响妊娠和未受影响妊娠之间的分布存在重叠。一般来说,分析过程中步骤越多,累积的不准密度就越高。对于cfDNA,基于计数的方法可以通过增加DNA片段计数的测序深度来提高精密度。另一个与试验相关的因素是FF。正如前面在低FF的样品中所讨论的,虽然超过实验室可接受的限度,但受影响妊娠的结果分布将更接近于整倍体妊娠,从而导致假阴性结果。对于个别病例,考虑不一致的可能的生物学原因可决定适当的临床管理。
(一)假阳性游离DNA结果的生物学原因
一个未被发现的双胎妊娠在早期妊娠时自然地减少为单胎妊娠是相当普遍的。当这样一个消失的双胞胎发生时,额外的胎盘组织可以持续存在并由cfDNA试验检测到。因为大部分非整倍体都是不能存活的,所以持续组织受到影响的几率会增加,导致假阳性结果。基于SNP的cfDNA方法可以识别可能表示一对双胞胎正在消失的其他单倍型。
一些表型正常的女性有不正常的核型。最常见的是SCAs,如47,XXX例或X染色体缺失的细胞系的嵌合病例。21、18或13三体低水平嵌合也是可能的,以及在其中一条染色体上也可能有少量拷贝数变异(copy number variant,CNV)。在所有这些情况下,都可能产生cfDNA阳性结果。另一个相关的问题,但更大的咨询挑战是隐蔽的母体恶性肿瘤。在这种情况下,可能会有不止一种非整倍体的cfDNA阳性结果,这种非整倍体本身可能会提醒实验室警惕可能的恶性肿瘤。
胎盘特异性嵌合体(Confined placental mosaicism,CPM)是细胞遗传学中众所周知的的现象,即胎盘与胎儿之间存在核型差异。由于胎儿cfDNA主要来源于胎盘,因此cfDNA结果可能与羊膜穿刺术胎儿核型不一致。当胎盘受到影响,胎儿正常时,这将产生假阳性的cfDNA结果36。
(二)假阴性游离DNA结果的生物学原因
与假阳性的位置类似,CPM也会导致假阴性,当胎盘是核型正常,胎儿受到影响时就会出现这种情况。Edwards、Patau和Turner综合征宫内病死率高;比较可行的病例有嵌合胎盘。因此,由CPM引起的假阴性cfDNA结果更有可能存在。原则上,不同细胞系克隆的胎儿真嵌合体比非嵌合病例更容易出现假阴性cfDNA结果。
十、双胞胎
在单绒毛膜双胎中,常规的筛查可以像单卵双生子一样使用该方案,尽管使用的风险算法考虑了增加的(大约两倍的)生化标志物浓度。在双绒毛膜双胞胎中,由于一个胎儿患有唐氏综合征,而另一个没有受到影响,因此生物化学标志物的区分力就降低了。为了避免这种情况,许多中心仅依靠超声标记物;即使是考虑到胎儿之间的相关性的最优算法,检测也相对较低。例如,在一项研究中,模型预测的唐氏综合征检出率为77%,假阳性率为5%37。
cfDNA筛查也出现了类似的问题。对于给定的FF,如果双胞胎中的一个受到影响,而另一个正常,那么每个基因组的21号染色体数目将减少到2.5×FF+2×(1-FF)。这一数字将在一定程度上被双胎妊娠总FF的普遍增加所抵消,在一项研究中估计为三分之一38,可能是因为胎盘体积更大。另一个问题是双胞胎的FF是来自两个胎儿的cfDNA的聚合体,所以受影响的双胞胎可能会造成较低的FF限值,但是聚合体大于该方法的限值,因此检测结果是可以接受的。
文献报道了957例cfDNA双胞胎的结果38-45。在一个胎儿受到影响的情况下,总体检出率如下: 唐氏综合征95.3%(41/43),Edwards综合征88.9%(8/9)和Patau综合征100%(2/2)。当两个胎儿都受到影响时,总体检出率如下:唐氏综合征100%(5/5)和Edwards综合征100%(2/2)。假阳性率为0.0%(0/897)。
虽然双胞胎的检出率低于单胎的估计值,但仍可以认为在这类妊娠中需要进行一级的cfDNA检测。首先,传统的筛查检出率也低于单胎。更重要的是,有过辅助生殖技术的女性中有很大一部分是双胞胎。这样的女性,尤其是那些长期不育的女性,不太可能经历有创性产前诊断的危险。
十一、亚染色体异常
cfDNA检测的商业提供者开始在panel中包括数量有限的与CNVs相关的相对常见的严重异常。这些异常包括如下:DiGeorge综合征(22q11.2缺失),1p36缺失综合征,Prader-Willi,Angelman和Cri-du-Chat综合征。到目前为止,最常见的是DiGeorge综合征,有人估计它的发生率和唐氏综合征一样频繁46。cfDNA检测对这些异常的鉴别能力是通过一项使用孕妇血浆样品和人工血浆混合物的研究得出的47。DiGeorge综合征的估计检出率为83.3%,假阳性率为0.71%,其他综合征合并检出率分别为45.5%和0.06%。因此,在panel中列入这种异常情况预计将使假阳性率几乎增加一倍。包括所有常见的非整倍体和这些亚染色体异常将导致1.6%的侵入性产前诊断率,接近常规筛查。在那些定期提供CVS和羊膜穿刺术样品微阵列分析的中心,患者需要接受咨询,通过侵入性检测将检测到大量的亚染色体异常48。
十二、颈部透明层和孕早期标志物的持续作用
如果cfDNA检测的单位成本大幅降低,包括公共卫生系统在内的第三方支付机构将负担得起一级cfDNA。到那时,卫生规划人员可能会考虑放弃孕早期的超声NT扫描,也许会用更简单的定期扫描代替它。然而,即使没有非整倍体,NT与结构异常和遗传综合征的风险增加有关。其中主要是心脏缺陷;仅这一方面就足以证明保留扫描的合理性。对20项研究的meta分析发现假阳性率为5.5%,检出率为44%49。
还可以保留了一些孕早期的生化标记物,专门用于筛查不良妊娠结局,如子痫前期和生长受限。这些结果比所有非整倍体组合要普遍得多。通过使用母体血清PAPP-A和PlGF早孕筛查,结合超声子宫动脉多普勒和血压测量50-52,以及每日低剂量可溶性阿司匹林的筛查阳性,可以预防很大一部分53。成本效益分析表明这种类型的筛查是可以负担得起的54。
参考文献
1. Cuckle H, Aitken D, Goodburn S, et al. Age-standardisation for monitoring performance in Down’s syndrome screening programmes. Prenat Diagn 2004;24(11):851-6.
2. Cuckle HS, Pergament E, Benn P. Multianalyte maternal serum screening for chromosomal abnormalities and neural tube defects. In: Milunsky A, Milunsky JM, editors. Genetic disorders and the fetus: diagnosis, prevention and treatment-7th edition. Hoboken (NJ): Wiley-Blackwell; 2016. p. 483-540.
3. Huang T, Dennis A, Meschino WS, et al. First trimester screening for Down syndrome using nuchal translucency, maternal serum pregnancy-associated plasma protein A, free-b human chorionic gonadotrophin, placental growth factor and a-fetoprotein. Prenat Diagn 2015;35(7): 709-16.
4. Chiu RW, Akolekar R, Zheng YW, et al. Non-invasive prenatal assessment of trisomy 21 by multiplexed maternal plasma DNA sequencing: large scale validation study. Br Med J 2011;342:c7401.
5. Ehrich M, Deciu C, Zweifellhofer T, et al. Noninvasive detection of fetal trisomy 21 by sequencing of DNA in maternal blood: a study in a clinical setting. Am J Obstet Gynecol 2011;204:205.e201-11.
6. Palomaki GE, Kloza EM, Lambert-Messerlian GM, et al. DNA sequencing of maternal plasma to detect Down syndrome: an international clinical validation study. Genet Med 2011;13:913-20.
7. Bianchi DW, Platt LD, Goldberg JD, et al, on behalf of the MatErnal Blood IS Source to Accurately diagnose fetal aneuploidy (MELISSA) Study Group. Genome-wide fetal aneuploidy detection by maternal plasma DNA sequencing. Obstet Gynecol 2012;119:890-901.
8. Sparks AB, Wang ET, Song K, et al. Noninvasive prenatal detection and selective analysis of cell-free DNA obtained from maternal blood: evaluation for trisomy 21 and trisomy 18. Am J Obstet Gynecol 2012;206:319.e1-9.
9. Ashoor G, Syngelaki A, Wagner M, et al. Chromosome-selective sequencing of maternal plasma cell-free DNA for first-trimester detection of trisomy 21 and trisomy 18. Am J Obstet Gynecol 2012;206:322.e1-5.
10. Norton ME, Brar H, Weiss J, et al. Non-invasive chromosomal evaluation (NICE) study: results of a multicenter prospective cohort study for detection of fetal trisomy 21 and trisomy 18. Am J Obstet Gynecol 2012;207:137.e1-8.
11. Verweij EJ, Jacobsson B, van Scheltema PA, et al. European non-invasive trisomy evaluation (EU-NITE) study: a multicenter prospective cohort study for noninvasive fetal trisomy 21 testing. Prenat Diagn 2013;33(10):996-1001.
12. Stumm M, Entezami M, Haug K, et al. Diagnostic accuracy of random massively parallel sequencing for non-invasive prenatal detection of common autosomal aneuploidies: a collaborative study in Europe. Prenat Diagn 2014;34(2):185-91.
13. Nicolaides KH, Syngelaki A, Gil M, et al. Validation of targeted sequencing of single-nucleotide polymorphisms for non-invasive prenatal detection of aneuploidy of chromosomes 13, 18, 21, X, and Y. Prenat Diagn 2013;33(6):575-9.
14. Zimmermann B, Hill M, Gemelos G, et al. Noninvasive prenatal aneuploidy testing of chromosomes 13, 18, 21, X, and Y, using targeted sequencing of polymorphic loci. Prenat Diagn 2012;32(13):1233-41.
15. Liang D, Lv W, Wang H, et al. Non-invasive prenatal testing of fetal whole chromosome aneuploidy by massively parallel sequencing. Prenat Diagn 2013;33(6):409-15.
16. Porreco RP, Garite TJ, Maurel K, et al. Noninvasive prenatal screening for fetal trisomies 21, 18, 13 and the common sex chromosome aneuploidies from maternal blood using massively parallel genomic sequencing of DNA. Am J Obstet Gynecol 2014;211(4):365.e1-12.
17. Dan S, Wang W, Ren J, et al. Clinical application of massively parallel sequencing-based prenatal noninvasive fetal trisomy test for trisomies 21 and 18 in 11,105 pregnancies with mixed risk factors. Prenat Diagn 2012;32:1225-32.
18. Nicolaides KH, Syngelaki A, Ashoor G, et al. Noninvasive prenatal testing for fetal trisomies in a routinely screened first-trimester population. Am J Obstet Gynecol 2012;207:374.e1-6.
19. Song Y, Liu C, Qi H, et al. Noninvasive prenatal testing of fetal aneuploidies by massively parallel sequencing in a prospective Chinese population. Prenat Diagn 2013;33(7):700-6.
20. Lau TK, Cheung SW, Lo PS, et al. Non-invasive prenatal testing for fetal chromosomal abnormalities by low-coverage whole-genome sequencing of maternal plasma DNA: review of 1982 consecutive cases in a single center. Ultrasound Obstet Gynecol 2014;43(3):254-64.
21. Gil MM, Quezada MS, Bregant B, et al. Implementation of maternal blood cellfree DNA testing in early screening for aneuploidies. Ultrasound Obstet Gynecol 2013;42(1):34-40.
22. Bianchi DW, Parker RL, Wentworth J, et al, CARE Study Group. DNA sequencing versus standard prenatal aneuploidy screening. N Engl J Med 2014;370(9):799-808.
23. Norton ME, Jacobsson B, Swami G, et al. Non-invasive examination of trisomy using directed cell free DNA analysis: the NEXT study. J Med 2015;372(17):1589-97.
24. Pergament E, Cuckle H, Zimmermann B, et al. Single-nucleotide polymorphismbased non-invasive prenatal testing in a high- and low-risk cohort. Obstet Gynecol 2014;124(2 Pt 1):210-8.
25. WHO Commission on MacroEconomics and Health. Macroeconomics and health: investing in health for economic development. Report of the commission on macroeconomics and health. Geneva, Switzerland: World Health Organization; 2001.
26. Cuckle H, Benn P, Pergament E. Maternal cfDNA screening for Down’s syndrome-a cost sensitivity analysis. Prenat Diagn 2013;33(7):636-42.
27. Dar P, Curnow KJ, Gross SJ, et al. Clinical experience and follow-up with large scale single-nucleotide polymorphism-based noninvasive prenatal aneuploidy testing. Am J Obstet Gynecol 2014;211(5):527.e1-17.
28. McCullough RM, Almasri EA, Guan X, et al. Non-invasive prenatal chromosomal aneuploidy testing - clinical experience: 100,000 clinical samples. PLoS One 2014;9(10):e109173.
29. Futch T, Spinosa J, Bhatt S, et al. Initial clinical laboratory experience in noninvasive prenatal testing for fetal aneuploidy from maternal plasma DNA samples. Prenat Diagn 2013;33(6):569-74.
30. Zhang H, Gao Y, Jiang F, et al. Noninvasive prenatal testing for trisomy 21, 18 and 13-clinical experience from 146,958 pregnancies. Ultrasound Obstet Gynecol 2015;46(1):130.
31. Davis C, Cuckle H, Yaron Y. Screening for Down syndrome -incidental diagnosis of other aneuploidies. Prenat Diagn 2014;34(11):1044-8.
32. Palomaki GE, Deciu C, Kloza EM, et al. DNA sequencing of maternal plasma reliably identifies trisomy 18 and trisomy 13 as well as Down syndrome: an international collaborative study. Genet Med 2012;14:296-305.
33. Hook EBH. Chromosomal abnormalities: prevalence, risks and recurrence. In: Brock DJH, Rodeck CH, Ferguson-Smith MA, editors. Prenatal diagnosis and screening. London: Churchill Livingstone; 1992. p. 351-92.
34. Gil MM, Quezada MS, Revello R, et al. Analysis of cell-free DNA in maternal blood in screening for fetal aneuploidies: updated meta-analysis. Ultrasound Obstet Gynecol 2015;45(3):249-66.
35. Hook EB, Warburton D. Turner syndrome revisited: review of new data supports the hypothesis that all viable 45X cases are cryptic mosaics with a rescue cell line implying an origin by mitotic loss. Hum Genet 2014;133:417-24.
36. Grati FR, Malvestiti F, Ferreira JC, et al. Fetoplacental mosaicism: potential implications for false-positive and false-negative noninvasive prenatal screening results. Genet Med 2014;16(8):620-4.
37. Maymon R, Rozen H, Baruchin O, et al. Model predicted Down’s syndrome detection rates for nuchal translucency screening in twin pregnancies. Prenat Diagn 2011;31(5):426-9.
38. Canick JA, Kloza EM, Lambert-Messerlian GM, et al. DNA sequencing of maternal plasma to identify Down syndrome and other trisomies in multiple gestations. Prenat Diagn 2012;32:1-5.
39. Gro¨mminger S, Yagmur E, Erkan S, et al. Fetal aneuploidy detection by cell-free DNA sequencing for multiple pregnancies and quality issues with vanishing twins. Clin Med 2014;3:679-92.
40. Leung TY, Qu JZZ, Liao GJW, et al. Noninvasive twin zygosity assessment and aneuploidy detection by maternal plasma DNA sequencing. Prenat Diagn 2013;33(7):675-81.
41. Lau TK, Jiang F, Chan MK, et al. Non-invasive prenatal screening of fetal Down syndrome by maternal plasma DNA sequencing in twin pregnancies. J Matern Fetal Neonatal Med 2013;26:434-7.
42. Srinivasan A, Bianchi D, Liao W, et al. Maternal plasma DNA sequencing: effects of multiple gestation on aneuploidy detection and the relative cell-free fetal DNA (cffDNA) per fetus. Am J Obstet Gynecol 2013;208:S31.
43. Huang X, Zheng J, Chen M, et al. Noninvasive prenatal testing of trisomies 21 and 18 by massively parallel sequencing of maternal plasma DNA in twin pregnancies. Prenat Diagn 2014;34(4):335-40.
44. del Mar Gil M, Quezada MS, Bregant B, et al. Cell-free DNA analysis for trisomy risk assessment in first-trimester twin pregnancies. Fetal Diagn Ther 2014;35(3):204-11.
45. Bevilacqua E, Gil MM, Nicolaides KH, et al. Performance of screening for aneuploidies by cell-free DNA analysis of maternal blood in twin pregnancies. Ultrasound Obstet Gynecol 2015;45(1):61-6.
46. Grati FR, Molina Gomes D, Ferreira JC, et al. Prevalence of recurrent pathogenic microdeletions and microduplications in over 9500 pregnancies. Prenat Diagn 2015;35(8):801-9.
47. Wapner RJ, Babiarz JE, Levy B, et al. Expanding the scope of non-invasive prenatal testing: detection if fetal microdeletion syndromes. Am J Obstet Gynecol 2015;212(3):332.e1-9.
48. Wapner RJ, Martin CL, Levy B, et al. Chromosomal microarray versus karyotyping for prenatal diagnosis. N Engl J Med 2012;367(23):2175-84.
49. Sotiriadis A, Papatheodorou S, Eleftheriades M, et al. Nuchal translucency and major congenital heart defects in fetuses with normal karyotype: a meta-analysis.Ultrasound Obstet Gynecol 2013;42(4):383-9.
50. Akolekar R, Syngelaki A, Poon L, et al. Competing risks model in early screening for preeclampsia by biophysical and biochemical markers. Fetal Diagn Ther 2013;33(1):8-15 [Erratum appears in Fetal Diagn Ther 2013;34(1):43].
51. Park F, Russo K, Pellosi M, et al. The impact of aspirin on the prevalence of early onset pre-eclampsia after first trimester screening. Prenat Diagn 2014;34(Suppl 1):e4.
52. Poon L, Syngelaki A, Akolekar R, et al. Combined screening for preeclampsia and small for gestational age at 11-13 weeks. Fetal Diagn Ther 2013;33:16-27.
53. Roberge S, Nicolaides KH, Demers H, et al. Prevention of perinatal death and adverse perinatal outcome using: a meta-analysis. Ultrasound Obstet Gynecol 2013;41:491-9.
54. Shmueli A, Meiri H, Gonen R. Economic assessment of screening for preeclampsia. Prenat Diagn 2012;32(1):29-38.




