三种人乳头瘤病毒DNA检测方法的比较: 下一代测序、多重PCR和巢式PCR结合Sanger测序
背 景
人乳头瘤病毒(HPV)是一种全球分部的,源于分类学乳头瘤病毒科的多样化双链DNA病毒,具有超过170种HPV基因分型,其中40种可以感染人体子宫颈阴道黏膜。它们的风险量级和临床表现多样。高危HPV在宫颈癌的发生中发挥这关键作用,是超过90%宫颈癌和多种头颈部肿瘤亚型的罪魁祸首。一些HPV可侵犯皮肤,具有皮肤趋向性,似乎在皮肤癌的发生方面发挥着作用。因此,定义HPV的类型比诊断HPV感染更重要,可以更准确地消除HPV的致癌风险。
目前尚无HPV的体外培养方法,也没有稳定的血清型反应。因此HPV的诊断仅仅依靠分子方法检测HPV DNA及其序列。HPV具有大约8000bp长度的环形双链DNA基因组,分为3个主要部分:早期区(E)、晚期区(L)和非编码部位(处于调节区上游,URR)。在E2和L2 ORF之间还存在高可变性的非编码小区。所有已知的HPV具有相似的基因结构,在HPV基因分型分类学定义的L1区中,核苷酸具有大于10%的可变性。
目前,一些实验室试验可以用于检测HPV-DNA。一些国家已经使用HPV-DNA多重PCR对与子宫颈阴道细胞学相关的宫颈癌进行预防和筛查。该方法是一种PCR技术,使用一些特定HPV类型(根据致癌风险分组)引物。尽管大部分的试剂盒目前不再受专利所有权的限制,但是生产商仍将他们所生产引物的精确序列作为商业机密。总之,同时使用4种引物扩增每个风险分组/类型不同大小的基因:HPV16、HPV18、高危HPV(除了16和18)以及HPV6/11。HPV多重PCR的灵敏度高于HC,还能检测不同分组HPV基因型的累积感染。因此,这种方法被医疗机构广泛采用,费用相对低廉,但是这种方法在科学研究的应用方面受到限制,因为除了内在的HPV-DNA非测序局限性之外,它还不能区分HPV基因分型或变体。
科学研究使用的最传统技术,多年以来的金标方法,是使用共同引物MY09和MY11(正反向)的PCR结合Sanger基因测序或采用类型特异性探针的液体杂交技术。这种经典的技术源于1991年,由Manos和Bauer提出,是一种独特的PCR。这种PCR可以在使用Sanger测序或特异类型探针杂交后,扩增HPV基因组L1区450bp长度的片段。随后Roda Husman等人发现了其他引物对(GP5+/GP6+),能够扩增L1区140bp-450bp长度的片段,叠加在MY引物上。比较了这些技术的研究报告称联合使用这些技术可以提高试验的准确度。称之为巢式PCR的技术具有较高的灵敏度,该试验由2个连续的PCR反应组成(在2个试管内)。第一阶段试验使用MY引物,第二阶段试验使用GP引物,接着使用电泳或Sanger测序分析。该技术非常灵敏,已经用于科学研究。但是,这种技术不能通过多个HPV基因型检测累积感染。
最终,一种具有前景的新技术—下一代测序(NGS)横空出世。NGS标志着现代基因组学的到来。NGS不同于Sanger方法,能以空前的速度对DNA进行测序,开启了其在生物医学研究应用领域的大门。测序系统可以在一次运行内以300千碱基值至1兆碱基的范围内生成并输出数据。它的主要区别是对每个独立样品使用特殊核苷酸初始序列标记的引物,这称作打上“条码”的引物或标记引物。在相同的PCR反应中可能使用多个引物对多个目标DNA进行测序(如有必要还可以对多个种类进行测序)。结果是巨大的平行序列和DNA片段,每个特殊样品(条码)全部具有共同的初始序列,逐个对分子进行分析。NGS支持累积感染和微生物组特征描述,在24小时内,单一测序装置可以让NGS读取百万计的DNA序列,相当于上千个Sanger测序装置同时运行。NGS尚未广泛应用,它的HPV诊断准确性也尚未确定。此次研究的目标是比较3种实验室方法:下一代测序、多重PCR和巢式PCR结合Sanger测序的HPV感染诊断性能。
方法
研究设计比较3中实验室方法对宫颈分泌物样品中HPV DNA感染和基因分型方面的性能。我们使用便于获得的样品,这些样品来源于一项旨在评估亚马逊地区妇女人群子宫颈阴道HPV感染患病率的研究,该研究涉及的妇女人群没有先前HPV感染诊断史,但是存在高宫颈癌风险。该研究得到了巴西国家IRB的批准,随机选择了95份样品。样品通过双盲方式处理。
全部95份样品通过NGS使用相同程序(见下文)分析2次。因此每份样品同时双盲分析4次,一次多重PCR分析,一次Sanger测序后巢式PCR分析和2次NGS分析(总共4次)。样品被编号保密。全部试验使用了阳性和阴性质控品。
标本采集和DNA提取
在原始研究中,研究参与者接受妇科检查包括窥镜检查,使用宫颈刷插入宫颈通道1-1.5厘米深旋转3次采集宫颈细胞。接着根据推荐的方法将样品洗脱到标本运输介质(STM®;Qiagen,CA)内并使用QIAamp DNA试剂盒(Qiagen,CA)分离DNA。全部样品在实验室生物安全柜内处理,实验室与执行PCR扩增的地点分开。
使用重新获得DNA样品的3uL等份执行多重PCR。根据生产商建议我们使用HPV4a ACE Screnning®试剂盒(Seegene,Seoul,South Korea)。PCR系统由4uL引物溶液(5x HPV4A ACE AM)、3uL8-MOP缓冲液和10uL 2X多重主混合溶液(包含聚合酶)组成,最终体积20uL。设置Eppendorf热循环仪–Gradient® Mastercycler(Eppendorf AG,Hamburg,Germany)的程序:初始变性作用,94℃,15分钟;接着在94℃的环境中的30秒循环;60℃环境中进行90秒循环;72℃环境中进行90秒循环,循环总数为40个,并在72℃环境中最终延长到10分钟。接着将1份5uL等份的PCR产物提交电泳处理,使用2.0%溴化乙锭(1.0ug/ml)染色的琼脂糖胶分析20分钟。根据扩增子的大小(见表I),执行HPV基因型定义分析(目测)。多重PCR试验能够检测27种黏膜HPV基因型:i)高危(HR)-HPV类型:16,18,31,33,35,39,45,51,52, 6,58,59,66,68;ii)可能的HR-HPV类型:26,53,67,70, 73,82;低风险(LR)-HPV类型:6,11。
按照Roda Husman等人的叙述连续执行2个阶段的PCR。第一阶段PCR使用的系统由重新获得的DNA溶液的5uL等份、5uL 10X缓冲液、1.5uL MgCl2 50mM、5uL正向引物MY11、5uL反向引物MY09、1.0uLdNTP 10mM、0.5uLTaq聚合酶 5U/uL和27uL Mili-Q水组成,最终体积50uL。Eppendorf 热循环仪–Gradient® Mastercycler的运行设置方法见表II。
表I. 电泳内HPV-DNA产物多重PCR的解析
a其他HR-HPV:26, 31, 33, 35, 39, 45, 51, 52, 53, 56, 58, 59, 66, 68,73 e 82。根据厂商建议,HR-HPV使用的引物可能通过交叉反应检测3种其他的HPV类型(39,52和68)
表II. HPV第1和第2阶段巢式PCR热循环仪协议
全部样品进入第2阶段PCR。第2阶段使用的PCR系统由1uL第1阶段的PCR产物(扩增的DNA样品)、5uL 10X缓冲液、1.5uL MgCl2 50mM、5uL GP5+引物、5uL GP6+引物、1.0uL dNTP 10mM、0.5uL Taq聚合酶5U/uL和31uL MiliQ水组成,最终体积50uL。使用相同的热循环仪方法。使用2.0%溴化乙锭(1.0ug/ml)染色的琼脂糖胶分析巢式PCR产物20分钟。响应扩增子150bp。
使用Exo-SAP纯化阳性样品并提交自动DNA测序1000®型设备,通过Sanger技术处理(MegaBace 1000®,Biociences Amersham,UK)。我们使用的反应系统:5.0uL扩增的DNA、4.0uL预混合DyEnamic ET Terminator Kit®(GE Healthcare,UK)、1.0uL GP5+引物50pmol/uL。我们使用下列协议:95℃,25秒;95℃,15秒;50℃,20秒;60℃,1分钟重复30个周期扩增。添加1uL醋酸铵沉淀和40uL纯酒精进行PCR产物测序,接着搅拌并在室温环境中避光培养20分钟。使用冷冻离心分离机5801R(Eppendorf®)以4000rpm的速度离心微孔板,去除上清液。根据生产商的标准执行毛细管聚丙烯酰胺凝胶电泳(使用3KV电压入射80秒,该运行在44℃的环境中以6KV电压执行200分钟)。使用生物信息学软件MEGA6®(Arizona)执行分析。为了校准序列,我们使用CLUSTAL W® BIOEDIT(Clustal,Dublin,Ireland)。为了鉴别和确认HPV类型,使用BLAST/BLASTN®程序将序列与世界数据库核苷酸基因库®存储的数据比较。同源性e值小于e-10的序列为有效序列。
NGS引物设计和样品源。由于人群研究中HPV类型呈现的光谱未知,我们利用Burk实验室开发的新型下一代测序试验(纽约阿尔伯特•爱因斯坦医学院)标定L1 ORF内的2个区。对每个试验使用一些针对不同HPV属、种或类型的简并引物,引物被混合。唯一的8bp Hamming DNA条码添加到每种引物的5’终端。每个条码至少2bp不同于全部其他引物。
每个NGS试验中每种DNA的1个等份使用打上8bp条码的寡核苷酸引物扩增。对于所有样品,通过正向和反向引物引入1个唯一的条码到PCR扩增子。简而言之,一个25uL反应内1uL DNA样品具有一份等量AmpliTaq Gold DNA聚合酶(Life Technologies,CA)和HotStart-IT FideliTaq DNA聚合酶(Affymetrix,CA),PCR条件包括95℃,5分钟的变性作用,接着是55℃,1分钟;68℃,1分钟;95℃25个循环1分钟;60℃,1分钟;以及68℃,1分钟,最终在68℃延长到10分钟。试验fapR退火温度调节到前15个循环57℃,后25个循环62℃。
将溴化乙锭凝胶的谱带强度与质控品对比,估计每个扩增子的预测HPV片段成功扩增;将全部样品打码的PCR产物混合,通过添加少量富PCR产物样品调整PCR产物的DNA浓度。如果溴化乙锭染色没有观测到扩增子,添加20uL反应物到混合物中。该PCR产物混合物使用QIAquick凝胶提取试剂盒(Qiagen,CA)纯化;使用Illumina TruSeq DNA样品制备试剂盒制备1等份纯化的DNA扩增子,并在阿尔伯特爱因斯坦表观基因组学共享设施Illumina HiSeq(Illumina Inc., San Diego,CA)内使用150bp配对末端测序技术测序。
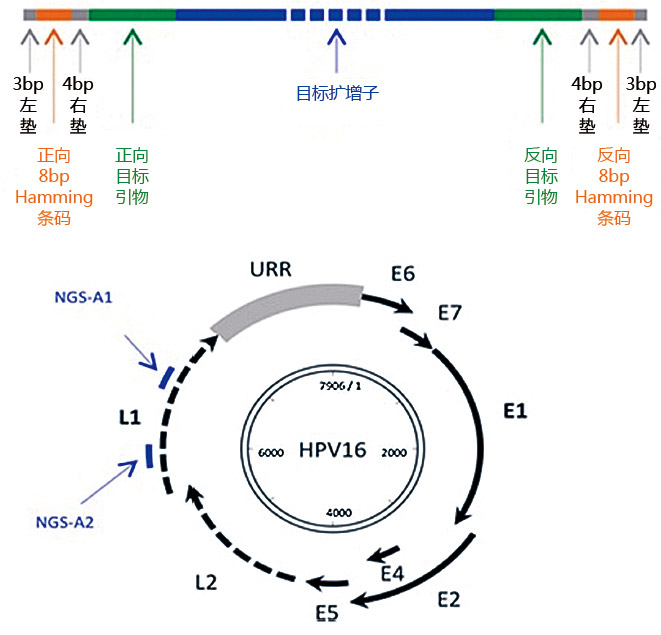
图1. 下一代测序的引物设计
配对末端短Illumina读数根据其样品特异性条码,使用NovobarcodeV1.00进行信号分离、过滤,获得低质量读数(Q20)并使用prinseq-lite V0.20.4缩短长度,使用FlASh V1.2.7合并为单一读数。附上不能合并的单端读数。提交这些序列使用USEARCH v 7.0.1001中的UChime参照“黄金”PV参考数据库挑选嵌合体。所有通过QC过滤器的读数被聚合到95%恒等运算分类单位(OTU)中并使用USEARCH分配HPV分类法,调整为PV数据集。观测并创建每份样品每种HPV的次数计数表;按照类型进行分类。
设置临界值去除“低频率合并”,如果总HPV读数数量低于1000,则认为样品为阴性。每种样品中,对于每个既定HPV类型,需要考虑的阴性或阳性,读数数量大于样品内总HPV读数数量的1%,还要大于另一个样品内该类型最大读数数量的1%。
每种实验室技术使用描述性分析。为了比较占比,我们对小样品使用x2试验或费舍尔精密试验。使用Cohen kappa指数评估观察者间一致性。将多重PCR、巢式PCR和NGS试验1的结果与NGS试验2的结果进行比较。HPV、HR-HPV、HPV16、HPV18型,HPV类型累积感染检测,样品中检测的HPV类型多样性,分析的问题具有积极性。根据世界卫生组织(WHO)国际癌症研究所的标准,我们假设HR-HPV类型为:16,18,26, 31, 33, 35, 39, 45, 51, 52, 53, 56, 58, 59, 66, 68,73 和82。
结 果
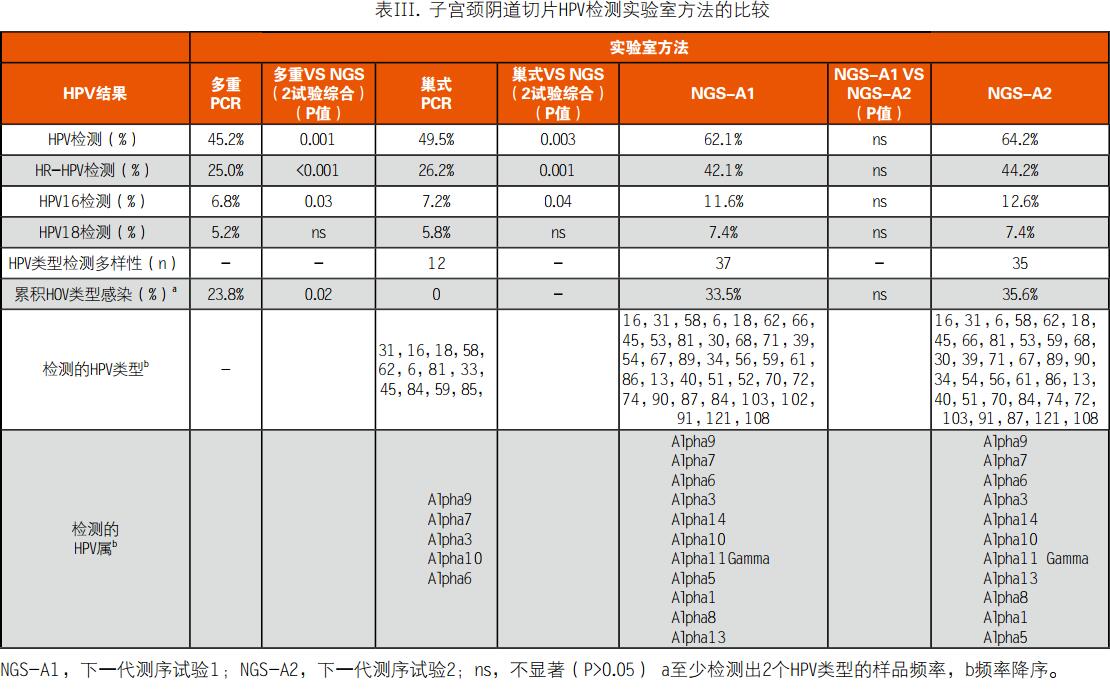
没有丢失样品,每个单独样品全部执行了4个试验。HPV检测随着实验室方法的不同而变化。NGS试验的HPV患病率高于多重PCR(分别为64.2% VS 45.2%,P=0.001)和巢式PCR(分别为64.2% VS 49.5%,P=0.003)确定的患病率。HR-HPV检测也可以看到相同的情况。比较2个HGS试验时,HPV和HR-HPV的患病率没有区别。NGS试验在于巢式PCR和多重PCR比较时,在检测HPV16方面,NGS性能更佳(12.6% VS 7.2% VS6.8%,P<0.05)。但是,3种实验室方法在检测HPV18的患病率方面的性能存在显著差别(表III)。
NGS方法中患病率最高的HPV类型是HPV16和HPV31,巢式PCR也观测到了类似结果但是顺序相反。在利用HPV类型检测累积感染方面,NGS试验也具有显著优势。但是使用NGS试验检测,三分之一的样品可以多检测出不止一个HPV类型,通过多重PCR检测,仅有四分之一的样品能检测出累积HOV类型。通过NGS试验检测,2名患者的子宫颈阴道切片中检测出9种HPV类型。巢式PCR不能检测出累积感染。NGS试验可以检测出更多的HPV类型多样性(35型和37型),巢式PCR仅能检测出12种HPV类型(表III)。
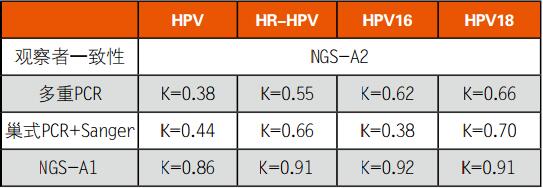
对于HPV感染诊断结果,多重PCR和巢式PCR相对于NGS(试验2)的观察者间一致性较低,且HR-HPV、HPV16和HPV18感染的诊断相关性中等(表IV)。NGS-A1与NGS-A2在检测HPV(k=0.86)、HR-HPV(k=.91)、HPV16(k=0.92)和HPV18(k=0.91)方面的相关性很强。
讨 论
下一代测序(也称为大型平行测序)是遗传学研究出现巨大转变的开始,因为这种技术有能力加强对基因组、转录组、表观基因组和微生物组学的描述。作为一种相对较新的方法,有必要验证其在HPV检测方面的性能。我们的研究证明:与临床最常用方法(多重PCR)和广泛应用于科学研究的传统Sanger测序相比,NGS具有更好的检测和基因分型性能。
与其他方法比较,NGS试验将HPV(全类型)诊断率提高了40%。关于HR-HPV的检测。NGS试验的性能更高,几乎提高了2倍的阳性率。NGS试验较高的灵敏度部分因为其较高的累积感染检测性能。Sangger测序生成每个阳性样品唯一的序列,大概是最丰富的HPV类型,疏漏了其他共感染类型即样品中丰度较低的类型。值得注意的是,此次研究中有的样品通过NGS试验检测出9种HPV类型,但是用传统方法(Sanger)只检测出1种类型。实验室方法检测HPV多样性时强调了这一点。另一个方法论方向有助于提高NGS的灵敏度,这个方向是定义HPV阳性但不根据电泳凝胶目测,目测是Sanger测序常用的方法。如前所述,第1扩增步骤内的全部阳性和阴性样品都使用NGS测序系统再评估。NGS电泳的目测仅作为确定用于测序的样品等份的大小。这些证据表明许多具有流行病学重要性的HPV类型不能通过使用传统分子生物学方法的经典流行病学研究检测出来。
表IV. HPV诊断试验和NGS(Cohen Kappa Index)之间的观察者一致性
众所周知,每次PCR获得的扩增子大小能影响试验的灵敏度。扩增子越小,灵敏度越高。尽管巢式PCR的第1反应已经使用了MY引物(生成了450bp扩增子),但是Sanger测序使用的该引物是GP5+,生成扩增子大小为140-150bp,比2个NGS试验生产的扩增子(150bp)小。但是,多重PCR生产的扩增子比较大(最大达到500bp),影响到了该试验的灵敏度。
此次研究的另一个贡献是比较了平行和双盲状态执行的2种NGS试验,在检测HPV患病率(和类型)方面没有显著区别,试验与主要分析预后存在较高的观察者一致性。我们的研究中,大部分和NGS试验不一致的样品与HPV非常低的读数计数有关(低载量病毒感染)。试验间较高的相关性确认了NGS在HPV诊断和基因分型方面方法学程序的稳定性。NGS试验生成的所有DNA序列(每个试验大约150万个序列)在混合扩增子等份后在一次测序反应中一起进行了测序。使用标记引物标志着明确每个序列源差异时代的到来。与NGS相关的一件有趣的事情是单一测序反应生成百万计的基因序列,因此巨大的信息量使得解析步骤受到了限制。这一分析“瓶颈”需要生物信息学专业训练和专业知识,还需要清晰的结果解析程序设计和分类任务。这一点目前还不是很重要,因为Sanger测序通常能提供唯一的序列。因此,我们的研究特别关注了对试验中使用的生物信息学分析程序的详尽描述。
我们断定NGS是一种子宫颈阴道切片灵敏的HPV诊断和基因分型方法,它比传统的多重PCR和Sanger测序更灵敏,在检测HPV类型累积感染和/或变异方面具有广泛的前景,打开了HPV流行病学综合研究的新视角,尤其在监测抗HPV疫苗效果方面具有特殊的效果。NGS具有成为传统HPV诊断和基因分型替代方法的潜力。但是NGS试验的实用性和成本仍是其广泛应用于临床实践和科学研究的障碍。
(摘自Journal of Medical Virology,版权归其所有,仅供内部参考)
编译:张凯
审校:胡济梁




