患者外周血LncRNA检测在胃癌诊断中的临床价值

作者:线海鹏 赵晓涛
单位:北京大学人民医院检验科
【摘要】长链非编码RNA(long non-coding RNA,LncRNA)是长度超过200个核苷酸的不“翻译”为蛋白质的RNA。在胃癌的发生发展过程中,有多种LncRNA表达量有显著变化,如HOTAIR、H19、LINC00152、MEG3、GAS5等。LncRNA在胃癌中的异常表达与胃癌的发病机制、临床诊断以及预后等有着密切的联系。本文旨在通过总结近年来外周血LncRNA在全球第二大致死肿瘤-胃癌领域的相关研究,为外周血LncRNA作为非侵袭性的胃癌诊断及预后标志物提供新的思路。
目前研究显示,超过90%的人类基因组转录为RNA,然而只有1.5%-2.0%人类基因组编码蛋白,转录但不翻译为蛋白质的称为非编码RNA(Non-coding RNA,ncRNA)[1]。LncRNA是指转录本长度大于200bp,但不具有编码蛋白质功能的长链非编码RNA分子,它具有高度保守的序列特征、特定的空间二级结构、复杂的亚细胞定位等特征。同时,大部分LncRNA在不同的组织中具有不同的分布特异性,其二级结构也不尽相同,这进一步说明了LncRNA拥有丰富的生物学功能[2]。根据相对位置可以将LncRNA分为五类:(1)基因内LncRNA(intronic LncRNA):指同一个基因中外显子之间,由内含子转录得到的LncRNA[3, 4];(2)基因间LncRNA(intergenic LncRNA,lincRNA):从不同非编码蛋白基因间转录得到的LncRNA;(3)正义LncRNA(sense LncRNA):转录方向与编码蛋白的基因转录方向相同的LncRNA;(4)反义LncRNA(antisense LncRNA):转录方向与编码蛋白的基因转录方向相反的LncRNA;(5)双向LncRNA(bidirectional LncRNA):转录方向与编码蛋白的基因转录方向相同或相反的LncRNA。相关研究应用多种技术和工具来探究LncRNA的相关功能,由于LncRNA种类多、缺少开放读码框(Opening Read Frame,ORF)、保守性较差等特点,所以LncRNA功能研究的难度要远大于microRNA、siRNA和snRNA等非编码RNA[5]。常用的LncRNA分析方法有:结合染色质免疫共沉淀的深度测序技术(Chromatin Immunoprecipitation Sequencing,ChIP-seq)、结合RNA-seq的LncRNA功能预测技术、非编码RNA沉默与定位分析技术(Combined knockdown and localization analysis of noncoding RNAs,c-KLAN)、荧光原位杂交技术(FISH)、RT-PCR技术等[6]。已经证实LncRNA可以对多种生物学相关功能进行调控:(1)肿瘤相关基因的沉默与过表达;(2)不同剪切体的功能调控与形式转换;(3)参与基因转录的上调与下调以及蛋白的甲基化和去甲基化;(4)参与调控细胞的凋亡与自噬。
一、LncRNA与胃癌
长期以来,人们对肿瘤基因组的研究主要集中在外显子组上,随着高通量测序技术的出现,测序深度不断提高,研究者发现,曾经被认为是转录“垃圾(junk)”的长链非编码RNA在生长发育、分化凋亡以及相关细胞代谢中有着不可或缺的作用,然而依旧有很多LncRNA的功能尚不明确[7, 8]。近年来,胃癌相关的LncRNA异常调节受到学界的广泛关注,这些LncRNA与肿瘤的进展、侵袭、转移与化疗耐受密切相关,最新报道显示在胃癌组织中存在135种异常表达的LncRNA,其中71种表达明显上调,64种表达下调,使得LncRNA有望成为胃癌新的治疗靶点[9]。本文总结(见表1)回顾和分析了近年来胃癌LncRNA的相关研究文献,为胃癌的研究提供了新的思路。
1. H19基因:H19是一种和细胞生长有关的“管家基因”,位于人类11号染色体短臂15区5亚带,长2322bp,拥有五个外显子和四个内含子。其表达含量受到差异表达区域(Differentially Methylated Domain,DMD)和印记控制区域(Imprinting Control Region,ICR)的调控[10]。H19由于其肿瘤基因的抑制作用,最初被认为肿瘤抑制因子,但最近的研究显示H19同时具有促肿瘤作用和抑肿瘤的作用[11]。最新研究显示,H19在胃癌组织和胃癌细胞中高表达,在贲门腺癌中表达量更是显著升高,同时与贲门腺癌TNM分析显著相关[9, 12-16]。此外,高表达的H19是胃癌患者生存期的独立危险因素。作为转录因子,细胞同源性禽类骨髓病毒致癌基因(Cellular Homolog of Avian Myelocytomatosis Virus Oncogene,c-myc)可以直接上调胃癌细胞中H19的表达含量,同时研究还显示,H19对p53活化起到一定作用,但其机制尚不明确[14]。一些研究还显示野生型p53对维护DNA甲基化非常重要,同时在p53突变或者野生型p53 cDNA增加的情况下,H19启动子的活性明显上升[12]。H19还被认为是某些mi-RNA的前体,如在胃癌细胞中,H19介导miR-675通过肿瘤抑制基因转录因子1(Tumor Suppressor Runt-Related Transcription Factor 1 Gene,RUNX1)来增殖为癌细胞[17, 18]。另外,该研究还发现在肿瘤细胞发生发展过程中的新的基因调节通路:H19/miR-675/RUNX1信号通路,该通路可能成为胃癌潜在的治疗靶点。
近年来相关研究还显示,H19在胃癌患者血浆表达量显著高于非癌人群,同时H19在血浆中非常稳定,且在胃切除后显著降低,这提示H19可能是早期胃癌诊断与评估的新型标志物。研究还显示,H19在缺氧状态下可以被p53/HHIF1-α通路介导,同时体外实验证实H19敲除可以显著抑制缺氧介导的癌细胞增殖[19]。此外,H19 siRNA在肝癌小鼠模型(Hepatic Cell Cancer,HCC)和膀胱癌细胞系中抑制肿瘤发生,而沉默H19可以增强体外缺氧恢复后肿瘤细胞的增殖能力[20]。因此,H19可以成为多种肿瘤(尤其是那些可以被缺氧压力促进生长的肿瘤)的治疗靶点。总的来说,H19可能成为胃癌新型的药物治疗靶点。
Table 1. 胃癌相关的LncRNA
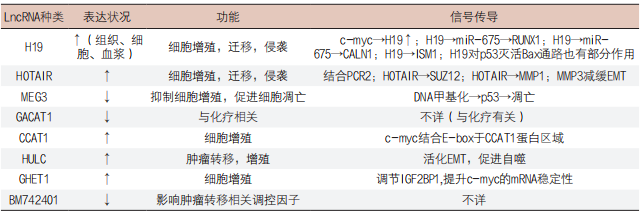
2. HOTAIR:HOX转录本基因间反义RNA(HOTAIR)是一个非编码RNA,其转录内容主要为典型HOXC基因的反义转录本。研究显示,HOTAIR长2158bp,位于HOXC所在的12号染色体长臂13区13亚带[21]。早期研究显示,尽管HOTAIR是一个非编码RNA,但其功能之多使学者们对非编码RNA的功能认识上升到一个新的高度[22]。HOTAIR与PRC 2(Polycomb Repressive Complex 2)、SUZ12(Suppressor of Zeste 12)、EZH2(Enhancer of Zeste Homolog 2)相互作用引起H3K27三甲基化[23, 24]。另外,其他研究报道显示,高表达的HOTAIR与包括乳腺癌、结直肠癌、前列腺癌在内的多种癌症有关[25]。
近来,文献报道显示,HOTAIR在胃癌组织中的高表达与胃癌进展及其TNM分期有关,特别是与肿瘤的淋巴结转移显著相关[26-28]。多篇研究显示过表达的SUZ12通过PRC2和H3K27的三甲基化途径提示,HOTAIR是一个非常重要的肿瘤转移相关因子[27]。另外,其他研究显示,EZH2可能结合HOTAIR相关位点,同时高水平的HOTAIR可以通过沉默PRC2抑制基因(如:Growth-Differentiation Factor 15,GDF15)进而介导细胞增殖[23, 29, 30]。此外,HOTAIR通过调节金属蛋白酶(Matrix Metalloproteinases,MMPs)介导上皮间充质转化(Epithelial-Mesenchymal Transition,EMT)进而在肿瘤细胞侵袭中起到重要作用[28]。miR-196a与HOTAIR同处HOX基因簇中,其上调在胃肠道间质瘤恶性转化中起到非常重要的作用。HOTAIR作为内生的RNA,它与miR-331-3p存在竞争性抑制进而影响人类表皮生长因子2及其转录相关的调节[31]。
总的来说,HOTAIR是一个肿瘤广谱表达的LncRNA,同时可以成为肿瘤患者诊断和预后评估的独立危险因素。更重要的是,HOTAIR为新一代分子靶向药物的开发提供方向。
二、胃癌相关的其它LncRNA
除了H19和HOTAIR,还有多种LncRNA在胃癌组织及细胞中异常表达,这些LncRNA可以大致分为两类:高表达的LncRNA和低表达的LncRNA。它与H19和HOTAIR类似,这些LncRNA中一部分也被证实与胃癌肿瘤大小、淋巴结转移和TNM分期有关[9, 13, 32, 33]。
1. 胃癌中高表达的LncRNA:上调的LncRNA与肿瘤的大小、侵袭深度、淋巴结转移有关,因此其可以成为潜在的胃癌患者TNM分期和预后指标。一项96人的配对研究显示,在这些上调的LncRNA中,SUMO1P3(Small Ubiquitin-Like Modifier 1 Pseudogene 3)的表达量与肿瘤大小、分化程度、淋巴结转移情况、侵袭深度显著相关。这意味着SUMO1P3可以成为胃癌患者潜在的预后生物标志物[34]。另外,它与人类T细胞凋亡和分裂有关,且乳腺癌中表达量下调的GAS5(Growth Arrest Specific Transcript 5)也在胃癌组织中表达上调[8, 13, 32]。这提示GAS5可能同时具备原癌基因和抑癌基因两种功能,这需要进一步的研究确认。
2. 胃癌中低表达的LncRNA:GACAT1(Gastric Cancer-Associated Transcript 1)是胃癌低表达的LncRNA中最具代表性的一个,其表达水平与胃癌淋巴结转移和胃癌分期显著相关[35]。BM742401是一个新发现的LncRNA,其表达水平在胃癌组织中显著降低且与III期胃癌患者的预后显著相关。同时BM742401与肿瘤的多种代谢过程有关,其中BM742401可以显著下调MMP9的现象受到学者们广泛关注,使得其成为胃癌潜在的治疗靶点[36]。uc001lsz也是胃癌细胞中新发现的LncRNA,在胃癌早期表达量较高,随着肿瘤的进展,表达量显著降低,使其可以成为肿瘤早期诊断的标志物[9]。此外,胃癌组织中MEG3(Maternally Expressed Gene 3)通过DNA甲基化导致其表达量下调,同时其功能通过活化p53基因介导细胞凋亡,抑制细胞增殖[37]。
还有多种LncRNA在胃癌中首次报道,其分子机制已经在其它肿瘤相关文献中阐明。例如,它与结肠癌相关的CCAT1(Colon Cancer-Associated Transcript 1)在胃癌组织中也出现明显上调,同时其与胃癌的肿瘤大小、淋巴结转移情况显著相关[38]。CCAT1通过调节启动子区域的c-myc与E-box之间的相互作用发挥原癌基因的功能。通过激活EMT进而促进肿瘤转移的HULC(Highly Upregulated In Liver Cancer)被发现在胃癌中通过激活自噬进而介导肿瘤细胞的增殖[39]。LncRNA-GHET1(Lncrna Gastric Carcinoma Highly Expressed Transcript 1)作为抗肿瘤药的潜在作用靶点与IGF2BP1(Insulin-Like Growth Factor 2 Mrna-Binding Protein 1)结合继而增强c-myc稳定性来促进细胞增殖[40]。此外LINC00152在BGC-823、MGC-803和SGC-7901等胃癌细胞株表达量显著高于胃组织正常细胞株GES-1,更为重要的是其在胃液中表达含量显著高于正常对照,这提示其具有预测胃癌发生的潜在能力[41]。
三、LncRNA在胃癌中的潜在临床应用价值
对于肿瘤的早期诊断来说,寻找新型的非侵袭性生物分子标志物是非常必要的,其有助于筛查早期可以治愈的肿瘤个体患者。胃癌作为全球第二大致死肿瘤一直备受人们的重视,更为重要的是多数患者在诊断时已经处于晚期,失去了手术治疗的最佳时机[42]。因此,如何对胃癌进行早期诊断成为临床亟待解决的问题。前期的深入研究表明,通过mRNA表达谱可以进行肿瘤的早期预测与分型评估,但LncRNA作为肿瘤生物标志物对诊断及预后的价值还有待研究。这可能是由于mRNA具有更多的生物学功能,因此可能对生物信号通路产生更大的影响。但是越来越多的研究表明,LncRNA在多种生物学功能中扮演着非常重要的角色。本文总结了可以用于胃癌的LncRNA的诊断价值,及其特异性与敏感度(表2)。
Table 2. 应用于胃癌诊断的LncRNA
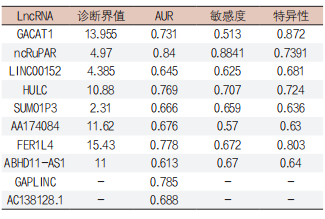
如上表所示,多个LncRNA对胃癌诊断的特异性与敏感度均超过传统的血清学肿瘤标志物,这进一步证明,LncRNA有成为新型胃癌肿瘤标志物的可行性[43]。虽然上述LncRNA的研究例数有限,且研究类型多为回顾性研究,但是随着越来越多的学者参与其中,相信大型的前瞻性研究很快就会公布,届时将有更有力的证据证明LncRNA可以作为胃癌诊断及预后的标志物。
随着研究的不断深入,多种胃癌相关的LncRNA被相继发现,其与胃癌发生发展的相关机制被不断明确,其作为胃癌诊断与预后标志物的可能性在不断增加,越来越多的学者们将LncRNA视为未来肿瘤诊断和治疗的工具之一。本文分析和论述了多种LncRNA在肿瘤尤其是胃癌中的作用,其中一些指标已经应用于临床试验,这为LncRNA真正用于临床检测及其广泛应用前景打下坚实基础。
参考文献
Hauptman, N. and D. Glavač, Long non-coding RNA in cancer. International journal of molecular sciences, 2013. 14(3): p. 4655-4669.
Rongjun, X., et al., LncRNA OIP5-AS1 facilitates gastric cancer cell growth by targeting the miR-422a/ANO1 axis. Acta Biochimica et Biophysica Sinica, 2020.
Rinn, J.L. and H.Y. Chang, Genome regulation by long noncoding RNAs. Annu Rev Biochem, 2012. 81: p. 145-66.
Jin, J., et al., Upregulation of BCAM and its sense lncRNA BAN are associated with gastric cancer metastasis and poor prognosis. Molecular Oncology, 2020.
Tang, J.Y., et al., Long noncoding RNAs-related diseases, cancers, and drugs. ScientificWorldJournal, 2013. 2013: p. 943539.
Shin, H.R., M.C. Carlos, and C. Varghese, Cancer control in the Asia Pacific region: current status and concerns. Jpn J Clin Oncol, 2012. 42(10): p. 867-81.
Kapranov, P., et al., RNA maps reveal new RNA classes and a possible function for pervasive transcription. Science, 2007. 316(5830): p. 1484-8.
Mourtada-Maarabouni, M., et al., GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer. Oncogene, 2009. 28(2): p. 195-208.
Song, H., et al., Long non-coding RNA expression profile in human gastric cancer and its clinical significances. J Transl Med, 2013. 11(1): p. 225.
Thorvaldsen, J.L., K.L. Duran, and M.S. Bartolomei, Deletion of the H19 differentially methylated domain results in loss of imprinted expression of H19 and Igf2. Genes Dev, 1998. 12(23): p. 3693-702.
Hao, Y., et al., Tumour-suppressor activity of H19 RNA. Nature, 1993. 365(6448): p. 764-7.
Yang, F., et al., Up-regulated long non-coding RNA H19 contributes to proliferation of gastric cancer cells. Febs j, 2012. 279(17): p. 3159-65.
Wang, Y., et al., Microarray expression profile analysis of long non-coding RNAs of advanced stage human gastric cardia adenocarcinoma. Mol Genet Genomics, 2014. 289(3): p. 291-302.
Zhang, E.B., et al., c-Myc-induced, long, noncoding H19 affects cell proliferation and predicts a poor prognosis in patients with gastric cancer. Med Oncol, 2014. 31(5): p. 914.
Zhang, Y., et al., LncRNA H19 induced by helicobacter pylori infection promotes gastric cancer cell growth via enhancing NF-κB-induced inflammation. Journal of Inflammation, 2019. 16(1): p. 1-8.
Liu, Y., et al., Potential Role of lncRNA H19 as a Cancer Biomarker in Human Cancers Detection and Diagnosis: A Pooled Analysis Based on 1585 Subjects. Biomed Research International, 2019. 2019: p. 1-11.
Cai, X. and B.R. Cullen, The imprinted H19 noncoding RNA is a primary microRNA precursor. Rna, 2007. 13(3): p. 313-6.
Zhuang, M., et al., The long non-coding RNA H19-derived miR-675 modulates human gastric cancer cell proliferation by targeting tumor suppressor RUNX1. Biochem Biophys Res Commun, 2014. 448(3): p. 315-22.
Matouk, I.J., et al., The oncofetal H19 RNA connection: hypoxia, p53 and cancer. Biochim Biophys Acta, 2010. 1803(4): p. 443-51.
Matouk, I.J., et al., The H19 non-coding RNA is essential for human tumor growth. PLoS One, 2007. 2(9): p. e845.
Rinn, J.L., et al., Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell, 2007. 129(7): p. 1311-23.
Woo, C.J. and R.E. Kingston, HOTAIR lifts noncoding RNAs to new levels. Cell, 2007. 129(7): p. 1257-9.
Gupta, R.A., et al., Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature, 2010. 464(7291): p. 1071-6.
Tsai, M.C., R.C. Spitale, and H.Y. Chang, Long intergenic noncoding RNAs: new links in cancer progression. Cancer Res, 2011. 71(1): p. 3-7.
Pan, W., et al., A Functional lncRNA HOTAIR Genetic Variant Contributes to Gastric Cancer Susceptibility. Mol Carcinog, 2015. 55(1).
Endo, H., et al., Enhanced expression of long non-coding RNA HOTAIR is associated with the development of gastric cancer. PLoS One, 2013. 8(10): p. e77070.
Hajjari, M., et al., Up-regulation of HOTAIR long non-coding RNA in human gastric adenocarcinoma tissues. Med Oncol, 2013. 30(3): p. 670.
Xu, Z.Y., et al., Knockdown of long non-coding RNA HOTAIR suppresses tumor invasion and reverses epithelial-mesenchymal transition in gastric cancer. Int J Biol Sci, 2013. 9(6): p. 587-97.
Kogo, R., et al., Long noncoding RNA HOTAIR regulates polycomb-dependent chromatin modification and is associated with poor prognosis in colorectal cancers. Cancer Res, 2011. 71(20): p. 6320-6.
Kim, K., et al., HOTAIR is a negative prognostic factor and exhibits pro-oncogenic activity in pancreatic cancer. Oncogene, 2013. 32(13): p. 1616-25.
Liu, W.J., et al., Expression of serum let-7c, let-7i, and let-7f microRNA with its target gene, pepsinogen C, in gastric cancer and precancerous disease. Tumour Biol, 2015. 36(5): p. 3337-43.
Cao, W.J., et al., Analysis of long non-coding RNA expression profiles in gastric cancer. World J Gastroenterol, 2013. 19(23): p. 3658-64.
Huang, S. and L. Chen, Overexpression of lncRNA LINC01793 acts as a potential predictor for progression and poor prognosis of gastric cancer. Histology & Histopathology Cellular & Molecular Biology, 2019. 34: p. págs. 233-239.
Mei, D., et al., Up-regulation of SUMO1 pseudogene 3 (SUMO1P3) in gastric cancer and its clinical association. Med Oncol, 2013. 30(4): p. 709.
Xiao, B. and J. Guo, Long noncoding RNA AC096655.1-002 has been officially named as gastric cancer-associated transcript 1, GACAT1. Tumour Biol, 2013. 34(5): p. 3271.
Park, S.M., et al., A known expressed sequence tag, BM742401, is a potent lincRNA inhibiting cancer metastasis. Exp Mol Med, 2013. 45: p. e31.
Sun, M., et al., Downregulated long noncoding RNA MEG3 is associated with poor prognosis and promotes cell proliferation in gastric cancer. Tumour Biol, 2014. 35(2): p. 1065-73.
Yang, F., et al., Long noncoding RNA CCAT1, which could be activated by c-Myc, promotes the progression of gastric carcinoma. J Cancer Res Clin Oncol, 2013. 139(3): p. 437-45.
Zhao, Y., et al., Role of long non-coding RNA HULC in cell proliferation, apoptosis and tumor metastasis of gastric cancer: a clinical and in vitro investigation. Oncol Rep, 2014. 31(1): p. 358-64.
Yang, F., et al., Long non-coding RNA GHET1 promotes gastric carcinoma cell proliferation by increasing c-Myc mRNA stability. Febs j, 2014. 281(3): p. 802-13.
Pang, Q., et al., Increased expression of long intergenic non-coding RNA LINC00152 in gastric cancer and its clinical significance. Tumor Biology, 2014. 35(6): p. 5441.
Zhang, Z.Y. and H.Y. Ge, Micrometastasis in gastric cancer. Cancer Lett, 2013. 336(1): p. 34-45.
Datta, J., et al., Poor survival after resection of early gastric cancer: extremes of survivorship analysis reveal distinct genomic profile. British Journal of Surgery, 2020. 3(129): p. 421-27.





