不同的MSI标志物组合在微卫星不稳定(MSI)检测中的应用

作者:倪崇锋 张瑶 张仑
微卫星(Microsatellite,MS)是指细胞基因组中以少数几个核苷酸(多为1~6个)为单位的串联重复DNA序列,又称短串联重复(short tandem repeat,STR)序列。微卫星不稳定(Microsatellite instable,MSI)被认为是由于DNA错配修复(Mismatch repair,MMR)功能异常,引起微卫星复制错误得不到纠正并不断积累,使微卫星序列长度及碱基组成发生变化。研究表明,MSI检测可以用于林奇综合征(Lynch Syndrome)的初筛、Ⅱ期结直肠癌预后、Ⅱ期结直肠癌辅助化疗疗效预测、晚期实体瘤免疫治疗疗效预测[1]。
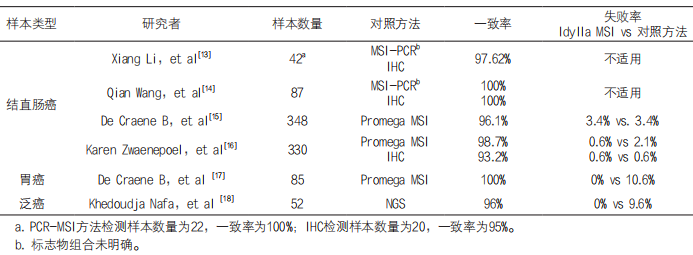
微卫星不稳定性生物标志物是用来指示微卫星状态的STR序列。1996年,美国国家癌症研究院(NCI)召开关于林奇综合征的研讨会。并于1997年制定了统一的MSI检测标准,即“Bethesda指南”(Bethesda guidelines)。本指南首次推荐BAT25、BAT26、D2S123、D5S346和D17S250标志物组合用于MSI状态的检测,并制定了微卫星高度不稳定(Microsatellite instable High,MSI-H)的判断标准,即5个标志物中有两个及以上存在突变可判断为MSI-H[2]。后续推出的MSI标志物组合均参考了Bethesda指南推荐的标志物组合和判定标准。2002年,NCI研讨会增加了一些建议,推荐对于只有一个不稳定位点的肿瘤增加额外的单核苷酸标记,因为单核苷酸重复标记在MSI-H型肿瘤诊断上更为可靠[3]。2019年,比利时的Biocartis公司推出了基于新的单核苷酸重复标志物组合的MSI产品(Idylla MSI Test,以下简称“Idylla MSI”),其MSI-H的判定标准与“Bethesda指南”相似。
一、传统MSI标志物组合
目前,临床和研究使用较多的MSI标志物是基于NCI推荐的传统标志物组合[4],表1所示的几种标志物组合均包括了BAT-25和BAT-26,这与它们在MSI-H肿瘤中总是高度不稳定有关[5]。不同标志物组合在MSI检测中的敏感性和特异性存在差异。为了提高检测的敏感性,Promega对其广泛使用的标志物组合进行了优化。2020年,Shafei Wu等使用ProDx® MSI analysis System(ProDx® MSI)、Promega MSI analysis System 1.2(以下简称Promega MSI)、基于NCI标志物组合的PCR试剂与MMR免疫组织化学(Immunohistochemistry,IHC)法对97例结直肠癌样本进行了对比研究,结果表明单核苷酸重复标志物(NR-21,NR-24,BAT-25,BAT-26,MONO-27,BAT-52,BAT-56,BAT-59和BAT-60)灵敏度高于二核苷酸重复标志物(D2S123, D5S346和D7S250)。作者还对比了不同长度的单核苷酸重复标志物,发现当标志物不稳定时,STR序列较长的单核苷酸重复标志物(BAT-52,BAT-56,BAT-59和BAT-60)序列长度的改变要大于STR序列长度较短的单核苷酸重复标志物(NR-21,NR-24,BAT-25,BAT-26和MONO-27),长度的改变增加了聚合酶链式反应(Polymerase Chain Reaction,PCR)毛细管电泳图谱中野生峰和突变峰的位置差异,利于MSI结果的判读[6]。在传统标志物组合中,BAT-25和 BAT-26是一种范围可变的近似单态(quasi-monomorphic variation range,QMVR)的单核苷酸重复标志物。其STR序列长度在不同种族人群中分布存在多态性。研究表明,在高加索人、非裔美国人、中国人群中都现存在此现象[7,8,9,10]。因此,使用该类标志物进行检测时需要正常组织作为对照。
二、Idylla MSI标志物组合
与传统的标志物组合不同,Idylla MSI所使用的标志物组合全部为单态性单核苷酸重复标记。该标志物组合筛选自Zhao等在2014年提出的59个MSI检测标志物[11],依据标志物在结直肠癌、胃癌和子宫内膜癌中的出现频率、与所用技术平台的兼容性等评价指标,最终从59个标志物中筛选确定了7个标志物:ACVR2A、BTBD7、DIDO1、MRE11、RYR3、SEC31A和SULF2。研究表明,Idylla MSI 标志物组合在非裔美国人、高加索人、东亚人、拉美裔和印度人群中保持稳定[12]。此外,不同的研究者对Idylla MSI标志物组合在多个癌种中的检测性能进行了验证,研究结果显示,Idylla MSI与对照方法的总体一致率在95%以上(见表2)。两项中国人群的研究数据显示,Idylla MSI与对照方法的总体一致率分别为97.62%和100%[13,14],说明Idylla MSI标志物组合在中国人群中的适用性。研究中还发现,Idylla MSI的扩增成功率明显高于对照方法,这可能与标志物的扩增片段长度有关(Idylla MSI扩增片段长度<90bp,Promega MSI扩增片段长度介于94-154bp之间)。
传统MSI标志物组合和Idylla MSI标志物组合均使用PCR方法进行检测,但两者在结果判读方式上存在差异。传统MSI标志物组合使用毛细管电泳法,通过比较待测样本和正常样本扩增产物片段长度差异,判断待测样本的MSI状态。而Idylla MSI结果判读基于扩增后的熔解曲线分析,直接判断待测样本的MSI状态,无需正常组织对照。不同结果判读方式与不同标志物单核苷酸重复序列长度和单态性有关。由于无需使用正常组织对照和毛细管电泳分析,相比传统的MSI-PCR方法,Idylla MSI在检测时间和操作便捷性上存在优势。Idylla MSI基于自动化的检测流程设计,减少了样本周转时间和结果判读主观差异。
表1. 基于NCI推荐标志物组合的PCR检测列表
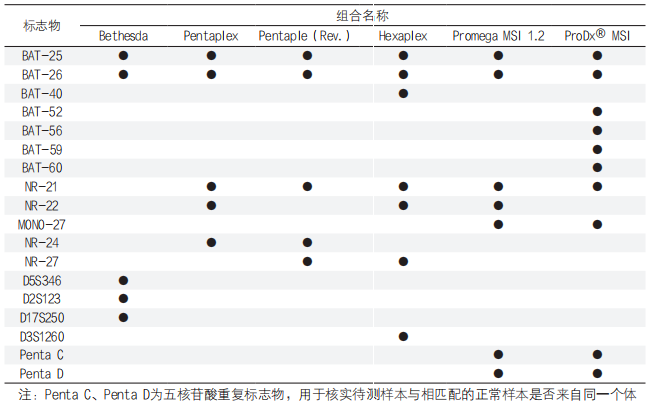
表2. 使用Idylla MSI检测盒研究不同类型的肿瘤样本
三、其他MSI标志物
传统的MSI标志物组合和Idylla MSI标志物组合均基于PCR方法。当前,MSI-PCR是MSI检测的金标准。除了MSI-PCR,基于生物学等效性评价的IHC标志物也同样可用于MSI检测。与MSI-PCR标志物组合不同,IHC使用MLH1、MSH2、MSH6和PMS2蛋白作为检测标志物,该组合直接检测MMR的功能是否存在缺陷,从而间接判断MSI的状态。但两者存在一定差异,在子宫内膜癌中为13%,其他癌种中为<4%[20]。一方面,这是由于并非所有的MMR突变都会导致抗原性的丧失[21]。另一方面,MSI检测方法所选择的有限标记物,具有肿瘤特异性[22],无法准确反应全部肿瘤中的MSI状态。因此将两种方法结合起来,可以避免因方法学的差异而造成的漏检。
目前NGS也逐渐应用于MSI检测,与MSI-PCR相比,NGS的检测标志物数量更多,覆盖范围更广,能够提高其在非结直肠癌中MSI检测的敏感性。NGS检测结果准确性除与其选择的标志物组合和数量有关,也与其设计的算法有关。研究结果显示,基于mSINGS 的算法,对91个前列腺癌样本的146个单核苷酸位点进行评估,其检测灵敏度为93.1%,高于传统MSI-PCR[23]。另一项包含12,288个泛癌种实体瘤的大样本研究提示,基于MSIsensor算法,MSK-IMPACT组合测试表现良好,在一些非结直肠癌/子宫内膜癌中,比传统MSI-PCR检测敏感度更高[24]。但同时,NGS MSI检测在进行代表位点筛选、算法设计和优化、收集验证所需的样本量、重复序列捕获和测序上也存在一定的挑战。
2016年,欧洲临床肿瘤协会指出MSI检测可用于转移性结直肠癌患者免疫检查点抑制剂治疗疗效预测[25]。进一步研究展示,MSI可以作为多种实体瘤免疫治疗受益的关键标志物[26]。随着国内免疫检查点抑制剂药物适应症的陆续获批,MSI检测可以使更多患者获益。
参考文献
袁瑛. 结直肠癌及其他相关实体瘤微卫星不稳定性检测中国专家共识[J]. 实用肿瘤杂志, 2019(5): 381-389.
Van Lier MG, Leenen CH, Wagner A, et al. Yield of routine molecular analyses in colorectal cancer patients ≤70 years to detect underlying Lynch syndrome[J]. Journal of Pathology 2012;226:764-74.
Umar A, Boland CR, Terdiman JP, et al. Revised Bethesda Guidelines for hereditary nonpolyposis colorectal cancer (Lynch syndrome) and microsatellite instability. J Natl Cancer Inst. 2004;96:261-268.
Buecher B, Cacheux W, Rouleau E, et al. Role of microsatellite instability in the management of colorectal cancers[J]. Digestive and Liver Disease, 2013, 45(6): 441-449.
Duval, A. Evolution of instability at coding and non-coding repeat sequences in human MSI-H colorectal cancers[J]. Human Molecular Genetics, 2001, 10(5): 513-518.
Wu S, Liu X, Wang J, et al. DNA Mismatch Repair Deficiency Detection in Colorectal Cancer by a New Microsatellite Instability Analysis System[J]. Interdisciplinary Sciences Computational Life Sciences, 2020(7).
Hoang JM, et al Hamelin R. BAT-26, an indicator of the replication error phenotype in colorectal cancers and cell lines [J]. Cancer Res 1997; 57: 300-303.
Zhou XP, et al. Determination of the replication error phenotype in human tumors without the requirement for matching normal DNA by analysis of mononucleotide repeat microsatellites[J]. Genes Chromosomes Cancer 1998; 21:101-107.
Pyatt R, Chadwick RB, Johnson CK, Adebamowo C, de la Chapelle A, Prior TW. Polymorphic variation at the BAT-25 and BAT-26 loci in individuals of African origin: implications for microsatellite instability testing[J]. Am J Pathol 1999;155:349-353.
Zheng Y, Liu L, Sun Y, et al. Comparative Polymorphism of BAT-26 between Healthy Individuals and Cancer Patients and Its Cancer Risk Implication for Local Chinese[J]. The International Journal of Biological Markers, 2016, 31(3):252-257.
Zhao H, et al. Mismatch repair deficiency endows tumors with a unique mutation signature and sensitivity to DNA double-strand breaks [J]. Elife. 2014 Aug 1;3:e02725.
Maertens G, et al. Detection of microsatellite instability (MSI) in colorectal cancer samples with the automated Idylla MSI Test. ESMO 2017 #138P.
Xiang Li, et al. Evaluation of a Fully Automated Idylla Test System for Microsatellite Instability in Colorectal Cancer. [J].Clin Colorectal Cancer. 2019 Dec;18(4):e316-e323
Qian Wang, et al. Validation of the fully automated idylla system for microsatellite instability detection in Chinese colorectal cancer patients. ASCO 2020 e13644.
De Craene B, et al. Detection of microsatellite instability (MSI) in colorectal cancer samples with a novel set of highly sensitive markers by means of the Idylla MSI Test prototype. ASCO 2018 #e15639;
Karen Zwaenepoel, et al. Clinical Performance of the Idylla MSI Test for a Rapid Assessment of the DNA Microsatellite Status in Human Colorectal Cancer[J]. The Journal of Molecular Diagnostics Volume 22, Issue 3, March 2020, Pages 386-395.
De Craene B, et al. Detection of microsatellite instability (MSI) with a novel panel of biomarkers in gastric cancer samples. ESMO 2017 #697P.
Khedoudja Nafa, et al. Rapid Assessment of Microsatellite Instability Status using the Idylla MSI Test AMP 2018 ST119.
Zhang, Liying. Immunohistochemistry versus Microsatellite Instability Testing for Screening Colorectal Cancer Patients at Risk for Hereditary Nonpolyposis Colorectal Cancer Syndrome: Part II. The Utility of Microsatellite Instability Testing[J]. Journal of Molecular Diagnostics, 2008, 10(4):301-307.
Trabucco S E, Gowen K, Maund S L, et al. A Novel Next-Generation Sequencing Approach to Detecting Microsatellite Instability (MSI) and Pan-Tumor Characterization of One Thousand MSI-High Cases in 67,000 Patient Samples[J]. Journal of Molecular Diagnostics, 2019, 21(6).
Lindor NM, Burgart LJ, Leontovich O, et al. Immunohistochemistry versus microsatellite instability testing in phenotyping colorectal tumors[J]. Journal of Clinical Oncology Official Journal of the American Society of Clinical Oncology, 2002, 20(4):1043.
Rowena D. Faulkner, et al. BAT-25 and BAT-26, two mononucleotide microsatellites, are not sensitive markers of microsatellite instability in acute myeloid leukaemia[J]. British Journal of Haematology, 2004.
Hempelmann J A, Lockwood C M, Konnick E Q, et al. Microsatellite instability in prostate cancer by PCR or next-generation sequencing[J]. Journal for ImmunoTherapy of Cancer, 2018, 6(1):29.
Middha S, Zhang L, Nafa K, et al. Reliable Pan-Cancer Microsatellite Instability Assessment by Using Targeted Next-Generation Sequencing Data[J]. JCO Precision Oncology, 2017(1):1-17.
ESMO consensus guidelines for the management of patients with metastatic colorectal cancer[J]. Annals of Oncology, 2016, 27(8):mdw235.
Le DT, et al. Mismatch repair deficiency predicts response of solid tumors to PD-1 blockade[J]. ence, 2017, 357(6349):409-413.





