线粒体检测技术在衰老及衰老相关疾病中的应用前景

作者:孙宇哲 宋志杰 聂超
【摘要】人类衰老的生物学基础仍然是最大的尚未解决的科学问题之一。然而,越来越多的证据表明,线粒体基因组突变积累和功能的减弱是衰老的重要因素和表现。本文,我们主要讨论了线粒体在衰老中扮演的角色,线粒体与衰老相关疾病的关系,以及线粒体检测的应用。
【关键词】衰老,线粒体,功能异常,线粒体检测
一、线粒体与衰老
衰老是以一系列生物变化累积为特征导致生物有机体随时间而功能退化的普遍过程。细胞衰老是个体衰老和老年病发病的基础,与个体衰老关系错综复杂,是机体生长发育中的正常现象。细胞衰老的机制有基因学说、ROS学说、端粒钟学说等。
在20世纪20年代,Raymond Pearl指出,人体的代谢率似乎与寿命成反比[1]。在此基础上,Denham Harman提出了衰老的自由基理论[2]。他认为来自线粒体的活性氧(ROS)的产生和积累,导致随后蛋白质、脂质和DNA的受损,随之而来的是一个不可避免的过程:细胞凋亡和人体衰老,并且提高年龄相关疾病的患病概率[2]。线粒体通过氧化还原作用产生ATP,供给细胞活动所需的能量,并调节细胞的新陈代谢。同时,还生产出了副产物ROS,ROS过度积累会导致细胞凋亡。在衰老进程中,线粒体动力学指标持续下降,从而引发线粒体功能的减退;而被认为与衰老密切相关的ROS的不断累积,会造成线粒体DNA、蛋白质和脂质的损伤,线粒体基因组(mtDNA)突变不断累积,而线粒体DNA却倾向于复制带有缺失的线粒体DNA片段,造成线粒体氧化磷酸化系统功能下降,继而加剧ROS生成,形成恶性循环。正常的细胞表达模式与凋亡都会随之变化,氧化应激压力也会加大,造成DNA、蛋白和质膜损伤,导致细胞衰老加速[3]。
线粒体功能和稳态的打破也会引发神经系统、心脏和肌肉的疾病[4]。一般认为,细胞衰老与老年性疾病如神经退行性疾病、心脑血管疾病、糖尿病及肿瘤等关联紧密,而线粒体则通过调控细胞衰老而影响衰老相关疾病[5,6]。
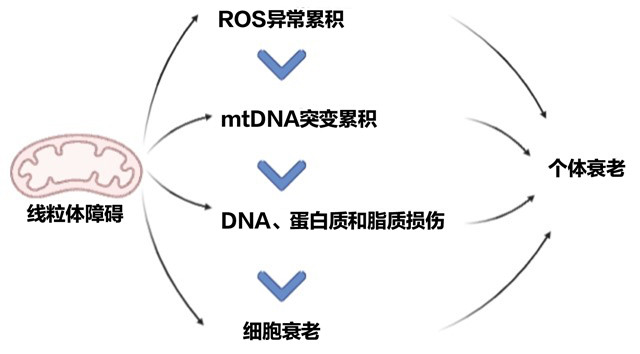
图1. 线粒体与衰老
二、线粒体疾病
虽然人类mtDNA只编码13种蛋白质,约相当于线粒体活动所需蛋白质总数的1%[4,7],但是这些蛋白在ATP合成和电子传递链中起着非常重要的作用[8]。由于遗传缺损导致线粒体代谢酶缺陷,致使ATP合成障碍、能量来源不足导致的异质性病变叫做线粒体病。线粒体基因组的缺失或者突变使得氧化代谢过程必需的酶或载体功能发生障碍,导致能量代谢紊乱、氧自由基不能及时被线粒体当中的超氧化物歧化酶清除,同时mtDNA匮乏修复机制使得上述mtDNA变异得已累积看,导致细胞能量代谢障碍,产生复杂的临床症状。
由于肌肉、心脏和大脑等组织需要线粒体提供相对大量的ATP,对能量敏感,易受线粒体问题所累,因此线粒体病的症状较多地表现为这些组织的异常病变,体现出较大异质性。根据受累组织器官的不同,线粒体疾病也可主要分为线粒体肌病、线粒体脑病、线粒体脑肌病三类[9-11]。中枢神经系统的线粒体病被称为线粒体脑病,常表现为脑卒中、癫痫、偏头痛、共济失调、智力障碍等。根据中国神经系统线粒体病的诊治指南,线粒体脑病又可细分为弥漫性进行性脑灰质变性综合征、弥漫性进行性脑灰质变性综合征、弥漫性进行性脑灰质变性综合征[11]。以骨骼肌为特征的线粒体病被称为线粒体肌病,临床表现为四肢无力和运动耐受力下降、肌肉绞痛、肌红蛋白尿症[11,12]。线粒体脑肌病则累及中枢神经系统和肌肉组织等多个系统和器官,基于多系统线粒体功能障碍,其主要伴发乳酸中毒和卒中样发作[11,13]。
三、线粒体与神经系统退行性疾病
阿尔茨海默病(Alzheimer's Disease,AD),帕金森病(Parkinson's Disease,PD)和肌萎缩性侧索硬化症(Amyotrophic Lateral Sclerosis,ALS)是最常见的神经退行性疾病。主要特征进行性功能障碍和选择性神经元群的丧失。为何会发生选择性神经元丢失仍然是尚未解决的问题,同时也是这一领域中关注的焦点,线粒体功能障碍作为一种常见的病理特征出现,被认为是研究神经退行性变机理的热点。
线粒体及其代谢过程的影响最终导致ROS/RNS(活性氮)水平升高,导致线粒体蛋白质,脂质和DNA氧化。氧化应激被认为与许多神经退行性疾病有关,例如阿尔茨海默氏病,肌萎缩性侧索硬化,亨廷顿病(Huntington’s Disease),多发性硬化症(Multiple sclerosis)和帕金森病。此外,引起蛋白质错误折叠的氧化应激可能会导致其他神经退行性疾病,包括牛海绵状脑病(疯牛病)和致死性家族失眠症(Fatal Familial Insomnia)[14]。
1. 线粒体与阿尔茨海默症:阿尔茨海默症是痴呆症的最常见形式,也是影响范围最广,人数最多的神经退行性疾病,其特征是选择性神经元死亡和两个病理学特征:即由淀粉样β(Aβ)肽的细胞外沉积形成的老年斑和tau蛋白的细胞内神经原纤维缠结(NFTs)[15]。
(1)阿尔茨海默症与线粒体DNA:2004年,Swerdlow和Khan等人提出AD的线粒体级联假说[16,17],后期随着后续研究的补充,得到了其主要内容是:一个人从母系遗传来的mtDNA决定了线粒体的基本功能,随着年龄的增长和暴露于各种环境下,线粒体基因组突变大量积累,线粒体异质性提高,线粒体功能下降则导致AD特异性病理。这个假说提出了线粒体功能老化阈值的概念,同时并不是所有mtDNA突变都能导致AD病情,事实上,大部分的mtDNA突变都是无害的“中性突变”。级联假说解释了散发性AD与年龄的显著关系,以及AD的母系遗传倾向,强调了“线粒体衰老”在AD中的作用,然而并没有清楚地解释线粒体功能障碍是如何导致Aβ和NFT积累。
(2)阿尔茨海默症与氧化应激:有大量证据表明了AD患者的神经元中线粒体的功能障碍:AD中的线粒体功能下降,线粒体编码的基因表达变化,ROS大量释放,导致神经元脂质和蛋白质的广泛氧化损伤,由于线粒体自身也容易受到氧化应激的影响,涉及线粒体功能障碍和氧化应激之间的相互作用的恶性循环下,糖代谢水平降低、线粒体酶减少和ROS增多,直接影响了脑细胞的能量代谢,最终导致神经元细胞死亡[18-20]。
AD病人神经元的氧化损伤水平大大超过了正常老年人,结构和功能受损的线粒体就是ROS的来源,同时线粒体功能障碍导致ATP生产减少,细胞生理生化过程功能不足,ROS的积累对于正常细胞来说是致命性的,ROS的大幅度升高可以破坏几乎所有的细胞内活动,对神经细胞有永久性的损害,随着ROS积累和释放,大规模的神经元细胞死亡,导致病人认知进行性的下降。
2. 线粒体与帕金森氏病:帕金森氏病较多晚年发病,是继阿尔茨海默氏病之后最常见的运动障碍和第二大最常见的神经退行性疾病,其主要特征是黑质致密部中多巴胺能神经元的缺失,导致纹状体中的多巴胺缺乏。60岁以上的人口中约有2%受此病影响。其临床症状主要包括运动迟缓、静止性震颤、驼背、运动失调、步态异常、易跌倒。除了典型的运动症状外,还会出现各种非运动功能,例如植物神经功能障碍,睡眠障碍,抑郁和认知障碍,这表明变性过程更加普遍[21]。PD涉及生物能缺陷,线粒体DNA突变,核DNA基因突变,线粒体动力学变化,运输/运输和线粒体运动变化,异常大小和形态,转录障碍以及与线粒体相关的突变蛋白的存在[22]。
(1)PARK2(Parkin)和PARK6(PINK1):1997年,一项遗传连锁分析显示,染色体6q25.2-27带有一个未知基因,该基因导致了13个日本家庭的隐性早发型帕金森病(AR-JP)[23]。一年后,清水研究小组克隆了并确定了基因PARK2(Parkin)。2001年,在意大利人群的研究中发现了1号染色体的PARK6基因,这些携带PARK6突变患者的临床症状与散发型PD患者相同[24]。
尽管PINK1定位于线粒体并且Parkin定位于细胞质中,但在研究中发现,两种蛋白都以相同的途径起作用[25]。然后,细胞生物学研究表明,PINK1积聚在功能失调线粒体的表面,PINK1检测线粒体功能障碍,然后发出信号提示Parkin泛素化特定的受损线粒体,并通过自噬促进其清除[26,27]。这表明PINK1和Parkin共同作用构成了线粒体质量控制体系。线粒体质量控制是PINK1和Parkin的必不可少的功能,进一步有助于支持人类帕金森病的预防。
(2)帕金森病中损伤线粒体的清除:呼吸链损伤也是散发性PD患者的关键特征,研究发现除了Parkin和PINK,多个核基因编码的蛋白质与线粒体功能障碍有关[28]。家族遗传的PD中, α-synuclein, DJ-1, UCHL-1, LRRK2, NURR1, VPS35和HtrA2等基因的突变会直接或间接导致致病性的线粒体功能障碍[22]。同时,环境中的百草枯,薄荷脑和鱼藤酮等杀虫剂也会增加PD的患病风险。
及时清理损伤线粒体对机体尤其是大脑至关重要,清除平衡的打破被认为是PD发病的重要原因。健康的神经元通过自噬来清除受损的线粒体,这是确保细胞存活的重要机制。过量的线粒体损害(来自百草枯、薄荷脑和鱼藤酮暴露,和衰老引起的功能障碍等)会使神经元死亡,从而导致PD发病。同样,降低细胞清除受损线粒体的能力(比如失去了Parkin或PINK1)也会使这些功能障碍的线粒体积聚,从而导致早发PD[27]。
PD的发病机制与线粒体功能障碍、线粒体清除和氧化应激密切相关。针对PD中线粒体的治疗方法包括:抗氧化疗法、补充肌酸、改善α-突触核蛋白、增强线粒体、调节钙稳态和基因治疗等方法。
3. 线粒体与肌萎缩测索硬化症:肌萎缩测索硬化症(ALS)是一种运动神经元疾病。患者运动神经元功能丧失或死亡,导致肌肉细胞功能减弱萎缩[29]。早期症状轻微,患者可能只是感到些许无力感、肩关节变得突出,容易发生半脱位、并且有分裂手现象,后期肌无力可能进展为呼吸衰竭[29]。
具体发病机制涉及神经毒性物质累积、自由基产生、线粒体异常[29]。遗传研究发现,铜/锌超氧化物歧化酶(SOD1)突变与ALS紧密相关,突变的SOD1产生细胞毒作用,引起线粒体功能失调[30],SOD1突变造成细胞内部各种生理功能障碍,如胞内氧化应激产生大量ROS,轴突转运障碍,RNA代谢失衡,线粒体功能失调[31-33]。利鲁唑片(Riluzole)可以通过作用前突触神经元Na离子通道减少谷氨酸释放来对抗谷氨酸兴奋性毒性,清除自由基,改善线粒体功能。2017年,FDA批准依达拉奉(Edaravone)作为ALS静脉输注疗法。依达拉奉能减少氧化应激,清除ROS,防止过度氧化,从而抑制细胞的氧化损伤,减少SOD1沉积。此外,熊去氧胆酸(Ursodeoxycholic Acid)可能阻碍神经元细胞凋亡途径,目前处在ALS治疗的临床试验阶段。细胞治疗的主要策略是利用病人体细胞生成IPS细胞再诱导分化为运动神经元和胶质细胞,然后输回病人体内以使其重新恢复运动功能[34]。
四、线粒体的检测技术
由于线粒体以及ROS在生理和病理过程中都起着重要作用。不同的ROS,例如超氧化物(O 2 •-),过氧化氢和过氧亚硝酸盐(ONOO •-),会刺激不同的细胞信号通路,并根据其数量和亚细胞定位而导致不同的结果[35]。现在已经开发出了多种用于检测线粒体和ROS的方法,这不仅有助于线粒体相关的科学研究也有助于临床诊断线粒体疾病。目前对线粒体的检测方法有线粒体基因组测序、呼吸控制法、线粒体ATP生成率、膜电位等方法,此外,通过电子显微镜还可以直接观察线粒体的形态、数量变化。
1. 线粒体基因组检测技术:传统的线粒体测序是基于Sanger法测序,利用线粒体特有的序列进行PCR,然后进行Sanger法测序,得到序列后与标准序列比较,这种方法由于通量低价格也相对较低,适用于线粒体遗传病的筛查[36]。但是,由于一个细胞中线粒体数量庞大,这种方法无法精确检查线粒体突变的情况,于是引入了高通量测序(NGS)。高通量测序灵敏度高,非常适合检测线粒体的异质性、多态性,识别导致线粒体疾病的点突变、缺失突变、插入突变、倒位突变、重复突变等基因结构的改变。所以线粒体异常疾病的直接诊断策略为高通量测序技术直接检出致病基因的突变[37]。
2. 线粒体氧化呼吸链检测:此项检测技术主要有传统的线粒体功能检测、用Seahorse检测线粒体功能和细胞代谢和线粒体基因组衰老水平的检测技术等。
(1)传统的线粒体功能检测:线粒体呼吸控制率(RCR)、ATP生成率和膜电位的检测都是通过电化学的方法,以评价线粒体结构和功能完整性以及氧化磷酸化效率的权威而经典的技术方法。RCR检测既可以反映线粒体的完整程度,也可以反应氧化磷酸化效率,RCR等于由ADP磷酸化时的耗氧量除以ADP用完后的耗氧量。一般需要提前分离完整的线粒体,在密闭环境下利用氧电极法检测。正常线粒体的RCR为3至10:RCR降低意味着线粒体ATP合成功能损伤,呼吸障碍;RCR增高意味着细胞活动旺盛,代谢加快。线粒体ATP生成率(MAPR)检测也是评价线粒体完整性和功能的重要指标。此方法是利用重组荧光素酶与ATP反应后根据激发荧光来检测ATP的生成率。膜电位的检测利用线粒体JC-1试剂盒,通过JC-1在线粒体内聚集(红色荧光)或散布在buffer中并未聚集(绿色荧光)来显示线粒体的膜电位变化,也可以直接通过膜片钳技术,实时的记录细胞膜上单一的或者多个离子通道活动[38]。
(2)用Seahorse检测线粒体功能和细胞代谢:Seahorse细胞能量代谢分析仪是检测线粒体功能和细胞代谢的有力工具,可以实时且不需要任何染料或标记物的非侵入性方式测量线粒体呼吸和糖酵解。Seahorse检测时将细胞培养在专用的微孔板上,实时检测加入不同药物后线粒体功能与糖酵解的关键参数:基础呼吸,ATP生成,质子漏,最大呼吸值,能量储备能力,糖酵解能力,糖酵解能量储备能力。仪器系统会发射激光,并测量激发荧光的变化。瞬态微腔所形成的很小的体积允许实时、灵敏、精确且无损地测量参数。自动计算氧气浓度和pH的变化,并以耗氧率(OCR)和细胞外酸化率(ECAR)为结果。
3. 线粒体基因组衰老水平的检测技术:除了线粒体直接导致的疾病,线粒体的基因组、生理生化水平也会随着年龄的增长发生变化。通常认为与年龄相关的体细胞mtDNA突变是由于损伤而产生的,这种损伤和突变在衰老过程中逐渐积累,并伴随着线粒体形态变化的出现。这一理论得到了大量相关数据的支持,这些数据显示,老年人中的体细胞mtDNA突变水平高于年轻人[36]。
人的体细胞通常包含数千个mtDNA拷贝,mtDNA的复制与细胞周期无关,因此特定的mtDNA分子可以在细胞分裂时复制很多次。此外,单个细胞经常包含一种以上基因型的mtDNA,这种情况称为异质性。在致病性突变的异质性情况下,突变的mtDNA的比例将决定是否发生线粒体功能障碍。突变的mtDNA损害功能时的阈值水平通常很高,但也取决于突变的类型和位置[39]。
体细胞线粒体DNA突变水平的测量技术上比较复杂,尚无对于单个非扩增mtDNA进行测序的标准方法,因此所有可用的方案均涉及PCR和/或克隆。因此,新技术的应用主要还是通过PCR扩增,然后再针对产物进行检测或者测序。针对mtDNA大片段缺失,可以通过定量PCR的方法,如果存在突变量高于总量的5%,则可以通过Southern blot来检测。针对mtDNA全局点突变的检测,主要是通过高通量测序得到大量的序列数据,通过生物信息算法去除PCR错误和测序错误,最后得到线粒体的全局点突变,并以此来计算线粒体异质性[36,39]。由于线粒体异质性与衰老以及遗传选择性有密切关系,以此来计算个体或组织的衰老程度。
4. 线粒体健康指数的评估:2014年,Balu Chacko等提出了生物能源健康指数(BHI)的概念,以检测血液白细胞的线粒体功能为核心,以综合评价目标个体的生物能功能障碍[40]。BHI指数转化生物学的重要结果。2018年,新的研究简化了指数检测的内容,整合了核DNA和线粒体DNA编码的呼吸链酶活性和线粒体DNA拷贝数,提出了线粒体健康指数(MHI)[41]。该方法定量检测了柠檬酸合酶(CS),细胞色素C氧化酶(COX,complex IV)和琥珀酸脱氢酶(SDH,complex II)的酶活性, 并进行了mtDNA和核DNA计数以归一化单个细胞中的线粒体数目。这项研究指出MHI可以反映出人的日常情绪和压力状态[41]。
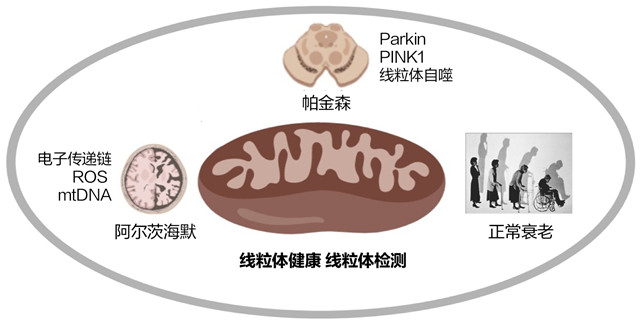
图2. 线粒体健康
五、展望
我们已经了解到,所有人类至少都具有一定程度的mtDNA异质性,从而导致与年龄相关的线粒体稳态的降低和受损线粒体比例增加。目前,许多研究正在尝试添加小分子药物以增强线粒体生物活性:提高老年小鼠的NAD+水平似乎可以恢复线粒体功能[42];基于PINK1和Parkin的分子机制,直接或间接地调节PINK1或许可以提高线粒体自噬,降低异质性[43]。来自人类和模式动物的证据完全支持mtDNA突变在衰老中的负面作用,减少mtDNA突变,降低线粒体异质性是否可以延长寿命并没有明确的答案,需要更多的研究才能清楚地理解mtDNA突变与衰老之间的联系。
综上所述,这些观察结果表明线粒体可以与多种与衰老相关的过程密切相关,包括mtDNA突变积累,ATP消耗下降、ROS水平上升、线粒体形态变化等,个体层面包括组织和器官功能的普遍的下降。由于线粒体与衰老的密切关系,使得线粒体代谢和DNA突变积累成为长寿研究的重要方向。随着测序和实验成本的不断降低,许多基于线粒体检测和线粒体靶向治疗的研究,为修补人类衰老和延长寿命提供了全新的思路。
参考文献
Speakman, J.R. Body size, energy metabolism and lifespan. J Exp Biol 2005, 208, 1717-1730, doi:10.1242/jeb.01556.
Harman, D. Aging: a theory based on free radical and radiation chemistry. J Gerontol 1956, 11, 298-300, doi:10.1093/geronj/11.3.298.
Sas, K.; Szabo, E.; Vecsei, L. Mitochondria, Oxidative Stress and the Kynurenine System, with a Focus on Ageing and Neuroprotection. Molecules 2018, 23, doi:10.3390/molecules23010191.
Sokol, A.M.; Sztolsztener, M.E.; Wasilewski, M.; Heinz, E.; Chacinska, A. Mitochondrial protein translocases for survival and wellbeing. FEBS Lett 2014, 588, 2484-2495, doi:10.1016/j.febslet.2014.05.028.
Calcinotto, A.; Kohli, J.; Zagato, E.; Pellegrini, L.; Demaria, M.; Alimonti, A. Cellular Senescence: Aging, Cancer, and Injury. Physiol Rev 2019, 99, 1047-1078, doi:10.1152/physrev.00020.2018.
Campisi, J.; Robert, L. Cell senescence: role in aging and age-related diseases. Interdiscip Top Gerontol 2014, 39, 45-61, doi:10.1159/000358899.
Becker, T.; Song, J.; Pfanner, N. Versatility of Preprotein Transfer from the Cytosol to Mitochondria. Trends Cell Biol 2019, 29, 534-548, doi:10.1016/j.tcb.2019.03.007.
Nunnari, J.; Suomalainen, A. Mitochondria: in sickness and in health. Cell 2012, 148, 1145-1159, doi:10.1016/j.cell.2012.02.035.
Craven, L.; Alston, C.L.; Taylor, R.W.; Turnbull, D.M. Recent Advances in Mitochondrial Disease. Annu Rev Genomics Hum Genet 2017, 18, 257-275, doi:10.1146/annurev-genom-091416-035426.
Alston, C.L.; Rocha, M.C.; Lax, N.Z.; Turnbull, D.M.; Taylor, R.W. The genetics and pathology of mitochondrial disease. J Pathol 2017, 241, 236-250, doi:10.1002/path.4809.
Chinnery, P.F. Mitochondrial Disorders Overview. In GeneReviews((R)), Adam, M.P., Ardinger, H.H., Pagon, R.A., Wallace, S.E., Bean, L.J.H., Stephens, K., Amemiya, A., Eds. Seattle (WA), 1993.
Dalakas, M.C.; Illa, I.; Pezeshkpour, G.H.; Laukaitis, J.P.; Cohen, B.; Griffin, J.L. Mitochondrial myopathy caused by long-term zidovudine therapy. N Engl J Med 1990, 322, 1098-1105, doi:10.1056/NEJM199004193221602.
Majamaa, K.; Moilanen, J.S.; Uimonen, S.; Remes, A.M.; Salmela, P.I.; Karppa, M.; Majamaa-Voltti, K.A.; Rusanen, H.; Sorri, M.; Peuhkurinen, K.J., et al. Epidemiology of A3243G, the mutation for mitochondrial encephalomyopathy, lactic acidosis, and strokelike episodes: prevalence of the mutation in an adult population. Am J Hum Genet 1998, 63, 447-454, doi:10.1086/301959.
Islam, M.T. Oxidative stress and mitochondrial dysfunction-linked neurodegenerative disorders. Neurol Res 2017, 39, 73-82, doi:10.1080/01616412.2016.1251711.
Smith, M.A. Alzheimer disease. Int Rev Neurobiol 1998, 42, 1-54, doi:10.1016/s0074-7742(08)60607-8.
Swerdlow, R.H.; Burns, J.M.; Khan, S.M. The Alzheimer's disease mitochondrial cascade hypothesis. J Alzheimers Dis 2010, 20 Suppl 2, S265-279, doi:10.3233/JAD-2010-100339.
Swerdlow, R.H.; Khan, S.M. A "mitochondrial cascade hypothesis" for sporadic Alzheimer's disease. Med Hypotheses 2004, 63, 8-20, doi:10.1016/j.mehy.2003.12.045.
Albensi, B.C. Dysfunction of mitochondria: Implications for Alzheimer's disease. Int Rev Neurobiol 2019, 145, 13-27, doi:10.1016/bs.irn.2019.03.001.
Schapira, A.H. Mitochondrial diseases. Lancet 2012, 379, 1825-1834, doi:10.1016/S0140-6736(11)61305-6.
Cheignon, C.; Tomas, M.; Bonnefont-Rousselot, D.; Faller, P.; Hureau, C.; Collin, F. Oxidative stress and the amyloid beta peptide in Alzheimer's disease. Redox Biol 2018, 14, 450-464, doi:10.1016/j.redox.2017.10.014.
Spillantini, M.G.; Schmidt, M.L.; Lee, V.M.; Trojanowski, J.Q.; Jakes, R.; Goedert, M. Alpha-synuclein in Lewy bodies. Nature 1997, 388, 839-840, doi:10.1038/42166.
Bose, A.; Beal, M.F. Mitochondrial dysfunction in Parkinson's disease. J Neurochem 2016, 139 Suppl 1, 216-231, doi:10.1111/jnc.13731.
Matsumine, H.; Saito, M.; Shimoda-Matsubayashi, S.; Tanaka, H.; Ishikawa, A.; Nakagawa-Hattori, Y.; Yokochi, M.; Kobayashi, T.; Igarashi, S.; Takano, H., et al. Localization of a gene for an autosomal recessive form of juvenile Parkinsonism to chromosome 6q25.2-27. Am J Hum Genet 1997, 60, 588-596.
Valente, E.M.; Bentivoglio, A.R.; Dixon, P.H.; Ferraris, A.; Ialongo, T.; Frontali, M.; Albanese, A.; Wood, N.W. Localization of a novel locus for autosomal recessive early-onset parkinsonism, PARK6, on human chromosome 1p35-p36. Am J Hum Genet 2001, 68, 895-900, doi:10.1086/319522.
Clark, I.E.; Dodson, M.W.; Jiang, C.; Cao, J.H.; Huh, J.R.; Seol, J.H.; Yoo, S.J.; Hay, B.A.; Guo, M. Drosophila pink1 is required for mitochondrial function and interacts genetically with parkin. Nature 2006, 441, 1162-1166, doi:10.1038/nature04779.
Matsuda, N.; Sato, S.; Shiba, K.; Okatsu, K.; Saisho, K.; Gautier, C.A.; Sou, Y.S.; Saiki, S.; Kawajiri, S.; Sato, F., et al. PINK1 stabilized by mitochondrial depolarization recruits Parkin to damaged mitochondria and activates latent Parkin for mitophagy. J Cell Biol 2010, 189, 211-221, doi:10.1083/jcb.200910140.
Pickrell, A.M.; Youle, R.J. The roles of PINK1, parkin, and mitochondrial fidelity in Parkinson's disease. Neuron 2015, 85, 257-273, doi:10.1016/j.neuron.2014.12.007.
Rocha, E.M.; De Miranda, B.; Sanders, L.H. Alpha-synuclein: Pathology, mitochondrial dysfunction and neuroinflammation in Parkinson's disease. Neurobiol Dis 2018, 109, 249-257, doi:10.1016/j.nbd.2017.04.004.
Swerdlow, R.H.; Parks, J.K.; Cassarino, D.S.; Trimmer, P.A.; Miller, S.W.; Maguire, D.J.; Sheehan, J.P.; Maguire, R.S.; Pattee, G.; Juel, V.C., et al. Mitochondria in sporadic amyotrophic lateral sclerosis. Exp Neurol 1998, 153, 135-142, doi:10.1006/exnr.1998.6866.
Pasinelli, P.; Belford, M.E.; Lennon, N.; Bacskai, B.J.; Hyman, B.T.; Trotti, D.; Brown, R.H., Jr. Amyotrophic lateral sclerosis-associated SOD1 mutant proteins bind and aggregate with Bcl-2 in spinal cord mitochondria. Neuron 2004, 43, 19-30, doi:10.1016/j.neuron.2004.06.021.
Trotti, D.; Rolfs, A.; Danbolt, N.C.; Brown, R.H., Jr.; Hediger, M.A. SOD1 mutants linked to amyotrophic lateral sclerosis selectively inactivate a glial glutamate transporter. Nat Neurosci 1999, 2, 848, doi:10.1038/12227.
Howland, D.S.; Liu, J.; She, Y.; Goad, B.; Maragakis, N.J.; Kim, B.; Erickson, J.; Kulik, J.; DeVito, L.; Psaltis, G., et al. Focal loss of the glutamate transporter EAAT2 in a transgenic rat model of SOD1 mutant-mediated amyotrophic lateral sclerosis (ALS). Proc Natl Acad Sci U S A 2002, 99, 1604-1609, doi:10.1073/pnas.032539299.
Miller, T.M.; Pestronk, A.; David, W.; Rothstein, J.; Simpson, E.; Appel, S.H.; Andres, P.L.; Mahoney, K.; Allred, P.; Alexander, K., et al. An antisense oligonucleotide against SOD1 delivered intrathecally for patients with SOD1 familial amyotrophic lateral sclerosis: a phase 1, randomised, first-in-man study. Lancet Neurol 2013, 12, 435-442, doi:10.1016/S1474-4422(13)70061-9.
Jaiswal, M.K. Riluzole and edaravone: A tale of two amyotrophic lateral sclerosis drugs. Med Res Rev 2019, 39, 733-748, doi:10.1002/med.21528.
Papa, S.; Skulachev, V.P. Reactive oxygen species, mitochondria, apoptosis and aging. Mol Cell Biochem 1997, 174, 305-319.
Larsson, N.G. Somatic mitochondrial DNA mutations in mammalian aging. Annu Rev Biochem 2010, 79, 683-706, doi:10.1146/annurev-biochem-060408-093701.
Sharma, P.; Sampath, H. Mitochondrial DNA Integrity: Role in Health and Disease. Cells 2019, 8, doi:10.3390/cells8020100.
Dikalov, S.I.; Harrison, D.G. Methods for detection of mitochondrial and cellular reactive oxygen species. Antioxid Redox Signal 2014, 20, 372-382, doi:10.1089/ars.2012.4886.
Sun, N.; Youle, R.J.; Finkel, T. The Mitochondrial Basis of Aging. Mol Cell 2016, 61, 654-666, doi:10.1016/j.molcel.2016.01.028.
Chacko, B.K.; Kramer, P.A.; Ravi, S.; Benavides, G.A.; Mitchell, T.; Dranka, B.P.; Ferrick, D.; Singal, A.K.; Ballinger, S.W.; Bailey, S.M., et al. The Bioenergetic Health Index: a new concept in mitochondrial translational research. Clin Sci (Lond) 2014, 127, 367-373, doi:10.1042/CS20140101.
Picard, M.; Prather, A.A.; Puterman, E.; Cuillerier, A.; Coccia, M.; Aschbacher, K.; Burelle, Y.; Epel, E.S. A Mitochondrial Health Index Sensitive to Mood and Caregiving Stress. Biol Psychiatry 2018, 84, 9-17, doi:10.1016/j.biopsych.2018.01.012.
Gomes, A.P.; Price, N.L.; Ling, A.J.; Moslehi, J.J.; Montgomery, M.K.; Rajman, L.; White, J.P.; Teodoro, J.S.; Wrann, C.D.; Hubbard, B.P., et al. Declining NAD(+) induces a pseudohypoxic state disrupting nuclear-mitochondrial communication during aging. Cell 2013, 155, 1624-1638, doi:10.1016/j.cell.2013.11.037.
Shi, R.; Guberman, M.; Kirshenbaum, L.A. Mitochondrial quality control: The role of mitophagy in aging. Trends Cardiovasc Med 2018, 28, 246-260, doi:10.1016/j.tcm.2017.11.008.





