代谢组学技术在衰老和衰老相关疾病研究中的应用

作者:王全会 孔双泉
【摘要】衰老是一个复杂的涉及多种因素和多种生理或病理反应的过程。为了探讨衰老过程及其机制,并发现衰老相关疾病的诊断标志物与治疗靶标,科学家们对衰老进行了多层次研究。过去的研究表明,基因组不稳定性、DNA突变、转录错配、蛋白质翻译异常等过程均可能导致衰老相关细胞器和细胞功能的异常。随着近年来组学技术的发展,科学家们开展了大量衰老相关基因组学、转录组学和蛋白质组学研究,试图从全局角度运用大数据对衰老进行阐明。代谢组学作为一种高通量组学技术,可以对生物样本中的代谢物谱进行全面表征,能够提供不同于基因组学和蛋白质组学的新视角,因而在衰老及相关疾病如神经退行性病变、脑卒中、心血管疾病等的研究中也有大量的应用。本文将对代谢组学技术及近年来代谢组在衰老相关领域的研究应用进行综述。
【关键词】代谢组学;衰老
一、代谢组技术
1. 代谢组的定义:代谢组学是通过考察生物体系(细胞、组织或生物体)受刺激或扰动后代谢产物图谱的动态变化,来研究生物体系整体代谢变化情况及其应答变化规律的一种技术,是继基因组学、转录组学和蛋白质组学之后发展起来的一门新兴“组学”。与其他三种组学研究的DNA、RNA和蛋白质等生物大分子不同,代谢组学是对生物体系中分子量在1000Da以下的小分子化合物进行定性定量研究。近年来,代谢组学相关技术发展迅速,已广泛应用到各种疾病研究、药物研发、植物学、微生物学等多学科研究领域。
2. 代谢组检测技术:组学技术,无论是基因组、蛋白质组还是代谢组学,要求对生物样品中的目标分子的检测覆盖度越大越好,只有这样才能获得系统的信息。然而,同蛋白质组一样,迄今为止尚没有一种技术可以在一个分析中对所有代谢物进行全面的定性和定量分析。人类代谢组数据库(HMDB)包含水溶性和脂溶性代谢物在内超过了50000多种,但是被定量检测到的代谢物仅有3000种左右[1]。目前对代谢物进行定性定量分析的两种常用的技术手段为液质联用技术(LC-MS/MS)与核磁共振技术(NMR)。NMR技术虽然定量重现性较好,但是灵敏度低,通常只能检测丰度比较高的代谢物,而质谱技术兼备色谱的高分离度、高通量及质谱的普适性、高灵敏度和特异性,因此,近年来基于质谱技术的代谢组学应用要多于NMR技术。
基于质谱技术的代谢组学又分为非靶向代谢组和靶向代谢组(图1)。一般来说,运用非靶向代谢组手段,可鉴定到其中约1000多种代谢物,运用靶向代谢组手段则可分析其中约500种左右。虽然靶向代谢组手段可分析的代谢物种类少一些,但这种策略具有更高的灵敏度并能提供更加准确的绝对定量而非相对定量结果,而且定量结果的重现性比较好[2,3]。对于临床大队列研究项目,往往动辄几百例乃至几千例样本,靶向代谢组技术具有显而易见的优势。因此,近年来有越来越多的研究采用靶向代谢组技术。靶向代谢组方法可以由实验室自建,目前也有商品化试剂盒,比较成熟的为Biocrates公司的靶向代谢组系列试剂盒,分别有激素靶向分析、胆汁酸靶向分析、P180、P500等,其中P500可对620种代谢物进行靶向绝对定量分析,覆盖了生物体内主要的代谢通路(图2)。这些试剂盒获得了欧洲代谢组学会白皮书的推荐,并且已经在肿瘤研究、心血管疾病研究、神经退行性疾病研究等领域获得了广泛应用。
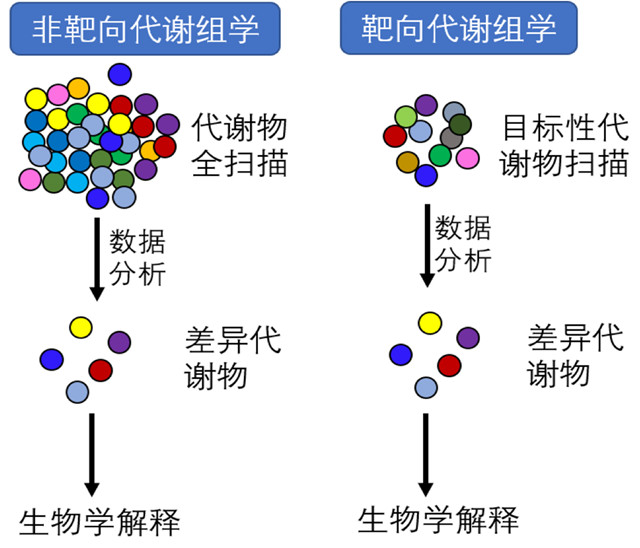
图1. 代谢组学分类
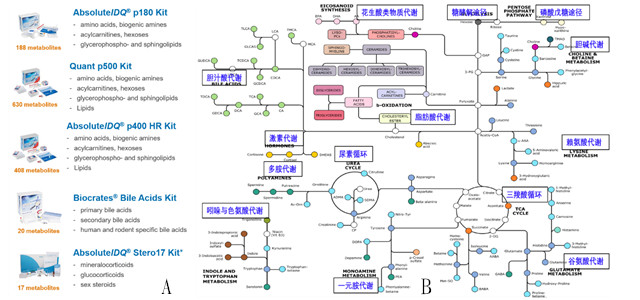
图2. Biocrates靶向代谢组试剂盒。
A为试剂盒种类,B为Quant P500代谢物覆盖的代谢通路。
3. 代谢组检测流程:其基本流程为样品的采集及制备、数据采集、数据分析等。
(1)样品的采集及制备:在采集样品之前,需要设计好样本的采集时间、种类、部位、样本量和采集样品的器材等。由于代谢组对外界扰动比较敏感,因此采集样品时要有严格的质量控制,尽可能在相同的条件下采集样本,使实验误差减到最小。处理样品时应抑制残留酶的活性,避免发生氧化,采用冰冻、液 氮降温或冷冻干燥技术保存样品。根据研究目的和分析技术的不同,样品预处理的方法也各不相同。磁共振(NMR)技术一般用有机溶剂抽提或进行蛋白沉淀的方式提取代谢物,提取后用重水或氘代有机溶剂溶解后上机检测。气相色谱质谱(GC-MS)技术一般检测具有挥发性的化合物,因此对挥发性较小的化合物需进行衍生化,以增加其挥发性,扩大化合物的检测范围。而液质联用质谱技术一般检测低挥发性或无挥发性化合物,通常根据相似相溶原理,利用水和有机溶剂(甲醇、己烷等)分别提取,从而把非极性和极性物质分开。由于特定的提取条件只适于某类化合物,目前尚无一种普适性的提取方法,故应根据检测手段及化合物的性质选择不同的样品制备方法。
(2)数据采集:如果采用非靶向代谢组技术进行研究,则预处理后的样本应采用QTOF或者Orbi一类的高精度质谱仪进行数据采集,如果采用靶向代谢组技术,则预处理后的样本一般选择三重四极杆类质谱进行数据采集。
(3)数据分析:由分析仪器获得的原始数据首先进行预处理,如滤噪、峰检测、峰匹配、标准化、归一化和重叠峰解析等,得到大量的代谢物Feature及其峰强度、色谱保留时间等多维信息,然后进一步借助数学计算方法从预处理后的信息中建立代谢物feature与样本分类信息的联系,并进行样本间代谢物feature的丰度比较,获得代谢物的差异变化信息及其与样本分类的关联。最后对差异代谢物进行主成分分析(PCA)、聚类分析(CA)、神经网络(NN)、偏最小二乘法(PLS)、偏最小二乘法-判别式分析(PLS-DA)等。其中PCA和PLSDA是代谢组学研究中最常用的模式识别方法,这两种方法通常以得分图获得对样品分类的信息,载荷图获得对分类有贡献变量及其贡献大小,从而用于发现可作为生物标记物的变量。代谢组学分析还需要借助一些生物化学数据库,如HMDB[1]、METLIN[4]、Lipid Maps[5]和代谢途径数据库如KEGG[6]、Reactome[7]、Cyc数据库[8]等,可以一方面对代谢物Feature进行鉴定和功能注释,同时对代谢途径或通路进行探索,进而了解疾病的发病机制。
二、代谢组与衰老
许多证据表明,由大量代谢过程产生的分子层面的损伤累积是衰老发生的驱动因素[9,10]。这些累积的损伤不仅局限于氧化损伤,还包括生物体内酶催化反应副产物的产生、基因表达的异常、转录和翻译的错误以及DNA突变等。而且,研究表明,损伤形式的多样性以及代谢过程的各种副产物会随着年龄的增长而增加[11]。因此,对老年人群代谢特征进行分析可以更好地理解衰老的机制。
第一个衰老相关代谢组研究于2008年开展。该研究在269例参与者血浆中发现了100种代谢物与衰老相关[12]。近年来,非靶向代谢组与靶向代谢组研究在衰老相关研究中均有开展,并且基于更大的人群队列。如Yu等人对2162例不同年龄段的健康人群进行采用Biocrates靶向代谢组方法进行了分析[13],发现线粒体脂肪酸氧化功能异常与老化相关;Menni等人对6055对双胞胎进行了非靶向代谢组分析[14],发现C-交联糖化色氨酸与衰老相关。有许多研究者对多种模式生物开展了衰老相关代谢组分析,如线虫、果蝇、小鼠[15-19]。这些研究表明,随着年龄的增长,在不同物种、不同组织器官中代谢组谱图均发生了显著变化。大部分代谢物具有器官或组织特异性,而有些代谢物则不论在哪种组织器官中均表现出显著的年龄相关性。除了组织器官特异性,某些代谢物在人类男性和女性中水平不同,但是有些代谢物不受性别限制,仅与年龄增长相关,提示这类代谢物可能作为预测生物衰老的可靠标志物。重要的是,有研究发现,随着年龄的增长,代谢物种类的多样性(即总代谢物的种类)增加,说明随着年龄的增长出现了一些新的代谢物种类,提示这些代谢物可能在老年生物体内出现了代谢副产物或其他损伤的积累[20]。
代谢组技术还被用于研究一些模式生物及人类长寿相关代谢组图谱[21-24]。如在对一组百岁老人的研究中发现,其氨基酸类和脂类代谢物的水平显示出与非长寿人群的显著不同,如血浆中色氨酸水平显著降低、神经鞘磷脂水平显著升高、单不饱和脂肪酸与多不饱和脂肪酸的比例升高以及氧化脂类水平降低等。由于多不饱和脂肪酸更容易被氧化,百岁老人血浆中多不饱和脂肪酸水平的降低,可能提示长寿老人对氧化损伤的耐受更强[22]。另外一项对1526例来自于90多岁老人的后代的中年人群的脂质组分析,发现19种脂类与家族性长寿相关[25]。这些研究表明抗氧化能力的提高及更有效的线粒体β-氧化功能可能与人类长寿相关。
饮食控制作为一种与寿命正相关的生活方式,可以显著改变代谢组图谱,使其表现为一种更“年轻”的状态。然而,饮食控制导致代谢组水平发生变化的确切机制尚未完全明确。一项针对果蝇这种模式生物的靶向代谢组研究表明,甲硫氨酸代谢相关代谢物的水平变化与年龄增长具有显著的相关性。使两种调控甲硫氨酸代谢的酶:同型S-腺苷甲硫氨酸水解酶类似蛋白dAhcyL1和 dAhcyL2缺失,可以使果蝇的寿命显著延长,并且组蛋白的三甲基化修饰(H3K4me3)水平被抑制[26]。另一项针对来自于韩国、英国等六个国家和地区的6组人群进行的靶向代谢组分析表明,白细胞端粒的长度与血清中的代谢物水平具有相关性。如溶血磷脂酰胆碱、磷脂酰胆碱PCae的水平在6个人群中均呈现与线粒体长度的正相关性,而磷脂酰胆碱PCaa、甲硫氨酸、酪氨酸的水平则与线粒体长度呈负相关性,通路分析发现,后三种代谢物参与同型半胱氨酸循环[27]。
这些研究结果进一步提示对衰老相关代谢组进行分析,有可能发现引起衰老过程重编程的关键代谢物。除了甲硫氨酸代谢相关代谢物,其他的代谢物小分子如烟酰胺腺嘌呤二核苷酸(NAD)、黄素腺嘌呤二核苷酸(FAD)、乙酰辅酶A、以及酮戊二酸盐等许多酶的辅酶或调控因子也可以通过染色质重排、转录调控及其他过程来影响老化进程。
然而,代谢组与衰老之间的关系仍有许多未解的问题。由于代谢组受到遗传因素和环境因素的多重影响,因此,将转录组、蛋白质组和代谢组进行整合分析可能能够对代谢组与长寿及衰老之间的关系提供更加深入的信息。而且,对于一些寿命差别较大的物种之间,其代谢组谱的变化是怎样的尚不清楚。也许对于不同物种之间的代谢组进行比较,能够揭示不同物种的自然寿命不同的一些原因[28,29]。
三、代谢组检测技术在衰老相关疾病中的应用
1. 神经系统退行性疾病:代谢组对环境变化的敏感性使得其成为生物标志物的理想来源。近年来,有许多研究运用代谢组学技术对神经退行性疾病进行了分析,以寻找疾病生物标志物。如有研究对阿尔兹海默病(AD)人群进行代谢组分析,发现血浆中的神经鞘脂、酰基肉碱和氨基酸类代谢物能够区分AD病人与正常健康人群[30-33]。有些研究则对稳定的中度认知障碍(MCI)病人及可转变为AD的中度认知障碍人群进行了代谢组分析,以发现AD发生的标志物分子。如Mapstone等人发现了一组代谢物能够区分可转化性MCI和稳定MCI,灵敏度和特异性达到90%,为AD的诊断提供了潜在标志物[34]。
帕金森病(PD)也是一种重要的神经退行性疾病,有许多研究者对PD的代谢组进行了分析,以发现PD诊断标志物。这些研究采用的仪器类型不尽相同,针对的病例来源不同,采用的样本类型也不同,有的为血浆,有的为脑脊液,但值得注意的是,有3种代谢物:5-酰氨基-6氨基-3甲基尿嘧啶、丙氨酸、谷氨酸在多个不同的研究中结果一致,表明这几种代谢物具有作为PD诊断标志物的潜力[35-40]。有些研究中对发现的PD相关代谢标志物进行了ROC分析,以评价这些代谢物用于PD诊断的特异性,大部分潜在标志物的ROC曲线下峰面积(AUC)均在0.83-0.95之间[34,35,41,42]。
犬尿氨酸途径是哺乳动物色氨酸分解的主要途径,可以通过犬尿氨酸-3-单氧化酶催化犬尿氨酸降解而产生3-羟基犬尿氨酸(3-HK)。大量研究表明,3-HK通过产生反应性自由基而具有神经毒性。在PD相关代谢组研究中发现3-HK及其下游产物喹啉酸在PD人群中较正常人群中升高,在PD人群脑脊液中也发现了3-HK的升高现象[36,41]。在AD病人血浆样本中同样发现了犬尿氨酸通路的异常[43]。此外,甲硫氨酸循环参与细胞浆中同型半胱氨酸的再甲基化进而产生甲硫氨酸。这一代谢通路的稳定与维生素B9及B12的水平相关,而代谢组研究发现,在PD病人及老年痴呆病人体内这两种维生素的水平通常是异常的,而且血浆中同型半胱氨酸水平的升高通常与认知能力的下降呈相关关系[44]。
2. 脑卒中:脑出血可以引起局部性及系统性的代谢变化和应激反应,如细胞能量代谢异常。基于这些发现,许多研究者对循环系统中的代谢组进行了研究,但多数研究集中在评价血液中代谢物是否能够判断脑卒中风险、是否能够进行脑卒中的诊断以及能否阐明不同脑卒中亚型的病因等几个方面,而研究代谢组对于脑卒中发生以后的转归预测的则较少。
代谢组与脑卒中风险。在2019年开展的一项包含了68659个病例的前瞻性研究种,分析了亚油酸(主要的膳食ω-6多不饱和脂肪酸)的水平与心血管疾病之间的相关性,结果表明循环系统中亚油酸的高水平与心血管疾病及出血性脑卒中的低风险呈相关性[45]。这一发现可以为人们提供饮食方面的建议。在2018年开展的一项研究中,针对685个参与者进行了脂质组研究,发现循环系统中短链双键三酰基甘油三酯和胆甾醇酯的水平升高对包括以中风为终点的复合型心血管疾病具有很强的预测作用,并且该结果在英国双胞胎(TwinsUK)队列项目中得到了验证[46]。2019年发表的另外一项研究中,对3904个动脉粥样硬化高危并且最终发生出血性中风的个体进行了代谢组分析,发现脂肪酸ω氧化的产物长链二羧酸十四烷酸盐与十六烷基二酸的水平与突发性出血性中风,特别是心脏栓塞性中风的高风险呈正相关[47]。
代谢组与脑卒中的诊断。代谢物由于其小分子量特征而较蛋白质更容易通过血脑屏障,因此许多研究对急性期出血性中风病人进行了代谢组分析,试图发现诊断标志物。在2016年开展的一项研究中,研究者对200多例出血性中风或ICH病人进行了基于质谱的代谢组分析,发现了由Asn、C5:1、Arg/Orn、Val/Phe、以及(C0+C2+C3+C16+C18:1)/Cit共5种变量构成的组合可以识别出79%的脑中风病人[48]。但是这一结果缺乏外部验证证据,具有一定的局限性。另外一项研究则基于灵长类动物脑中风模型实验结果,同时考虑到神经鞘磷脂类代谢物在脑中丰度较高且容易穿过血脑屏障,研究者采用靶向代谢组技术对神经酰胺 42:1和神经鞘磷脂36:0进行了分析,研究表明这些代谢物可以很好地将出血性脑中风组与对照组区分开来,曲线下峰面积为0.8712[49]。然而这项研究仅对14个病人进行研究,病例数比较有限,因而结论有待进一步验证。
代谢组与脑卒中的病因。许多研究评价了代谢组是否可用于区分病因学不同的脑卒中亚型。一项研究对669个急性脑出血中风病例的血浆样本进行了代谢组分析,发现心源性栓塞性脑中风病人血浆中游离脂肪酸的水平明显高于其他类型的中风病人[50]。另外一项研究则对367例急性出血性脑卒中病人的血液样本进行了基于质谱的代谢组分析,发现三羧酸循环的中间产物琥珀酸、α-酮戊二酸以及苹果酸的水平在心源性栓塞性脑中风病人血液中的水平明显高于非心源性脑中风病人[51]。但是这些发现是否可用于指导临床做出相关的预防性治疗尚不明确。
3. 心衰:越来越多的证据表明,除了蛋白质标志物和基因标志物以外,循环系统中代谢物分子的变化与心血管疾病如心衰的风险及预后密切相关。一项在美国弗雷明翰和欧洲波美拉尼亚地区开展的项目对接近6000名参与者的神经酰胺C24:0、C22:0和C16:0的水平及其比值进行了分析,研究发现神经酰胺C24:0/C16:0的比值与突发性心衰呈负相关[52]。针对神经酰胺在心衰中的功能的更深入的研究目前仍在进行。另外一项针对HF-ACTION试验人群样本及进行左心室辅助装置(LVAD)手术人群样本的靶向代谢组研究表明,血浆中长链酰基肉碱与心衰患者的死亡率正相关,而当进行LVAD置换后,这些代谢物的水平逐渐恢复正常[53]。值得注意的是,在2018年发表的一项对左心室重构病人的研究中,同样发现长链酰基肉碱在左心室重构后显著下降[54]。长链酰基肉碱是脂肪酸在线粒体内β氧化的中间产物,心衰患者血液中长链酰基肉碱水平的提高说明其线粒体β氧化功能异常。
同蛋白质或基因类的标志物一样,代谢组的标志物也建议由多个标志物组合在一起,以做出更为准确和全面的判断。比如,Cheng ML等人对一个包含心衰患者及其对照人群共515例样本进行了代谢组分析,其中234例为发现组,281例为验证组,另外有一组独立的样本包含了32例心衰恢复者。研究发现由组氨酸、苯丙氨酸、亚精胺和磷脂酰胆碱C34:4构成的代谢指标组合具有心衰诊断功能,而由二甲精氨酸/精氨酸比值、亚精胺、丁酰肉碱及总必需氨基酸构成的代谢物组合在心衰的预后方面具有很好的潜力[55]。在另外一项类似的研究中,有1288个参与者,研究者对血浆样本中的氨基酸及脂质组进行了分析,并在另外一组人群中进行了验证。结果表明,氨基酸类代谢物与突发性心衰的发生相关,其中亮氨酸和苯基丙氨酸具有预测心衰发病率的潜力,根据这两个指标预测中等风险组的发病率较低风险组加倍,而高风险组的发病率则高达8倍[56]。有意思的是,该研究组又开展了另外一个有890个病例的项目,同样对氨基酸进行了分析,结果表明组氨酸、苯基丙氨酸和鸟氨酸构成的指标组合能够对不同的发病风险组进行很好的区分,可能用于心衰病人的风险评价[57]。
Lanfear DE等人开展的一项研究,则运用靶向代谢组技术对1000多例射血分数降低的心衰病例血浆中的代谢组进行了分析[58]。研究者将病例随机分为实验组和验证组,发现了由氨基酸、酰基肉碱、有机酸等13种代谢物组成的组合指标,能够很好地区分低射血分数人群和高射血分数人群,以及预测心衰患者的生存。但是关于这些代谢物的相关机制研究及其在临床诊断中的应用需要更进一步的研究。
四、展望
衰老是一个复杂的生物学过程,受到遗传及环境因素的多重作用。虽然已有大量研究发现在衰老过程中发生了许多生理过程的变化,但是衰老的精确分子机制仍然不够明确。鉴于衰老涉及到复杂的生物学过程和遗传过程,对其机制的阐明需要进行系统的研究。
近年来,高通量技术如二代测序技术、蛋白质组技术、代谢组技术等的发展,可以实现对衰老过程中的生物分子进行更为精准、特异和高通量的分析,从而为研究者了解衰老机制提供了新的机会。然而,技术的进步使得我们产生大数据的能力飞速提升,但是如何将这些大数据进行整合分析,以阐明衰老过程中的生物调控因子及衰老的原因仍然是一项挑战。而且,代谢组并非独立存在,而是与基因组和蛋白质组之间存在内在联系的,因此,未来的方向应该是将代谢组学与蛋白质组或基因组数据进行整合,构建相互作用网络,来研究衰老或疾病因素对这个网络的影响。
此外,对衰老相关疾病的代谢组研究中发现了许多代谢标志物,单独的或由几种代谢物构成的组合,表现出对相关疾病的良好的诊断或预后效果。然而,关于这些代谢物与疾病发生或进展之间的关系及其内在机制仍然缺乏研究,而且在临床上如何应用这些生物标志物也尚不明确。因此,未来还应该对所发现的代谢物分子进行更加深入的研究,以明确其应用方向。
参考文献
Wishart DS, Feunang YD, Marcu A, et.al. HMDB 4.0: the human metabolome database for 2018. Nucleic Acids Res. 2018 Jan 4; 46(D1): D608-D617.
Patti GJ, Yanes O, Siuzdak G. Innovation: Metabolomics: the apogee of the omics trilogy. Nat Rev Mol Cell Biol. 2012 Mar 22; 13(4): 263-9.
Tzoulaki I, Ebbels TM, Valdes A, et.al. Design and analysis of metabolomics studies in epidemiologic research: a primer on -omic technologies. Am J Epidemiol. 2014 Jul 15; 180 (2): 129-39.
Montenegro-Burke JR, Guijas C, Siuzdak G. METLIN: A Tandem Mass Spectral Library of Standards. Methods Mol Biol. 2020; 2104: 149-163.
Sud M, Fahy E, Cotter D, et.al. LMSD: LIPID MAPS structure database. Nucleic Acids Res. 2007 Jan; 35 (Database issue): D527-32. pii: gkl838. doi: 10.1093/nar/gkl838. PubMed PMID: 17098933.
Kanehisa M, Furumichi M, Tanabe M, et.al. KEGG: new perspectives on genomes, pathways, diseases and drugs. Nucleic Acids Res. 2017 Jan 4; 45 (D1): D353-D361.
Jassal B, Matthews L, Viteri G, et.al. The reactome pathway knowledgebase. Nucleic Acids Res. 2019 Nov 6.
Caspi R, Billington R, Keseler IM,e.al. The MetaCyc database of metabolic pathways and enzymes-a 2019 update. Nucleic Acids Res. 2019 Oct 5. pii: 5581728.
Golubev A, Hanson AD, Gladyshev VN. Non-enzymatic molecular damage as a prototypic driver of aging. J Biol Chem. 2017 Apr 14; 292(15): 6029-6038..
Yin D, Chen K. The essential mechanisms of aging: Irreparable damage accumulation of biochemical side-reactions. Exp Gerontol. 2005 Jun; 40 (6): 455-65. pii: S0531-5565(05)00079-3.
Gladyshev VN. The origin of aging: imperfectness-driven non-random damage defines the aging process and control of lifespan. Trends Genet. 2013 Sep; 29 (9): 506-12.
Lawton KA, Berger A, Mitchell M, et.al. Analysis of the adult human plasma metabolome. Pharmacogenomics. 2008 Apr; 9(4): 383-97.
Yu Z, Zhai G, Singmann P,et.al. Human serum metabolic profiles are age dependent. Aging Cell. 2012 Dec; 11(6): 960-7. doi: 10.1111/j.1474-9726.2012.00865.x. PubMed PMID: 22834969.
Menni C, Kastenmüller G, Petersen AK, et.al. Metabolomic markers reveal novel pathways of ageing and early development in human populations. Int J Epidemiol. 2013 Aug; 42(4): 1111-9.
Fuchs S, Bundy JG, Davies SK, et.al. A metabolic signature of long life in Caenorhabditis elegans. BMC Biol. 2010 Feb 10; 8: 14.
Hoffman JM, Soltow QA, Li S, et.al. Effects of age, sex, and genotype on high-sensitivity metabolomic profiles in the fruit fly, Drosophila melanogaster. Aging Cell. 2014 Aug; 13(4): 596-604.
Houtkooper RH, Argmann C, Houten SM, et.al. The metabolic footprint of aging in mice. Sci Rep. 2011; 1: 134.
Tomás-Loba A, Bernardes de Jesus B, Mato JM,et.al. A metabolic signature predicts biological age in mice. Aging Cell. 2013 Feb; 12(1): 93-101.
Jové M, Maté I, Naudí A, et.al. Human Aging Is a Metabolome-related Matter of Gender. J Gerontol A Biol Sci Med Sci. 2016 May; 71(5): 578-85.
Avanesov AS, Ma S, Pierce KA, et.al. Age- and diet-associated metabolome remodeling characterizes the aging process driven by damage accumulation. Elife. 2014 Apr 29; 3: e02077.
Kaya A, Ma S, Wasko B, et.al. Defining Molecular Basis for Longevity Traits in Natural Yeast Isolates. NPJ Aging Mech Dis. 2015; 1: e148.
Patti GJ, Tautenhahn R, Johannsen D, et.al. Meta-analysis of global metabolomic data identifies metabolites associated with life-span extension. Metabolomics. 2014 Aug 1; 10(4): 737-743.
Collino S, Montoliu I, Martin FP, et.al. Metabolic signatures of extreme longevity in northern Italian centenarians reveal a complex remodeling of lipids, amino acids, and gut microbiota metabolism. PLoS ONE. 2013; 8(3): e56564.
Jové M, Naudí A, Gambini J, et.al. A Stress-Resistant Lipidomic Signature Confers Extreme Longevity to Humans. J Gerontol A Biol Sci Med Sci. 2017 Jan; 72(1): 30-37.
Gonzalez-Covarrubias V, Beekman M, Uh HW, et.al. Aging Cell. 2013 Jun; 12(3): 426-34. doi: 10.1111/acel.12064.
Parkhitko AA, Binari R, Zhang N, et.al. Tissue-specific down-regulation of S-adenosyl-homocysteine via suppression of dAhcyL1/dAhcyL2 extends health span and life span in Drosophila. Genes Dev. 2016 Jun 15; 30(12): 1409-22.
van der Spek A, Broer L, Draisma HHM,et.al. Metabolomics reveals a link between homocysteine and lipid metabolism and leukocyte telomere length: the ENGAGE consortium. Sci Rep. 2019 Aug 12; 9(1): 11623.
Ma S, Upneja A, Galecki A, et.al. Cell culture-based profiling across mammals reveals DNA repair and metabolism as determinants of species longevity. Elife. 2016 Nov 22; 5.
Ma S, Yim SH, Lee SG, et.al. Organization of the Mammalian Metabolome according to Organ Function, Lineage Specialization, and Longevity. Cell Metab. 2015 Aug 4; 22(2): 332-43.
Han X, Rozen S, Boyle SH, et.al. Metabolomics in early Alzheimer's disease: identification of altered plasma sphingolipidome using shotgun lipidomics. PLoS ONE. 2011; 6(7): e21643.
Orešič M, Hyötyläinen T, Herukka SK, et.al. Metabolome in progression to Alzheimer's disease. Transl Psychiatry. 2011 Dec 13; 1: e57.
Trushina E, Dutta T, Persson XM, et.al. Identification of altered metabolic pathways in plasma and CSF in mild cognitive impairment and Alzheimer's disease using metabolomics. PLoS ONE. 2013; 8(5): e63644.
Kaddurah-Daouk R, Rozen S, Matson W,et.al. Metabolomic changes in autopsy-confirmed Alzheimer's disease. Alzheimers Dement. 2011 May; 7(3): 309-17.
Mapstone M, Cheema AK, Fiandaca MS, et.al. Plasma phospholipids identify antecedent memory impairment in older adults. Nat Med. 2014 Apr; 20(4): 415-8.
Fiandaca MS, Gross TJ, Johnson TM, et.al. Potential Metabolomic Linkage in Blood between Parkinson's Disease and Traumatic Brain Injury. Metabolites. 2018 Sep 7; 8(3) .
Han W, Sapkota S, Camicioli R, et.al. Profiling novel metabolic biomarkers for Parkinson's disease using in-depth metabolomic analysis. Mov Disord. 2017 Dec; 32(12): 1720-1728.
Hatano T, Saiki S, Okuzumi A, et.al. Identification of novel biomarkers for Parkinson's disease by metabolomic technologies. J Neurol Neurosurg Psychiatry. 2016 Mar; 87(3): 295-301.
Trupp M, Jonsson P, Ohrfelt A, et.al. Metabolite and peptide levels in plasma and CSF differentiating healthy controls from patients with newly diagnosed Parkinson's disease. J Parkinsons Dis. 2014;4(3):549-60.
Wuolikainen A, Jonsson P, Ahnlund M, et.al. Multi-platform mass spectrometry analysis of the CSF and plasma metabolomes of rigorously matched amyotrophic lateral sclerosis, Parkinson's disease and control subjects. Mol Biosyst. 2016 Apr; 12(4): 1287-98.
Nagesh Babu G, Gupta M, Paliwal VK, et.al. Serum metabolomics study in a group of Parkinson's disease patients from northern India. Clin Chim Acta. 2018 May; 480: 214-219.
Chang KH, Cheng ML, Tang HY, et.al. Alternations of Metabolic Profile and Kynurenine Metabolism in the Plasma of Parkinson's Disease. Mol Neurobiol. 2018 Aug; 55(8): 6319-6328.
Zhao H, Wang C, Zhao N,et.al. Potential biomarkers of Parkinson's disease revealed by plasma metabolic profiling. J Chromatogr B Analyt Technol Biomed Life Sci. 2018 Apr 1; 1081-1082: 101-108.
Schwarz MJ, Guillemin GJ, Teipel SJ, et.al. Increased 3-hydroxykynurenine serum concentrations differentiate Alzheimer's disease patients from controls. Eur Arch Psychiatry Clin Neurosci. 2013 Jun; 263(4): 345-52.
Sleeman I, Lawson RA, Yarnall AJ, et.al. Urate and Homocysteine: Predicting Motor and Cognitive Changes in Newly Diagnosed Parkinson's Disease. J Parkinsons Dis. 2019;9(2):351-359.
Marklund M, Wu JHY, Imamura F, et.al. Cohorts for Heart and Aging Research in Genomic Epidemiology (CHARGE) Fatty Acids and Outcomes Research Consortium (FORCE). Biomarkers of Dietary Omega-6 Fatty Acids and Incident Cardiovascular Disease and Mortality. Circulation. 2019 May 21; 139(21): 2422-2436.
McGarrah RW, Crown SB, Zhang GF,et.al. Cardiovascular Metabolomics. Circ Res. 2018 Apr 27; 122(9): 1238-1258.
Sun D, Tiedt S, Yu B, et.al. A prospective study of serum metabolites and risk of ischemic stroke. Neurology. 2019 Apr 16; 92(16): e1890-e1898.
Hu Z, Zhu Z, Cao Y,et.al. Rapid and Sensitive Differentiating Ischemic and Hemorrhagic Strokes by Dried Blood Spot Based Direct Injection Mass Spectrometry Metabolomics Analysis. J Clin Lab Anal. 2016 Nov; 30(6): 823-830.
Sheth SA, Iavarone AT, Liebeskind DS, et.al. Targeted Lipid Profiling Discovers Plasma Biomarkers of Acute Brain Injury. PLoS ONE. 2015; 10(6): e0129735.
Choi JY, Kim JS, Kim JH, et.al. High free fatty acid level is associated with recurrent stroke in cardioembolic stroke patients. Neurology. 2014 Apr 1; 82(13): 1142-8.
Ay H, Furie KL, Singhal A, et.al. An evidence-based causative classification system for acute ischemic stroke. Ann Neurol. 2005 Nov; 58(5): 688-97.
Peterson LR, Xanthakis V, Duncan MS, et.al. Ceramide Remodeling and Risk of Cardiovascular Events and Mortality. J Am Heart Assoc. 2018 May 3; 7(10) .
Ahmad T, Kelly JP, McGarrah RW, et.al. Prognostic Implications of Long-Chain Acylcarnitines in Heart Failure and Reversibility With Mechanical Circulatory Support. J Am Coll Cardiol. 2016 Jan 26; 67(3): 291-9.
Elmariah S, Farrell LA, Furman D, et.al. Association of Acylcarnitines With Left Ventricular Remodeling in Patients With Severe Aortic Stenosis Undergoing Transcatheter Aortic Valve Replacement. JAMA Cardiol. 2018 Mar 1; 3(3): 242-246.
Cheng ML, Wang CH, Shiao MS, et.al. Metabolic disturbances identified in plasma are associated with outcomes in patients with heart failure: diagnostic and prognostic value of metabolomics. J Am Coll Cardiol. 2015 Apr 21;65(15):1509-20.
Wang CH, Cheng ML, Liu MH. Amino Acid-Based Metabolic Panel Provides Robust Prognostic Value Additive to B-Natriuretic Peptide and Traditional Risk Factors in Heart Failure. Dis Markers. 2018; 018:3784589.
Wang CH, Cheng ML, Liu MH, et.al. Amino Acid-Based Metabolic Profile Provides Functional Assessment and Prognostic Value for Heart Failure Outpatients. Dis Markers. 2019; 019: 8632726..
Lanfear DE, Gibbs JJ, Li J, et.al. Targeted Metabolomic Profiling of Plasma and Survival in Heart Failure Patients. JACC Heart Fail. 2017 Nov; 5(11): 823-832.





