肠道菌群在衰老过程中的作用与抗衰老的干预效果

作者:李志明 聂超
单位:深圳华大生命科学研究院精准健康研究所
【摘要】近年来由于测序技术的发展,涌现大量报道,清晰的描述了肠道微生物群的组成,多样性和肠道微生物功能与衰老之间的关系。它们支持了肠道微生物与年龄相关的免疫功能下降和一些慢性炎症有关,从而产生病理性的衰老。通过饮食和益生菌干预微生物可以增强抗氧化活性,维持免疫稳态,抑制慢性炎症,并且能够调节脂肪代谢和预防胰岛素抵抗,从而有益于宿主的健康衰老。在预防和治疗与年龄有关的疾病方面,包括动脉粥样硬化、2型糖尿病和帕金森氏病等疗法中,粪便微生物菌群移植具有很高的有效性和安全性占据了极大的优势。在这篇综述中,我们总结了肠道微生物在衰老过程中的作用以及对常用的微生物检测方法进行描述,重点是针对微生物的抗衰老干预措施和治疗潜力。
【关键词】微生物;衰老;干预;健康;检测
在过去的几十年里,世界各地的预期寿命显著增加。虽然医疗水平的提升可以改善他们的健康状况,但是这也造成许多社会和经济问题。在发达国家人口中,老年人比例显著上升,很可能的原因是,公众和专业人士致力于发展延长寿命方法和老年生物学研究的兴趣。
人们普遍认为,人类健康是由遗传、表观遗传和环境(主要是饮食和生活方式)因素以及他们之间复杂的相互作用决定的[1]。近年来,研究证实了肠道菌群在调节健康状况和寿命方面的重要作用[2-4]。一组研究报告称,肠道微生物在防御病原体、提供能量和营养以及维持宿主健康方面有着重要的作用[5]。研究表明,肠道微生物可以影响宿主机体中的几个重要生理和代谢功能,并且在早期发育过程中就会推动免疫反应的成熟,从而有助于成长过程中体内免疫的平衡。一个健康的肠道菌群在体内代谢、抵抗感染和炎症、预防自身免疫和癌症以及调节脑-肠轴等方面发挥着关键作用[6]。此外,肠道菌群能够影响胃肠道疾病,如结肠直肠癌、炎症性肠病和肠易激综合征,以及一些肠道外疾病,包括那些影响肝脏和呼吸道的疾病(支气管哮喘、过敏、囊性纤维化等)[7-11]。在健康人肠道中存在的双歧杆菌属和种的含量、组成的改变具有预防和治疗上述疾病以及其他疾病的潜力[12,13]。最近,微生物靶向治疗有望成为年龄相关性代谢和神经退行性疾病的一种有前景的治疗模式[14]。在这篇综述中,根据最近的研究,微生物靶向干预不仅对特定的年龄和相关疾病具有治疗潜力,而且对减缓衰老过程本身以及促进人类健康和寿命也具有潜力,本文对此进行了总结和讨论。
一、肠道菌群的检测技术
传统上以培养为基础的方法通常用于鉴定肠道细菌。培养的主要限制是,绝大多数的肠道微生物不能通过体外培养的方法而获得,因此低估了实际的物种数量和高估了物种的重要性[15]。尽管方法上存在的局限性,但目前大多数知识都是自基于培养的研究,在某些情况下,这些方法很重要。例如,用于确定抗生素耐药性和培养丰度很低微生物(如艰难梭菌)。细菌培养是指培养大量细菌的过程,方法是使用多种不同的培养基类型和条件(即需氧和厌氧),有时将副产物加入培养基中以模拟原生环境。尽管目前培养仍然存在适应培养条件的细菌大量增长的弊端,但最近的技术能够恢复存在于人类肠道中的大多数细菌[16]。近年来,培养组学的应用使新细菌的发现取得了重大进展[17],但它仍然主要是用于研究的工具,临床可用性有限。虽然大多数人将注意力放在细菌上,但也需要识别其他生命形式(古生菌、真菌、病毒、真核生物),其中许多是难以或不可能用实验室培养技术来评估的。
由于肠道中的部分细菌无法培养,因此宏基因组测序应运而生。宏基因组分析,也称为“散弹枪测序”,用于随机测序环境中存在的DNA片段(如粪便)。该方法直接从样本中提取DNA序列,它绕过了PCR扩增步骤,减少了PCR偏倚的潜在影响,但由于与测序深度的影响对较低丰度的细菌识别有所影响,因此这对宏基因组的生物信息分析是一个挑战。这些数据可用于功能分析,在功能分析中,根据基因的代谢(如细胞壁的构建、抗生素耐药性、碳水化合物的代谢)将基因分为大类。然后可以在两个或更多的群落之间比较这种代谢情况。在宏基因组的研究也提供会提供一些分类信息,因为在测序的过程中,会有用于分类的基因被测序(例如16SrRNA基因,cpm60基因等)[18,19]。
用于分类的基因也可以通过使用靶向保守区域的引物和NGS测序的扩增子进行PCR扩增。这些基因包含高度保守区域(即在大多数细菌物种中是相似的)和物种之间差异很大的高变区域,从而可以区分单个DNA链所属的分类单元(如种、属、科等)。到目前为止,16SrRNA基因在这方面的应用最为广泛,根据其复杂性和测序工作的不同,它可以提供接近100%的细菌组成的深度数据。
二、年龄相关的肠道微生物组成的变化
先前的研究表明,早期的不良环境可能会造成终身健康和疾病风险[20]。在过去的几年里,有证据表明肠道菌群在人类健康和疾病的发展中扮演着重要的角色。成年人肠道菌群在生命的早期建立,在余下的生命中保持相对稳定,但可能会因生活方式、饮食、细菌感染、抗生素或手术等而改变。肠道中早期微生物的定殖可以影响宿主与细菌的相互作用,并影响晚年疾病的发生,如肥胖、2型糖尿病、慢性肠道炎症、自身免疫性疾病、过敏、肠易激综合征、过敏性肠胃炎、坏死性小肠结肠炎[21,22]。
在此背景下,重要的一点是,肠道菌群的组成随着衰老和相关疾病而发生显著变化[23]。通过依赖于细菌培养和不依赖于细菌培养的方法均揭示了年轻人和老年人肠道菌群的差异[24]。 随年龄增长而引起内脏中生理变化(如胃动力障碍、肠神经系统退化等)对肠道微生物的多样性、组成和功能特征都有显著的影响[6]。这些与年龄相关的差异,是由于免疫系统的长期刺激导致免疫系统功能的下降导致的(“免疫衰老”)。与此同时,慢性炎症伴随着许多衰老相关的疾病,包括胃肠道疾病、动脉粥样硬化、癌症、脂肪肝、代谢综合征、2型糖尿病等[6,14,25]。这种炎症状态可能使宿主对肠道细菌更敏感,反之亦然,因为肠道菌群组成的改变与老年人各种病理状况的进展都有关系。
随着年龄的增长,肠道菌群的变化包括菌群多样性的下降,糖溶菌的减少和蛋白水解菌的增加[14],核心物种丰度降低,次优势物种丰度增加[2,26],某些变形菌门增加,双歧杆菌计数减少,厚壁菌门与拟杆菌的比值降低(F/B)等[21]。例如,在哺乳期分娩的婴儿中,双歧杆菌数量在整个生命周期中都在下降,从出生后肠道菌群总数的90%下降到成人结肠中不足5%,这些数字在老年人或者某些疾病患者中甚至更小[27]。
肠道菌群的显著变化发生在从成年到老年的过渡期。它与成年人相比,在老年人(大于65岁)肠道微生物表现出多样性的减少和更大的个体差异以及双歧杆菌、厚壁菌门、柔嫩梭菌的减少[28]。然而,应该指出的是,与年龄相关的微生物组成变化的数据在不同的人群中是不同的。与成年人相比,百岁老人的微生物菌群多样性较低,并且双歧杆菌、拟杆菌和肠杆菌水平较低,梭状芽胞杆菌水平较高[2,29]。这种与年龄相关的微生物的差异不一定是由年龄引起的,但它们是由年龄引起的可能性与健康状况普遍下降、营养不良和药物(包括非甾体抗炎药和抗生素)的需求增加有关。在讨论微生物多样性随年龄变化时,核心微生物多样性的丧失与其说与实际年龄本身有关,不如说是与年龄相关的体内炎症有关[24,28,30,31]。
年龄相关的肠道菌群失调被证明是年龄相关病理状态的重要因素,如慢性炎症[32]、神经退行性变[33]、认知能力下降[34]、1型和2型糖尿病[35]以及非酒精性脂肪肝和心血管疾病[36]。肠道菌群也被认为是调节癌症发展风险的重要因素,据推测是通过特定因素的作用,在癌变过程中特定毒素、代谢产物或微生物结构,以及诱导的免疫反应对癌症的发生作用[37]。此外,瘦人与肥胖受试者之间的肠道微生物也存在显著差异[38],例如,肥胖个体的微生物主要以拟杆菌减少和厚壁菌门增加为特征,在获取食物能量方面比正常体重和瘦人更有效。因此,肠道菌群可以作为代谢健康的一个标准[39]。
三、肠道菌群在宿主衰老中的作用
有证据表明微生物组成可能影响宿主衰老速率[40,41],并没有发现随着年龄的变化微生物组成会发生剧烈变化;相反,这些变化是随着时间逐渐发生的[24]。衰老过程中宿主和肠道菌群之间相互作用年龄受特定因素的影响,如生活方式、营养、炎症的改变[40]。
肠道微生物的某些变异认为是人类长寿的预测因子。Biagi等人[42]证明,在年轻人和70岁的老年人中,微生物组成高度相似,但与百岁老人有显著差异。100岁以上的个体肠道菌群的特征是肠道内微生物的重排,厚壁菌门和变形菌门在含量上富集。根据一系列外周血炎症标志物,百岁老人体内微生物-宿主稳态的降低与炎症状态有关。百岁老人微生物的重塑还伴随着柔嫩梭菌及其株的大量减少,它们是具有明显的抗炎活性的共生物种。此外,粘液真细菌及其株被鉴定为长寿的特征细菌,因为它们在百岁老人中增加了十倍以上。在最近对半超级老人(105-109岁)进行的系统发育微生物分析中,与成年人、老年人和百岁老人相比,特定健康群体中观察到Akkermansia、双歧杆菌和Christensenellaceae的增加[3]。在另一个百岁老人研究中,老年人与年轻人相比,肠道微生物年龄有关特点是短链脂肪酸合成相关基因的减少,而蛋白水解作用更丰富[43]。这种功能变化与条件致病菌的富集有关,即在成人肠道生态系统中存在较少的条件致病菌。此外,116个与衰老显著相关的微生物基因被鉴定为长寿的标志[44]。
微生物可能影响宿主健康和衰老过程的分子机制,目前在临床试验中有深入研究,这些试验使用了益生菌和益生元,益生元已被证明能够有效地预防老年人群的特殊病理状况。在这些机制中,最重要的是抑制慢性炎症,在老年人中上调的促炎细胞因子的合成减少,如白细胞介素IL-6、IL-8和IL-10,以及肿瘤坏死因子(TNF),增加了坏死淋巴细胞、自然杀伤细胞[25,45]。调节肠道菌群可以减少炎症反应,提高适应性免疫反应,从而对抗免疫衰老。其他机制是调节碳水化合物的降解、提高抗氧化活性、产维生素B和共轭亚油酸[27]、调节脂肪沉积和新陈代谢[46]、预防胰岛素抵抗[47]等。此外,这些机制还包括改善肠道屏障功能,减少短链脂肪酸的产生,增加脂质代谢和葡萄糖稳态中重要作用的酶,限制热量摄入[48]。此外,肠道菌群可以通过调节宿主的一些基因表达来影响宿主的衰老[49]。
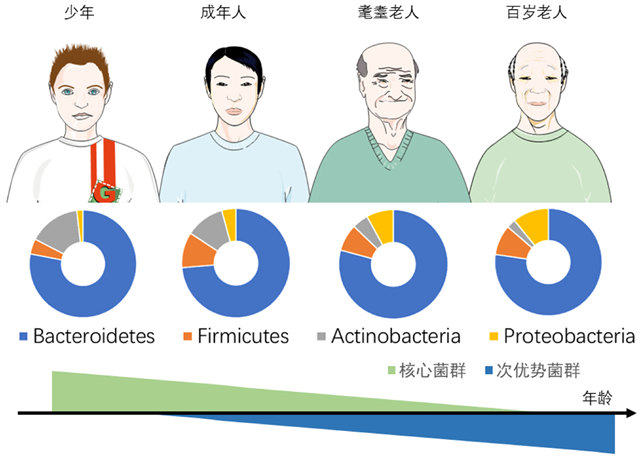
图1. 与年龄相关菌群变化趋势
(来自于文章DOI:https://doi.org/10.1016/j.arr.2017.01.001)
四、肠道菌群干预促进健康
各种促进健康的干预措施均揭示了肠道菌群组成的改变。多项研究表明,肠道菌群与热量限制(CR,目前最可行且有效的抗衰老手段之一[50])所导致的体重减轻之间存在关联。特别地,F/B比例在肥胖患者中一直被发现是增加的,而通过基于CR的减肥干预可以降低F/B比例[51]。例如,在针对肥胖的手术减肥和饮食减肥疗法中,在腹腔镜胃切除术后,F/B比值随着CR的增加而降低[52]。值得注意的是,在上述两组报道中,厚壁菌门的变化都会伴随着产丁酸盐细菌种类的变化。在接受减肥饮食干预的肥胖个体中,F/B比率也显著下降[53]。导致体重增加的细菌反过来可以诱导与碳水化合物和脂质代谢相关的基因表达,从而影响膳食能量的摄入[54]。在动物模型中,CR诱导的寿命延长伴随着肠道菌群结构的调整。例如,终生低脂饮食显著改变了C57BL/6J小鼠肠道菌群的整体结构。CR可以丰富与寿命呈正相关的细菌,减少与寿命负相关的细菌[55]。伴随上述一致变化的是血清脂多糖结合蛋白水平的显著降低,提示肠道微生物的结构平衡可能是通过CR建立的。作者认为,CR可以通过减少肠道抗原负荷而对宿主有益。由于营养物质的供应和转化高度依赖于肠道菌群[56]。因此可以假设,某些抗衰老干预可能导致肠道菌群的特殊变化,从而促进健康并延长寿命。
世界卫生组织把益生菌定义为“活的微生物,给予适量时,对宿主的健康有益。”它们通过改善屏障功能、免疫调节和产生神经递质对宿主健康状况产生有益。此外,它们还可以影响宿主的微生物和肠-脑轴的细胞组分[57]。考虑到益生菌可以减弱与年龄相关的微生物组成的变化,因此益生菌可以应用于老年人治疗各种胃肠和呼吸系统疾病就不足为奇了[14]。以改善老年人健康状况为目标的微生物干预措施中,益生元和益生菌,特别是含有双歧杆菌和乳酸菌的益生元和益生菌在临床试验中调查和记录最多[14,25,41]。人们发现,通常健康的人食用益生菌的益处与他们的健康有关。例如,益生菌鼠糖糖乳杆菌被证明能够促进特定微生物的抗炎[58]。一些人认为,它们是通过调节促进肠道微生物内稳态,而不是改变其组成[59]。
在小鼠模型中,口服含有双歧杆菌和植物乳杆菌可以有效预防慢性紫外线照射引起的皮肤光老化[60,61],植物乳杆菌可以减少紫外线诱导的皮肤光老化症状[62]。口服短乳杆菌可抑制肠道微生物脂多糖的产生、p16的表达和NF-B的激活,改善衰老相关的结肠炎症和记忆障碍[63]。此外,强有力的实验和临床证据表明,补充益生菌可以通过调节在衰老过程中发挥关键作用的指标来改善代谢和心血管健康状况。益生菌治疗可以降低低密度脂蛋白和胆固醇水平,改善低密度/高密度脂蛋白比率,还可以减少炎症发生、血糖水平、血压[64]。
在实验研究中,益生菌也被证明能够延长寿命。在线虫中发现补充益生菌有显著的延长寿命的作用,他们促进长寿是通过刺激先天免疫应答[65]、提高抗氧化应激能力[66]、降低脂褐素积累[67]来实现的。还有证据表明,益生菌可以促进小鼠延长寿命,可能是通过抑制结肠中的慢性炎症过程[68]。大多数现有的实验和临床试验数据都支持益生菌对大多数人群是安全的。然而,在特殊的病人群体中使用应谨慎。益生菌治疗的潜在风险包括胃肠道副作用、不良代谢状况、易感个体的过度免疫刺激和全身感染[69]。此外,由于不同益生菌的特性可能具有菌株特异性,因此,某些菌株的益生菌效果不应在单独的研究中未经确认而传播给其他菌株[70]。因此,需要进一步的调查,以全面评价与益生菌有关的不利事件以及它的严重程度。
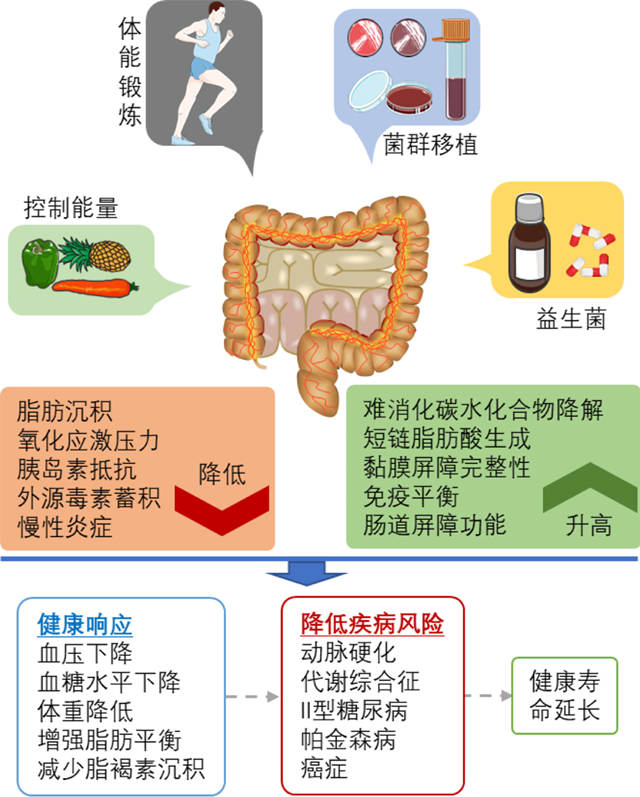
图2. 肠道菌群延长寿命的潜在途径
五、结论和展望
近年来,在认识肠道菌群在人类健康、衰老和长寿中的作用取得了很大进展。但这一新兴领域仍存在诸多局限性和挑战,仍需要进一步研究和完善。它的局限性大多与这些研究中普遍采用的研究方法有关。事实上,尽管越来越多的证据表明肠道菌群与年龄相关的病理状况之间存在关系,但在因果推论时必须谨慎,因为该领域常用的横断面研究仅仅提供相关而非因果的证据[71]。由于微生物的组成会受到多个混杂因素的严重影响,因此对这些数据的解释应谨慎。特别是必须考虑微生物-药物的相互作用上,越来越多的证据表明,使用各种药物治疗可能显著影响微生物基因的表达水平[72]。其他混杂因素包括饮食[73]、心理压力[74]和宿主基因[75]等。然而,在大多数情况下,正确控制混淆因素、是一项非常困难的任务。例如,很难找到足够多的未接受过医学治疗的患者来为调查提供足够的统计力。在进一步的研究中,识别和控制混杂因素是非常重要的。
微生物靶向治疗模式的发展和临床实施作为最有希望的干预途径时,根据上面回顾的数据,可以假定FMT在微生物靶向干预措施中、在预防和治疗年龄相关疾病包括代谢和神经退行性病变方面具有巨大的治疗潜力。然而,尽管FMT手术具有很高的成功率和安全性,但其在临床实践中的实施仍存在一些问题。这些问题包括捐赠者筛选、新鲜粪便样本的细菌生存能力有限、准备和使用方面的问题、对病原体传播的担忧、缺乏标准化的治疗方案,以及患者是否同意接受治疗等。为了解决这些问题,正在开发新的方法[76]。另一种合理的替代治疗方法是使用粪便替代物,即多株细菌的组合,制备从单一供体中分离的多菌种菌群。伴随这些有利的治疗效果的是肠道微生物组成的显著改变,这些变化反映在合成混合物中分离出的菌株。
重要的是,一些研究结果表明,肠道微生物能够调节治疗特定的年龄相关疾病,而且可以作为对抗衰老过程的治疗方案。口服从健康百岁老人中分离出的双歧杆菌亚株可以改善细胞、体液和非特异性免疫功能和小鼠肠道中的免疫屏障功能[77]。因此,分离与人类超常寿命相关的肠内细菌纯培养物,以及使用这种培养物进行FMT治疗,似乎有希望提高老年人的寿命。
参考文献
Passarino, G.; De Rango, F.; Montesanto, A.J.I.; Ageing. Human longevity: Genetics or Lifestyle? It takes two to tango. Immunity & Ageing 2016, 13, 12.
Biagi, E.; Nylund, L.; Candela, M.; Ostan, R.; Bucci, L.; Pini, E.; Nikkïla, J.; Monti, D.; Satokari, R.; Franceschi, C.J.P.o. Through ageing, and beyond: gut microbiota and inflammatory status in seniors and centenarians. PloS one 2010, 5.
Biagi, E.; Franceschi, C.; Rampelli, S.; Severgnini, M.; Ostan, R.; Turroni, S.; Consolandi, C.; Quercia, S.; Scurti, M.; Monti, D.J.C.B. Gut microbiota and extreme longevity. Current Biology 2016, 26, 1480-1485.
Kong, F.; Hua, Y.; Zeng, B.; Ning, R.; Li, Y.; Zhao, J.J.C.B. Gut microbiota signatures of longevity. Current Biology 2016, 26, R832-R833.
Lakshminarayanan, B.; Stanton, C.; O’Toole, P.; Ross, R.P.J.T.j.o.n., health; aging. Compositional dynamics of the human intestinal microbiota with aging: implications for health. The journal of nutrition, health & aging 2014, 18, 773-786.
Konturek, P.; Haziri, D.; Brzozowski, T.; Hess, T.; Heyman, S.; Kwiecien, S.; Konturek, S.; Koziel, J.J.J.P.p. Emerging role of fecal microbiota therapy in the treatment of gastrointestinal and extra-gastrointestinal diseases. J Physiol pharmacol 2015, 66, 483-491.
Wirbel, J.; Pyl, P.T.; Kartal, E.; Zych, K.; Kashani, A.; Milanese, A.; Fleck, J.S.; Voigt, A.Y.; Palleja, A.; Ponnudurai, R.J.N.m. Meta-analysis of fecal metagenomes reveals global microbial signatures that are specific for colorectal cancer. Nature medicine 2019, 25, 679-689.
Thomas, A.M.; Manghi, P.; Asnicar, F.; Pasolli, E.; Armanini, F.; Zolfo, M.; Beghini, F.; Manara, S.; Karcher, N.; Pozzi, C.J.N.m. Metagenomic analysis of colorectal cancer datasets identifies cross-cohort microbial diagnostic signatures and a link with choline degradation. Nature medicine 2019, 25, 667-678.
Lloyd-Price, J.; Arze, C.; Ananthakrishnan, A.N.; Schirmer, M.; Avila-Pacheco, J.; Poon, T.W.; Andrews, E.; Ajami, N.J.; Bonham, K.S.; Brislawn, C.J.J.N. Multi-omics of the gut microbial ecosystem in inflammatory bowel diseases. Nature 2019, 569, 655-662.
Honda, K.; Littman, D.R.J.N. The microbiota in adaptive immune homeostasis and disease. Nature 2016, 535, 75-84.
Iqbal, S.; Quigley, E.M.J.C.g.r. Progress in our understanding of the gut microbiome: implications for the clinician. Current gastroenterology reports 2016, 18, 49.
Arboleya, S.; Watkins, C.; Stanton, C.; Ross, R.P.J.F.i.m. Gut bifidobacteria populations in human health and aging. Frontiers in microbiology 2016, 7, 1204.
Tojo, R.; Suárez, A.; Clemente, M.G.; de los Reyes-Gavilán, C.G.; Margolles, A.; Gueimonde, M.; Ruas-Madiedo, P.J.W.j.o.g.W. Intestinal microbiota in health and disease: role of bifidobacteria in gut homeostasis. World journal of gastroenterology: WJG 2014, 20, 15163.
Bischoff, S.C.J.C.O.i.C.N.; Care, M. Microbiota and aging. Current Opinion in Clinical Nutrition & Metabolic Care 2016, 19, 26-30.
Eckburg, P.B.; Bik, E.M.; Bernstein, C.N.; Purdom, E.; Dethlefsen, L.; Sargent, M.; Gill, S.R.; Nelson, K.E.; Relman, D.A.J.s. Diversity of the human intestinal microbial flora. science 2005, 308, 1635-1638.
Lau, J.T.; Whelan, F.J.; Herath, I.; Lee, C.H.; Collins, S.M.; Bercik, P.; Surette, M.G.J.G.m. Capturing the diversity of the human gut microbiota through culture-enriched molecular profiling. Genome medicine 2016, 8, 72.
Lagier, J.-C.; Hugon, P.; Khelaifia, S.; Fournier, P.-E.; La Scola, B.; Raoult, D.J.C.m.r. The rebirth of culture in microbiology through the example of culturomics to study human gut microbiota. Clinical microbiology reviews 2015, 28, 237-264.
Hill, J.E.; Fernando, W.U.; Zello, G.A.; Tyler, R.T.; Dahl, W.J.; Van Kessel, A.G.J.A.E.M. Improvement of the representation of bifidobacteria in fecal microbiota metagenomic libraries by application of the cpn60 universal primer cocktail. Appl. Environ. Microbiol 2010, 76, 4550-4552.
Petrosino, J.F.; Highlander, S.; Luna, R.A.; Gibbs, R.A.; Versalovic, J.J.C.c. Metagenomic pyrosequencing and microbial identification. Clinical chemistry 2009, 55, 856-866.
Hanson, M.J.J.o.d.o.o.h.; disease. The birth and future health of DOHaD. Journal of developmental origins of health and disease 2015, 6, 434-437.
Goulet, O.J.N.r. Potential role of the intestinal microbiota in programming health and disease. Nutrition reviews 2015, 73, 32-40.
Wallace, J.; Gohir, W.; Sloboda, D.J.J.o.d.o.o.h.; disease. The impact of early life gut colonization on metabolic and obesogenic outcomes: what have animal models shown us? Journal of developmental origins of health and disease 2016, 7, 15-24.
Lakshminarayanan, B.; Stanton, C.; O’Toole, P.; Ross, R.P.J.T.j.o.n., health; aging. Compositional dynamics of the human intestinal microbiota with aging: implications for health. The journal of nutrition, health & aging 2014, 18, 773-786.
O’Toole, P.W.; Jeffery, I.B.J.S. Gut microbiota and aging. Science 2015, 350, 1214-1215.
Perez Martinez, G.; Bäuerl, C.; Collado, M.J.B.M. Understanding gut microbiota in elderly's health will enable intervention through probiotics. Beneficial Microbes 2014, 5, 235-246.
Rondanelli, M.; Giacosa, A.; Faliva, M.A.; Perna, S.; Allieri, F.; Castellazzi, A.M.J.W.J.o.C.C.W. Review on microbiota and effectiveness of probiotics use in older. World Journal of Clinical Cases: WJCC 2015, 3, 156.
Rivière, A.; Selak, M.; Lantin, D.; Leroy, F.; De Vuyst, L.J.F.i.m. Bifidobacteria and butyrate-producing colon bacteria: importance and strategies for their stimulation in the human gut. Frontiers in microbiology 2016, 7, 979.
Claesson, M.J.; O'Sullivan, O.; Wang, Q.; Nikkilä, J.; Marchesi, J.R.; Smidt, H.; De Vos, W.M.; Ross, R.P.; O'Toole, P.W.J.P.o. Comparative analysis of pyrosequencing and a phylogenetic microarray for exploring microbial community structures in the human distal intestine. PloS one 2009, 4.
Drago, L.; Toscano, M.; Rodighiero, V.; De Vecchi, E.; Mogna, G.J.J.o.c.g. Cultivable and pyrosequenced fecal microflora in centenarians and young subjects. Journal of clinical gastroenterology 2012, 46, S81-S84.
Jackson, M.A.; Jeffery, I.B.; Beaumont, M.; Bell, J.T.; Clark, A.G.; Ley, R.E.; O’Toole, P.W.; Spector, T.D.; Steves, C.J.J.G.m. Signatures of early frailty in the gut microbiota. Genome medicine 2016, 8, 8.
Jeffery, I.B.; Lynch, D.B.; O'toole, P.W.J.T.I.j. Composition and temporal stability of the gut microbiota in older persons. The ISME journal 2016, 10, 170.
Rehman, T.J.E., Metabolic; Targets, I.D.-D. Role of the gut microbiota in age-related chronic inflammation. Endocrine, Metabolic & Immune Disorders-Drug Targets (Formerly Current Drug Targets-Immune, Endocrine & Metabolic Disorders) 2012, 12, 361-367.
Friedland, R.P.J.J.o.A.s.D. Mechanisms of molecular mimicry involving the microbiota in neurodegeneration. Journal of Alzheimer's Disease 2015, 45, 349-362.
Magnusson, K.; Hauck, L.; Jeffrey, B.; Elias, V.; Humphrey, A.; Nath, R.; Perrone, A.; Bermudez, L.J.N. Relationships between diet-related changes in the gut microbiome and cognitive flexibility. Neuroscience 2015, 300, 128-140.
Paun, A.; Danska, J.S.J.P.d. Modulation of type 1 and type 2 diabetes risk by the intestinal microbiome. Pediatric diabetes 2016, 17, 469-477.
Sanduzzi Zamparelli, M.; Compare, D.; Coccoli, P.; Rocco, A.; Nardone, O.M.; Marrone, G.; Gasbarrini, A.; Grieco, A.; Nardone, G.; Miele, L.J.I.j.o.m.s. The metabolic role of gut microbiota in the development of nonalcoholic fatty liver disease and cardiovascular disease. International journal of molecular sciences 2016, 17, 1225.
Pope, J.L.; Tomkovich, S.; Yang, Y.; Jobin, C.J.T.R. Microbiota as a mediator of cancer progression and therapy. Translational Research 2017, 179, 139-154.
Graham, C.; Mullen, A.; Whelan, K.J.N.r. Obesity and the gastrointestinal microbiota: a review of associations and mechanisms. 2015, 73, 376-385.
Janssen, A.W.; Kersten, S.J.T.F.J. The role of the gut microbiota in metabolic health. The FASEB Journal 2015, 29, 3111-3123.
Candela, M.; Biagi, E.; Brigidi, P.; O’Toole, P.W.; De Vos, W.M.J.M.o.a.; development. Maintenance of a healthy trajectory of the intestinal microbiome during aging: a dietary approach. Mechanisms of ageing and development 2014, 136, 70-75.
Saraswati, S.; Sitaraman, R.J.F.i.m. Aging and the human gut microbiota—from correlation to causality. Frontiers in microbiology 2015, 5, 764.
Biagi, E.; Nylund, L.; Candela, M.; Ostan, R.; Bucci, L.; Pini, E.; Nikkïla, J.; Monti, D.; Satokari, R.; Franceschi, C.J.P.o. Through ageing, and beyond: gut microbiota and inflammatory status in seniors and centenarians. PloS one 2010, 5.
Rampelli, S.; Candela, M.; Turroni, S.; Biagi, E.; Collino, S.; Franceschi, C.; O'Toole, P.W.; Brigidi, P.J.A. Functional metagenomic profiling of intestinal microbiome in extreme ageing. Aging 2013, 5, 902.
Park, S.-H.; Kim, K.-A.; Ahn, Y.-T.; Jeong, J.-J.; Huh, C.-S.; Kim, D.-H.J.B.m. Comparative analysis of gut microbiota in elderly people of urbanized towns and longevity villages. BMC microbiology 2015, 15, 49.
Lowry, C.A.; Smith, D.G.; Siebler, P.H.; Schmidt, D.; Stamper, C.E.; Hassell, J.E.; Yamashita, P.S.; Fox, J.H.; Reber, S.O.; Brenner, L.A.J.C.e.h.r. The microbiota, immunoregulation, and mental health: implications for public health. Current environmental health reports 2016, 3, 270-286.
Nehra, V.; Allen, J.M.; Mailing, L.J.; Kashyap, P.C.; Woods, J.A.J.P. Gut microbiota: modulation of host physiology in obesity. Physiology 2016, 31, 327-335.
Zhang, X.; Zheng, X.; Yuan, Y.J.D.d.t. Treatment of insulin resistance: straight from the gut. Drug discovery today 2016, 21, 1284-1290.
Keenan, M.J.; Marco, M.L.; Ingram, D.K.; Martin, R.J.J.A. Improving healthspan via changes in gut microbiota and fermentation. Age 2015, 37, 98.
Patrignani, P.; Tacconelli, S.; Bruno, A.J.J.o.c.g. Gut microbiota, host gene expression, and aging. Journal of clinical gastroenterology 2014, 48, S28-S31.
Ma, S.; Sun, S.; Geng, L.; Song, M.; Wang, W.; Ye, Y.; Ji, Q.; Zou, Z.; Wang, S.; He, X.J.C. Caloric restriction reprograms the single-cell transcriptional landscape of Rattus norvegicus aging. Cell 2020.
Mathur, R.; Barlow, G.M.J.E.R.o.G.; Hepatology. Obesity and the microbiome. Expert Review of Gastroenterology & Hepatology 2015, 9, 1087-1099.
Damms-Machado, A.; Mitra, S.; Schollenberger, A.E.; Kramer, K.M.; Meile, T.; Königsrainer, A.; Huson, D.H.; Bischoff, S.C.J.B.r.i. Effects of surgical and dietary weight loss therapy for obesity on gut microbiota composition and nutrient absorption. BioMed research international 2015, 2015.
Remely, M.; Tesar, I.; Hippe, B.; Gnauer, S.; Rust, P.; Haslberger, A.J.B.m. Gut microbiota composition correlates with changes in body fat content due to weight loss. Beneficial microbes 2015, 6, 431-439.
John, G.K.; Mullin, G.E.J.C.o.r. The gut microbiome and obesity. Current oncology reports 2016, 18, 45.
Zhang, C.; Li, S.; Yang, L.; Huang, P.; Li, W.; Wang, S.; Zhao, G.; Zhang, M.; Pang, X.; Yan, Z.J.N.c. Structural modulation of gut microbiota in life-long calorie-restricted mice. Nature communications 2013, 4, 1-10.
Jonkers, D.M.J.B.p.; gastroenterology, r.C. Microbial perturbations and modulation in conditions associated with malnutrition and malabsorption. Best practice & research Clinical gastroenterology 2016, 30, 161-172.
Sánchez, B.; Delgado, S.; Blanco-Míguez, A.; Lourenço, A.; Gueimonde, M.; Margolles, A.J.M.n.; research, f. Probiotics, gut microbiota, and their influence on host health and disease. Molecular nutrition & food research 2017, 61, 1600240.
Eloe-Fadrosh, E.A.; Brady, A.; Crabtree, J.; Drabek, E.F.; Ma, B.; Mahurkar, A.; Ravel, J.; Haverkamp, M.; Fiorino, A.-M.; Botelho, C.J.M. Functional dynamics of the gut microbiome in elderly people during probiotic consumption. MBio 2015, 6, e00231-00215.
Sanders, M.E.J.B.m. Probiotics and microbiota composition. BMC medicine 2016, 14, 82.
Kim, H.M.; Lee, D.E.; Park, S.D.; Kim, Y.-T.; Kim, Y.J.; Jeong, J.W.; Jang, S.S.; Ahn, Y.-T.; Sim, J.-H.; Huh, C.-S.J.J.M.B. Oral administration of Lactobacillus plantarum HY7714 protects hairless mouse against ultraviolet B-induced photoaging. J. Microbiol. Biotechnol 2014, 24, 1583-1591.
Satoh, T.; Murata, M.; Iwabuchi, N.; Odamaki, T.; Wakabayashi, H.; Yamauchi, K.; Abe, F.; Xiao, J.J.B.M. Effect of Bifidobacterium breve B-3 on skin photoaging induced by chronic UV irradiation in mice. Beneficial Microbes 2015, 6, 497-504.
Lee, D.E.; Huh, C.-S.; Ra, J.; Choi, I.-D.; Jeong, J.-W.; Kim, S.-H.; Ryu, J.H.; Seo, Y.K.; Koh, J.S.; Lee, J.-H.J.J.M.B. Clinical evidence of effects of Lactobacillus plantarum HY7714 on skin aging: a randomized, double blind, placebo-controlled study. J Microbiol Biotechnol 2015, 25, 2160-2168.
Jeong, J.-J.; Kim, K.; Hwang, Y.-J.; Han, M.; Kim, D.-H.J.B.M. Anti-inflammaging effects of Lactobacillus brevis OW38 in aged mice. Beneficial Microbes 2016, 7, 707-718.
Thushara, R.M.; Gangadaran, S.; Solati, Z.; Moghadasian, M.H.J.F.; function. Cardiovascular benefits of probiotics: a review of experimental and clinical studies. Food & function 2016, 7, 632-642.
Nakagawa, H.; Shiozaki, T.; Kobatake, E.; Hosoya, T.; Moriya, T.; Sakai, F.; Taru, H.; Miyazaki, T.J.A.c. Effects and mechanisms of prolongevity induced by Lactobacillus gasseri SBT2055 in Caenorhabditis elegans. Aging cell 2016, 15, 227-236.
Grompone, G.; Martorell, P.; Llopis, S.; González, N.; Genovés, S.; Mulet, A.P.; Fernández-Calero, T.; Tiscornia, I.; Bollati-Fogolín, M.; Chambaud, I.J.P.o. Anti-inflammatory Lactobacillus rhamnosus CNCM I-3690 strain protects against oxidative stress and increases lifespan in Caenorhabditis elegans. PloS one 2012, 7.
Komura, T.; Ikeda, T.; Yasui, C.; Saeki, S.; Nishikawa, Y.J.B. Mechanism underlying prolongevity induced by bifidobacteria in Caenorhabditis elegans. Biogerontology 2013, 14, 73-87.
Matsumoto, M.; Kurihara, S.; Kibe, R.; Ashida, H.; Benno, Y.J.P.o. Longevity in mice is promoted by probiotic-induced suppression of colonic senescence dependent on upregulation of gut bacterial polyamine production. PloS one 2011, 6.
Doron, S.; Snydman, D.R.J.C.I.D. Risk and safety of probiotics. Clinical Infectious Diseases 2015, 60, S129-S134.
Boyle, R.J.; Robins-Browne, R.M.; Tang, M.L.J.T.A.j.o.c.n. Probiotic use in clinical practice: what are the risks? The American journal of clinical nutrition 2006, 83, 1256-1264.
Devkota, S.J.S. Prescription drugs obscure microbiome analyses. Science 2016, 351, 452-453.
Maurice, C.F.; Haiser, H.J.; Turnbaugh, P.J.J.C. Xenobiotics shape the physiology and gene expression of the active human gut microbiome. Cell 2013, 152, 39-50.
Sandhu, K.V.; Sherwin, E.; Schellekens, H.; Stanton, C.; Dinan, T.G.; Cryan, J.F.J.T.R. Feeding the microbiota-gut-brain axis: diet, microbiome, and neuropsychiatry. Translational Research 2017, 179, 223-244.
Gupta, S.; Allen-Vercoe, E.; Petrof, E.O.J.T.a.i.g. Fecal microbiota transplantation: in perspective. Therapeutic advances in gastroenterology 2016, 9, 229-239.
Dąbrowska, K.; Witkiewicz, W.J.F.i.m. Correlations of host genetics and gut microbiome composition. Frontiers in microbiology 2016, 7, 1357.
Youngster, I.; Sauk, J.; Pindar, C.; Wilson, R.G.; Kaplan, J.L.; Smith, M.B.; Alm, E.J.; Gevers, D.; Russell, G.H.; Hohmann, E.L.J.C.I.D. Fecal microbiota transplant for relapsing Clostridium difficile infection using a frozen inoculum from unrelated donors: a randomized, open-label, controlled pilot study. Clinical Infectious Diseases 2014, 58, 1515-1522.
Yang, H.; Liu, A.; Zhang, M.; Ibrahim, S.A.; Pang, Z.; Leng, X.; Ren, F.J.C.m. Oral administration of live Bifidobacterium substrains isolated from centenarians enhances intestinal function in mice. Current microbiology 2009, 59, 439-445.





