艰难梭菌感染的实验室诊断与检测方法
作者:林珊 宋小军 宋广忠 金大智
【摘要】艰难梭菌是一种专性厌氧革兰阳性芽孢菌,也是抗生素相关腹泻的主要病原菌。长期或不规范使用抗生素、免疫抑制剂等造成产毒型艰难梭菌过度繁殖并释放毒素是引起机体感染的主要原因。近年来,随着高毒力菌株在世界范围内的暴发和流行,艰难梭菌感染的发病率与病死率均呈明显上升趋势。我国临床对艰难梭菌感染仍未引起足够重视,大部分临床实验室尚未开展检测,且实验室检测方法各不相同,使得临床艰难梭菌感染病例的诊断面临较大挑战。本文从艰难梭菌感染的致病因子、实验室诊断和检测方法等角度进行综述,为后续进一步推进我国艰难梭菌感染临床实验室诊断及防控提供参考。
【关键词】艰难梭菌;诊断标准;实验室检测方法
艰难梭菌(Clostridium difficile,CD)是一种专性厌氧革兰阳性芽孢菌,广泛分布于人和动物的肠道以及环境中[1]。近年来,由于高毒力株NAP1/RT027/BI(脉冲场凝胶电泳分型NAP1,PCR核糖体分型027,限制性内切酶分型BI)的出现,艰难梭菌感染(Clostridium difficileinfection,CDI)呈现出高发病率与高死亡率态势[2]。2013年美国疾病预防与控制中心(Center for disease control and prevention,CDC)发布美国抗生素耐药性威胁报告,将CD列为“极危(Urgent)”等级[3],其警示级别高于“超级细菌”的“严重(Serious)”等级。目前,CD已对多种抗生素产生耐药性[4],同时当前的分子流行病学研究数据表明我国CDI流行病学特征具有自身的特点[5]。虽然地区间的临床发病率存在差异,但是不可否认临床存在一定比例的CDI病例。而且随着研究的深度及广度不断被提升,CDI将越来越会受到重视。因此,临床快速、准确地诊断CDI,及时采取有效的治疗措施,对改善患者预后状况及节省国家医疗支出具有重要意义。本文对CDI的致病因子、实验室诊断和检测方法等方面进行综述,以加深临床及实验室对于CDI的理解并引起重视。
一、艰难梭菌的致病因子
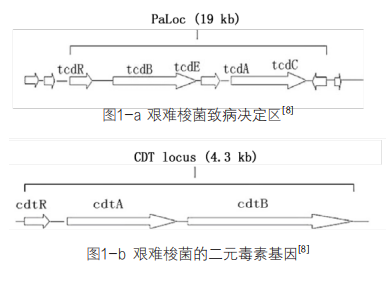
CD可分为产毒型和非产毒型菌株,其中产毒型菌株主要产生两种毒素:毒素A(tcdA)和毒素B(tcdB)。tcdA与tcdB分别为肠毒素与细胞毒素,二者主要通过破坏肠黏膜的完整性导致疾病的发生,感染时可出现腹泻和假膜性结肠炎[6-7]。编码毒素A、B的tcdA、tcdB基因分别位于具有19.6kb长度的致病性决定区域(Pathogenicity locus,PaLoc)[8](图1-a),并且该功能区还含有两个调节基因(tcdC和tcdD)以及一个孔蛋白基因(tcdE)。
尽管tcdA/B可单独引起CDI,但临床上一些“高毒力”的菌株如RT027(ST1)和/或 RT078(ST11)型还会产生二元毒素(Binary toxin)或将其称为艰难梭菌转移酶(Clostridium difficile transferase,CDT)[9],CDT位于具有4.3kb长度的CDT基因簇(图1-b)并由两个亚基(cdtA和cdtB)组成[8]。CDT可能是通过增强细胞毒性和CD在肠道的黏附力使其毒力增强[10]。粪便中CD的毒素水平与疾病严重程度之间存在潜在的相关性[11];高龄(>65岁)、长期暴露于广谱抗菌药物、使用免疫抑制剂、长期住院[12-15]等因素造成机体免疫力低下,会加大CD定植与感染的风险,是患者出现CDI的易感因素。
二、艰难梭菌感染的诊断标准
2010年美国卫生保健流行病学会与传染病学会(Society for Healthcare Epidemiology of America,SHEA)联合发布《成人艰难梭菌感染临床实践指南》。指南表明CDI的诊断原则包含临床表现和实验室检测结果两部分,其中临床表现为:24小时或更短时间内腹泻(稀便、黏液便或水样便等不成形便)3次或3次以上;实验室检测结果包含(1)粪便检测出产毒型CD或CD毒素检测结果呈阳性(tcd A-B+或tcd A+B+);(2)结肠镜或组织病理学检查发现假膜并证实为伪膜性肠炎[16]。
本指南为临床实验室诊断提供了重要参考依据,也为我国CDI的防控与治疗带来很好的启示与指导作用。2017年我国发布了《中国成人艰难梭菌感染诊断和治疗专家共识》[5],将CDI诊断标准更新为:患者出现中至重度腹泻或肠梗阻,并满足以下任一条件:(1)粪便检测CD毒素或产毒型CD结果阳性;(2)内镜下或组织病理检查显示伪膜性肠炎。
三、艰难梭菌的实验室检测方法
(一)艰难梭菌的菌株检测
1. 厌氧培养:1979年George等研制出CCFA培养基(Cefoxitin cycloserine fructose agar,CCFA),经厌氧培养48~72小时后,培养基出现“马粪”气味等典型特征,并且在紫外光照射下菌落呈现出黄绿色荧光[17]。加入胆红素盐(尤其是牛磺胆酸钠)可以克服培养难度大、分离困难等特点,促进CD芽孢萌发,提高阳性检出率[18]。ChromIDC. difficileagar(CDIF),是一种用牛磺胆酸钠与显色剂制成的新型培养基,在24小时内可以分离并准确鉴定CD菌株,现已被商业化使用[19-20]。
目前,大多数显色培养基来源于国外,售价昂贵且常无法连续供货。本实验室研发出目前国内唯一具有自主知识产权的C. difficile Chromogenic Agar(CDCA)显色培养基,具备良好的灵敏度与特异性、可以更快速地分离CD,并且其成本与国外产品的价格相比节省了约1/3[21],在CD临床粪便标本分离与培养方面具有广阔的应用前景。虽然厌氧培养法的灵敏度与特异性较高,但操作繁琐、耗时长,不能快速有效地为临床诊断提供依据,常需与其他检测方法配合使用。
2. 谷氨酸脱氢酶检测:谷氨酸脱氢酶(Glutamate dehydrogenase,GDH)是CD细胞壁的抗原性蛋白质之一,这种具高特异性且大量表达的代谢酶可协助CD在肠道内定植[22]。Barbut等[23]于2000年首次将GDH应用于CD相关性疾病的诊断,并对304份疑似CDI的粪便标本展开检测,研究结果指出GDH呈阴性结果可准确地排除CDI(阴性预测值为99.6%)。采用Meta分析针对共计2852份病例的GDH抗原检测结果进行研究,结果表明GDH检测法的灵敏度与特异度皆为96%[24]。Yoo等[25]开展150份粪便标本的GDH抗原检测试验,结果显示GDH的灵敏度与特异性均高于CD培养法。
通常使用酶免疫方法直接检测粪便标本中的GDH抗原,具有快速,价格低廉且阴性预测值高等优点,适合进行CDI初筛试验[26-27]。我国临床实验室通过原核表达GDH,建立GDH胶体金免疫层析检测法,成功研制出胶体金试纸,可以快速、准确、方便地诊断CDI[28]。GDH检测结果阴性即可排除CDI[23],但由于GDH是CD菌株表面的一种共同抗原,阳性结果还需经过毒素检测进一步确认是否为产毒型菌株。
(二)艰难梭菌毒性检测
1.细胞毒性试验:细胞毒性试验(Cell cytotoxicity assay,CCTA)即细胞毒性中和试验(Cell cytotoxin neutralization assay,CCNA),一般与CD的产毒素培养(Toxigenic culture,TC)同时进行,二者被认为是30年来CDI诊断的“金标准”[29]。TC可以评估CD的产毒素能力,CCTA则可以直接判断粪便标本中是否含有CD毒素[5]。毒性试验分为两步:(1)将CD分离并接种至选择性培养基上生长,(2)分离生长物置于肉汤中再次培养,取上清液过滤后,加入Vero或Hep2等细胞共同孵育,评估毒素是否引起细胞病变作用(Cytopathic effect,CPE),最后再加入抗tcdA、tcdB的“中和抗毒素”;若超过50%的病变细胞在48小时内被中和,则判定为CCTA阳性[5、30、31]。
在一项研究的数据显示,通过CCTA检测CD的灵敏度与特异度分别为80%~100%、98%~99%[32]。然而由于CCTA的灵敏度不高,若将其视为唯一的诊断方法,那么有相当一部分CDI病例(31.9%)包括重型病例将会被漏诊[33]。细胞培养对设备要求十分苛刻,需要可靠的抗毒素才能开展中和试验,并且检测可能需要几天才能完成,因此该诊断方法不适合在临床实验室中被常规使用[34]。
2. 毒素免疫检测:目前国内外最常用的毒素免疫检测方法包括酶免疫分析(Enzyme immunoassay,EIA)与酶联免疫吸附试验法(Enzyme linked immunosorbent assay,EIASA)。这两种方法通过抗原-抗体反应直接检测腹泻粪便标本中的tcdA/B[35],具有特异性高,可以区分出产毒型与非产毒型菌株等优势[16],操作简便,可在数分钟至1小时内即可得出检测结果[36]。
但是多项研究显示,EIAs诊断方法的稳定性不佳,Mohan等的实验结果显示EIAs的灵敏度在50%至90%间[37]。Sandlund等[38]针对PCR检测结果呈阳性的45份疑似CDI病例的粪便标本再进行EIAs试验,结果表明其灵敏度与特异度分别为57.6%、100.0%。灵敏度不稳定的原因可能是由于不同菌株毒素的抗原变异、标本储存和运输条件的影响以及不同实验室操作技术的差异等多种因素造成[39]。若单独使用此法进行CDI诊断病例,可能会造成轻中度患者漏诊。
(三)艰难梭菌毒素的核酸检测
核酸分子诊断目前在临床微生物诊断领域已被广泛使用,具有特异性好、灵敏度高、检测结果稳定等特点[40]。近年来,通过核酸扩增技术(Nucleic acid amplification test,NAAT)诊断CDI病例的方法也备受青睐,多项研究结果表明NAATs不但提高了灵敏度而且缩短了检测周期[41]。本节主要介绍目前临床常用的技术如PCR、LAMP等。
1. PCR法:PCR法主要针对位于PaLoc中的tcdA、tcdB、tcdC基因、二元毒素基因CDT以及磷酸丙糖异构酶(Triosephosphate isomerase,tpi)基因来诊断CDI。实时荧光定量PCR(Quantitative Real-time PCR,qRT-PCR)实现了传统PCR技术从定性到定量的飞跃并可避免凝胶电泳产物污染标本,目前国内已有多个临床实验室使用此法诊断CDI病例[42-44]。本实验室于2013年开发出具有自主知识产权的多重qRT-PCR诊断产毒型毒素检测和鉴定方法[45]。采用QF-PCR结合Allglo新型探针技术,对浙江杭州地区180份腹泻患者的粪便标本进行检测,其结果较直接测序法准确、可靠且重复性好。在此基础上王等[46]使用成本更低的Taqman替代Allglo探针,并与BDMAX C.difficile Assay试剂盒进行临床应用性能对比评估,结果表明两种方法的一致性较好,且符合率高(Kappa=0.922,P<0.01),为我国CDI的快速诊断提供技术储备。
普通PCR操作繁琐且易污染,qRT-PCR灵敏度高、特异性强、重复性好。但荧光PCR仪售价较为昂贵,并且较高的灵敏度高也带来了一些问题如:无法区分CD定植与感染、不必要地使用抗菌药物、过高估计医院CDI 的发病率等[47]。故采用此方法检测时,还应该结合患者的临床症状作出诊断。
2. LAMP:环介导等温扩增(Loop-mediated isothermalamplification,LAMP)是一种新型核酸等温扩增技术,利用高置换链活性的DNA聚合酶和一组专门设计的引物,在等温条件下高效地扩增目标基因,目前已被用作传染性和非传染性疾病诊断的工具[48-49]。针对PubMed等数据库中应用LAMP技术诊断CDI病例的文献进行Meta分析,Wei等[50]认为LAMP适合作为快速、有效且可靠的CDI独立诊断方法。Neuendorf等[51]使用LAMP的延展技术检测了989个粪便标本,与TC检测法相比其敏感度与特异度皆为91%。LAMP检测CD耗时短,操作简便,不需要大型仪器,检测周期可被缩减至2小时内,为及时隔离CDI患者提供了基础[52]。但在此方法建立过程中,为避免出现非特异性扩增而降低效率,需要人工参与引物设计并且要求极高。
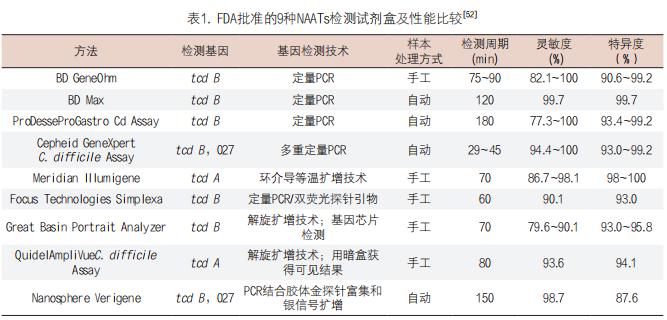
3. NAATs:2013年《美国胃肠病学杂志》在关于CDI的诊断指南中提出,NAATs可作为一种独立的检测方法开展产毒性CD检测[53]。截至目前, 美国食品药品监督管理局(Food and Drug Administration,FDA)已批准9种CD分子检测平台上市(见表1),包括Cepheid GeneXpert C. difficile Assay、BD GeneOhm等常用的高效检测系统[54]。
GeneXpert C.difficile Assay是被中国FDA批准的唯一用于CDI临床诊断的试剂盒,现已被国内临床及实验室应用[55]。尽管NAATs的灵敏度及特异性非常高,总体上优于其他CDI诊断方法。但是临床收费过高、仪器成本昂贵等因素是目前制约NAATs大规模应用的主要原因。使用本实验室开发的qRT-PCR检测法分析176份腹泻患者粪便标本中的CD毒素,并以XpertC. dffile/Epi检测法为标准进行临床应用性能对比评估,研究数据表明qRT-PCR检测法的灵敏度与特异度分别为91.7%、100.0%,同时两种方法检测方法的一致性较好(Kappa =0.950,P<0.001)[55]。qRT-PCR检测法诊断CDI病例成本低、耗时短,可提高临床报告的效率,作为国产化的检测技术极具发展前景。
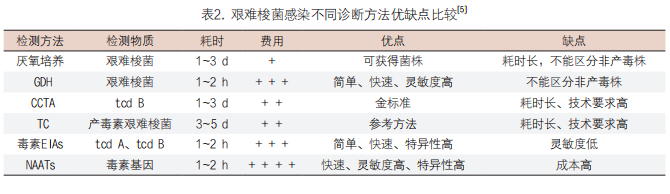
4. 联合检测:不同的诊断方法之间各有优缺点[5](见表2),由于无法区分CDI与无症状携带者以及评估CDI病例的严重程度,目前对于最佳的实验室检测方法仍具争议[56]。综合考虑不同方法的灵敏度、特异性、耗时、费用等因素,欧洲临床微生物与感染性疾病学会(艰难梭菌研究组)[57]和中国医师协会(检验医学专家委员会)皆将两步法或三步法列入临床标准检测流程[5]。
(1)两步法:两步法[5](图2)是指对临床标本同时进行GDH和tcdA/B EIAs检测。双阳性结果判定为CDI阳性,双阴性结果判定为CDI阴性;对于GDH阳性、tcdA/B EIAs呈阴性的标本需应用CCTA、TC或NAATs进一步确证,阴性结果可排除CDI,阳性结果需结合病例的临床症状作出诊断。
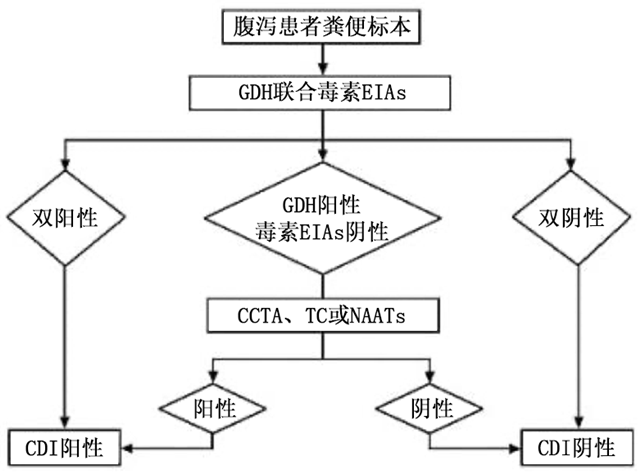
图2. 二步法诊断流程[5]
(2)三步法:三步法[5](图3)同样使用了高灵敏度的GDH进行初筛试验,阴性结果即可排除CDI。对于结果为阳性的标本进行tcdA/B EIAs检测,tcdA/B EIAs阳性判定为CDI,阴性的标本需再运用CCTA、TC或NAATs等检测方法进行评估,以确认病例是CDI或CD携带者。相较于两步法,三步法似乎显得有点繁琐,但是一些毒素量低于阈值或tcdA/B EIAs检测结果为假阴性的CDI病例可以被准确诊断,及时给予相应的临床治疗。
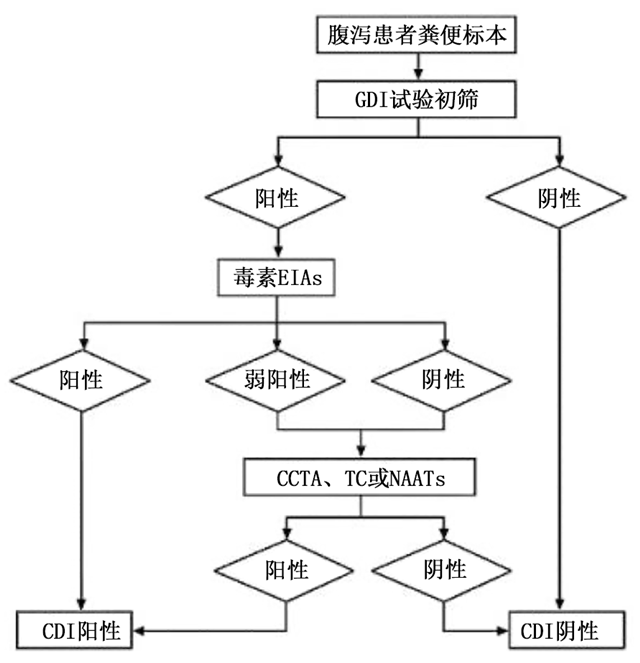
图3. 三步法诊断流程[5]
四、艰难梭菌检测的新方法
目前,CD已对多种抗生素产生耐药性,并且我国的CDI流行病学特征具有多样性,使CDI的诊断与防控形势更加严峻。本文提供几种CD检测的新方法,以期为基层临床实验室快速诊断病例提供新的思路。
1. 电子鼻:电子鼻(ElectronicNose,e-Nose)又称气味扫描仪,是一种由传感系统和自动化识别系统组成的电子感官仪器,具有分析速度快、识别效果准确、评价结果客观等优点,已成功在食品工业、环境监测、医学诊断等领域开展商业化检测[58-59]。在医疗领域主要通过e-Nose中的传感器阵列分析细菌培养液中微生物代谢的挥发性产物(Volatile Organic Compounds,VOCs)及人体的代谢产物,可以了解伤口内病原菌的类型进而开展疾病的诊断与治疗[60]。2014年,英国莱斯特大学研究者提出可通过鉴别CD独特的VOCs快速诊断CDI[61]。Chan等[62]于2015年首次使用e-Nose技术诊断CDI,结果显示灵敏度、特异度及准确度分别为80%、85%、84%。国内临床实验室尚未有使用e-Nose检测CD的报道,虽然此技术的灵敏度与特异性较高,但均低于分子检测技术等常规方法。期待未来改进此技术能更快速、准确地诊断出CDI病例,协助临床医生采取更有针对性的治疗。
2. 基质辅助激光解析电离飞行时间质谱:每种微生物都由自身独特的肽或蛋白质组成,可以理解为是这些微生物专属的“指纹”。通过基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)检测指纹图谱,经软件处理并与微生物数据库进行比对分析,可在几分钟之内完成对微生物种、属水平的鉴定[63]。Reil等[64]于2011年首次将此技术应用于CD分型,并指出此方法能够有效鉴定RT001、027和078型菌株。目前国内CD主要的流行型别为RT017型菌株,Li等[65]认为MALDI-TOF MS是鉴定RT017型菌株快速且可靠的工具。以基因测序作为参比方法进行对比时,此技术的鉴定能力要强于传统的生化鉴定方法,但如何控制不同系统在鉴定性能上可能出现的差异等问题,仍需研究者们进一步探索。
3. 实时细胞分析技术:实时细胞分析技术(Real-time cellular analysis,RTCA)通过细胞培养板中的感受器检测细胞电阻变化来检测细胞毒性[66]。2010年,Ryder等[67]通过RTCA检测了300份腹泻粪便标本中的tcdB,数据显示其灵敏度与特异度分别为87.5%、99.6%,并且检测结果高于EIAs但低于PCR检测法。
本实验室基于此研究,于2014年开发出RTCA二代检测系统[68],以TC检测法为参考标准,针对401份腹泻粪便标本中的tcdB进行检测,研究结果表明第二代RTCA的灵敏度与特异度分别为96.2%、99.7%。该系统更容易操作,检测tcdB的灵敏度(0.12ng/mL)较上代(0.2ng/mL)提高了约2倍,并且与RT-PCR检测法的一致性较好。同时检测结果显示CD的毒素浓度与临床诊断程度呈正相关,因此可为CDI伴随性诊断及临床严重程度的评估提供有价值的工具。
五、展望
如何快速、准确地诊断CDI病例仍是临床面临的巨大挑战,不同的诊断方法均具有明显的优点和缺点。本文主要介绍了一些常见的CDI实验室诊断及检测方法。近年来,高产毒株在世界范围内的流行与临床上严重病例的增加,给医疗保健体系带来了巨大的经济负担。目前,国外已建立起系统的CDI监测网络并且拥有完善的CDI临床诊疗指南。我国临床艰难梭菌研究起步较晚,检测技术及防控措施皆未得到共识。迄今为止,国内绝大多数医院检验科仍未开展CDI的实验室检测,临床标本数据采集困难,给相关科研工作带来了不少考验。
临床实验室选择合适的检测方法对快速、准确诊断出CDI有着重大影响,也是我国CDI防控的关键步骤之一。结合国内人口众多、抗菌药物滥用严重、人口老龄化趋势加快、医疗卫生条件仍有待改善的基本情况,可推测我国CDI现状不容乐观。综上,通过本文的综述,未来期望(1)通过大规模分子流行病研究获得更加完整的我国CDI分子流行病学特征,为临床诊断技术的使用提供充足的佐证;(2)通过多项基础研究深入地了解我国临床CD的致病机制,不断寻求更合理、更经济、更有效、更快速的检测技术;(3)临床医务人员应提高对CDI认识,早发现、早治疗,减轻患者的痛苦及经济负担;(4)临床检验科应尽快开展CD的检测工作并全面提升检测能力,为我国CDI的防控及治疗奠定基础。
参考文献
CzepielJ,DróżdżM,Pituch H, et al. Clostridium difficile infection: review.[J]. Eur J Clin Microbiol Infect Dis. 2019, 38(7):1211-1221
Imad Kansau,AmiraBarketi-Klai,MarcMonot, et al. Deciphering Adaptation Strategies of the Epidemic Clostridium difficile 027 Strain during Infection through In Vivo Transcriptional Analysis.[J]. PLoS ONE, 2017,11(6).
Evans CharlesnikaT, SafdarNasia. Current Trends in the Epidemiology and Outcomes of Clostridium difficileInfection.[J]. Clinical infectious diseases: an official publication of the Infectious Diseases Society of America, 2015, 60 Suppl 2.
BanawasSS.Clostridium difficile Infections: A Global Overview of Drug Sensitivity and Resistance Mechanisms.[J]. Biomed Res Int. 2018; 2018:8414257
程敬伟, 刘文恩, 马小军, 等. 中国成人艰难梭菌感染诊断和治疗专家共识[J]. 协和医学杂志, 2017, 8(Z1):131-138.
Aktories K, Schwan C, Jank T. Clostridium difficile Toxin Biology.[J]. Annu Rev Microbiol. 2017; 71:281-307.
Peng Z, Liu S, Meng X, et al. Genome characterization of a novel binary toxin-positive strain of Clostridium difficile and comparison with the epidemic 027 and 078 strains.[J]. Gut Pathog. 2017;9:42.
Rupnik M, Wilcox MH, Gerding DN. Clostridium difficile infection: new developments in epidemiology and pathogenesis.[J]. Nat Rev Microbiol. 2009; 7(7):526-536.
Abughanimeh O, Qasrawi A, Kaddourah O, et al. Clostridium difficile infection in oncology patients: epidemiology, pathophysiology, risk factors, diagnosis, and treatment.[J]. Hosp Pract(1995). 2018;46(5):266-277.
杨靖, 赵建宏. 艰难梭菌感染实验室诊断的研究进展[J]. 临床荟萃, 2018, 33(05):373-376.
Carroll KC, Mizusawa M. Laboratory Tests for the Diagnosis of Clostridium difficile.[J]. Clin Colon Rectal Surg. 2020; 33(2):73-81.
Leibovici-Weissman Y, Atamna A, Schlesinger A, et al. Risk factors for short- and long-term mortality in very old patients with Clostridium difficile infection: A retrospective study.[J]. GeriatrGerontol Int. 2017; 17(10):1378-1383.
Balram B, Battat R, Al-Khoury A, et al. Risk Factors Associated with Clostridium difficile Infection in Inflammatory Bowel Disease: A Systematic Review and Meta-Analysis.[J]. J Crohns Colitis. 2019; 13(1):27-38.
Pouch SM, Friedman-Moraco RJ. Prevention and Treatment of Clostridium difficile-Associated Diarrhea in Solid Organ Transplant Recipients.[J]. Infect Dis Clin North Am. 2018; 32(3):733-748.
Prasad N, Labaze G, Kopacz J, et al. Asymptomatic rectal colonization with carbapenem-resistant Enterobacteriaceae and Clostridium difficile among residents of a long-term care facility in New York City.[J].Am J Infect Control. 2016; 44(5):525-532.
Cohen S H, Gerding D N, Johnson S, et al. Clinical practice guidelines for Clostridium difficile infection in adults: 2010 update by the society for healthcare epidemiology of America (SHEA) and the infectious diseases society of America (IDSA)[J]. Infection Control & Hospital Epidemiology, 2010, 31(5): 431-455.
George WL, Sutter VL, Citron D, et al. Selective and differential medium for isolation of Clostridium difficile.[J]. J Clin Microbiol. 1979; 9(2):214-219.
Connor MC, McGrath JW, McMullan G, et al. Development of an optimized broth enrichment culture medium for the isolation of Clostridium difficile.[J]. Anaerobe. 2018; 54:92-99.
Reigadas E, AlcaláL, Marín M, et al. C. difficile PCR-ribotype 023 might go undetected when using ChromIdC. difficileagar.[J]. Anaerobe. 2017; 44:34-35.
Park KS, Ki CS, Lee NY. Isolation and Identification of Clostridium difficile Using ChromIDC. difficile Medium Combined With Gram Staining and PRO Disc Testing: A Proposal for a Simple Culture Process.[J]. Ann Lab Med. 2015; 35(4):404-409.
边俏, 罗芸, 许星星, 等. 一种用于艰难梭菌分离培养的新型显色培养基的性能评价[J]. 临床检验杂志, 2019, 37(04):301-304.
Girinathan Brintha Parasumanna, BraunSterling, Sirigireddy Apoorva Reddy, et al. Importance of Glutamate Dehydrogenase (GDH) in Clostridium difficile Colonization In Vivo.[J]. PloS one, 2016; 11(7):e0160107.
Barbut F, Lalande V, Daprey G, et al. Usefulness of simultaneous detection of toxin A and glutamate dehydrogenase for the diagnosis of Clostridium difficile-associated diseases.[J]. Eur J Clin Microbiol Infect Dis. 2000; 19(6):481-484.
祁琪, 茅一萍, 李阳, 等. 艰难梭菌谷氨酸脱氢酶抗原及毒素检测试剂盒对艰难梭菌感染诊断价值的Meta分析[J]. 中国感染控制杂志, 2019, 18(06):511-518.
Yoo IY, Song DJ, Huh HJ, et al. Simultaneous Detection of Clostridioides difficile Glutamate Dehydrogenase and Toxin A/B: Comparison of the C. DIFF QUIK CHEK COMPLETE and RIDASCREEN Assays.[J]. Ann Lab Med. 2019; 39(2):214-217.
Cheng JW, Xiao M, Kudinha T, et al. The Role of Glutamate Dehydrogenase (GDH)Testing Assay in the Diagnosis of Clostridium difficile Infections: A High Sensitive Screening Test and an Essential Step in the Proposed Laboratory Diagnosis Workflow for Developing Countries like China.[J]. PLoS One. 2015; 10(12): e0144604.
Gateau C, Couturier J, Coia J, et al. How to: diagnose infection caused by Clostridium difficile.[J].Clin Microbiol Infect. 2018; 24(5):463-468.
任永鑫, 孙斌, 徐广贤. 艰难梭菌谷氨酸脱氢酶的原核表达及其胶体金免疫层析检测方法的建立[J]. 中国生物制品学杂志, 2019, 32(03):303-310.
Peng Z, Ling L, Stratton CW, et al. Advances in the diagnosis and treatment of Clostridium difficileinfections.[J]. Emerg Microbes Infect. 2018;7(1):15.
Martínez-Meléndez A, Camacho-Ortiz A, Morfin-Otero R, et al. Current knowledge on the laboratory diagnosis of Clostridium difficileinfection.[J]. World J Gastroenterol. 2017; 23(9):1552-1567.
Reller ME, Lema CA, Perl TM, et al. Yield of stool culture with isolate toxin testing versus a two-step algorithm including stool toxin testing for detection of toxigenic Clostridium difficile.[J]. J Clin Microbiol. 2007; 45(11): 3601-3605.
Kachrimanidou M, Malisiovas N. Clostridium difficile infection: a comprehensive review.[J]. Crit Rev Microbiol. 2011; 37(3):178-187.
Reigadas E, Alcalá L, Marín M, et al. Clinical significance of direct cytotoxicity and toxigenic culture in Clostridium difficileinfection.[J].Anaerobe. 2016; 37:38-42.
Mirzaei EZ, Rajabnia M, Sadeghi F, et al. Diagnosis of Clostridium difficile infection by toxigenic culture and PCR assay. Iran J Microbiol.[J]. 2018; 10(5):287-293.
Ignatius R, Neuber R, Kietzmann H, et al. Clostridioides difficile in Outpatients: Application of a Diagnostic Algorithm Recommended by the European Society of Clinical Microbiology and Infectious Diseases.[J]. Eur J Microbiol Immunol (Bp). 2019; 9(3):88-90.
Cançado GGL, Silva ROS, Nader AP, et al. Impact of simultaneous glutamate dehydrogenase and toxin A/B rapid immunoassay on Clostridium difficile diagnosis and treatment in hospitalized patients with antibiotic-associated diarrhea in a university hospital of Brazil.[J]. J Gastroenterol Hepatol. 2018; 33(2):393-396.
Mohan SS, McDermott BP, Parchuri S, Cunha BA. Lack of value of repeat stool testing for Clostridium difficiletoxin.[J]. Am J Med. 2006; 119(4):356-e7.
Sandlund J, Mills R, Griego-Fullbright C, et al. Laboratory comparison between cell cytotoxicity neutralization assay and ultrasensitive single molecule counting technology for detection of Clostridioides difficile toxins A and B, PCR, enzyme immunoassays, and multistep algorithms.[J]. Diagn Microbiol Infect Dis. 2019; 95(1):20-24.
郭旭光, 王婷, 汤昕, 等. 艰难梭菌实验室诊断研究进展[J]. 国际检验医学杂志, 2018, 39(16):2043-2045.
Boyanton BL Jr, Sural P, Loomis CR, et al. Loop-mediated isothermal amplification compared to real-time PCR and enzyme immunoassay for toxigenic Clostridium difficiledetection.[J]. J Clin Microbiol. 2012; 50(3):640-645.
Banz A, Lantz A, Riou B, et al. Sensitivity of Single-Molecule Array Assays for Detection of Clostridium difficile Toxins in Comparison to Conventional Laboratory Testing Algorithms.[J]. J Clin Microbiol. 2018; 56(8):e00452-18.
储琼芳, 李先平, 华玉婷, 等. TaqMan实时荧光定量PCR检测艰难梭菌[J]. 疾病监测, 2018, 33(05):417-422.
Song PH, Min JH, Kim YS, et al. Rapid and accurate diagnosis of Clostridium difficile infection by real-time polymerase chain reaction.[J]. Intest Res. 2018; 16(1):109-115.
Wang LZ, Chen LD, Xiao B, et al. Efficacy of real-time PCR for detecting Clostridium difficile infection: comparison with enzyme-linked fluorescent spectroscopy-based approaches.[J]. Nan Fang Yi Ke Da XueXue Bao. 2017; 37(12):1648-1653.
金大智, 罗芸, 罗丽, 等. 多重荧光PCR结合Allglo探针技术检测艰难梭菌相关毒素基因[J]. 中华检验医学杂志, 2014(01):32-35.
王丽倩, 罗芸, 黄忱, 等. 一种艰难梭菌毒素基因检测方法的临床应用评价[J]. 中华检验医学杂志, 2017, 40(07):511-514.
Polage CR, Gyorke CE, Kennedy MA, et al. Overdiagnosis of Clostridium difficile Infection in the Molecular Test Era.[J]. JAMA Intern Med. 2015; 175(11):1792-1801.
Mori Y, Notomi T. Loop-mediated isothermal amplification (LAMP): Expansion of its practical application as a tool to achieve universal health coverage.[J]. J Infect Chemother. 2020; 26(1):13-17.
Wong YP, Othman S,LauYL,et al. Loop-mediated isothermal amplification (LAMP): a versatile technique for detection of micro-organisms.[J]. J Appl Microbiol. 2018; 124(3):626-643.
Wei C, Wen-En L, Yang-Ming L, et al. Diagnostic accuracy of loop-mediated isothermal amplification in detection of Clostridium difficile in stool samples: a meta-analysis.[J]. Arch Med Sci. 2015; 11(5):927-936.
Neuendorf M, Guadarrama-Gonzalez R, Lamik B, et al. A prospective study of two isothermal amplification assays compared with real-time PCR, CCNA and toxigenic culture for the diagnosis of Clostridium difficileinfection.[J].BMC Microbiol. 2016; 16:19.
Norén T, Unemo M, Magnusson C, et al. Evaluation of the rapid loop-mediated isothermal amplification assay Illumigene for diagnosis of Clostridium difficile in an outbreak situation.[J]. APMIS. 2014; 122(2):155-160.
Surawicz CM, Brandt LJ, Binion DG, et al. Guidelines for diagnosis, treatment, and prevention of Clostridium difficileinfections.[J]. Am J Gastroenterol. 2013; 108(4):478-499.
Burnham Carey-Ann D,Carroll Karen C. Diagnosis of Clostridium difficile infection: an ongoing conundrum for clinicians and for clinical laboratories.[J]. Clinical microbiology reviews, 2013, 26(3):604-630.
庄国华, 宋小军, 许星星, 等. 两种产毒型艰难梭菌分子诊断方法的临床应用评价[J]. 中华检验医学杂志, 2018, 41(01):35-40.
Garvey MI, Bradley CW, Wilkinson MAC, et al. Can a toxin gene NAAT be used to predict toxin EIA and the severity of Clostridium difficileinfection?.[J]. Antimicrob Resist Infect Control. 2017; 6:127.
Terhes G, Urbán E, Sóki J, et al. Comparison of a rapid molecular method, the BD GeneOhmC.diff assay, to the most frequently used laboratory tests for detection of toxin-producing Clostridium difficile in diarrheal feces.[J]. J Clin Microbiol. 2009; 47(11):3478-3481.
Dragonieri S, Pennazza G, Carratu P, et al.Electronic Nose Technology in Respiratory Diseases.[J]. Lung. 2017; 195(2):157-165.
Shi H, Zhang M, Adhikari B. Advances of electronic nose and its application in fresh foods: A review.[J]. Crit Rev Food Sci Nutr. 2018; 58(16):2700-2710.
Liang Z, Tian F, Zhang C, et al. A correlated information removing based interference suppression technique in electronic nose for detection of bacteria.[J].Anal Chim Acta. 2017; 986:145-152.
刘燕庐. 质谱仪“电子鼻”可快速嗅出超级细菌.[N]. 科技日报, 2014-09-30(002).
Chan D K, Anderson M, Lynch D T, et al. Su1352 Detection of Clostridium difficile-Infected Stool by Electronic-Nose Analysis of Fecal Headspace Volatile Organic Compounds.[J]. Gastroenterology, 2015, 148(4):S-483-S-483.
胡继红, 马筱玲, 王辉, 等. MALDI-TOF MS在临床微生物鉴定中的标准化操作专家共识[J]. 中华检验医学杂志, 2019(04):241-249.
Reil M, Erhard M, Kuijper EJ, et al. Recognition of Clostridium difficile PCR-ribotypes 001, 027 and 126/078 using an extended MALDI-TOF MS system.[J]. Eur J Clin Microbiol Infect Dis. 2011; 30(11):1431-1436.
Li R, Xiao D, Yang J, et al. Identification and Characterization of Clostridium difficile Sequence Type 37 Genotype by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry.[J]. J Clin Microbiol. 2018; 56(5):e01990-17.
Huang B, Li H, Jin D, et al. Real-time cellular analysis for quantitative detection of functional Clostridium difficile toxin in stool.[J]. Expert Rev Mol Diagn. 2014; 14(3):281-291.
Ryder AB, Huang Y, Li H, et al. Assessment of Clostridium difficile infections by quantitative detection of tcdB toxin by use of a real-time cell analysis system.[J]. J Clin Microbiol. 2010; 48(11):4129-4134.
Huang B, Jin D, Zhang J, et al. Real-time cellular analysis coupled with a specimen enrichment accurately detects and quantifies Clostridium difficile toxins in stool.[J]. J Clin Microbiol. 2014; 52(4):1105-1111.





