DNA纳米结构触发的等温扩增检测新技术与生物分子检测应用策略
【摘要】实用性强、成本低廉、操作简单的生物分子检测技术在医学诊断、食品分析等方面发挥着重要作用。近年来,随着分析化学、材料科学及临床检验诊断学等多学科的交叉融合发展,一系列基于DNA纳米结构及等温扩增技术开发的具有高灵敏、高特异性、自动化程度高且快捷的检测新方法受到研究者的广泛关注。在本文中,我们将总结过去二十年来以DNA纳米结构为基本元件辅助等温扩增技术所发展的检测新技术、新方法;并总结这些新技术、新方法在不同生物标志物检测中的应用及不足。同时,在检测过程中,依据靶标分子和信号探针的参与输出比例可主要分为N:N扩增、1:N扩增和1:N2扩增这三种模式。基于此,本文将针对这三种扩增模式分别探讨和总结。
【关键词】脱氧核糖核酸;DNA纳米结构;等温扩增技术;生物分子检测
脱氧核糖核酸(Deoxyribonucleic acid,DNA)[1]作为生命的遗传物质的储存者,在生命的发育与机能运作中起着关键的调节作用。随着纳米技术和生命科学的发展,研究者发现DNA分子具有刚性结构、编码性强等特点[2-5],可以作为纳米级材料并依据Watson-Crick碱基配对原则构建各种纳米结构,在生物医学、化学以及材料等领域具有潜在的应用价值。随着医学检测技术的发展,基于不同尺寸的DNA纳米结构已经被应用于构建各种高效的生物检测方法。其中,等温扩增技术(Isothermal amplification technology)[6-9]因其简单的实际操作、不需精良的温控仪器,且实用性强、成本低等优点,已被越来越多的研究人员用于开发各种新的基于DNA纳米结构的生物分子的检测。
一、DNA纳米结构各种等温检测新方法分类
目前,基于DNA纳米结构开发的各种等温检测新方法根据靶标分子和信号分子的在检测反应中参与的比例,主要被分为三类:N:N,1:N和1:N2型扩增反应(图1)[10-12],且这些检测新方法在提高生物分子检测的灵敏度、特异性和准确度等方面皆具有不同的意义。
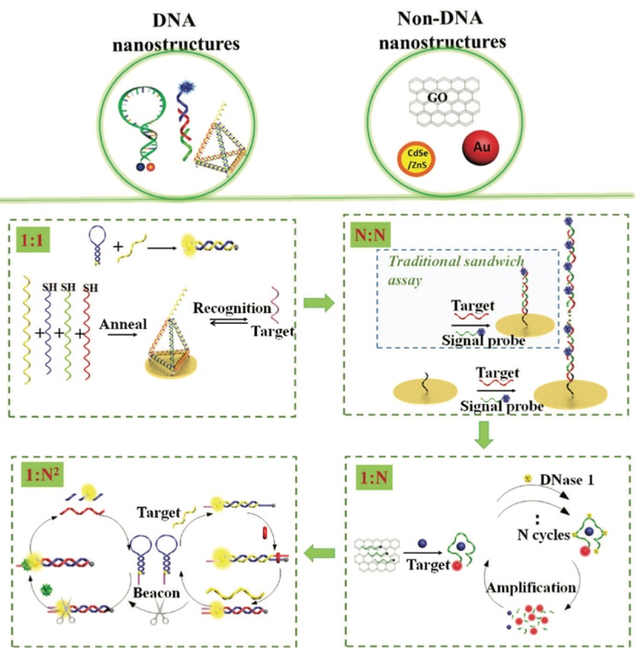
图1. 基于DNA纳米结构发展的不同靶标-信号检测模式的等温扩增检测策略
Fig. 1 Schematic illustration of isothermal amplification based on nanostructure. [13]
最初,DNA线性探针(Linear probe)[14, 15]是最早被开发出的信号分子探针,它是一种最简单的DNA纳米结构,它通过Watson-Crick碱基配对原则[16]可以很好的用于靶标分子的特异性识别和检测[17]。然而,在检测过程中,这些DNA线性探针可以非特异性地吸附在检测介质表面及非互补的DNA片段,因此,需要使用洗涤步骤去除多余的未特异性结合的DNA线性探针,从而大大地降低了检测速度及灵敏度[18]。1996年,分子信标(Molecular beacon,MB)[19-21]被报道继而广泛地应用于生物分子的检测中。MB分子是一种荧光标记的寡核苷酸链,在没有靶标时,分子信标的荧光和猝灭基团相互靠近,荧光被猝灭;而当靶标存在时,可以触发分子信标改变其构象,从而发出荧光信号报告靶标的存在(图2 A-B)[22, 23]。另外,基于识别探针-靶标-信号探针的“三明治”夹心结构[24-26]也逐渐被研究者用于制造新型的生物传感器。由于靶标分子需要同时杂交、结合两个识别元件——识别探针和信号探针才能触发检测信号的改变,因此,这种“三明治”夹心结构具有很高的特异性(图2 C)[27]。随着对DNA纳米结构的深入研究,Fan课题组[28]通过自组装技术利用4条单链DNA设计了一个具有四面体构象的DNA四面体(DNA tetrahedron)探针(图2 D),这种DNA四面体探针具有良好的空间可控性和高度的稳定性,在生物检测过程中可以极大地降低背景信号。然而,尽管这些基于分子信标、“三明治”夹心结构或四面体结构的DNA探针在生物传感检测过程中发挥着或增强特异性或降低背景信号的作用,但是由于没有扩增反应的参与,导致靶标与信号分子比为1:1[29, 30],因此,基于这些DNA纳米结构的检测结果灵敏度较低,不能很好的为现代化高灵敏的生物分子检测发挥作用。
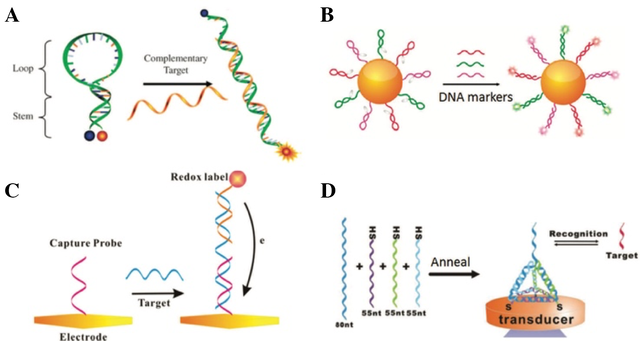
图2 A,分子信标结构示意图[22];B,多种分子信标修饰的金纳米[23];C,三交联结构用于靶标DNA的检测[27];D,四面体DNA纳米结构的形成及在DNA检测中的应用[28]
Fig. 2 A) Schematic illustration of molecular beacon (MB)[22]; B) Various MB modified gold nanoparticle[23]; C) Sandwich structure for DNA detection[27]; D) The construction of DNA tetrahedron and its application in DNA detection [28]
聚合酶链式反应(Polymerase chain reaction,PCR)[31, 32]是一种指数扩增方法,已被广泛的用于生物标志物的检测。然而,检测过程需要热循环仪器辅助快速的温度调节,以及检测结果易污染、操作复杂限制了它在基层医院及检测机构的广泛应用。此外,生物体内的恒温环境也极大的限制了PCR在细胞内或动物体内原位检测应用。近年新发展起来的等温扩增技术在临床和现场快速诊断中显示了良好的应用前景。等温扩增技术主要分为酶促等温扩增和无酶等温扩增两大类。酶促等温扩增策略主要是基于上述提到的DNA纳米结构,辅助利用链置换聚合酶(Strand displacement specific nuclease)[33, 34]、核酸外切酶(Exonuclease)[35, 36]、限制性内切酶(Nicking enzyme)[37]或者是双链特异性核酸酶(Duplex-specificnuclease,DSN)[38, 39]等促使靶标分子再循环参与扩增反应,从而提高检测的灵敏度。而无酶的等温扩增策略则主要通过利用一些超级三明治结构、条形码探针、杂交链式反应(Hybridization chain reaction,HCR)[40-42]以及发夹DNA级联放大反应(Hairpin DNA cascade amplification)[43-45]等实现生物体内的高灵敏、特异性检测。尽管这些基于不同DNA纳米结构的等温检测策略可以极大的缩短反应时间、简化操作步骤,靶标-信号比可以实现1:N,N:N等扩增模式,但是在检测过程中依然存在非特异性扩增以及高背景信号等问题。
在本文中,作者将从以下几方面探讨并总结近年来基于DNA纳米结构的等温扩增检测策略的最新发展。首先,将探讨以DNA超级“三明治”结构为基础发展的一系列等温扩增策略在靶标分子检测中的应用。这些扩增策略依赖多种靶标参与、诱导不同的信号探针分子在检测过程中自组装形成一个整体DNA纳米结构,其中靶标-信号输出比为N:N,相较于传统的1:1检测模式可以极大地提高灵敏度核特异性。其次,将探讨基于条形码技术、HCR反应等发展的一系列线性等温扩增检测策略在生物分子检测中的应用。这些扩增策略可以实现靶标-信号1:N的扩增,实现信号的放大输出。最后,将探讨基于功能化分子信标、改进的线性探针等发展的一系列指数次扩增反应在生物分子检测中的作用,可以实现靶标和信号1:N2的扩增反应。
二、基于靶标与信号N:N的等温扩增检测策略
应用研究表明,“三明治”夹心结构的DNA纳米结构[46]可以降低非特异性杂交产生的背景信号,在DNA和蛋白质检测分析中发挥重要作用。然而,由于待检的靶标DNA仅与单个信号探针发生杂交反应,导致灵敏度有限,从而极大地限制了该类DNA纳米结构的广泛应用。Xia课题组[47]通过合理的设计发展了一种超级夹心结构,在这种结构里,信号探针的5’端区域和3’端区域分别与靶DNA的3’端和5’端的两个区域互补,从而与靶DNA发生连续的杂交反应,形成含有多个信号分子标记的超级“三明治”结构,这种靶标与信号探针N:N参与检测过程可以实现信号放大,最终极大地提高了检测灵敏度(图3)。最近,这种超级夹心结构已被广泛用于检测其它生物分子,如蛋白质和离子[48-51]。例如,Li报道[52]了一种基于电化学平台的检测策略用于检测血小板衍生生长因子(Platelet-derived growth factor-BB,PDGF-BB)。在该策略中,DNA适配体和待测蛋白PDGF-BB特异性结合,形成一种纳米管结构,继而大量的氧化还原物质[Ru(NH3)6]3+可嵌入到纳米管中,实现信号放大,最终检测灵敏度可低至100fM。在另一项研究中,Xu等人[53]基于Hg2+可嵌入T-T碱基对形成T-Hg2+-T的DNA超级纳米结构,最终,基于这种DNA交联结构,研发了一种用于Hg2+的电化学发光检测策略。该策略表现出较高的灵敏度和选择性,其检测限可低至0.25nM。值得提出的是,这种以电化学为分析平台的检测过程,虽然靶标和信号比为N:N,实际上得到的信号输出不等同于传统的1:1非扩增检测模式。这主要是由于DNA纳米结构的形成导致大量的信号探针被整合到一个大的整体并附着在检测表面,可以极大地增强检测信号的输出。在这种情况下,DNA纳米结构有效地捕获、并高效的整合信号探针,最终实现信号的放大增强。
综上,这种N:N检测模式其信号输出更来源于每一个靶标的参与,而传统的1:1非扩增检测模式的信号输出则依赖一个靶标,因此,N:N的检测模式具有更高的特异性。而且,同传统1:1非扩增检测模式相比较,基于N:N扩增的检测模式是一种群体反应,每一个信号探针都参与到靶标的准确识别过程,因此具有更高的选择性和检测灵敏度。
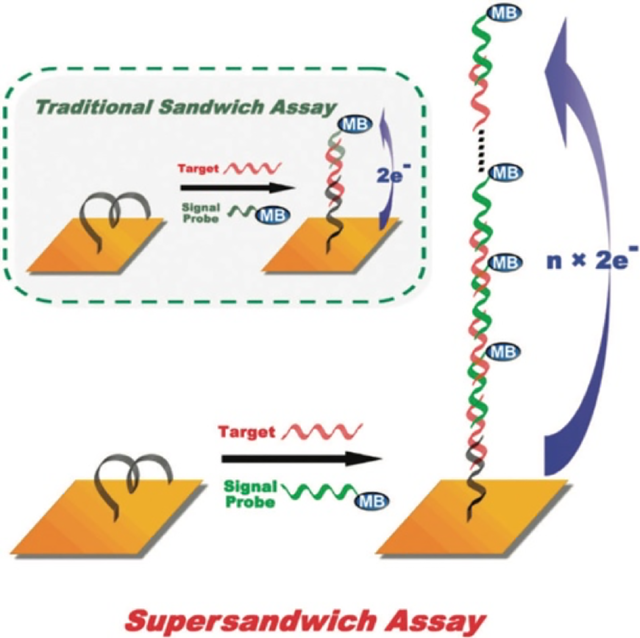
图3. 传统的“三明治”夹心检测及超级夹心检测原理图[47]
Fig. 3 Schematic illustration of a sandwich method
and a super-sandwich method[47]
三、基于靶标与信号1:N的等温检测策略
1. 基于DNA纳米结构的酶促扩增策略:分子信标(Molecularbeacon,MB)[54, 55]是一种荧光标记的寡核苷酸链,具有环状区、茎干区结构,在寡核苷酸链的3’和5’末端分别具有荧光基团和猝灭基团。如图2 A所示,在没有靶标存在时,MB分子的茎干区为dsDNA结构,其末端的荧光和猝灭基团相互靠近,荧光猝灭。而当与靶标结合后,MB的空间构象发生改变,导致荧光和猝灭基团远离,荧光信号得以恢复。MB分子具有高特异性、合成成本低、设计简单等优点,目前已广泛适用于各种均相体系中靶标以及活细胞内靶标的检测。但是由于一个靶标只能对应结合一个MB探针分子实现荧光信号的输出。这种靶标-信号探针1:1的化学计量比限制了该类检测方法的灵敏度,其检测限仅为纳摩尔范围。基于此,利用生物酶的靶标循环反应策略已被研发用于提高检测灵敏度。例如,Xie等人[56]通过引入限制性内切酶(Nicking enzyme)发展了一种检测策略可提高检测灵敏度。当MB探针分子识别并与靶标DNA互补杂交后,产生的双链结构可被内切酶特异性识别并剪切MB分子,从而靶标DNA被释放,进而可参与到新的MB信号分子的识别、杂交过程,实现靶标在检测体系中的循环。而且,通过内切酶不断剪切MB信号分子,可释放无数的荧光信号(图4)。这种依赖于内切酶辅助靶标再循环的扩增检测策略[57, 58]可实现待测靶标-信号的1:N放大,较于传统的1:1非扩增检测策略,检测限得到极大改善。
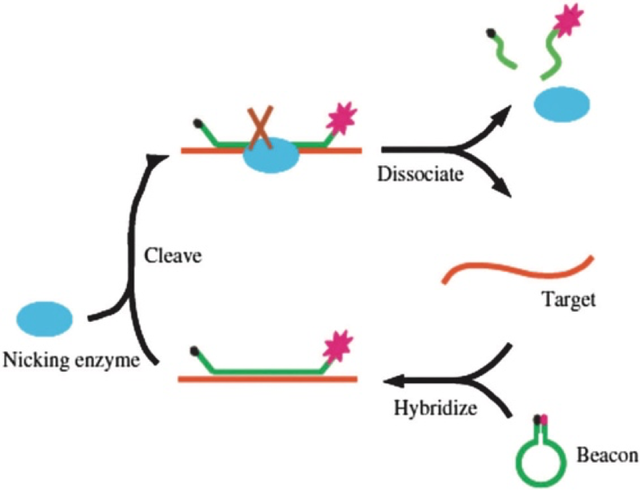
图4. 基于分子信标和限制性内切酶的等温扩增反应用于
DNA的检测,其扩增模式为1:N[56]
Fig. 4 The isothermal amplification based on MB and nicking enzyme for DNA detection, and the target to signal was 1:N [56]
虽然基于内切酶辅助的信号放大策略可极大提高靶标的检测灵敏度,但是,由于切口酶具有序列特异性,因此在靶序列的选择上存在诸多限制。为了克服上述问题,核酸外切酶III(Exonuclease III,Exo III)[35, 59, 60]也作为扩增反应的生物催化剂应用于生物标志物的检测中。Exo III可以特异性地消化dsDNA的平端3’末端,从而释放未被消化的另一条DNA单链。Plaxco等[61]基于MB信号探针,并利用外切酶III驱动靶标分子发生循环反应,发展了一种通用型的序列-非依赖性扩增DNA检测策略。在此策略的检测过程中,MB分子和靶标分子杂交后,构象发生改变并打开发卡结构,释放出荧光信号,并且暴露出平端3’末端。随后,外切酶III可以识别此平端3’末端并进行消化,最终导致靶标被释放进而参与到下一个新的循环反应中(图5)。这种序列-非依赖性扩增DNA检测策略展示了较高的灵敏度,其最低检测限可低至20 aM。
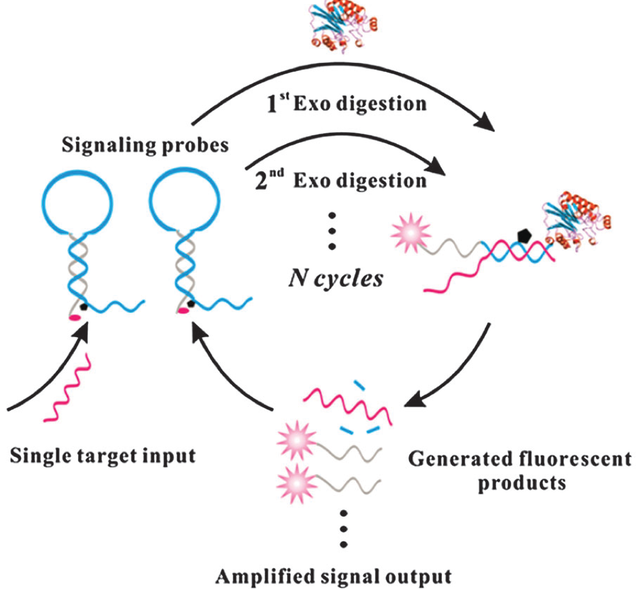
图5. 基于分子信标和外切酶III的等温扩增应用于靶标
DNA的检测,其扩增模式为1:N[61]
Fig. 5 The isothermal amplification based on MB and exonuclease III for DNA detection, and the target to signal was 1:N [61]
除了核酸外切酶III,双链特异性核酸酶(Duplex-specificnuclease,DSN)[62, 63]可以特异性降解双链DNA和DNA:RNA杂交体中的DNA,而对单链核酸分子和双链RNA没有反应活性,并且具有区分碱基错配的功能,因此也已被广泛应用于依赖酶促的靶标检测反应中。例如,Ye等人[64]发展了一种依赖双链特异性核酸酶的信号放大策略用于微小RNA (microRNA,miRNA)的灵敏检测。在这一策略中,设计的DNA线性探针Taqman探针其5’端携带荧光基团,3’端携带猝灭基团,当和靶标miRNA识别、结合后形成DNA:RNA双连体,随后可被DSN酶特异性识别。DSN酶选择性的消化DNA Taqman探针并释放出靶标miRNA;并且,消化的Taqman探针导致荧光基团和猝灭基团分离,释放出荧光信号。由于靶标miRNA被释放后可继续参与与新的Taqman探针的杂交反应,最终基于DSN酶的消化可诱导数千个Taqman探针被消化、荧光信号释放,导致荧光信号的显著扩增。基于此策略,最终可实现飞摩尔浓度范围的miRNA的高灵敏检测。
DNA链置换聚合酶(DNA strand-displacement polymerase)[65, 66]是另一种用于靶标再循环的重要生物酶。它可以催化dsDNA中的单链发生置换并催化聚合扩增反应。Wang等人[67]报道了一种基于DNA链置换聚合酶和分子信标的扩增方法用于靶标DNA的灵敏检测。在这一方法中,靶标DNA可以和MB分子的环状区域杂交从而打开茎干的dsDNA结构。随后,引物可与MB分子的茎端发生杂交进而引发聚合反应。在这一过程中,靶标DNA被引物置换下来,然后与另一个MB探针杂交,产生聚合反应并产生相应的荧光信号,最终可以用于痕量的DNA高灵敏检测。基于此方法,检测限可低至6.4×1015 M。
随着DNA纳米结构的发展以及其在等温扩增反应的广泛参与,随之发展了多种多样的生物分子检测技术。例如,为了实现低成本,高特异性和快速检测,Liu等[68]通过设计一种与靶标DNA互补的接头DNA(Linker DNA)[69],以及一种DNA修饰的金纳米探针发展了一种检测技术(图6)。在这一技术中,当有靶标DNA存在时,接头DNA与靶标DNA相结合随后被内切酶识别、剪切。随后,靶标DNA与接头DNA解离并进入到下一个循环。最终可以获得对靶标低至10pM的检测限。为了进一步克服切口酶对靶序列的选择特异性,Xia课题组[70]报道了一种通用型检测策略用于端粒酶的灵敏检测。当端粒酶存在时,可以触发单链DNA(ssDNA)探针延伸,延伸产物为TTAGGG的重复序列。继而,这种重复序列可以和不同的信号探针相结合,形成复杂的DNA网状结构。由于端粒酶可以产生不同重复数量的序列片段,这种检测模式可以定义为1:N的信号扩增模式。
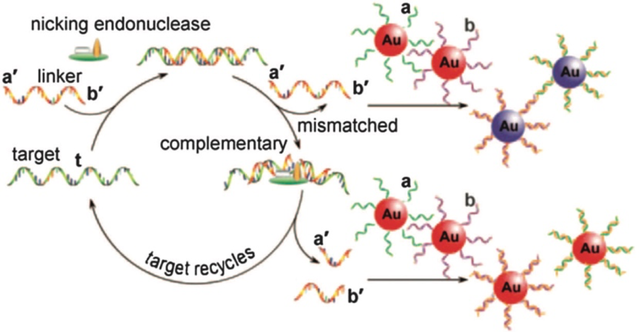
图6. 基于纳米金的等温扩增反应应用于靶标DNA的检测,其扩增模式为1:N [68]
Fig. 6 The isothermal amplification based on gold nanoparticles for DNA detection, and the target to signal was 1:N [68]
2. 基于DNA纳米结构的无酶扩增策略:尽管DNA超级纳米结构已经引起人们的广泛关注,但由于很大一部分检测反应的靶标和信号为N:N,因此提高检测限仍是目前检测技术进步面临的主要问题。在Xia课题组的另一项研究中[71],他们设计并改进了DNA超级夹心结构,这种结构可以在只有一种靶标DNA的情况下自组装形成,实现真正的靶标和信号1:N的信号扩增模式,从而极大地增强了检测限(图7)。有意思的是,通过将这种纳米结构应用于构成纳米级的分子探针,研究者们又相继开发了用于检测亚纳摩尔DNA和亚微摩尔ATP的装置[72]。
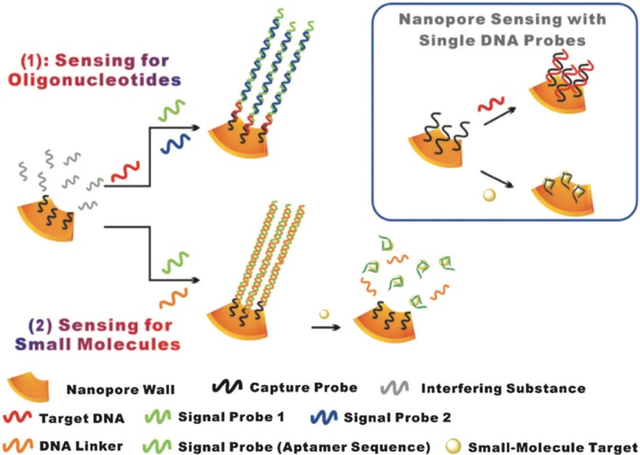
图7. 基于纳米孔技术和超级夹心结构的等温扩增反应应用于靶标DNA的检测[71]
Fig. 7 The isothermal amplification based on nanopore and supersandwich structure for DNA and ATP detection[71]
HCR反应是一种新型的无酶参与的核算聚合反应,通过靶标分子触发若干自身结构稳定的DNA茎环结构发生级联反应,从而产生于类似这种DNA超级夹心结构的超长链DNA结构,实现靶标分子的信号放大(图8)[73]。
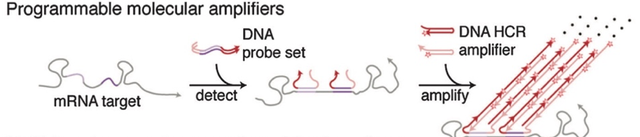
图8. 杂交链式反应的原理示意图[74]
Fig. 8 Schematic illustration of hybridization chain
reaction (HCR) [74]
目前,HCR技术已被应用于生物体靶标分子的检测。Tan等[74]通过引入两个发夹结构开发了一种发夹DNA级联放大器(Hairpin DNA cascade amplifier,HDCA)用来成像活细胞中的miRNA的表达(图9)。在细胞内,当靶标miRNA存在时,可触发发夹H1和H2双链体的组装(H1-H2),释放出荧光信号。更重要的是,在这一发夹DNA级联放大器介导的检测过程中,可实现靶标miRNA的循环再利用,从而产生更多的H1-H2双链体结构,一个miRNA靶标分子相对应的可以实现多个信号的输出,因此这种检测模式实现了1:N的信号放大。尽管这种基于DNA自组装的反应比基于酶的反应需要更长的时间,由于不需要将任何商业酶转染到活细胞中,因此更适合于活细胞中的检测,且检测程序简单、成本低廉。

图9. 基于发夹级联放大器开发的等温检测技术用于
细胞内靶标RNA的检测[74]
Fig. 9 The isothermal amplification based on hairpin DNA cascade amplifier for mRNA detection in living cells [74]
从以上实例中我们可以看到,1:N的信号放大策略可以实现一个靶标触发多个信号探针的释放,从而发出强检测信号,极大地增强检测灵敏度。与1:1和N:N的检测策略相比,这种基于1:N的信号放大策略更具有通用性,应用更广泛,其高灵敏的检测性能使得其更适应于生物体内痕量生物标志物的检测。
四、基于1:N2的信号放大检测策略
基于1:N信号扩增模式的等温核酸扩增技术作为一种潜在的扩增技术已逐步应用于快速、低成本的生物靶标分子检测。然而,目前技术检测市场依然对生物标志物检测存在普适性,灵敏度和特异性的需求。近年来,指数型扩增模式已被很多研发者采用以制备新型的生物分子检测策略。通过合理设计DNA序列并使其功能化磁性粒子纳米探针,研发者开发了一种发夹介导的指数酶促扩增策略(Hairpin-mediated quadratic enzymatic amplification,HQEA)[75-76],用于快速的miRNA高灵敏检测(图10)。基于HQEA检测策略,可以实现最低检测限10fM的靶标检测。并且,这种检测策略还表现出高度的特异性,可以很好的用于区分靶标miRNA家族成员。更重要的是,研究者将HQEA用于MCF-7,PC3细胞系和乳腺癌患者组织中miRNA提取样本的检测,其结果也证实该策略具有很高的检测灵敏度和特异性,表面该策略具有潜在的临床研究应用价值。Cheng等[78]通过合理的设计扩增模板,使其携带两个与靶miRNA互补的重复序列区域,通过内切酶辅助的双重识别、剪切,并结合聚合酶的扩增反应,在靶标miRNA分子的检测过程中可同时实现两种指数型的扩增反应(图11),最终实现靶标miRNA低至0.1zM的检测限,其动态检测范围可跨10个数量级。以上基于一个靶标分子可同时触发两个指数型的扩增反应,实现了1:N2的信号扩增,显著地提高了待测靶标生物分子的超高灵敏检测。
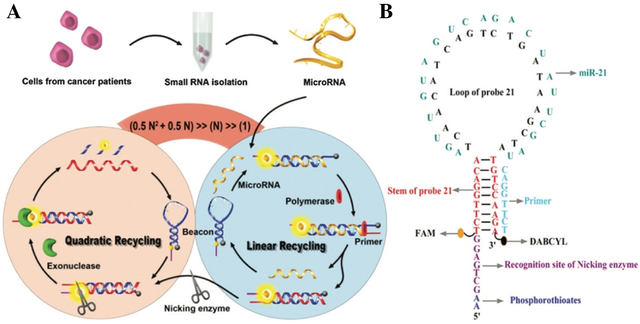
图10 A,基于发夹介导的指数酶促扩增策略用于靶标miRNA的检测,其扩增模式为1:N2 [75];B,分子信标结构示意图[76]
Fig. 10 A) Schematic representation of HQEA for miRNA detection, and the target to signal was 1:N2 [75]; B) the structure of molecular beacon [76]
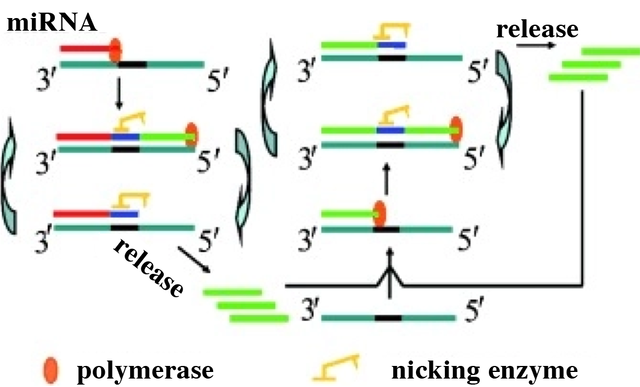
图11. 基于指数酶促扩增策略应用于靶标miRNA的检测示意图[77]
Fig. 11 Schematic of exponential isothermal amplification for miRNA detection [77]
目前,随着等温扩增技术的发展,一系列依赖各种等温扩增聚合酶发展起来的等温扩增技术[78-79]如链替代等温扩增、滚环等温扩增、依赖解旋酶等温扩增、依赖核酸序列等温扩增、单引物等温扩增和核酸快速等温检测放大等技术,也已得到了研究者的广泛关注和应用。这些技术易于操作,可以在恒定温度下实现指数型核酸扩增,已成为PCR的替代方案。
五、结论与展望
在本文中,重点介绍了基于不同种类的DNA纳米结构辅助等温扩增技术发展的一系列生物分子检测方法;并且总结了不同种类检测方法在信号放大技术上的弊端和改进;还展示了如何利用具有不同尺寸的各种DNA纳米结构,例如夹心结构,DNA超级“三明治”夹心结构,生物条形码探针,改进的MB分子和DNA线性探针等实现生物分子的高效扩增。DNA纳米结构具有很强的可编程能力,可用于在复杂环境中构建多重生物检测方法。然而,对于生物分子检测的选择性,速度,特异性,准确性和自动化方面也依然是我们所要面临的挑战。我们期望可以开发更实用、有效的DNA纳米结构用以辅助等温扩增技术,以研发出靶标-信号分子1:Nn的扩增检测方法,可以实现痕量生物分子的高灵敏、高特异的快速检测,从而更准确、精准的用于临床疾病的辅助诊断。
参考文献
Baisnée P F, Baldi P, Brunak S, et al. Flexibility of the genetic code with respect to DNA structure[J]. Bioinformatics, 2001, 17(3):237-248.
Seeman N C. DNA Nanotechnology: novel DNA constructions[J]. Ann. Rev. Biophys. &Biomol. Struct, 1998, 27(1):225-248.
Seeman N C. Structural DNA nanotechnology: an overview[J]. Methods in Molecular Biology, 2005, 303(3):143-166.
Zhang D Y, Seelig G. Dynamic DNA nanotechnology using strand-displacement reactions[J]. Nature Chemistry, 2011, 3(2):103-113.
Bandy T J, Brewer A, Burns J R, et al. DNA as supramolecular scaffold for functional molecules: progress in DNA nanotechnology[J]. Chemical Society Reviews, 2010, 40(1):138-148.
PooriaGill, AmirGhaemi. Nucleic acid isothermal amplification technologies—a review[J]. Nucleosides Nucleotides & Nucleic Acids, 2008, 27(3):224-243.
Zhong J, Zhao X. Isothermal amplification technologies for the detection of foodborne pathogens[J]. Food Analytical Methods, 2018, (22):1-18.
Chen Z, Liu Y, Xin C, et al. A cascade autocatalytic strand displacement amplification and hybridization chain reaction event for label-free and ultrasensitive electrochemical nucleic acid biosensing[J]. Biosensors & Bioelectronics, 2018, 113:1-8.
[Qi H, Yue S, Bi S, et al. Isothermal exponential amplification techniques: From basic principles to applications in electrochemical biosensors[J]. Biosensors & Bioelectronics, 2018, 110:207-217.
Sun Y, Peng P, Guo R, et al. Exonuclease III-boosted cascade reactions for ultrasensitive SERS detection of nucleic acids[J]. Biosensors & Bioelectronics, 2018, 104:32-38.
Park K S. Nucleic acid aptamer-based methods for diagnosis of infections[J]. Biosensors & Bioelectronics, 2018, 102:179-188.
Goto M, Honda E, Ogura A, et al. Colorimetric detection of loop-mediated isothermal amplification reaction by using hydroxy naphthol blue[J]. Biotechniques, 2009, 46(3):167-172.
Duan R, Lou X, Xia F. The development of nanostructure assisted isothermal amplification in biosensors[J]. Chemical Society Reviews, 2016, 45(6):1738-1749.
Sena E P, Zarling D A. Targeting in linear DNA duplexes with two complementary probe strands for hybrid stability[J]. Nature Genetics, 1993, 3(4):365-372.
Lu W, Jin Y, Wang G, et al. Enhanced photoelectrochemical method for linear DNA hybridization detection using Au-nanopaticle labeled DNA as probe onto titanium dioxide electrode[J]. Biosensors & Bioelectronics, 2008, 23(10):1534-1539.
Xia T, Jr S L J, Burkard M E, et al. Thermodynamic parameters for an expanded nearest-neighbor model for formation of RNA duplexes with Watson-Crick base pairs[J]. Biochemistry, 1998, 37(42):14719-14735.
Riccelli P V, Merante F, Leung K T, et al. Hybridization of single-stranded DNA targets to immobilized complementary DNA probes: comparison of hairpin versus linear capture probes[J]. Nucleic Acids Research, 2001, 29(4):996-1004.
Ricci F, Lai R Y, Plaxco K W. Linear, redox modified DNA probes as electrochemical DNA sensors[J]. Chemical Communications, 2007, 36(36):3768-3770.
Piatek A S, Tyagi S, Pol A C, et al. Molecular beacon sequence analysis for detecting drug resistance in Mycobacterium tuberculosis[J]. Nature Biotechnology, 1998, 16(4):359-363.
Fang X, Liu X, Sheldon Schuster A, et al. Designing a novel molecular beacon for surface-immobilized DNA hybridization studies[J]. Journal of the American Chemical Society, 1999, 121(12):2921-2922.
Leone G, Van S H, Van G B, et al. Molecular beacon probes combined with amplification by NASBA enable homogeneous, real-time detection of RNA[J]. Nucleic Acids Research, 1998, 26(9):2150-2155.
Wang K, Tang Z, Yang C J, et al. Molecular engineering of DNA: molecular beacons[J].AngewandteChemie, 2010, 48(5):856-870.
Song S, Liang Z, Zhang J, et al. Gold-nanoparticle-based multicolor nanobeacons for sequence-specific DNA analysis[J]. Angew Chem Int Ed Engl., 2009;48(46):8670-86744.
Pandey P C, Upadhyay S, Pathak H C. A new glucose biosensor based on sandwich configuration of organically modified sol-gel glass[J]. Electroanalysis, 2015, 11(1):59-64.
Mo Z, Long X, Fu W. A new sandwich-type assay of estrogen using piezoelectric biosensor immobilized with estrogen response element[J]. Analytical Communications, 1999, 36(36):281-283.
Xia N, Deng D, Zhang L, et al. Sandwich-type electrochemical biosensor for glycoproteins detection based on dual-amplification of boronic acid-gold nanoparticles and dopamine-gold nanoparticles[J]. Biosensors & Bioelectronics, 2013, 43(1):155-159.
Shen J, Li Y, Gu H, et al. Recent development of sandwich assay based on the nanobiotechnologies for proteins, nucleic acids, small molecules, and ions[J]. Chemical Reviews, 2014, 114(15):7631-7677.
Pei H, Lu N, Wen Y, et al. A DNA Nanostructure-based biomolecular probe carrier platform for electrochemical biosensing[J]. Advanced Materials, 2010, 22(42):4754-4758.
Zuo X, Yang X, Wang K, et al. A novel sandwich assay with molecular beacon as report probe for nucleic acids detection on one-dimensional microfluidic beads array[J]. Analytica Chimica Acta, 2007, 587(1):9-13.
Meng H M, Fu T, Zhang X B, et al. Efficient fluorescence turn-on probe for zirconium via a target-triggered DNA molecular beacon strategy[J]. Analytical Chemistry, 2012, 84(5):2124-2128.
Orita M, Suzuki Y, Sekiya T, et al. Rapid and sensitive detection of point mutations and DNA polymorphisms using the polymerase chain reaction[J]. Genomics, 1989, 5(4):874-879.
Mullis K, Faloona F, Scharf S, et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction[J]. Cold Spring HarbSymp Quant Biol, 1986, 51(1):263-273.
Walker G T. Empirical aspects of strand displacement amplification. [J]. PCR Methods Appl. 1993, 3(1):1-6.
Walker G T, Fraiser M S, Schram J L, et al. Strand displacement amplification-an isothermal, in vitro DNA amplification technique[J]. Nucleic Acids Research, 1992, 20(7):1691-1696.
Xuan F, Luo X, Hsing I M. Ultrasensitive solution-phase electrochemical molecular beacon-based DNA detection with signal amplification by exonuclease III-assisted target recycling[J]. Analytical Chemistry, 2012, 84(84):5216-5220.
Ju E, Yang X, Lin Y, et al. Exonuclease-aided amplification for label-free and fluorescence turn-on DNA detection based on aggregation-induced quenching[J]. Chemical Communications, 2012, 48(95):11662-11664.
Tan E, Erwin B, Dames S, et al. Specific versus nonspecific isothermal DNA amplification through thermophilic polymerase and nicking enzyme activities[J]. Biochemistry, 2008, 47(38):9987-9999.
Tao Y, Yin D, Jin M, et al. Double-loop hairpin probe and doxorubicin-loaded gold nanoparticles for the ultrasensitive electrochemical sensing of microRNA[J]. Biosensors & Bioelectronics, 2017, 96:99-105.
Lin X, Zhang C, Huang Y, et al. Backbone-modified molecular beacons for highly sensitive and selective detection of microRNAs based on duplex specific nuclease signal amplification[J]. Chemical Communications, 2013, 49(65):7243-7245.
Evanko D. Hybridization chain reaction[J]. Nature Methods, 2004, 1(3):186-187.
Dirks R M, Pierce N A. Triggered amplification by hybridization chain reaction[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(43):15275-15278.
Zhang B, Liu B, Tang D, et al. DNA-based hybridization chain reaction for amplified bioelectronic signal and ultrasensitive detection of proteins[J]. Analytical Chemistry, 2012, 84(12):5392-5399.
Liu J, Chen L, Lie P, et al. A universal biosensor for multiplex DNA detection based on hairpin probe assisted cascade signal amplification[J]. Chemical Communications, 2013, 49(45):5165-5167.
Wang W, Yuan X, Zhang W, et al. Cascade signal amplification for ultra-sensitive impedimetric detection of DNA hybridization using a hairpin DNA as probe[J]. Electrochimica Acta, 2012, 78(9):377-383.
Liu Y L, Wu H P, Zhou Q, et al. Controllable extension of hairpin-structured flaps to allow low-background cascade invasive reaction for a sensitive DNA logic sensor for mutation detection[J]. Chemical Science, 2018, 9(6):1666-1673.
Li Z, Zhao B, Wang D, et al. DNA Nanostructure-based universal microarray platform for high-efficiency multiplex bioanalysis in biofluids[J]. Acs Applied Materials & Interfaces, 2014, 6(20):17944-17953.
An electrochemical supersandwich assay for sensitive and selective DNA detection in complex matrices[J]. Journal of the American Chemical Society, 2010, 132(41):14346-14348.
Huang R, He N, Li Z. Recent progresses in DNA nanostructure-based biosensors for detection of tumor markers[J]. Biosensors & Bioelectronics, 2018, 109:27-34.
Wang D, Liu Q, Wu D, et al. Isothermal self-assembly of spermidine/DNA nanostructure complex as a functional platform for cancer therapy[J]. Acs Appl Mater Interfaces, 2018, 10(18):15504-15516.
Hu Q, Wang S, Wang L, et al. DNA nanostructure-based systems for intelligent delivery of therapeutic oligonucleotides[J]. Advanced Healthcare Materials, 2018. 7(20):e1701153.
Chen Y, Yang M, Xiang Y, et al. Binding-induced autonomous disassembly of aptamer-DNAzymesupersandwich nanostructures for sensitive electrochemiluminescence turn-on detection of ochratoxin A[J]. Nanoscale, 2013, 6(2):1099-1104.
Li C, Wang Z, Gao T, et al. Fabrication of hand-in-hand nanostructure for one-step protein detection[J]. Chemical Communications, 2013, 49(36):3760-3762.
Yuan T, Liu Z Y, Hu L Z, et al. Label-free supersandwich electrochemiluminescence assay for detection of sub-nanomolar Hg2+[J]. Chemical Communications, 2011, 47(43):11951-11953.
Goel G, Kumar A, Puniya A K, et al. Molecular beacon: a multitask probe[J]. Journal of Applied Microbiology, 2010, 99(3):435-442.
Xu H, Hepel M. “Molecular beacon”-based fluorescent assay for selective detection of glutathione and cysteine[J]. Analytical Chemistry, 2011, 83(3):813-819.
Li J J, Chu Y, Lee Y H, et al. Enzymatic signal amplification of molecular beacons for sensitive DNA detection[J]. Nucleic Acids Research, 2008, 36(6):e36.
Zheng A X, Wang J R, Li J, et al. Nicking enzyme based homogeneous aptasensors for amplification detection of protein[J]. Chemical Communications, 2011, 48(3):374-376.
Wang F, Freage L, Orbach R, et al. Autonomous replication of nucleic acids by polymerization/nicking enzyme/DNAzyme cascades for the amplified detection of DNA and the aptamer-cocaine complex[J]. Analytical Chemistry, 2013, 85(17):8196-8203.
Zhang M, Guan Y M, Ye B C. Ultrasensitive fluorescence polarization DNA detection by target assisted exonuclease III-catalyzed signal amplification[J]. Chemical Communications, 2011, 47(12):3478-3480.
Bao B, Pan Y, Gu B, et al. Highly sensitive detection of nucleic acids using a cascade amplification strategy based on exonuclease III-assisted target recycling and conjugated polyelectrolytes[J]. Analyst. 2018, 143(18):4267-4272.
Zuo X, Xia F, Xiao Y, et al. Sensitive and selective amplified fluorescence DNA detection based on exonuclease III-aided target recycling[J]. Journal of the American Chemical Society, 2010, 132(6):1816-1818.
Yi H, Cho Y J, Won S, et al. Duplex-specific nuclease efficiently removes rRNA for prokaryotic RNA-seq[J]. Nucleic Acids Research, 2011, 39(20):e140.
Shagina I, Bogdanova E, Mamedov I Z, et al. Normalization of genomic DNA using duplex-specific nuclease[J]. Biotechniques, 2010, 48(6):455-459.
Yin B C, Liu Y Q, Ye B C. One-step, multiplexed fluorescence detection of microRNAs based on duplex-specific nuclease signal amplification[J]. Journal of the American Chemical Society, 2012, 134(11):5064-5067.
Maga G, Villani G, Tillement V, et al. Okazaki fragment processing: modulation of the strand displacement activity of DNA polymerase delta by the concerted action of replication protein A, proliferating cell nuclear antigen, and flap endonuclease-1[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(25):14298-14303.
Rodríguez I, Lázaro J M, Blanco L, et al. A specific subdomain in phi29 DNA polymerase confers both processivity and strand-displacement capacity[J]. Proc Natl Acad Sci U S A, 2005, 102(18):6407-6412.
Guo Q, Yang X, Wang K, et al. Sensitive fluorescence detection of nucleic acids based on isothermal circular strand-displacement polymerization reaction[J]. Nucleic Acids Research, 2009, 37(3):e20.
Xu W, Xue X, Li T, et al. Ultrasensitive and selective colorimetric DNA detection by nicking endonuclease assisted nanoparticle amplification[J]. Angew Chem Int Ed Engl, 2010, 48(37):6742-6742.
Zhao Y, Hu S, Wang H, et al. DNA dendrimer-streptavidin nanocomplex: an efficient signal amplifier for construction of biosensing platforms[J]. Analytical Chemistry, 2017, 89(12):6907-6914.
Duan R, Wang B, Zhang T, et al. Sensitive and bidirectional detection of urine telomerase based on the four detection-color states of difunctional gold nanoparticle probe[J]. Analytical Chemistry, 2014, 86(19):9781-9785.
Liu N1, Jiang Y, Zhou Y, et al. Two-way nanopore sensing of sequence-specific oligonucleotides and small-molecule targets in complex matrices using integrated DNA supersandwich structures[J]. Angew Chem Int Ed Engl. 2013, 52(7):2007-2011.
Ge Z, Lin M, Wang P, et al. Hybridization chain reaction amplification of microRNA detection with a tetrahedral DNA nanostructure-based electrochemical biosensor[J]. Analytical Chemistry, 2014, 86(4):2124-2130.
Choi H M, Beck V A, Pierce N A. Next-generation in situ hybridization chain reaction: higher gain, lower cost, greater durability[J]. Acs Nano, 2014, 8(5):4284-4294.
Wu C, Cansiz S, Zhang L, et al. A nonenzymatic hairpin DNA cascade reaction provides high signal gain of mRNA imaging inside live cells[J]. Journal of the American Chemical Society, 2015, 137(15):4900-4903.
Duan R, Zuo X, Wang S, et al. Lab in a tube: ultrasensitive detection of microRNAs at the single-cell level and in breast cancer patients using quadratic isothermal amplification[J]. Journal of the American Chemical Society, 2013, 135(12):4604-4607.
Duan R, Zuo X, Wang S, et al. Quadratic isothermal amplification for the detection of microRNA[J]. Nature Protocols, 2014, 9(3):597-607.
Jia H, Li Z, Liu C, et al. Ultrasensitive detection of microRNAs by exponential isothermal amplification[J]. Angew Chem Int Ed Engl., 2010, 49(32):5498-5501.
Nimitphak T, Kiatpathomchai W, Flegel T W. Loop-mediated isothermal amplification of DNA[J]. Nucleic Acids Research, 2000, 28(12):e63.
Deng R, Zhang K, Li J. Isothermal amplification for microRNA detection: From the Test Tube to the Cell[J]. Accounts of Chemical Research, 2017, 50(4):1059-1068.





