新型冠状病毒及其分子生物学诊断新技术
作者:朱 镭 朱庆义
【摘要】冠状病毒(Corona virus)为正向单链RNA病毒,目前已知引起人类疾病的冠状病毒有7种。新冠病毒(COVID-19)是2019年底在我国武汉突发性不明原因肺炎病例中发现的一种新型冠状病毒,为β属冠状病毒,主要引起新冠病毒肺炎。目前,COVID-19在世界范围内快速传播和蔓延,已成为一种常态化防控的新发传染病。本文对COVID-19的基因分型和实验室诊断新技术,包括实时荧光定量聚合酶链反应(FQ-PCR)、环介导等温扩增(LAMP)、POCT免疫层析快速诊断试验、核酸质谱分析(MALDI-TOF)和基因测序等作了简要概述。
【关键词】新型冠状病毒;分子分型;基因诊断新技术
冠状病毒(Corona virus)为正向单链RNA病毒,是一个大型病毒家族,根据系统发育树,冠状病毒可分为4个属:α、β、γ、δ,其中β属冠状病毒又可分为4个独立的亚群A、B、C和D群,目前已知引起人类疾病的冠状病毒有7种,分别是HCoV-229E、HCoV-OC43、HCoV-NL63、HCoV-HKU1、SARS-CoV、MERS-CoV、COVID-19[1]。
新冠病毒(COVID-19)是2019年底在我国武汉突发性不明原因肺炎病例中发现的一种新型冠状病毒[2],其基因特征与SARS-CoV(Severe acute respiratory syndrome-coronavirus)严重急性呼吸综合征冠状病毒和MERS-CoV(Middle east respiratory syndrome-coronavirus)中东呼吸综合征冠状病毒有明显区别,与蝙蝠SARS样冠状病毒(bat-SL-CoVZC45)同源性达85%以上。入侵时的细胞受体都是ACE2(angiotensin converting enzyme 2)血管紧张素转换酶[3]。2020年2月11日,世卫组织(WHO)正式命名为2019-新型冠状病毒(COVID-19)[4],国际病毒分类委员会命名为“SARS-CoV-2”(Severe Acute Respiratory Syndrome-Coronavirus-2),我国卫健委将“新型冠状病毒肺炎”英文名称修订为“COVID-19”,与WHO命名保持一致,中文名保持不变。
一、COVID-19生物学特性与致病性
1. 生物学特性:COVID-19属于β属新冠病毒[5],有包膜,刺突,病毒颗粒呈圆形或椭圆形,常为多形性,直径60~140nm,体外分离培养96h左右即可在人呼吸道上皮细胞内出现,如在VeroE6和Huh-7细胞系中分离培养约需6d。病毒对紫外线和热敏感,56℃ 30min、乙醚、75%乙醇、含氯消毒剂、过氧乙酸和氯仿等脂溶剂均可有效灭活病毒。
2. 致病性:新冠病毒传染源为COVID-19感染者,主要经呼吸道飞沫和接触传播,故人群普遍易感,引起新冠病毒肺炎,潜伏期一般为1~14d[6],多数患者预后良好,儿童症状相对较轻,一般感染COVID-19后患者症状并无特异性,因此对其诊断还需结合流行病学史、实验室诊断、影像学检查。
3. 临床特征:COVID-19主要引起新冠病毒肺炎,重症患者多出现呼吸困难和/或低氧血症,严重者可快速进展为急性呼吸窘迫综合征、脓毒症休克、难以纠正的代谢性酸中毒和出凝血功能障碍及多器官功能衰竭而导致死亡[7]。在重型或危重型患者中也可表现为中低热,甚至无明显发热;轻型患者仅表现为低热、轻微乏力,无肺炎表现;无症状感染者没有明显的发热、咳嗽等临床症状,但仍有其传染性。多数患者预后良好,少数患者病情危重,老年人和有慢性基础疾病者预后较差。
二、COVID-19分子结构
COVID-19基因组为正向单链RNA病毒,其主要成员是蝙蝠冠状病毒(Bat coronavirus),但病毒表面用来入侵细胞的刺突蛋白(Spike Protein,S)被替换(同源重组)变成一种新的刺突蛋白,成为一个新的未知的病毒[8],WHO命名为COVID-19,依赖ACE2(angiotensin-converting enzyme 2)血管紧张素转换酶感染细胞。ACE2位于下呼吸道,理论上COVID-19病毒吸附侵染下呼吸道,引起肺炎[9]。
1. 基因组构成:COVID-19基因组全长29,891bp,G+C含量38%,5'端约2/3为重叠的开放读框(Open reading frame,ORF)ORF1a和ORF1b,主要负责编码与病毒复制和转录相关的酶等非结构蛋白;后面1/3基因组负责编码4种主要结构蛋白:包括刺突蛋白(Spike protein,S)、包膜蛋白(Envelope membrane protein,E)、膜蛋白(Membrane protein,M)和核衣壳蛋白(Nucleocapsid protein,N),少数毒种还有血凝素酯酶(Haemaglutinin-esterase,HE),另外还有嵌套在3'端基因组中的一系列基因编码的辅助性蛋白。这其中结构蛋白至关重要,决定了病毒的组装、稳定性和侵袭力。关于COVID-19遗传物质,根据现已测定的COVID-19全基因组序列,序列间高度保守,少量序列差异可能是早期在人群传播过程中适应性突变产生的。
2. 基因组结构蛋白:通过对我国首次公布的COVID-19基因组序列分析[11],发现其基因组编码至少有27种蛋白质,其中包括15种非结构蛋白(nsp1-nsp10,nsp12-nsp16),4种结构蛋白(S,E,M和N)以及8种附属蛋白(3a,3b,p6,7a,7b,8b,9b和14)。基因组类似SARS-CoV,但明显存在一些编码蛋白序列的插入和缺失,尤其是S蛋白。COVID-19编码的S蛋白序列组成和SARS以及MERS相比差异很大。新冠病毒S蛋白可分为S1和S2两个结构域。在病毒入侵宿主细胞过程中,宿主源组织蛋白酶切割S蛋白后得到一个S1受体结构域,内含受体结合区(receptor binding domain,RBD)以及来自不同动物的ACE2。在人体主要分布于呼吸道上皮细胞、肺脏、心脏、肾脏和消化道等位置,作为受体感染宿主,此过程激活了病毒感染力并决定了宿主范围。另外得到一个主要介导病毒包膜与细胞膜融合的S2结构域。COVID-19和SARS-CoV在S蛋白的RBD区与宿主受体之间结合的5个关键位点中,有4个是不同的,这些不同的位点是COVID-19在致病能力上与SARS-COV有差异的潜在原因[12]。
三、分子诊断技术
COVID-19的实验室诊断,包括病毒分离培养,免疫血清学诊断,分子生物学诊断等多种方法。临床上比较实用的是分子生物学诊断新技术,特别是荧光定量PCR(FQ-PCR)具有快速、敏感,特异之特点。WHO发布了各国对COVID-19分子诊断试验法[13]:我国CDC病毒所开发了COVID-19 ORF1ab和N基因2个区,法国巴斯德研究所采用RdRp gene/nCoV_IP2RdRp gene/nCoV_IP4 3个基因区,美国CDC设计了2019-nCoV_N1、N2、N3和RNAse P等4个基因位点,检测COVID-19(表1),引物和探针序列见表2[14]。
表1. WHO报告各国建立RT-PCR法检测COVID-19的基因位点
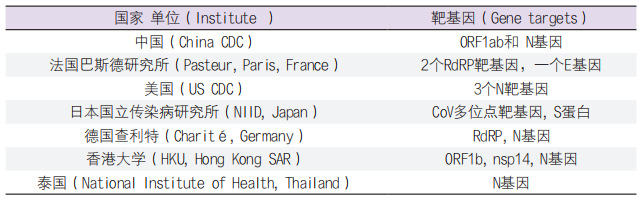
表2. WHO报告各国采用PCR试验检测COVID-19基因位点及其引物设计
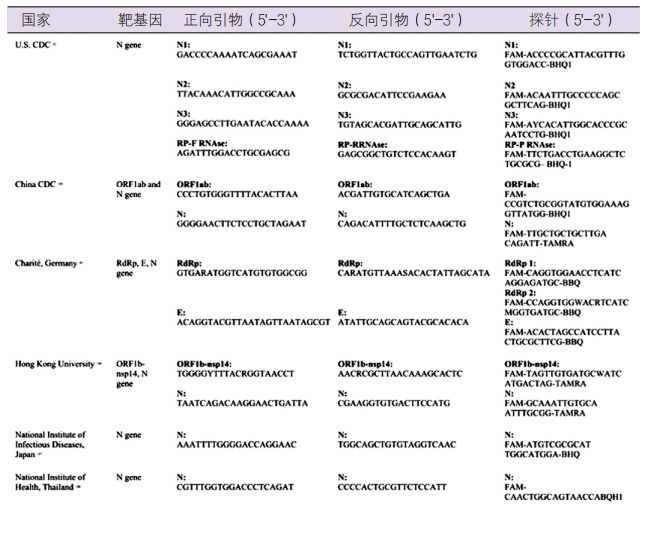
1. 聚合酶链反应(PCR):COVID-19为正向单链RNA(+ssRNA),PCR扩增需先通过反转录酶合成互补的cDNA链,再以cDNA链为基础进行RT-PCR[15]。目前已知COVID-19完整的基因序列,采集疑似COVID-19患者的上呼吸道(口咽和鼻咽)和下呼吸道(气管内吸痰、咳痰或支气管肺泡灌洗)样本进行RT-PCR试验,可对疑似感染COVID-19患者进行早期诊断。PCR试验方法有多种,如实时定量逆转录PCR(qRT-PCR)、实时RT-PCR(rtRT-PCR)、一步法RT-PCR等,均为RT-PCR的进一步优化。最初,我国Ren等针对COVID-19基因组ORF 1ab、N和E3个靶标域,设计引物和探针,建立了RT-PCR检测法[16]。Yip等研发了两个一步实时定量RT-PCR法[17]检测ORF 1b和N两个基因,可快速检测人体样本中的COVID-19。在临床样本中用RT-PCR检测N基因的灵敏度较高。目前国内有80多家企业研发出核酸检测试剂盒,经国家药品监督管理局(IFDA)应急审批并发放国家医疗器械注册证的公司有20多个COVID-19检测产品上市:如达安基因、上海捷诺、上海之江、上海伯杰、圣湘生物、华大生物(武汉)等。由于各大厂家产品研发上市时间短,导致市场上的检测试剂盒质量良莠不齐。因此,在目前需求量极大而企业缺少时间进行必要的研发优化及用已知的临床样本进行性能验证。郭元元等[18]对6种国产SARS-CoV-2(COVID-19)核酸检测试剂盒的检测性能进行了比较与分析,其结果显示弱阳性患者的核酸可能不易检出,各试剂的结果存在一定的差异。Chu等[19]参照Genbank我国首次公布的COVID-19序列(MN908947),选择β-冠状病毒塞尔维亚病毒(sarbecovirus)亚属中高度保守的两个序列区(ORF1b和N)设计引物和探针,建立两种针对新冠病毒ORF1b和N基因区的单基因实时RT-PCR检测法,对2例北京地区疑似COVID-19感染者,在症状出现后第3d~5d采集痰液和咽拭子样本,检测COVID-19 RNA均为阳性。陈炜等[20]研究显示痰标本中的病毒核酸量高于咽拭子标本,检测效果优于咽拭子,这可能与COVID-19侵袭感染下呼吸道和肺有关。对于COVID-19核酸检测结果的准确性,需要从样本类型、标本采集保存与运输(RNA易降解)以及患者感染周期等影响因素来综合分析,了解在不同标本中病毒的含量以及出现的时间,是确诊COVID-19感染患者及判断患者治疗恢复的关键点。
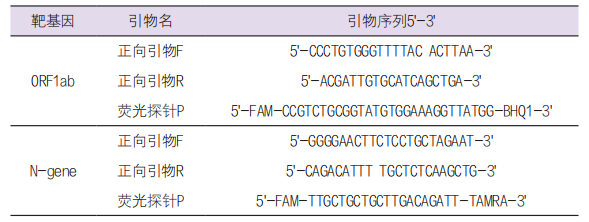
2. 实时荧光定量(FQ-PCR)试验法:我国疾病预防控制中心(CDC)病毒所最初建立测定COVID-19核酸的FQ-PCR法,选用COVID-19开放读码框ORF-1ab和核壳蛋白N基因设计引物和探针,建立多个位点RT-PCR试验方法。(1)引物和探针设计:选用COVID-19序列中开放读框ORF1ab基因和核壳蛋白N基因设计引物与探针(见表3)。
表3. ORF1ab和N基因引物和探针序列
(2)核酸提取和实时荧光RT-PCR反应体系:参考相关厂家试剂盒说明。(3)结果判断阴性:无Ct值或Ct为40;阳性:Ct值<37,可报告阳性;可疑:Ct值在37~40之间,建议重做试验,若重做结果, Ct值<40,扩增曲线有明显起峰,该样本判断为阳性,否则判断为阴性。
3. 环介导等温扩增(LAMP):LAMP技术所需设备简单,检测性能与实时荧光RT-PCR相近,检测时间短[21]。目前,我国已有多个团队应用LAMP技术完成了COVID-19检测试剂的研发。Lu等报告采用不匹配耐受扩增技术,建立了一种简单、快速、灵敏、直观的基于N基因的反转录环介导等温扩增(RT-LAMP)检测法[22]。该法特异性强,灵敏度高,重现性好,结果可通过FQ-PCR仪检测,也可通过比色由红色变为黄色进行可视化,其检出限为每25μL反应118.6copies RNA。当每25μL反应中,输入模板超过200copies,反应可在30min内完成实时荧光监测,或在40min内完成目视检测,检测方法如下:
(1)引物设计:根据COVID-19基因组序列,利用open access Primer Explorer V.5软件(http://primerexplorer.jp/)设计多套针对N、S和RdRp基因特异性LAMP引物。首先通过与其他6种人冠状病毒序列比对和使用BLAST工具在NCBI上进行同源性搜索,然后通过非模板对照(NTC)进行RT-LAMP反应,评估引物特异性。在NTC反应中剔除非特异性扩增引物后,发现其中一组针对N基因的引物扩增效率更高,因此选择该引物用于RT-PCR-LAMP检测(表4)。
表4. COVID-19 RT-LAMP检测法引物序列

(2)RNA提取:使用商售RNA提取试剂盒(Liferiver,China,Shanghai)从300μL疑似COVID-19患者的咽拭子中提取病毒RNA,并在90μL无核酸水中洗脱,立即使用或在-80℃保存。
(3)扩增体系:RT-LAMP反应体系总容量25µL,含1x等温扩增缓冲液,6mmol/L MgSO4,1.4mmol/L dNTPsl,8单位Warm Start Bst 3.0 DNA聚合酶,7.5单位Warm Start R RT,0.15单位Q5高保真DNA聚合酶,内引物FIP和BIP各1.6μmol/L,外引物F3、B3各0.2μmol/L,0.4μmol/L LAMP LB引物,0.4mmol/L SYTO9(Life technologies,Carlsbad,CA,USA)。每次RT-LAMP反应中加入3μL RNA标准品或模板。采用德国曼海姆(Roche Diagnostics公司)LightCycler 96 rt-PCR系统,在63℃反应50min,进行实时监测,报告结果。
4. 核酸质谱技术:是一种新型软电离生物质谱分析技术, 具有高灵敏度、高通量、操作简单,可同时检测多种病原体等特点,适用于呼吸系统感染性疾病病原体的鉴定[23, 24]。目前,国内已有企业如达瑞生物等研发出可同时检测20多种病原体(包括COVID-19)的核酸质谱试剂盒。王俊等报告通过对PCR-核酸飞行时间质谱(MALDI-TOF MS)检测法的优化[25],把PCR-TOF MS检测法结合多重PCR技术、单碱基延伸技术,设计SNP(single nucleotide polymorphism)单核苷酸多态性位点,对延伸后不同片段的核酸序列进行分子量检测,无须进行荧光信号的采集。此外,PCR-TOF-MS系统还可同时检测出延伸探针与延伸产物之间只有一个碱基的分子量差异,检测范围在1000~10000 Da,达到中等通量检测目的,提高了对低COVID-19含量病例检测的灵敏度。(1)仪器和试剂:采用国产Clin-ToF Ⅱ(北京毅新博创生物科技有限公司)。(2)试验方法:PCR-TOF MS技术方法的建立参考美国Agena Bioscience 核酸质谱法,检测步骤包括:PCR扩增、虾碱性磷酸酶(Shrimp Alkaline Phosphatase,SAP)消化、延伸反应、纯化和点样,以及飞行时间质谱系统对微阵列芯片进行的信号读取和判别,并对加入模版的量进行优化,根据阳性质粒质谱峰的信噪比值(signal-to-noise ratio,SNR),形成判断标准。(3)实验结果分析:利用受试者工作特征曲线法(ROC)确定ORF1ab基因和N基因参考值均为Ct值=38。通过对PCR-TOF MS模板条件的优化,可对COVID-19 ORF1ab和N基因进行有效扩增。该法对临床疑似病例样本的检出率为90.00%,高于FQ-PCR法(75.00%)。与此同时,检出了FQ-PCR 法中存在25.00%的可疑结果。对痰液和咽拭子两种样本的检出率分别为100.00%和85.45%,高于FQ-PCR法(64.00%和43.64%),阴性样本两法均未检出。通过PCR技术与MALDI-TOF MS结合,使得检测COVID-19目标片段指数级扩大,从而提高样本检测的灵敏度,准确性和特异性。同时证实了痰液样本优于咽拭子样本。作为FQ-PCR法的补充和协同,在一定程度上有利于弥补假阴性的问题,从而降低临床漏诊率,提高检测效率,为疫情的研判缩短时间。
5. 核酸POCT技术:POCT(Point-of-Care Testing)现场快速检验,简称即时检验,是使用POCT的免疫诊断新技术,可以直接快速检测临床标本中的COVID-19[26]。一台POCT仪及相关配套的卡、板、条等试剂,集合了多学科的新技术,可视为高新技术的微缩。使用小型便携式POCT测定仪,无需肉眼观察定性结果,是一种简化、便携式的检测方式,操作者可在短时间内完成核酸扩增、信号收集与结果分析,并有效防止交叉污染。
目前,国内已有企业,如中山达安等研发出AGS8830 COVID-19快速核酸检测系统,28min就可做出报告;中科院苏州医工所开发出“基于杂交捕获免疫荧光法(HC-IFA)对新冠病毒核酸现场快速检测系统”可在45min左右做出定性结果。美康生物研发的COVID-19蛋白快速检测试剂盒,采用免疫层析法,通过分别标记在纳米(nm)颗粒上和固定在硝酸纤维膜检测区带上的特异性抗体,与样本中的COVID-19蛋白抗原形成双抗体夹心的免疫反应,并运用纳米颗粒的显色作用,从而实现对特异性的COVID-19蛋白的定性检测,用于COVID-19初步快速筛查,10min可出结果。美国雅培(Abbott Laboratories)研制的检测COVID-19分子的即时检测(POCT)已获美国FDA紧急使用授权,该法可在5min内给出阳性结果,13min内给出阴性结果。Wolters等研究报告[27],建立Xpert Xpress COVID-19 POCT检测法与常规rt-PCR检测结果相当,检出限为8.26copies/mL,在临床样本检测中,一致性达到100%,对快速、准确的诊断COVID-19,是一个有价值的补充。Augustine等报告[28],把LAMP技术结合POCT,建立一个快速、敏感、特异、成本低廉的POCT快速测试法,直接从临床样本中提取,而不需要分离RNA。此外,LAMP干试剂的有效使用,简化了样品的储存和处理,而不影响灵敏度和特异性。据邓均等报告[29],我国对POCT的质量保证体系和管理规范化正在不断完善,拟定了《现场快速检测(POCT)专家共识》,通过对POCT临床应用进行规范化管理,充分发挥临床使用效益。
6. 基因测序技术:基因测序能直接对各种病原微生物或从患者呼吸道分泌物中对其分析测定出各基因组全序列,用于鉴定各种病原微生物或对罹患各种疾病做出早期诊断。基因测序在COVID-19病原学研究、鉴定、早期诊断、流行病学调查、及其突变株新种的产生和对人类疾病的影响等各个方面得到广泛的应用[30]。
(1)宏基因组测序(mNGS)临床应用: 我国科学家利用宏基因组二代测序技术(mNGS)于2020年1月7日在短时间内从患者体内分离出COVID-19,并进行全基因组测序,当时命名为2019-nCoV,并向世卫组织(WHO)提供报告。Lu等[31]利用英国第2代测序仪(纳米孔测序仪Min ION)从9名患者的支气管肺泡灌洗液样本培养分离物中获得10条(COVID-19)几乎相同的序列,这些序列与2018年在中国东部舟山采集的两种蝙蝠衍生的bat-SL-CoVZC45和bat-SL-CoVZXC21一致性为88%,与SARS-CoV(约79%)和MERS-CoV-2(约50%)相差较大。Chen等[32]使用低输入mNGS法,从支气管肺泡灌洗液(BALF)中提取RNA,可确保有较高的丰度值,并迅速鉴定出样本中唯一的病原体。最近,Di等开发了一种基于Tn5转座酶的转录组测序快速建库方法[33],对建库过程作了优化,同时对样本量的需求量较小,可用于高通量单细胞转录组测序,提升COVID-19等样本测序的质量和速度,适用于最初病毒的鉴定和后期病毒的进一步研究。当测序结果与已知COVID-19序列高度同源时便可作出确诊[34]。最近,Kirby报告在英国出现一种COVID-19新变种毒株[35],据英国冠状病毒新变种命名法,新变种指定为VUI 202012/01,基因序列为B.1.1.7亚型,经审核被重新指定为VOC 202012/01。目前,COVID-19新变种(VU1)在欧盟、美、加、亚、非等世界范围内传播和蔓延。中国CDC报告,我国也有3省发现英国新冠变异毒株感染的确诊患者。其基因序列与英国报道的新变异病毒株相类似。
(2)第二代宏基因组高通量测序:mNGS是一种基于二代测序技术,无需培养,可对临床样本中直接提取DNA或RNA进行高通量测序,再经数据库比对与生物信息学分析,一次性完成对病毒、细菌等多种病原体检测[36]。有关mNGS测序流程大致分为5个步骤:核酸提取、文库构建、上机测序、生物信息化分析与报告解读,在目前临床上对COVID-19这类新发病原体的快速鉴定、疑似病例的鉴别诊断和确诊患者的混合感染中发挥重要作用。
(3)第三代纳米孔高通量测序:李梦婷等报告[37],第三代纳米孔高通量测序以牛津纳米孔公司的设备(Oxford Nanopore Technologies),可以对DNA和RNA进行测序,并提供快速分析。纳米孔(nanopore)测序技术可直接对核酸片段进行长全长读取,同时具有极高的精确度,高样本检测效率,被应用于许多学科方面的研究。目前,国内外已有多个团队采用纳米孔基因测序技术对新型冠状病毒进行高通量基因测序。据Chan等报告[38], 使用纳米孔测序技术检测COVID-19,样本来源于确诊的2名患者及其他5名家族成员,做全基因组测序。直接从样本中提取核酸,去除宿主DNA后,应用不依赖序列的单引物扩增法,用特异性引物A(5' -GTTTCCCACTGGAGGATA-N9-3')将DNA酶处理的RNA进行逆转录为cDNA,利用Klenow片段(3' → 5' exo-)进行cDNA 第二条链的合成(新英格兰生物实验室,马萨诸塞州伊普斯维奇)。用引物B(5' -GTTTCCCAC TGGAGGATA-3')进行PCR扩增,获得扩增的cDNA文库。进行纳米孔测序文库构建,文库准备完成即可进行测序,采用R9.4.1进行12~48h测序。利用MEGAX软件构建进化树,进行数据分析。他们发现了一种与蝙蝠SARS样冠状病毒bat-SL-CoVZXC21(NCBI登录号MG772934)和bat-SL-CoVZC45(NCBI登录号MG772933)关系最为密切的新型冠状病毒,且其中2名患者的病毒株基因组序列完全一致,提示为同一病毒株感染。2名患者家属也存在同一病毒株感染情况,故提示该病毒具有传染性。实验发现,痰样本来源的循环阈值比咽拭子样本来源早8~13个循环,表明下呼吸道病毒载量较高。为了更好地对新型冠状病毒进行测序研究,纳米孔公司发布了一个关于2019-nCoV测序的实验流程,利用针对COVID-19特异性的98对引物对患者核酸样本的cDNA进行PCR扩增。纳米孔技术具有长读长优势,对未知新型肺炎病原体检测、鉴定具有重要意义,有利于临床初筛阴性样本,对疫情控制具有重要作用。
小结:针对COVID-19新冠肺炎的疫情暴发,目前临床上病原检测存在假阴性、病毒分离鉴定时间较长,病毒变异的潜在性等问题,应用高通量测序技术很好地解决了这些问题,对于COVID-19能更好地鉴定和分析其来源、新种变异情况,减少临床检查的盲目性,且检出速率较高,特别是采用纳米孔基因测序技术对COVID-19基因组进行高通量测序,发现其与参考基因组一致性为100%,并可在8h内完成从样本到结果的获取,进一步体现了高通量测序在COVID-19鉴定应用中的优势,对提高处理重大暴发事件的应急能力,保障人类生命安全有重大意义。
参考文献
Dhama K, Khan S, Tiwari R,et al. Coronavirus Disease 2019-COVID-19. Clin Microbiol Rev. 2020; 33(4): e00028-20.
Zhu N, Zhang D, Wang W, et al. A Novel coronavirus from patients with pneumonia in China, 2019[J]. N Engl J Med, 2020. 382, 727-733.
Ge XY, Li JL, Yang XL, et al. Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor. Nature. 2013,503:535-538. https://doi.org/10.1038/nature12711.
WHO Director-General's remarks at the media briefing on 2019-nCoV on 11 February 2020. https://www.who.int/dg/speeches/detail/who-director-general-s-remarks-at-the-media-briefing-on-2019-ncov-on-11-february-2020
Woo PC, Lau SK, Lam CS, et al. Discovery of seven novel mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus. J Virol 2012, 86:3995-4008.
Guan WJ, Ni ZY, Hu Y, et al. Clinical Characteristics of Coronavirus Disease 2019 in China. N Engl J Med. 2020, 382(18):1708-1720.
Ge H, Wang X, Yuan X, et al. The epidemiology and clinical information about COVID-19. Eur J Clin Microbiol Infect Dis. 2020 Jun; 39(6): 1011-1019.
Wu F, Zhao S, Yu B,et al. A new coronavirus associated with human respiratory disease in China. Nature. 2020, 579(3): 265-269.
Khailany RA, Safdar M, Ozaslan M. Genomic characterization of a novel SARS-CoV-2. Gene Rep. 2020, 19: 100682.
Zhang C, Zheng W, Huang X, et al. Protein Structure and Sequence Reanalysis of 2019-nCoV Genome Refutes Snakes as Its Intermediate Host and the Unique Similarity between Its Spike Protein Insertions and HIV-1. J Proteome Res. 2020, 19(4): 1351-1360.
Rabaan AA, Al-Ahmed SH, Haque S, et al. SARS-CoV-2, SARS-CoV, and MERS-CoV: a comparative overview. Infez Med. 2020, 28(2): 174-184.
Li F,. Structure, function, and evolution of coronavirus spike proteins. Annu Rev Virol. 2016, 3: 237-261.
World Health Organization. Molecular assays to diagnose COVID-19: Summary table of available protocols. 2020. https://www.who.int/who-documents-detail/molecular-assays-to-diagnose-covid-19-summary-table-of-available-protocols
Udugama B, Kadhiresan P, Kozlowski HN, et al. Diagnosing COVID-19: The Disease and Tools for Detection . ACS Nano 2020, 14, 4, 3822-3835
Li Y, Yao L, Li J, et al. Stability issues of RT-PCR testing of SARS-CoV-2 for hospitalized patients clinically diagnosed with COVID-19. J Med Virol. 2020, 92(7): 903-908.
Ren LL, Wang YM, Wu ZQ, et al. Identification of a novel coronavirus causing severe pneumonia in human: a descriptive study. Chin Med J. 2020, 133: 1015-1024.
Yip CC, Sridhar S, Leung KH, et al. Development and Evaluation of Novel and Highly Sensitive Single-Tube Nested Real-Time RT-PCR Assays for SARS-CoV-2 Detection. Int J Mol Sci. 2020, 21(16): 5674.
郭元元, 王昆, 张宇, 等. 6种国产新型冠状病毒核酸检测试剂检测性能比较与分析. 重庆医学2020, 49(15): 2435-2439.
Chu DKW, Pan Y, Cheng SMS, et al. Molecular Diagnosis of a Novel Coronavirus (2019-nCoV) Causing an Outbreak of Pneumonia. Clin Chem, 2020, 66(4): 549-555.
陈炜, 张春阳, 朱颖, 等. 4例新型冠状病毒感染病例咽拭子与痰标本病毒核酸检测的比较. 中国人兽共患病学报, 2020, 36(5): 354-358.
Lu R, Wu X , Wan Z, et al. A Novel Reverse Transcription Loop-Mediated Isothermal Amplification Method for Rapid Detection of SARS-CoV-2. Int J Mol Sci. 2020, 18; 21(8): 2826.
Shirato K, Semba S, Kafrawy SA, et al. Development of fluorescent reverse transcription loopmediated isothermal amplification (RT-LAMP) using quenching probes for the detection of the Middle East respiratory syndrome coronavirus[J]. J Virol Methods, 2018, 258: 41-8.
23. Rocca MF, Zintgraff JC, et al. Dattero ME,A combined approach of MALDI-TOF mass spectrometry and multivariate analysis as a potential tool for the detection of SARS-CoV-2 virus in nasopharyngeal swabs.J Virol Methods. 2020, 286: 113991.
Xiu L,Zhang C,Wu Z,. Establishment and Application of a Universal Coronavirus Screening Method using MALDI-TOF mass spectrometry. Front Microbiol. 2017, 8: 1510.
王俊, 张栋, 陈晴晴, 等. PCR-核酸飞行时间质谱系统检测新型冠状病毒方法的建立及应用研究. 中国全科医学2020, 23(35): 4430-4435.
Yang T, Wang YC, Shen CF, et al. Point-of-care RNA-based diagnostic device for COVID-19. Diagnostics 2020, 10: 165.
Wolters F, van de Bovenkamp J, van den Bosch B, et al. Multi-center evaluation of cepheid xpert xpress SARS-CoV-2 point-of-care test during the SARS-CoV-2 pandemic. J Clin Virol. 2020, 128: 104426.
Augustine R, Hasan A, Suvarthi Das S, et al. Loop-Mediated Isothermal Amplification (LAMP): A Rapid, Sensitive, Specific, and Cost-Effective Point-of-Care Test for Corona-viruses in the Context of COVID-19 Pandemic. Biology (Basel). 2020, 9(8): 182.
邓均, 宋世平,郑峻松. 我国POCT发展现状与展望. 临床检验杂志2015, 33(11): 844-845.
Wang H, Li X, Li T, et al. The genetic sequence, origin, and diagnosis of SARS-CoV-2. Eur J Clin Microbiol Infect Dis, 2020, 39: 1629-1635.
Lu R, Zhao X, Li J, et al. Genomic characterization and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding[J]. Lancet, 2020, 395(10224): 565-574.
Chen L, Liu W, Zhang Q, et al. RNA based mNGS approach identifies a novel human coronavirus from two individual pneumonia cases in 2019 Wuhan outbreak[J]. Emerg Microbes Infect, 2020, 9(1): 313-9.
Di L, Fu Y, Sun Y, et al. RNA sequencing by direct tagmentation of RNA/DNA hybrids[J]. Proc Natl Acad Sci USA, 2020, 117(6): 2886-93.
Chan JF, Kok KH, Zhu Z, et al. Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan. Emerg Microbes Infect. 2020, 9: 221-236
Kirby T. New variant of SARS-CoV-2 in UK causes surge of COVID-19. Lancet Respir Med. 2021, S2213-2600 (21) 00005-9.
Li B, Si HR, Zhu Y, et al. Discovery of bat coronaviruses through surveillance and probe capture-based next-generation sequencing. MSphere. 2020, 5: e00807-19.
李梦婷, 祝小荐, 万绍贵. 高通量基因测序技术在新型冠状病毒检测中的应用. 赣南医学院学报. 2020, 40(03): 225-228.
Chan WM, Ip JD, Chu AW, et al. Identification of nsp1 gene as the target of SARS-CoV-2 real-time RT-PCR using nanopore whole-genome sequencing. J Med Virol. 2020, 92(11): 2725-2734.





