血浆微生物游离DNA宏基因组高通量测序在重症监护室脓毒症 诊断中的应用
【摘要】目的 评价重症监护室脓毒症患者中血浆微生物游离DNA(mcfDNA)宏基因组高通量测序(mNGS)的临床应用价值。方法 纳入2020年1月至2020年6月疑似脓毒症病例199例,同时进行血培养和血浆mcfDNA mNGS,以检测血流感染病原体。同时进行其他传统的临床微生物检测,包括涂片、相关感染部位的样本培养和β-D-葡聚糖/半乳甘露聚糖试验等。结果 以临床诊疗小组最终诊断为参考标准,血浆mcfDNA测序诊断脓毒症的敏感性和特异性分别为68.1%和63.2%。对于引起医院感染常见细菌的检出,血浆mcfDNA测序明显优于血培养,如鲍曼不动杆菌(p<0.01)、肺炎克雷伯菌(p<0.01);仅血浆mcfDNA测序可检测到病毒。在抗生素未暴露的病例中,血浆mcfDNA测序与血培养的阳性率分别为43.6%和30.9%,差异无统计学意义(p=0.17);抗生素暴露后,血浆mcfDNA测序阳性率显著高于血培养(52.8%,25.0%,p<0.01)。在非脓毒症组中,44.8%(13/29)的病例通过血浆mcfDNA测序发现了其他部位感染,如下呼吸道感染、腹腔内感染和中枢神经系统感染。随着疾病进展,血浆mcfDNA测序可以检测到微生物种类的显著动态变化。结论 血浆微生物游离DNA宏基因组高通量测序(mcfDNA mNGS)可作为重症监护室脓毒症患者病原学诊断技术的有益补充,有效提升临床诊治水平。
脓毒症是重症监护室(intensive care unit,ICU)最致命的疾病之一,其死亡率约占住院期间死亡人数的三分之一[1]。因此,需要早期诊断和治疗来降低全因死亡率,且拯救脓毒症运动(Surviving SepsisCampaign,SSC)指南提出推荐使用“1h集束化”治疗策略[2]。然而,最初的经验性抗生素治疗效果不佳,导致了不必要的广谱抗生素暴露。此外,对于培养阴性的脓毒症患者,准确识别病原体仍然是一个挑战[3]。
在纯培养物中分离微生物是识别导致脓毒症病原体的最常见方法[4]。常见的传统检测方法如生化表型检测和基质辅助激光解吸/电离飞行时间质谱(MALDI-TOF MS)等,阳性率仅达到30-40%[4]。因此,需要不依赖于培养的技术来早期诊断并指导抗菌药物的合理使用[5]。作为一种分子诊断技术,多重聚合酶链反应被广泛应用于脓毒症的病原体鉴定,但由于病原体预设不明确,不能满足病原学不明的复杂临床标本的要求[4]。在过去十年中,宏基因组下一代测序(metagenomic next-generation sequencing,mNGS)技术通过检测微生物核酸来诊断感染性疾病,但其结果仍是复杂且不明确的[6, 7]。因此,迫切需要一种理想的诊断方法,能够在多器官功能障碍综合征(MODS)发生之前快速确定病原体并指导抗生素治疗。
血浆游离DNA(cell-free DNA,cfDNA)最早发现于上个世纪。循环中的cfDNA分子来自死亡细胞和定植/入侵的微生物,它们在分解时将核酸释放到血液中[8]。目前,作为一项新兴技术,血浆mcfDNA测序技术在各种侵袭性感染中的诊断性能尚不清楚。因此,本研究比较血浆mcfDNA测序和血培养对ICU患者脓毒症的诊断能力,以证明血浆mcfDNA测序在预测和监控感染性疾病方面具有广阔前景。
一、研究对象与研究方法
1. 研究对象:前瞻性研究,研究对象为2020年1月至2020年6月上海市东方医院ICU的疑似脓毒症患者。纳入标准:(1)疑似血流感染的患者(发烧或低温、白细胞增多或减少和PCT升高);(2)样本的相关信息完整,包括年龄、性别、采集日期、临床诊断、既往病史、抗菌药物使用情况等。排除标准:(1)拒绝行相应病原微生物学证据采集;(2)病情危重,预计入院后48h内死亡;(3)年龄<18岁或>80岁。收集的临床数据来自电子病历系统,包括基线人口学特征、临床诊断、预后以及相关的实验室检查结果。根据ICU专家对疾病的最终诊断,将患者分为感染性疾病组(ID组)和非感染性疾病组(NID组)。此外,根据sepsis-3.0的定义,感染性疾病组(ID组)被分为两个亚组,分别是脓毒症组和非脓毒症组[9]。对采集的血液样本同时进行常规血培养(Bact/alert Virtuo,BioMérieux,France)和血浆mcfDNA测序(BGI,China)。其他临床微生物检测包括涂片、其他感染部位的样本培养以及β-D-葡聚糖/半乳甘露聚糖(BDG/GM)试验等。该研究由上海市东方医院伦理委员会批准(2018-099号),并根据《赫尔辛基宣言》(2013年修订)进行。所有患者或其近亲均有知情权并签署知情同意书。
2. 研究方法:血浆mcfDNA测序与分析:(1)样本处理和DNA提取:取3-5mL血液样本,将采血管置于4℃,3000rpm离心10分钟,取300微升血浆于新的离心管中。若不及时检测,需冻存于-80℃,以防止核酸降解。使用TIANamp Micro DNA Kit(DP316,TIANGEN BIOTECH,Beijing,China),根据试剂盒说明书提取DNA,提取的DNA用于DNA文库构建。(2)构建DNA文库:对提取的核酸进行末端修复、接头连接和PCR扩增,以制备DNA文库。使用Qubit dsDNA HS分析试剂盒(Thermo Fisher Science,USA)对DNA文库进行定量,并将文库等量混合。将合格的DNA文库进行环化,形成单链环状结构,经滚环复制(rolling circle amplification,RCA)制备DNA纳米球(DNA nanoball,DNB)。将制备好的DNB加载到测序芯片上,使用BGISEQ-50测序平台进行测序。(3)生物信息学分析:测序数据下机后,去除低质量的数据以获得高质量的测序数据。通过Burrows-Wheeler比对,去除高质量数据中与人类参考基因组(hg19)匹配的序列。去除低复杂性数据后,其余数据同时与四个微生物基因组数据库(PMSEQ® 商业数据库)进行比对,包括细菌、真菌、病毒和寄生虫。RefSeq包含6350个细菌基因组,1064个真菌,1798个DNA病毒和234个寄生虫。
3. 病原体具体报告规则:(1)细菌(不包括分枝杆菌)、真菌、寄生虫、支原体/衣原体/立克次体:对样本中鉴定的微生物按Standard Genus Stringly MAP Reads Number(SDG_SMRN)进行排序,对排名前五的属进一步分析。根据Standard Species Stringently Mapped ReadsNumber(Latin SMRN)对种进行排序,每个属不超过两个种,且严格比对的序列数需≥3。(2)病毒:对样本中鉴定的微生物按Standard Genus Stringly MAP Reads Number(SDG_SMRN)进行排序,对排名前五的属进一步分析,且严格比对的序列数需≥3。(3)分枝杆菌:当严格比对的序列数≥1时,结核分枝杆菌为阳性。非结核分枝杆菌鉴定原理与上述原理(1)相同。(4)除SMRN外,还综合考虑了覆盖度、深度和Shannon指数等参数。
4. 统计分析:数据分析采用SPSS 22.0软件。比较分析采用Pearson χ2检验、McNemar检验或Fisher精确检验。p<0.05具有显著性意义,所有检验均为双侧检验。
二、研究结果与分析
1. 患者和样本基本特征:根据纳入和排除标准,199例病例纳入该研究。181例病例被诊断为感染性病(图1)。其中男性占比64.8%。ICU平均住院时间为13天,28天死亡率为27.6%(补充表S1)。连续采样共102次,涉及44例患者。该研究中,血流感染是最常见的感染性疾病(94,47.2%),其次是下呼吸道感染(LRT)。在非脓毒症组中[87/181(48.1%)],下呼吸道感染最常见[68/87(78.2%)],其次是泌尿系感染(UTIs)[14/87(16.1%)]和腹腔感染(IAIs)[13/87(14.9%)](图2)。在脓毒症组中,64(68.1%)个样本通过两种方法都检测出病原体(补充表S2)。此外,对11例患者进行了多次mNGS检测(采样≥3次),其中8例被诊断为脓毒症。

图1. 研究流程图(ICU,重症监护室)
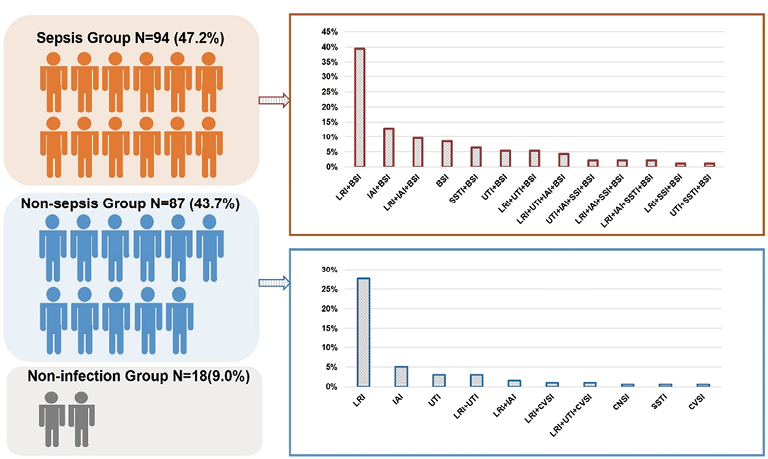
注:BSI,血液感染;CVSI,心血管感染;LRI,下呼吸道感染;SSTI,皮肤或软组织感染;UTI,尿路感染;IAI,腹内感染;CNSI,中枢神经系统感染。
图2. 本研究中脓毒症组和非脓毒症组的分布
2. 血浆mcfDNA测序与血培养的关系:(1)脓毒症与非脓毒症的鉴别诊断性能:在脓毒症组,94例mcfDNA测序试验中有64例至少检测出一种微生物(补充表S2)。细菌是最常检测到的物种,58例检出了至少一种细菌。排除NID组(n=18),181例病例纳入该诊断性能研究。脓毒症组和非脓毒症组的血浆mcfDNA测序阳性率均显著高于血培养(p<0.01)(图3A)。血浆mcfDNA测序诊断脓毒症的阳性预测值(PPV)为66.7%,阴性预测值(NPV)为64.7%,阳性似然比为1.85,阴性似然比为0.50。血浆mcfDNA测序的敏感性显著高于血培养(68.1% vs.40.4%,p<0.01),但与血培养相比,血浆mcfDNA测序的特异性降低了约20%(63.2% vs. 82.8%,p<0.01)(图3B)。
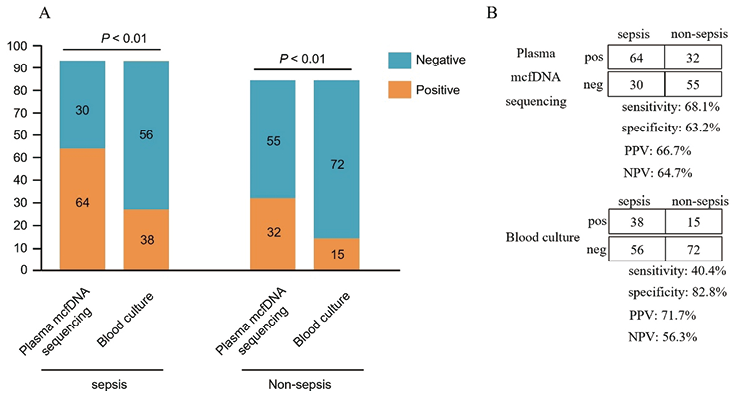
注:A、针对脓毒症和非脓毒症的血浆mcfDNA和血培养检测两种方法的检出率比较。B、血浆mcfDNA测序和血培养区分脓毒症组和非脓毒症组的诊断性能分析。NPV,阴性预测值;PPV,阳性预测值。pos,阳性;neg,阴性。
图3. 脓毒症组和非脓毒症组血浆微生物游离DNA(mcfDNA)测序和血培养的比较
(2)血浆mcfDNA测序与血培养的相关性和一致性:199例病例中,41例(20.6%)血浆mcfDNA测序和血培养均阳性,87例(43.7%)均阴性。59例(29.6%)仅血浆mcfDNA测序阳性,12例(6.0%)仅血培养检测阳性。双阳性的案例中(n=41),16例病原体完全匹配,12例完全不匹配。其余13例是“部分匹配”的,即至少有一种病原体重叠(图4)。在非脓毒症组中,血浆mcfDNA测序检测到的微生物可能不是引起脓毒症的原因,包括一些重新激活的疱疹病毒(4/29)、幽门螺杆菌等慢性感染(2/29)、可能共生的微生物(9/29)以及非脓毒症相关的急性感染病因(13/29)(补充表S3)。
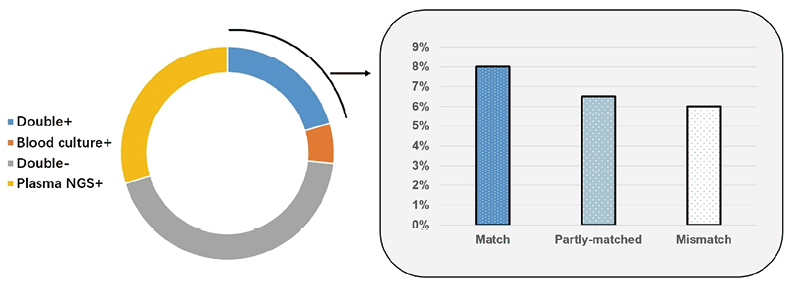
图4. 所有样本血浆微生物游离DNA(mcfDNA)测序与血培养之间的相关性(n=199)
(3)抗生素暴露对病原菌检测的影响:纳入的所有病例均来自ICU,由于大多数患者入院后需进行呼吸支持,因此在血浆mcfDNA测序和血培养前抗生素暴露率较高(144/199,72.4%)。在抗生素未暴露的患者(n=55)中,血浆mcfDNA阳性率与对照组无显著性差异(43.6% vs. 30.9%,p=0.17)。因既往使用抗生素(n=144),血浆mcfDNA测序阳性率显著高于血培养(52.8% vs.25.0%,p<0.01)(图5)。
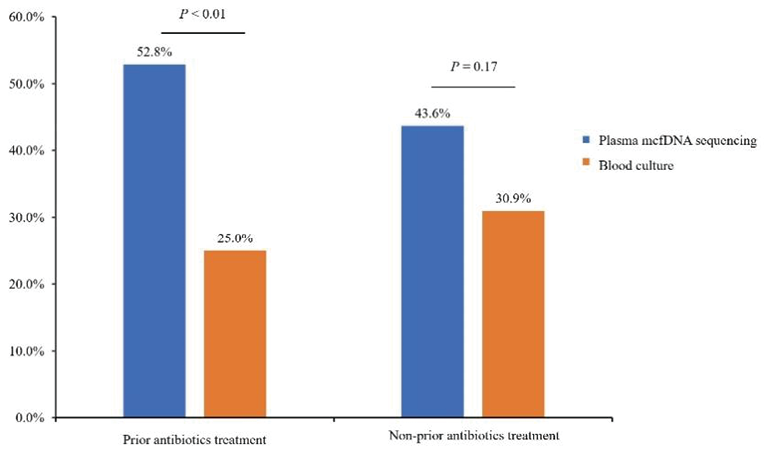
图5. 既往抗生素暴露对血浆微生物游离DNA(mcfDNA)测序阳性率的影响
3. 血浆mcfDNA测序与血培养在时效性和病原学方面的比较:(1)时效性的比较分析:表1列出了血培养中最常见的8种病原体及其周转时间(TAT)。利用我们实验室平台,从血液样本核酸提取到完成测序分析的周转时间(TAT3)约为26h。相比之下,病原体不同,血培养周转时间有所不同。在8种病原体中,鲍曼不动杆菌的TAT1(从血培养瓶上机到下机的周转时间)最短,仅为9.4h。厌氧菌的检测时间最长,为34.1h。对于念珠菌,血培养(TAT1)与血浆mcfDNA测序(TAT3)时长相当。TAT2(从血培养瓶上机到结果报告的周转时间)明显长于TAT3(p<0.01)。
表. 血浆mcfDNA测序与血培养在时效性方面的比较
|
Pathogens |
TAT1a |
P1b |
TAT2a |
P2b |
|
Acinetobacter baumannii |
9.4 (9.2-10.3) |
< 0.01 |
66.9 (49.0-77.8) |
< 0.01 |
|
Klebsiella pneumoniae |
11.1 (10.2-14.4) |
< 0.01 |
66.3 (47.8-74.5) |
< 0.01 |
|
Escherichia Coli |
10.4 (7.9-13.2) |
< 0.01 |
66.5 (53.2-67.5) |
< 0.01 |
|
Anaerobes |
34.1 (32.9-35.2) |
0.33 |
87.2 (80.3-94.2) |
< 0.01 |
|
Staphylococcus aureus |
15.9 (15.2-19.4) |
< 0.01 |
70.2 (50.2-87.2) |
< 0.01 |
|
Enterococcus |
13.3 (12.1-15.1) |
< 0.01 |
64.2 (51.5-68.4) |
< 0.01 |
|
Staphylococcus |
20.7 (17.7-29.7) |
< 0.01 |
73.2 (63.2-95.3) |
< 0.01 |
|
epidermidis/ capitis/hominis/haemolyticus |
|
|
|
|
|
Candida |
25.3 (18.6-28.5) |
0.15 |
83.1 (73.1-92.8) |
< 0.01 |
注:a由于非正态分布,数据以中位数(IQR)表示。b两组之间的比较是使用Mann-Whitney U检验进行的。TAT1,从血培养瓶上机到下机的周转时间;TAT2,从血培养瓶上机到结果报告的周转时间;TAT3,我们的平台从血液样本核酸提取到完成测序分析的实验室周转时间约为26h。P1,比较TAT1和TAT3;P2,比较TAT2和TAT3。IQR,四分位间距。TAT,周转时间。
(2)病原菌的比较分析:如图6所示,这两种方法最常检测到的微生物包括鲍曼不动杆菌、肺炎克雷伯菌和大肠埃希菌。总体而言,血浆mcfDNA测序的阳性检出率明显高于血培养([OR]=4.8,95%可信区间 (CI)=2.6-9.0,p<0.01),鲍曼不动杆菌(p<0.01),肺炎克雷伯菌(p<0.01),肠球菌(p=0.02),真菌(p=0.03)和病毒(仅血浆mcfDNA测序检测到,p<0.01)。对于厌氧菌,血浆mcfDNA测序的检出率高于血培养,但由于样本量小,差异不显著(p=0.25)。
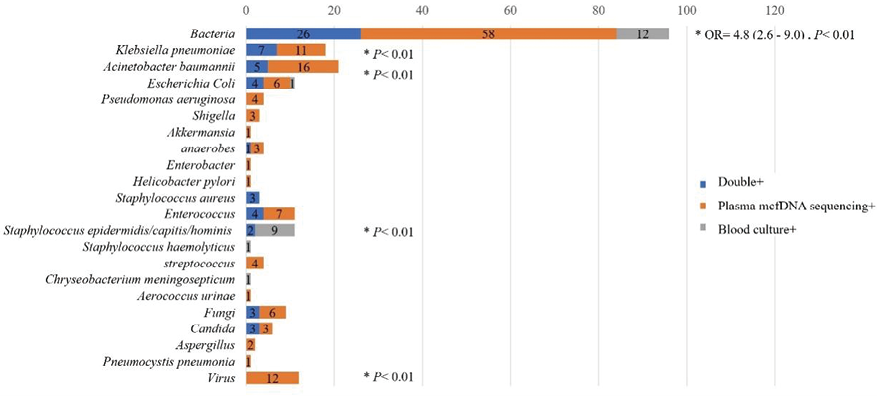
图6. 血浆微生物游离DNA(mcfDNA)测序和血培养检测的比较
4. 连续采样的案例分析:在临床上,11例患者处于复杂感染状态,如补充表S4所述,对其进行动态血浆mcfDNA测序,以确定这些患者在治疗过程中存在的微生物种类变化。在PT001患者重症肺炎早期,血浆mcfDNA测序快速、准确地检测出血液的肺炎克雷伯菌,与BALF培养结果具有良好的一致性。随着疾病进展,血液中肺炎克雷伯菌的载量增加,血浆mcfDNA测序显示肺炎克雷伯菌、铜绿假单胞菌和鲍曼不动杆菌的混合感染,导致重症肺炎加重,进一步发展为脓毒血症。PT043患者因鼻咽癌继发呼吸衰竭住进ICU,经治疗血液中EBV核酸逐渐清除。然而,化疗导致患者免疫功能降低,发展为鲍曼不动杆菌感染。
三、研究结论
宏基因组下一代测序已被广泛应用于病原体检测[10]。血浆mcfDNA测序的最大优点是能检测出难以培养的病原体,从而克服了传统微生物检测的缺点。作为一种非侵入性的疾病诊断方法,它可以通过捕获和鉴定血液中高度片段化的mcfDNA来诊断可能的感染。由于72.4%的ICU患者在采样前使用了抗生素,血浆mcfDNA测序对肺炎克雷伯菌、鲍曼不动杆菌等引起医院感染的常见菌的检出率明显高于血培养。血浆mcfDNA测序检出凝固酶阴性葡萄球菌的阳性率明显低于血培养(p<0.01),这可能与葡萄球菌的SDG_SMRN不在报告的前五位有关。
在本研究中,常规血培养和血浆mcfDNA测序同时检测ICU患者的潜在病原体,血浆mcfDNA测序诊断脓毒症的敏感性高(68.1%),比传统的血培养方法高28%。一定程度上是由于血培养的技术缺陷,以及局部病灶、挑剔的病原体或者血液中活微生物比例低。由于细菌直接释放DNA或吞噬细胞共释放DNA,脓毒症患者的cfDNA含量明显高于健康人[11]。
血浆mcfDNA测序的特异性低于血培养(63.2% vs.82.8%),假阳性可能是粘膜屏障受损或免疫抑制造成的,特别是在癌症患者和移植受体[12]。在多数情况下,侵入性采样技术对于患有各种严重细菌、真菌或病毒感染的ICU患者来说是难以接受的。一项单中心研究报告显示,在免疫受损的宿主中,61%的血浆mcfDNA测序报告临床相关病原体阳性,总共避免了34项侵入性技术[13]。即使感染限于特定部位,血浆mcfDNA测序也可以检测病原体核酸[14]。在该研究中,在非脓毒症组,仅通过血浆mcfDNA测序检测到的44.8%(13/29)病原体提示了身体其他部位的感染,如LRI、IAI和CNSI。检测到的mcfDNA可能是由于组织粘膜受损时,侵袭性病原体机会性地进入血液,严重时会导致菌血症或病毒血症。然而,血液中细菌DNA载量通常很低。根据Gu等人的研究,体液中病原体cfDNA的载量显著高于血液[15]。
除了诊断人体大部分来源的感染,血浆mcfDNA测序可以检测到比血培养更广泛的病原体谱。血浆mcfDNA测序在32例(32/94,34%)脓毒症病例中检出两种以上病原体。相比之下,血培养往往只能识别一个物种,这可能是由于不同类型的微生物在同一环境中存在竞争[16]。血浆mcfDNA测序也被用来检测病毒。对于血浆mcfDNA测序检测到的病毒是否会引起感染,目前还没有统一的标准,需要根据患者的病史来确定。例如,在PT132的血液中检测到人类细小病毒B19核酸序列(补充表S5)。患者临床表现为心律失常、心力衰竭、心源性休克,与心肌病诊断一致。B19表现出心肌趋向性,是心肌炎的常见原因[17]。然而,一项多中心回顾性队列研究报告了Karius结果呈阳性的病例,mNGS的临床影响不涉及DNA病毒或寄生虫,只涉及细菌和/或真菌[18]。
在中国,下呼吸道感染是最严重的公共卫生问题之一。中国的一项多中心临床调查显示,呼吸科的普通病房和ICU的医院获得性肺炎(HAP)的平均发病率分别为0.9%和15.3%[19]。值得注意的是,我们的数据显示,在重症肺炎患者中,血流感染引起并发症的概率很高,值得关注。最近的大量证据表明,血流感染是重症肺炎患者预后恶化的重要预测因子[20],早期识别重症肺炎患者的脓毒症或血流感染将有助于指导精确抗菌治疗。此外,Chen等人研究发现,除了宏基因组DNA测序外,RNA-seq还能够识别广泛的下呼吸道病原体,且提高对病毒和真菌检出的敏感性[21]。
本研究在以下几个方面存在局限性:1)由于血浆mcfDNA测序没有统一的参考标准,因此感染性疾病诊断的金标准是基于对感染人群的综合评价,包括所有微生物结果的解释、相关的临床生物标志物和临床诊断。2)未对样本进行细菌16SrDNA PCR或真菌28SrDNA-ITS PCR等验证性试验。3)本研究主要比较血培养和血浆mcfDNA测序的诊断性能差异。由于血培养无法检测病毒,本研究设计中未包括RNA检测。上述局限性将在后续的临床研究和实践中逐步完善,但整体来说,血浆微生物游离DNA宏基因组高通量测序(mcfDNA mNGS)可作为重症监护室脓毒症患者病原学诊断技术的有益补充,有效提升临床诊治水平。
参考文献
Zhang Z, Smischney NJ, Zhang H, et al. AME evidence series 001-The Society for Translational Medicine: clinical practice guidelines for diagnosis and early identification of sepsis in the hospital [J]. J Thorac Dis, 2016, 8(9): 2654-65.
Levy MM, Evans LE, Rhodes A. The Surviving Sepsis Campaign Bundle: 2018 update [J]. Intensive Care Med, 2018, 44(6): 925-8.
Thorndike J, Kollef MH. Culture-negative sepsis [J]. Curr Opin Crit Care, 2020, 26(5): 473-7.
Rajapaksha P, Elbourne A, Gangadoo S, et al. A review of methods for the detection of pathogenic microorganisms [J]. Analyst, 2019, 144(2): 396-411.
Rhee C, Chiotos K, Cosgrove SE, et al. Infectious Diseases Society of America Position Paper: Recommended Revisions to the National Severe Sepsis and Septic Shock Early Management Bundle(SEP-1) Sepsis Quality Measure[J]. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America, 2021, 72(4): 541-52.
Cummings LA, Kurosawa K, Hoogestraat DR, et al. Clinical Next Generation Sequencing Outperforms Standard Microbiological Culture for Characterizing Polymicrobial Samples [J]. Clin Chem, 2016, 62(11): 1465-73.
Simner PJ, Miller S, Carroll KC. Understanding the Promises and Hurdles of metagenomic Next-Generation Sequencing as a Diagnostic Tool for Infectious Diseases [J]. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America, 2018, 66(5): 778-88.
Lee RA, Al Dhaheri F, Pollock NR, et al. Assessment of the Clinical Utility of Plasma metagenomic Next-Generation Sequencing in a Pediatric Hospital Population [J]. J Clin Microbiol, 2020, 58(7).
Rhodes A, Evans LE, Alhazzani W, et al. Surviving Sepsis Campaign: International Guidelines for Management of Sepsis and Septic Shock: 2016 [J]. Intensive Care Med, 2017, 43(3): 304-77.
Wilson MR, Sample HA, Zorn KC, et al. Clinical metagenomic Sequencing for Diagnosis of Meningitis and Encephalitis [J]. N Engl J Med, 2019, 380(24): 2327-40.
Grumaz S, Stevens P, Grumaz C, et al. Next-generation sequencing diagnostics of bacteremia in septic patients [J]. Genome Med, 2016, 8(1): 73.
Han D, Li R, Shi J, et al. Liquid biopsy for infectious diseases: a focus on microbial cell-free DNA sequencing [J]. Theranostics, 2020, 10(12): 5501-13.
Rossoff J, Chaudhury S, Soneji M, et al. Noninvasive Diagnosis of Infection Using Plasma Next-Generation Sequencing: A Single-Center Experience [J]. Open Forum Infect Dis, 2019, 6(8).
O'Grady J. A powerful, non-invasive test to rule out infection [J]. Nat Microbiol, 2019, 4(4): 554-5.
Gu W, Deng X, Lee M, et al. Rapid pathogen detection by metagenomic next-generation sequencing of infected body fluids [J]. Nat Med, 2021, 27(1): 115-24.
Wang J, Zeng Y, Wang S, et al. Swine-Derived Probiotic Lactobacillus plantarum Inhibits Growth and Adhesion of Enterotoxigenic Escherichia coli and Mediates Host Defense [J]. Front Microbiol, 2018, 9: 1364.
Pollack A, Kontorovich AR, Fuster V, et al. Viral myocarditis--diagnosis, treatment options, and current controversies [J]. Nat Rev Cardiol, 2015, 12(11): 670-80.
Hogan CA, Yang S, Garner OB, et al. Clinical Impact of metagenomic Next-Generation Sequencing of Plasma Cell-Free DNA for the Diagnosis of Infectious Diseases: A Multicenter Retrospective Cohort Study [J]. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America, 2021, 72(2): 239-45.
Xie J, Wang H, Kang Y, et al. The Epidemiology of Sepsis in Chinese ICUs: A National Cross-Sectional Survey [J]. Crit Care Med, 2020, 48(3): e209-e18.
Zhou X, Wu H, Ruan Q, et al. Clinical evaluation of Diagnosis Efficacy of Active Mycobacterium tuberculosis Complex Infection via metagenomic Next-Generation Sequencing of Direct Clinical Samples [J]. Front Cell Infect Microbiol, 2019, 9: 351.
Chen H, Yin Y, Gao H, et al. Clinical Utility of In-house metagenomic Next-generation Sequencing for the Diagnosis of Lower Respiratory Tract Infections and Analysis of the Host Immune Response [J]. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America, 2020, 71(Supplement_4): S416-S26.




