宏基因组高通量测序技术在血流感染领域的临床应用与质量控制问题

许四宏,中国食品药品检定研究院研究员,主要从事传染病诊断试剂的质量控制。作为负责人主持完成或改进了五项HIV诊断试剂国家参考品/标准品的研究工作、制定了2项HIV诊断试剂药典标准和5项HIV诊断试剂的行业标准。先后主持国家级课题1项、参与省部级课题4项。获北京市科学技术奖一等奖1项、国家科学技术进步奖二等奖1项。作为副主编编写《艾滋病实验室检测技术与质量保证》,参与编写专著3部。发表论文61篇,其中第一作者或通讯作者论文30篇,国际SCI杂志上发表学术论文13篇。
【摘要】血流感染是临床常见感染之一,尽早发现和鉴别致病性微生物对于尽快启动或调整抗生素治疗方案至关重要。血液样本作为一种易获取、非侵入性样本,其中含有血流感染及局部病灶感染相关的病原微生物及其细胞游离脱氧核糖核酸,是临床感染重要的样本类型之一。相比于传统检测方法,近年来快速发展的宏基因组高通量测序技术具有检测周期短、灵敏度高及病原谱广等优势,在血流感染检测领域正得到越来越广泛的关注和应用。本文就宏基因组高通量测序技术在血流感染领域的临床应用现状及发展进行综述。
【关键字】宏基因组高通量测序技术;微生物细胞游离脱氧核糖核酸;血流感染
血流感染(bloodstream infection,BSI)是重要的临床感染问题,包括感染性心内膜炎、中心静脉导管相关血流感染、原发性菌血症以及由局部感染(如脓肿、骨髓炎、尿路感染或肺炎)引起的血流感染,其高发病率、高致死率及耐药细菌和真菌等因素,已成为急危重症患者的严重威胁之一[1, 2]。重症血流感染发病急、进展快、致病性复杂,能否及时接受适当的抗菌治疗是决定患者预后和死亡率的关键因素[2]。目前,临床感染检测方法仍以基于微生物培养的方法为“金标准”,如镜检、特殊染色及质谱等,但受限于较低的培养阳性率(≤50%)和较长的培养周期(≥48h)[3]。尽管基于聚合酶链反应(polymerase chain reaction,PCR)的分子检测技术具有较好的灵敏度和特异性,但由于血液中的微生物浓度较低(1~10CFU/mL)[4],绝大多数血流感染分子检测产品是以血培养阳性样本为待测样本,同样受上述局限的影响。
近年来,以血浆中的微生物细胞游离脱氧核糖核酸(microbial cell-free deoxyribonucleic acid,mcfDNA)为待测靶标的宏基因组高通量测序(metagenomic next-generation sequencing,mNGS)检测方法,下文简称mNGS-mcfDNA,通过对血流感染样本的cfDNA进行测序,理论上能“无偏倚”地检出全部潜在微生物,逐渐受到临床关注[5]。一般认为,血液中mcfDNA有两个来源,一是血液中死亡的微生物破裂后释放的核酸;二是当局部组织发生感染时,中性粒细胞在炎症部位趋化因子作用下渗出到病变部位,吞噬并杀死微生物,释放的产物通过血浆与组织液的交换进入血液[6, 7]。存在于血液中的mcfDNA的片段长度显著短于人源cfDNA(40~100bp vs. 166bp),GC含量约43.5%[8]。目前,已有多个研究报道了mNGS-mcfDNA检测方法的临床有用性和可行性,并从相较于传统检测方法的经济效益和患者受益等方面进行了分析。由于mNGS-mcfDNA检测方法具有不依赖微生物培养、检测周期较短(约24h)、灵敏度高及病原谱广等特点,能够较好地满足血流感染的临床检测需求,本文综述了该技术的相关临床应用现状及发展。
一、mNGS-mcfDNA检测流程
一般的,mNGS-mcfDNA检测流程包括湿实验和干实验两部分,可分为血浆分离、血浆总cfDNA提取及纯化、测序文库制备、上机测序、生信分析和报告解读等步骤,见图1。
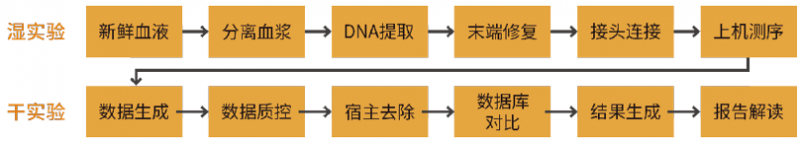
图1. mNGS-mcfDNA检测流程
不同于其他类型的临床样本,血浆样本中的待测物(即mcfDNA)更易降解及受到人源宿主DNA污染。因此,新鲜血液样本前处理的及时性和规范性,对于mNGS-mcfDNA检测的可靠性和稳定性至关重要。新鲜血液应在加入抗凝剂的真空采血管中保存,并及时通过低温离心的方式使血液分层,吸取上层血浆,吸取时应小心操作以防吸入位于“白膜层”的白细胞。Murugesan等人研究发现[9],使用EDTA抗凝管保存血液,室温下应在采血后4h以内完成血浆分离操作,在4℃冷藏的保存条件下不应超过24h。分离得到的血浆应确认未发生溶血后,才能进入下一步实验环节。常用的血浆cfDNA提取方法包括磁珠法和提取柱法。由于mcfDNA的片段长度显著短于人源cfDNA,且浓度可能较低,因此建议针对mcfDNA优化提取及纯化方法,如提取时裂解液中加入核酸酶抑制剂,使用carrier RNA等核酸助沉剂从而提高mcfDNA提取效率[10]。由于血浆cfDNA已经是核酸片段,因此不需额外的片段化处理,提取后的产物可直接用于测序文库构建。需要注意的是,由于血浆cfDNA的浓度一般低于其他临床样本,因此对于mNGS-mcfDNA的湿实验过程,应采取更严格的试验环境污染防控措施,并使用阴性质控品监测实验流程及环境背景菌。
测序下机的原始数据需要经过生物信息学分析(即干实验),才能完成最终的结果报告。干实验的一般过程包括:(1)数据质控,采用数据质控软件(如Trimmomatic[11]、fastp[12]、fastQC[13]等)对原始测序数据进行分析和控制,从而去除低质量序列、接头污染序列、低复杂度序列以及质控后序列长度过短的序列,由于mcfDNA片段长度分布范围较宽(40~100bp),存在部分序列短于50bp,在兼顾序列特异性的前提下,为了实现更高的灵敏度(即获取更多的微生物序列),对于短序列过滤的长度参数可适当的设置短一些,如35bp、40bp等。(2)人源核酸序列去除,对于质控后的高质量序列数据,采用比对软件(如Bowtie[14]、BWA[15]、SNAP[16]等)将其比对至人源参考基因组数据库,去除其中的人源宿主核酸序列后,用于病原微生物的序列分析。(3)病原分类鉴定,将得到非宿主序列与事先建立好的微生物数据库比对后进行序列的分类鉴定。(4)报告解读,由于血浆属于无菌体液样本,核酸浓度较低,因此在检测过程中更易受到检测试剂内源性以及检测环境外源性背景菌的影响。建议应建立并监测相适应的背景菌库,辅助mNGS-mcfDNA的检测结果解读和报告[17]。
二、mNGS-mcfDNA检测的优势
尽管与其他传统检测方法都可以应用于血流感染的辅助诊断,但mNGS-mcfDNA方法在技术原理上与其他检测方法具有显著差异,其检测靶标为血浆中的cfDNA,而基于血培养的检测方法则是以入血的具有活性的病原菌为检测靶标。血培养报告阳性对于血流感染的临床诊断具有非常明确的意义,且绝大多数时候为单个微生物阳性,有助于确定引起感染的病原菌。然而,基于血培养的检测方法也存在对样本中的病原微生物本身的活性依赖较高,且受抗生素使用的影响较大的因素所限。
相对而言,mNGS-mcfDNA检测方法则不依赖微生物培养,不仅具有更短的检测周期和更广的病原菌谱,对于厌氧、苛养菌的检测也更加友好,还具有分子诊断技术的高灵敏度和高特异性。Jing等人报道了一种改进的mNGS-mcfDNA检测方法的临床性能[24],血培养的阳性检出率为27.3%,而经临床小组判定为mNGS方法检出明确或可能感染病原体的阳性率为38.8%,其临床敏感性为87.1%,临床特异性为80.2%,阳性预测值为77.9%,阴性预测值为88.6%。该研究进一步提示了mNGS-mcfDNA检测方法在血流感染诊断领域的可行性及可靠性。
相对于基于血培养的检测方法,mNGS-mcfDNA方法具有更高的阳性检出率。一般地,对于临床常见可培养微生物,mNGS可提升一倍以上的阳性率,从而可能降低不适当的初始抗生素治疗(inappropriate initial antibiotic treatment,IIAT)导致的死亡率。两项血液病中性粒细胞缺乏合并革兰氏阴性杆菌血流感染的研究[25, 26],均表明接受IIAT后7d内病死率可高达29.9%~37.7%,提示精准的抗生素治疗方案对于提升感染者治愈几率和降低死亡率具有重要意义。Wang等人关于脓毒血症诊断的研究表明[27],就细菌而言,血浆游离核酸阳性样本显著高于血培养阳性(优势比[OR]=4.8,p<0.01),对于肺炎克雷伯菌、鲍曼不动杆菌、大肠埃希菌、肠球菌等可培养细菌,血培养和mNGS-mcfDNA的阳性检出比例分别为7:18、5:21、5:10及4:11,mNGS方法具有显著的优势比,阳性检出率提升近2倍。
相对于基于血培养的检测方法,mNGS-mcfDNA方法的检测时效性更高且受抗生素治疗影响更小。Wang等人分析了血培养中常见的8种病原菌的检测周转时间(turn-around time,TAT)[27],mNGS方法平均需要约26h,而不同病原菌的血培养报告阳性结果的平均时间约为9.4~34h,还要加上阳性血培养样本的鉴定报告时间。此外,该研究还发现72.4%的入组患者使用过抗生素,对使用过抗生素的患者,mNGS的阳性检出率显著高于血培养(52.8% vs. 25.0%,p<0.01);而对于未使用抗生素的患者,阳性检出率则无明显差异(43.6% vs. 30.9%,p=0.17)。Miao等人的研究表明[28],抗生素暴露后mNGS阳性检出率显著高于微生物培养(52.5% vs. 34.2%,p<0.1)。Blauwkamp等人的研究也表明[29],对于在检测两周内接受过抗菌治疗的患者样本,mNGS的阳性检出率远高于血培养(47.9% vs. 19.6%)。
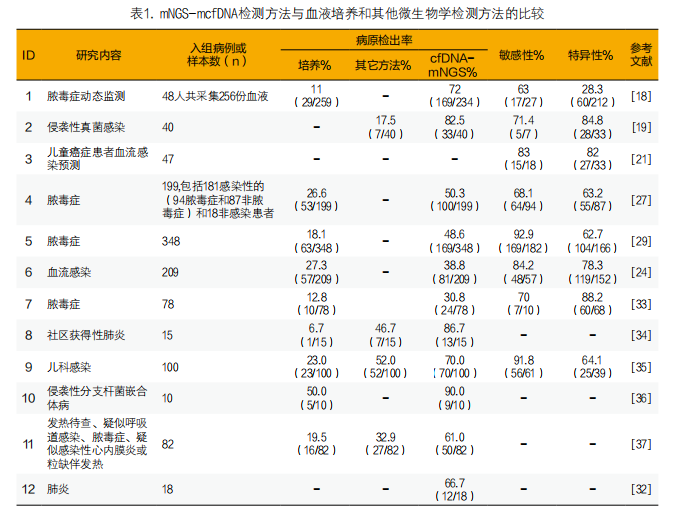
mNGS-mcfDNA方法对于病原体突破皮肤或呼吸道、消化道、泌尿生殖道等黏膜入血造成的不明原因发热和感染[1, 30],能够提供更全面的辅助诊断信息。Jiang等人关于耶氏肺孢子菌的回顾性研究[31]显示,在28例患者的肺泡灌洗液和血浆配对样本中均同时检出了耶氏肺孢子菌,提示血液样本能够检测到肺部感染的可能性。Langelier等人的一项针对25名危重呼吸道疾病患者的研究中,血培养和mNGS-mcfDNA方法均确认了3例菌血症和1例巨细胞病毒血症。对于经呼吸道样本培养确认阳性的病例中,67%(12/18)病例的血浆中同样检出了相关病原体,此外在44%(8/18)病例的血浆中检测出了更多病原体。该研究提示在难以获得下呼吸道样本的情况下,血浆样本对肺炎相关病原体的检测具有潜在的替代价值[32]。表1汇总了近几年来mNGS-mcfDNA检测方法与血液培养和其他微生物学检测方法的比较研究。
三、mNGS-mcfDNA检测的临床应用现状
mNGS-mcfDNA检测方法具有识别全身广泛感染的潜在优势,能够在使用过抗生素治疗的患者检出更多的病原体,对一些常规微生物检测技术难以覆盖的厌氧、苛养微生物的检测也有较大优势。Grumaz等人针对48例脓毒血症患者的256份血浆样本进行连续监测研究[18],发现与检测阳性率为33%的血培养相比,mNGS-mcfDNA检测的阳性率为72%,其中阳性报告中53%的检测结果经过专家组评估对临床治疗方案的调整产生了积极影响。Armstrong等人针对侵袭性真菌病(invasive fungal diseases,IFD)高危患者的研究中[19],研究人员使用mNGS-mcfDNA检测方法对40例儿童肿瘤患者或造血干细胞移植患者的血浆样本进行检测,检出7例真菌阳性,其中3例和非血液样品的微生物培养结果一致,1例和血培养结果一致。
对于血流感染预测及治疗效果监测领域,mNGS-mcfDNA检测可能发挥重要作用。Grumaz S等人使用mNGS技术监测脓毒血症患者的血浆mcfDNA[20],发现患者发生感染性休克(197.23ng/ml)和脓毒血症发病时(377.3ng/ml)血浆mcfDNA的浓度水平是健康人(55.43ng/ml)的3倍和6倍以上。后续的持续监测表明,BSI患者的血浆mcfDNA浓度变化与其病情发展变化情况密切相关,提示可以使用mNGS-mcfDNA的检测结果监测脓毒血症患者的治疗效果。Goggin等人开展了一项包括对47例可能发生血流感染复发的儿童癌症患者进行前瞻性试点队列研究[21],通过mNGS-mcfDNA方法监测患者的血液样本,约75%的患者在感染复发前3天即可通过监测结果得到提示,总体特异性为82%(95%CI,66%~91%)。研究结果提示mNGS-mcfDNA检测具有预测血流感染的潜力,从而可能使患者及时甚至提前获得相应的预防性治疗,从而降低高危免疫缺陷患者感染的发病率和死亡率。
对于干细胞或器官移植患者进行血流感染临床监测,mNGS-mcfDNA检测具有潜在应用价值。Fung等人对3位造血干细胞移植患者进行了持续监测[22],研究人员发现mNGS-mcfDNA检测方法能鉴定罕见病原引起的血流感染,同时相比传统血培养方法能更快检出致病性微生物,如对沙眼衣原体感染的检出时间可以提前19天和30天。同时,在治疗期间,mNGS-mcfDNA检测结果持续报告了引起复发感染的病原体,甚至在患者出现复发感染症状前就报告了样本中存在感染相关病原体,而血培养结果则持续为阴性,提示mNGS-mcfDNA方法受抗生素使用的干扰小于传统血培养,且具有较高的敏感性。Vlaminck等人针对肺移植患者的排异反应和感染症状的监测研究中[23],发现mNGS在血浆mcfDNA中检测到的巨细胞病毒(cytomegalovirus,CMV)和患者的排异反应之间存在相互激活的双向关系,提示mNGS-mcfDNA检测结果可以为预测患者排异反应的严重程度提供更多潜在指标。
四、mNGS-mcfDNA检测的质量控制
对于mNGS-mcfDNA检测流程,湿实验和干实验两部分流程众多、可变因素复杂且均可直接影响检测结果,因此,应进行严格的质量控制。质量控制的关键指标包括但不限于检测范围、准确性、特异性、检出限、精密度、干扰和稳定性等,且可以通过设置相应的标准品或参考品开展质量控制工作[38]。
参考品在设计时,应充分考虑到mNGS技术的特点、mcfDNA片段的核酸特征以及血浆样本的基质特征,制备参考品时才能尽可能模拟真实的临床样本。在选择血浆基质来源时,应通过超深度的mNGS检测对候选基质样本进行测序分析,并绘制其微生物核酸丰度谱。原则上候选基质中不应含有临床常见的病原微生物,同时,内源性和外源性背景菌应在可控范围,且不能影响后续参考品的检测结果。同其他mNGS检测方法一样,血浆样本中人源宿主核酸,即人源cfDNA的浓度直接影响检出限性能,因此参考品设计时需要考虑到人群中血浆人源cfDNA浓度的分布范围。如,Blauwkamp等人对一种mNGS-mcfDNA检测方法的分析性能进行评估研究时[29],将提取后的微生物基因组按40~100bp长度进行打断模拟mcfDNA,将提取后的人源基因组按166bp长度进行打断模拟人源cfDNA,同时分别设置了高、中、低三个水平的人源cfDNA浓度梯度,从而实现评估不同人源宿主核酸背景下该检测方法的检出限性能。
对于mNGS-mcfDNA检测方法的分析性能,可按准确性、特异性、精密性及检出限等关键指标进行质量控制。准确性是指检测结果与被测量真值的一致性,可以以临床“金标准”血培养作为细菌和真菌检测的对比方法,病毒则可以选择实时荧光定量PCR方法或Sanger测序等技术作为对比方法。特异性包括交叉反应和干扰两个方面,交叉反应指核酸序列具有同源性、易引起相同或相似的临床症状的其他微生物,血浆样品还需要考虑采血和检测过程中易引入的污染菌,如表皮葡萄球菌、痤疮丙酸杆菌等;血浆样品的干扰因素一般包括血红蛋白、甘油三酯、胆红素和EDTA等,mNGS方法还应考虑人源核酸浓度或丰度对检出限的影响。精密性是指使用相同的样本在各种特定条件下进行检测后得到测量值间的一致性,目前mNGS检测方法一般为定性检测,因此精密性可考察对临界值附近的样本在不同批次、人员、仪器、日期等不同因素下阴阳性判定结果的一致性和准确性。检出限(Limit of detection,LOD)是指某个病原体在95%置信度下的最低可检出的浓度或滴度(或核酸浓度),通常可以通过重复检测一定浓度梯度的系列稀释样本进行验证。对于单个病原体,mNGS技术对病原体的检测性能取决于病原体的核酸总量在整个样本中的占比,简单来讲,病原体核酸越高、人源核酸越低,检出限性能越好;病原体核酸越低、人源核酸越高,检出限性能越差。
五、展望
本文综述了近几年mNGS-mcfDNA检测技术在血流感染诊断领域的临床应用。尽管越来越多的临床研究表明,mNGS-mcfDNA技术的临床应用,已经能够充分发挥其自身技术特点和优势,并实现较为稳定的分析性能和可靠的临床性能。然而仍需认识到,现阶段mNGS技术本身的复杂性与局限性,特别是生物信息学分析流程的透明性,以及公认权威的质量控制与评价方法及相关质量标准的缺失,必将限制mNGS-mcfDNA技术的进一步推广和发展。因此,需要技术开发者、使用者及监管者等相关方面共同努力,把患者的利益放在首位,在保证科学的前提下稳步推动mNGS技术在血流感染领域发挥越来越重要的作用。
参考文献
刘军. 对血流感染若干问题的思考[J]. 中华医学杂志, 2019, 99(31):4.
宏基因组分析和诊断技术在急危重症感染应用专家共识组. 宏基因组分析和诊断技术在急危重症感染应用的专家共识. 中华急诊医学杂志 2019 , 28(2): 151-155.
O, Opota, A, et al. Blood culture-based diagnosis of bacteraemia: state of the art[J]. Clinical Microbiology & Infection, 2015.
Opota O, Jaton K, Greub G. Microbial diagnosis of bloodstream infection: towards molecular diagnosis directly from blood. Clin Microbiol Infect. 2015 Apr;2 1(4): 323-31. doi: 10.1016/j.cmi.2015.02.005. PMID: 25686695.
Han D, Li R, et al. Liquid biopsy for infectious diseases: a focus on microbial cell-free DNA sequencing. Theranostics. 2020; 10(12): 5501-5513. doi:10.7150/thno.45554.
Minasyan HA. Erythrocyte and leukocyte: two partners in bacteria killing. Int Rev Immunol. 2014 Nov-Dec;33(6):490-7. doi: 10.3109/08830185.2014.956359. PMID: 25259410.
Hayk Minasyan, Erythrocyte: Bacteria Killer and Bacteria Pray, International Journal of Immunology. Special Issue: Antibacterial Cellular and Humoral Immunity. Vol. 3, No. 1-1, 2015, pp. 1-7. doi: 10.11648/j.iji.2015030101.11.
Burnham P, Kim MS, Agbor-Enoh S, Luikart H, Valantine HA, Khush KK, et al. Single-stranded DNA library preparation uncovers the origin and diversity of ultrashort cell-free DNA in plasma. Sci Rep-Uk. 2016; 6.
Murugesan K, Hogan CA, Palmer Z, et al. Investigation of Preanalytical Variables Impacting Pathogen Cell-Free DNA in Blood and Urine. J Clin Microbiol. 2019 Oct 23;57(11):e00782-19. doi: 10.1128/JCM.00782-19. PMID: 31511335.
Sorber L, Zwaenepoel K, Deschoolmeester V, et al. A Comparison of Cell-Free DNA Isolation Kits: Isolation and Quantification of Cell-Free DNA in Plasma. J Mol Diagn. 2017 Jan;19(1):162-168. doi: 10.1016/j.jmoldx.2016.09.009. PMID: 27865784.
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014 Aug 1;30(15):2114-20. doi: 10.1093/bioinformatics/btu170. PMID: 24695404.
Hen S, Zhou Y, Chen Y, Gu J. fastp: an ultra-fast all-in-one FASTQ preprocessor. Bioinformatics. 2018 Sep 1;34(17):i884-i890. doi: 10.1093/bioinformatics/bty560. PMID: 30423086.
http://www.bioinformatics.babraham.ac. uk/projects/fastqc/
Langmead B, Trapnell C, Pop M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 2009;10(3):R25. doi: 10.1186/gb-2009-10-3-r25. PMID: 19261174.
Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 2009 Jul 15;25(14):1754-60. doi: 10.1093/bioinformatics/btp324. Epub 2009 May 18. PMID: 19451168.
Zaharia M, Bolosky WJ, Curtis K, et al. Faster and more accurate sequence alignment with SNAP[J/OL]. arXiv preprint arXiv: 1111.5572 (2011). [2021-03-01]. http:// citeseerx. ist. psu. edu/viewdoc/citations; jsessionid= 8A17973B6D27511A76971A667A444200? doi= 10.1.1.412.4273.
中华医学会检验医学分会临床微生物学组, 中华医学会微生物学与免疫学分会临床微生物学组, 中国医疗保健国际交流促进会临床微生物与感染分会. 宏基因组高通量测序技术应用于感染性疾病病原检测中国专家共识[J]. 中华检验医学杂志, 2021, 44(02): 107-120.
Grumaz S, Grumaz C, Vainshtein Y, et al. Enhanced Performance of Next-Generation Sequencing Diagnostics Compared With Standard of Care Microbiological Diagnostics in Patients Suffering From Septic Shock. Crit Care Med. 2019 May;47(5):e394-e402. doi:10.1097/CCM.0000000000003658. PMID: 30720537.
Armstrong AE, Rossoff J, Hollemon D, et al. Cell-free DNA next-generation sequencing successfully detects infectious pathogens in pediatric oncology and hematopoietic stem cell transplant patients at risk for invasive fungal disease. Pediatr Blood Cancer. 2019 Jul;66(7):e27734. doi: 10.1002/pbc.27734. PMID: 30941906.
Grumaz S, Stevens P, Grumaz C, Decker SO, Weigand MA, Hofer S, et al. Next-generation sequencing diagnostics of bacteremia in septic patients. Genome Med. 2016; 8: 73.40.
Goggin KP, Gonzalez-Pena V, Inaba Y, et al. evaluation of Plasma Microbial Cell-Free DNA Sequencing to Predict Bloodstream Infection in Pediatric Patients With Relapsed or Refractory Cancer. JAMA Oncol. 2020 Apr 1;6(4):552-556. doi: 10.1001/jamaoncol.2019.4120. PMID: 31855231.
Fung M, Zompi S, Seng H, et al. Plasma Cell-Free DNA Next-Generation Sequencing to Diagnose and Monitor Infections in Allogeneic Hematopoietic Stem Cell Transplant Patients. Open Forum Infect Dis. 2018 Nov 16;5(12):ofy301. doi: 10.1093/ofid/ofy301. PMID: 30581881.
De Vlaminck I, Martin L, Kertesz M, et al. Noninvasive monitoring of infection and rejection after lung transplantation. Proc Natl Acad Sci U S A. 2015 Oct 27;112(43):13336-41. doi: 10.1073/pnas.1517494112. PMID: 26460048.
Jing C, Chen H, Liang Y, et al. Clinical evaluation of an Improved metagenomic Next-Generation Sequencing Test for the Diagnosis of Bloodstream Infections. Clin Chem. 2021 Aug 5;67(8):1133-1143. doi: 10.1093/clinchem/hvab061. PMID: 34060627.
Tang Y, Wu X, Cheng Q, Li X. Inappropriate initial antimicrobial therapy for hematological malignancies patients with Gram-negative bloodstream infections. Infection. 2020 Feb;48(1):109-116. doi: 10.1007/s15010-019-01370-x. PMID: 31677085.
Zhang Y, Zheng Y, Dong F, et al. Epidemiology of Febrile Neutropenia Episodes with Gram-Negative Bacteria Infection in Patients Who Have Undergone Chemotherapy for Hematologic Malignancies: A Retrospective Study of 10 Years' Data from a Single Center. Infect Drug Resist. 2020 Mar 26;13:903-910. doi: 10.2147/IDR.S241263. PMID: 32273735; PMCID: PMC7105367.
Wang L, Guo W, Shen H, et al. Plasma Microbial Cell-Free DNA Sequencing Technology for the Diagnosis of Sepsis in the ICU. Front Mol Biosci. 2021 May 28; 8: 659390. doi:
Miao Q, Ma Y, Wang Q, et al. Microbiological Diagnostic Performance of metagenomic Next-generation Sequencing When Applied to Clinical Practice. Clin Infect Dis. 2018 Nov 13; 67(suppl_2):S231-S240. doi: 10.1093/cid/ciy693. PMID: 30423048.
Blauwkamp TA, Thair S, Rosen MJ, et al. Analytical and clinical validation of a microbial cell-free DNA sequencing test for infectious disease. Nat Microbiol. 2019 Apr;4(4):663-674. doi: 10.1038/s41564-018-0349-6. PMID: 30742071.
中国中性粒细胞缺乏伴发热患者抗菌药物临床应用指南 (2020年版). 中华血液学杂志, 2020, 41(12): 969-978.
Jiang J, Bai L, Yang W, et al. metagenomic Next-Generation Sequencing for the Diagnosis of Pneumocystis jirovecii Pneumonia in Non-HIV-Infected Patients: A Retrospective Study. Infect Dis Ther. 2021 Sep;10(3):1733-1745. doi: 10.1007/s40121-021-00482-y. PMID: 34244957.
Langelier C, Fung M, Caldera S, Deiss T, Lyden A, Prince BC, et al. Detection of pneumonia pathogens from plasma cell-free DNA. Am J Resp Crit Care. 2020; 201: 491-5.
Long Y, Zhang Y, Gong Y, Sun R, Su L, Lin X, et al. Diagnosis of sepsis with cell-free DNA by next-generation sequencing technology in ICU patients. Arch Med Res. 2016; 47: 365-71.
Farnaes L, Wilke J, Ryan Loker K, Bradley JS, Cannavino CR, Hong DK, et al. Community-acquired pneumonia in children: cell-free plasma sequencing for diagnosis and management. Diagn Micr Infec Dis. 2019; 94: 188-91.
Rossoff J, Chaudhury S, Soneji M, Patel SJ, Kwon S, Armstrong A, et al. Non-invasive diagnosis of infection using plasma next-generation sequencing: a single center experience. Open Forum Infect Di. 2019.
Nomura J, Rieg G, Bluestone G, Tsai T, Lai A, Terashita D, et al. Rapid detection of invasive Mycobacterium chimaera disease via a novel plasma-based next-generation sequencing test. BMC Infect Dis. 2019; 19.
Hogan CA, Yang S, Garner OB, Green DA, Gomez CA, Dien Bard J, et al. Clinical impact of metagenomic next-generation sequencing of plasma cell-free DNA for the diagnosis of infectious diseases: a multicenter retrospective cohort study. Clin Infect Dis. 2020.
刘东来, 张春涛, 王佑春, 等. 病原宏基因组高通量测序技术质量控制与评价的挑战和思考[J]. 生物工程学报, 2020, 36(12): 2598-2609.




