数字PCR在血流感染病原学诊断中的应用

余皓,杭州市领军人才,美国康涅狄格大学博士,中国科学院遗传与发育生物学研究所博士后。专注于功能基因组学的研究,先后发表SCI论文10多篇,获得2项发明专利。2018年加入领航基因科技(杭州)有限公司,负责液滴式数字PCR血流感染诊断试剂的开发工作。
血流感染是细菌、真菌等病原微生物入侵血液所致的一种感染性疾病,部分可发展为脓毒症,是ICU危重疾病之一[1-3]。全世界每年有4700至5000万例脓毒症患者,至少有1100万例死亡,占每年全球死亡人数的19.7%,是住院患者死亡的主要原因之一[4-6]。近年来,血流感染和脓毒症的发生率迅速上升[7, 8],医疗成本居高不下,2019年美国在脓毒症相关的医疗花费超过620亿美元[6]。早期诊断和针对性治疗可极大降低血流感染和脓毒症患者的死亡率[10-12]。据估算,血流感染每延迟治疗1个小时,患者死亡率增加7.8%[10];延迟治疗6小时,死亡率达到58%[11]。因而,建立快速准确的血流感染病原学诊断技术刻不容缓。
一、血流感染病原学诊断技术的现状
血培养是血流感染诊断的金标准,也是当前临床最常见的检测方法。常见细菌的血培养及鉴定一般需要2-3天,分枝杆菌和真菌等病原的检测周期更长。血培养阳性率受采血量、采集时间、采集方法及操作人员技术水平等多因素影响。因而,血培养技术灵敏度相对较低、检测周期长,制约临床诊疗效果。
除了常规血培养以外,还可以直接对病原体的核酸进行分子检测,以提高检测敏感性和缩减报告时间。目前,对病原菌核酸进行分子检测的方法大致分为两类(表1):(1)对阳性血培养物进行检测,(2)直接对血液中的病原菌核酸进行检测。
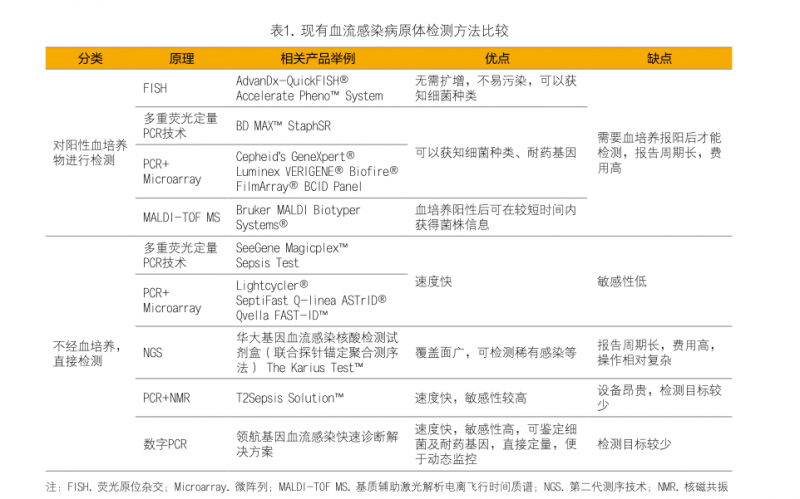
对阳性血培养物进行检测主要包括基于FISH、PCR和qPCR扩增、微阵列、MALDI-TOF MS等技术的检测方法。该类产品需要在血培养阳性后才能进行检测,虽然检测的敏感性和特异性较高,但总体报告时间长的问题仍未得到解决。尽管如此,与血培养报阳后传统的鉴定方法相比,此类方法已经表现出不少明显的优势。目前临床上常用的MALDI-TOF MS可在1小时内完成阳性血培养物的微生物鉴定。FilmArray® BCID产品可以将血培养之后传统的微生物及药敏鉴定时间由平均的57小时缩短到17小时,再结合用药管理,可以有效降低抗生素的使用[13];此外,快速的微生物鉴定还能够缩短病人的住院时间,将死亡率由15%降低至6%,并且能够使每百个病人的花费降低大约30000美元[14]。
直接对全血进行病原菌检测可以极大的缩短时间,并提高病原检出率。该类方法包括基于多重荧光定量PCR、PCR结合微阵列技术、NGS的检测方法等。
基于多重荧光定量PCR或PCR结合微阵列技术的产品可以减少致病菌鉴定时间,但血流感染患者的血液载菌量通常<10CFU/mL,回收富集难度较大,现有产品的敏感性都较低,如罗氏Lightcycler® SeptiFast敏感性只有50%[15],韩国SeeGene公司的Magicplex™敏感性仅为29%[16],远不能满足临床需求。
NGS作为新兴技术可以获得丰富的致病菌信息,但是费用高、周期长、需要专业生信团队,尤其是报告时间1-2天,不能真正反应患者当时的感染状况,因而在血流感染检测方面并无明显优势,而在少见感染、疑难杂症诊断方面可能更具潜力。
美国T2 Biosystems公司的T2Sepsis Solution™,可以利用PCR结合核磁共振(NMR)技术直接检测全血中的致病菌,耗时3-5小时,且表现出较高的临床敏感性[17]。并且T2Dx®仪器、T2Bacteria®和T2Candida®试剂现已获FDA批准在美国上市。但其检测菌种目前还相对较少,设备也较昂贵。
二、数字PCR技术
数字PCR(dPCR)技术是继普通PCR和qPCR之后的第三代核酸扩增与检测技术。其原理是将含有核酸分子的反应体系制成成千上万个纳升级的微反应单元,其中每个反应单元不含待检核酸靶分子,或者含有一个至数个待检核酸靶分子,且每个反应单元都作为一个独立的PCR反应器,经PCR扩增后对各个反应单元的荧光信号进行检测。有荧光信号的反应单元判读为1;没有荧光信号的反应单元判读为0,因此该技术被称为数字PCR。最终根据泊松分布原理以及阳性反应单元的比例,可以计算给出待检靶分子的浓度或拷贝数(图1)。数字PCR具有单分子扩增、不依赖标准曲线、直接绝对定量、对背景核酸和抑制剂耐受性高的优点,非常适合对qPCR等常规检测方法解决不了的极低含量目标片段进行定量或定性检测。
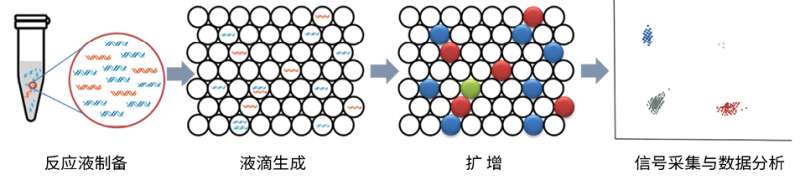
图1. 微滴式数字PCR原理和流程
三、数字PCR在血流感染病原学诊断中的应用
数字PCR比传统qPCR更加出色的灵敏度和特异性、精确性使其迅速得到广泛的应用,在复杂背景下稀有突变检测和表达量微小差异鉴定方面表现出的优势已被普遍认可,近几年应用于血流感染病原菌鉴定的相关报道也逐渐多起来,与其它方法相比也表现出一定的优势(表1)。
不少研究者将数字PCR应用于儿童血流感染的研究。金黄色葡萄球菌(Staphylococcus aureus)是儿童血流感染的重要细菌之一。陈卫华等[18]比较了数字PCR法和血培养法在检测儿童耐甲氧西林金黄色葡萄球菌血流感染中的应用效果。以血培养为金标准,数字PCR的敏感性为91.40%,特异性为94.15%,且数字PCR法平均检测时间为2.5小时,远少于血培养法的48小时。侵袭性真菌感染(invasive fungal infection,IFI)是新生儿重症监护室(neonatal intensive care unit,NICU)院内感染的重要原因。李辉桃等[19]采用血培养及微滴式数字PCR(ddPCR)对83例有IFI高危因素和/或临床症状的新生儿的研究表明,ddPCR的敏感性为86%,特异性为97%,且仅需要0.4mL血量。数字PCR法表现出了快捷、灵敏度高、特异性好的特点。
在基于核酸的病原菌检测方法中,16S rRNA和28S rRNA被广泛用于细菌和真菌的检测。Ziegler[20]等开发了基于16S rRNA引物探针的ddPCR血流感染检测方法,在83个血流感染疑似病人中的总体阳性率达到60%。在Wolters等[21]的研究中,通过16S rRNA检测细菌和28S rRNA检测真菌的最低检测限达到1-2个细菌或真菌每ddPCR反应。该ddPCR方法总体的灵敏性达到80%,特异性达到87%,整个检测可以在4小时内完成。
虽然通过检测16S rRNA和28S rRNA的ddPCR方法能够快速鉴定有无细菌或真菌造成的血流感染,但如果能够明确知道是哪种病原菌引起的感染以及携带何种耐药基因,对于临床针对性的使用抗生素治疗能够提供更精准的信息。Hu等人[22]使用多重ddPCR,分成6个检测Panel,能够检测临床上常见的16种细菌、4种真菌和4个耐药基因。他们比较了数字PCR、mNGS及血培养在ICU疑似血流感染患者中的应用,结果表明,mNGS能够检测更多种类的病原微生物,但对于ddPCR检测Panel所覆盖的病原菌,ddPCR比mNGS有更高的灵敏性,并且ddPCR的检测可在4小时内完成,相对于mNGS的2天有明显优势。
文献报道显示出了数字PCR技术用于血流感染病原学诊断具有较高的灵敏度和特异性,由于其不需要经过培养,在抽血之后几个小时之内完成检测,所以能够为早期诊断和针对性治疗提供有效帮助。结合其绝对定量的特点,有希望对患者在治疗过程的感染状况进行动态监控,从而可以及时调整抗生素的使用,确定治疗终点。随着数字PCR仪器设备自动化程度的提高以及检测成本的下降,数字PCR有希望在血流感染病原学的快速灵敏诊断中发挥重要作用。
参考文献
Bearman GM, Wenzel RP. Bacteremias: a leading cause of death. Arch Med Res. 2005, 36(6): 646-59.
Beekmann SE, Diekema DJ, Chapin KC, Doern GV. Effects of rapid detection of bloodstream infections on length of hospitalization and hospital charges. J Clin Microbiol. 2003, 41(7): 3119-25.
Marchetti O, Calandra T. Infections in neutropenic cancer patients. Lancet. 2002, 359(9308): 723-725.
Rudd KE, Johnson SC, Agesa KM, Shackelford KA, Tsoi D, Kievlan DR, Colombara DV, Ikuta KS, Kissoon N, Finfer S, Fleischmann-Struzek C, Machado FR, Reinhart KK, Rowan K, Seymour CW, Watson RS, West TE, Marinho F, Hay SI, Lozano R, Lopez AD, Angus DC, Murray CJL, Naghavi M. Global, regional, and national sepsis incidence and mortality, 1990-2017: analysis for the Global Burden of Disease Study. Lancet 2020, 395(10219): 200-211.
WSD fact sheet 2021. https://www.worldsepsisday.org/sepsisfacts (Access date: October 25, 2021).
Buchman TG, Simpson SQ, Sciarretta KL, Finne KP, Sowers N, Collier M, Chavan S, Oke I, Pennini ME, Santhosh A, Wax M, Woodbury R, Chu S, Merkeley TG, Disbrow GL, Bright RA, MaCurdy TE, Kelman JA. Sepsis among medicare beneficiaries: 3. The Methods, Models, and Forecasts of Sepsis, 2012-2018. Crit Care Med. 2020, 48(3): 302-318.
Rhodes A, Evans LE, Alhazzani W, Levy MM, Antonelli M, Ferrer R, Kumar A, Sevransky JE, Sprung CL, Nunnally ME, Rochwerg B, Rubenfeld GD, Angus DC, Annane D, Beale RJ, Bellinghan GJ, Bernard GR, Chiche JD, Coopersmith C, De Backer DP, French CJ, Fujishima S, Gerlach H, Hidalgo JL, Hollenberg SM, Jones AE, Karnad DR, Kleinpell RM, Koh Y, Lisboa TC, Machado FR, Marini JJ, Marshall JC, Mazuski JE, McIntyre LA, McLean AS, Mehta S, Moreno RP, Myburgh J, Navalesi P, Nishida O, Osborn TM, Perner A, Plunkett CM, Ranieri M, Schorr CA, Seckel MA, Seymour CW, Shieh L, Shukri KA, Simpson SQ, Singer M, Thompson BT, Townsend SR, Van der Poll T, Vincent JL, Wiersinga WJ, Zimmerman JL, Dellinger RP. Surviving Sepsis Campaign: International Guidelines for Management of Sepsis and Septic Shock: 2016. Intensive Care Med. 2017, 43(3):304-377.
Hall MJ, Williams SN, DeFrances C, Golosinskiy A. Inpatient care for septicemia or sepsis: a challenge for patients and hospitals. NCHS Data Brief. 2011, 62:1-8.
Lueangarun S, Leelarasamee A. Impact of inappropriate empiric antimicrobial therapy on mortality of septic patients with bacteremia: a retrospective study. Interdiscip Perspect Infect Dis. 2012:765205
Kumar A, Roberts D, Wood KE, Light B, Parrillo JE, Sharma S, Suppes R, Feinstein D, Zanotti S, Taiberg L, Gurka D, Kumar A, Cheang M. Duration of hypotension before initiation of effective antimicrobial therapy is the critical determinant of survival in human septic shock. Crit Care Med. 2006, 34(6): 1589-1596.
Kumar A. Antimicrobial delay and outcome in severe sepsis. Crit Care Med. 2014, 42(12):e802.
Gaieski DF, Mikkelsen ME, Band RA, Pines JM, Massone R, Furia FF, Shofer FS, Goyal M. Impact of time to antibiotics on survival in patients with severe sepsis or septic shock in whom early goal-directed therapy was initiated in the emergency department. Crit Care Med. 2010, 38(4):1045-1053.
MacVane SH, Nolte FS. Benefits of adding a rapid PCR-based blood culture identification panel to an established antimicrobial stewardship program. J Clin Microbiol. 2016, 54(10): 2455-63.
Pardo J, Klinker KP, Borgert SJ, Butler BM, Giglio PG, Rand KH. Clinical and economic impact of antimicrobial stewardship interventions with the FilmArray blood culture identification panel. Diagn Microbiol Infect Dis. 2016, 84(2): 159-164
Warhurst G, Dunn G, Chadwick P, Blackwood B, McAuley D, Perkins GD, McMullan R, Gates S, Bentley A, Young D, Carlson GL, Dark P. Rapid detection of healthcare-associated bloodstream infection in critical care using multipathogen real-time polymerase chain reaction technology: a diagnostic accuracy study and systematic review. Health Technol Assess. 2015, 19(35): 1-142.
Zboromyrska Y, Cillóniz C, Cobos-Trigueros N, Almela M, Hurtado JC, Vergara A, Mata C, Soriano A, Mensa J, Marco F, Vila J. evaluation of the Magicplex™ Sepsis Real-Time Test for the Rapid Diagnosis of Bloodstream Infections in Adults. Front. Cell. Infect. Microbiol. 2019, 9(56).
Clancy CJ, Pappas PG, Vazquez J, Judson MA, Kontoyiannis DP, Thompson GR 3rd, Garey KW, Reboli A, Greenberg RN, Apewokin S, Lyon GM 3rd, Ostrosky-Zeichner L, Wu AHB, Tobin E, Nguyen MH, Caliendo AM. Detecting Infections Rapidly and Easily for Candidemia Trial, Part 2 (DIRECT2): A Prospective, Multicenter Study of the T2Candida Panel. Clin. Infect. Dis. 2018, 66(11): 1678-1686
徐卫华, 田克印, 李小双. 数字PCR和血培养检测儿童MRSA血流感染应用价值比较[J]. 中山大学学报(医学科学版). 2020, 41(6): 930-936.
李辉桃, 林冰纯, 黄智峰, 杨传忠, 黄为民. 应用微滴式数字PCR技术快速诊断新生儿侵袭性真菌病. 中国当代儿科杂志 2019, 21(1): 45-51.
Ziegler I, Lindström S, Källgren M, Strålin K, Mölling P. 16S rDNA droplet digital PCR for monitoring bacterial DNAemia in bloodstream infections. PLoS One. 2019, 14(11): e0224656.
Wouters Y, Dalloyaux D, Christenhusz A, Roelofs HMJ, Wertheim HF, Bleeker-Rovers CP, Te Morsche RH, Wanten GJA. Droplet digital polymerase chain reaction for rapid broad-spectrum detection of bloodstream infections. Microb Biotechnol. 2020, 13(3): 657-668.
Hu B, Tao Y, Shao Z, Zheng Y, Zhang R, Yang X, Liu J, Li X, Sun R. A Comparison of Blood Pathogen Detection Among Droplet Digital PCR, metagenomic Next-Generation Sequencing, and Blood Culture in Critically Ill Patients With Suspected Bloodstream Infections. Front. Microbiol. 2021, 12(1056).




