TorrentMLPA新技术在临床拷贝数变异检测中的应用

李晓天,硕士,毕业于黑龙江大学。现任北京安智因生物技术有限公司分子诊断高级产品经理。
拷贝数变异(Copy Number Variants,CNVs)指基因组DNA中大小为几十个碱基(>50bp)到几Mb的DNA拷贝数变异的现象,包括基因组重复、缺失、倒置和异位等,其中重复和缺失是最常见的[1]。CNVs产生于不同的突变机制,包括DNA重组,复制和修复相关过程[2]。其形成的确切机制尚不清楚,目前主要由非等位同源性重组(non-allelic homologous recombination,NAHR)、非同源末端连接机制(non-homologous end joining,NHEJ)、复制叉停滞(replication fork stagnation)、模块转换(fork stalling and template switchng,FoSTeS)和DNA移动原件的活动(DNA mobile component activity)等5种形式引起的变异[3-5]。CNVs被确认是广泛存在于人类基因组DNA的重要变异形式,通过扰乱基因活性和改变基因剂量来影响基因表达、表型差异和表型适应[6-7],从而影响个体表型,在人类发病机制中发挥关键作用。目前已经发现致病性的CNV可引起孟德尔遗传的基因组疾病或与复杂的多基因疾病有关,包括癌症、心血管、自身免疫、神经退行性疾病等[8]。因此临床上准确检测出患者可能存在的CNV变异,对于患者疾病风险、治疗方案和用药指导等有着重要的意义。
常见的检测CNV的方法有荧光原位杂交技术(Fluorescence in situ hybridization,FISH)及其衍生技术,定量聚合酶链式反应及其衍生技术,高通量芯片技术以及高通量测序技术(Next Generation Sequencing,NGS)[9]。每种方法都有各自的优缺点,FISH是检测细胞核基因组CNV的传统方法,但是受限于荧光探针价格昂贵,检测位点有限,过程繁琐。NGS是遗传病检测领域的一项革新技术,靶向测序和全外显子组测序(whole exome sequencing,WES)得到广泛认可[10], 然而测序技术主要依赖片段读取,因此通过信息学方法从NGS中检测CNV结果会不尽人如意。三代测序共同的特点,不依赖PCR扩增构建文库,最大还原基因组DNA本来的面貌,长读长测序方法对于CNV检测准确,但是鉴于昂贵的价格还没有大量投入临床,还处于科研阶段。多重连接探针扩增技术(multiplex ligation-dependent probe amplification,MLPA),该技术高效、特异、快速和简便,是目前CNV检测的金标准,许多临床基因检测例如BRCA1/2数据解读中国专家共识(2021版)[11],遗传性胸主动脉瘤/夹层基因检测及临床诊疗专家共识[12]等指南中推荐查找基因大片段插入/缺失的方法。
鉴于以上各方法学的优缺点和CNV在临床检测中的重要意义,我们开发了一种新的方法TorrentMLPA用于检测CNV变异情况。
一、TorrentMLPA技术概况
1. 技术背景:CNV检测方法众多,各有优缺点,MLPA技术是众多专家共识指南中推荐的CNV检测方法,但是鉴于其一次只能检测45个位点,检测通量上还是受到限制。半导体测序技术[13]是基于核酸合成过程中释放出的氢离子(H+)的探测,通过检测这些离子的变化来确定DNA序列,无需进行基于光的碱基检测,因此不需要昂贵的摄影机和仪器,耗时短、测序成本低使其成为个人和小型实验室理想的选择。本方法通过试剂和流程优化将两种方法的优点结合,开发了TorrentMLPA检测方法,可高通量检测目的基因的拷贝数情况。
2. 技术原理:传统MLPA是根据不同长度的探针填充序列进行毛细管电泳片段分析,进而区分不同目标位置,毛细管电泳基线漂移和泵输液不稳定会导致数据处理相对复杂。TorrentMLPA探针长度一致,根据探针序列的特异性比对到不同目标区域,同时探针上携带通用引物序列,相同的引物和相似的扩增片段长度极大提高了连接探针的扩增效率使结果更加准确和稳定。不仅如此,传统MLPA一次至多检测45个位点,如果检测多个基因需要多个panel,耗时且成本高,而TorrentMLAP采用半导体测序的方法,一个反应可以检测几百到几千个位点,设计和使用更加灵活。将MPLA和IonTorrentNGS两种技术平台高效结合,使得CNV检测基因数目更多,检测成本更低,检测周期更短。
二、临床TorrentMLPA检测结果
1. 方法学流程:(1)基因组提取:使用满足条件的提取试剂盒进行基因组提取。例如安智因核酸提取试剂,京大械备2019011号。(2)模板变性与探针杂交:样本基因组DNA 98℃变性,加入杂交探针混合液60℃过夜。(3)杂交探针链连接与连接探针扩增:杂交过夜16-20h后加入ligase-65连接酶混合液,该酶特异性连接与模板匹配的两个紧挨着的杂交探针。待连接反应结束,加入扩增试剂混合液进行放大扩增。(4)半导体测序接头连接:取连接探针扩增产物与半导体测序接头试剂混合,连接测序接头。(5)测序与数据分析:根据样本个数和测序芯片规格稀释混合样本进行测序。下机数据使用安智因自主开发的TorrentMLPA数据分析软件进行数据分析和结果判读。整体实验流程见图1.TorrentMLPA方法学流程所示。
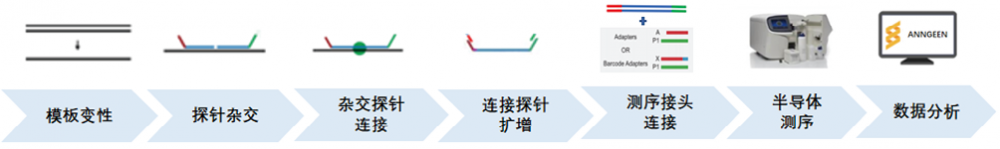
图1. TorrentMLPA方法学流程
2. 结果:选择BRCA1/2基因和FBN1基因已知CNV位点缺失的临床样本,使用TorrentMLPA和MLPA方法平行测试。
(1)BRCA1上游和exon1~2 del检测结果:图2为TorrentMLPA BRCA1上游和exon1~2 del结果,图3为MLPA BRCA1上游和exon1~2 del结果。可以看出两种方法检出结果均一致。
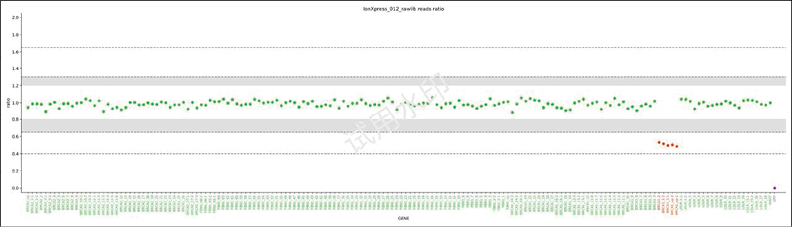
图2. TorrentMLPA BRCA1上游和exon1~2 del结果

图3. MLPA BRCA1上游和exon1~2 del结果
(2)BRCA2标准品exon21~27 del检测结果:图4为TorrentMLPA BRCA2标准品上游和exon21~27 del结果,此样本为BRCA2 del标准品,与已知基因型一致。
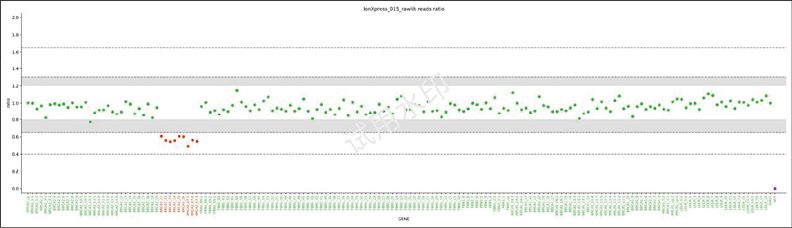
图4. TorrentMLPA BRCA2标准品上游和exon21~27 del结果
(3)FBN1 exon3~4 del检测结果:图5为TorrentMLPA FBN1 exon3~4 del结果,与已知基因型一致。

图5. 为TorrentMLPA FBN1 exon3~4 del结果
(4)DNA投入测试:考虑到本方法的技术原理为先捕获杂交再扩增放大,传统捕获的方法DNA投入起始量300-500ng/单样本,为了评估TorrentMLPA方法的最低DNA投入量,选取临床已知BRCA1上游和exon1~2 del样本进行20ng、30ng、50ng和100ng测试,结果如图6不同DNA投入量测试结果比较所示,20ng投入可以获得准确检测结果,但是位点分散较严重,随着DNA投入量增多,结果的散点会越来越集中,考虑到结果的准确性和稳定性,日常检测至少需要100ng左右的DNA投入。

图6. 不同DNA投入量测试结果比较
三、TorrentMLPA检测技术的应用前景
基因组拷贝数变异(copy number variants,CNVs)是人类遗传病的重要致病原因,也是多种疾病如产前胎儿超声异常、多种先天性畸形和发育障碍必检项目[14]。人类基因组中存在大量的CNVs,其遗传效应远大于SNPs,准确的检测和研究CNV有助于对基因组变异与疾病间的关系的深入理解。尽管目前检测CNV的方法有很多,都各有优缺点。TorrentMLPA是集低成本高效率的半导体测序技术与检测CNV金标准MLPA技术的高效结合,同时在通量与检测准确度上弥补了两个平台的短板。随着社会的进步,基因检测技术的普及,临床对于CNV的检测需求越来越大,要求也越来越高。TorrentMLPA一次可检测众多基因,成本低,准确率高,报告周期短,相信在未来的临床检测CNV的应用会更多。
参考文献
Hu L, Yao X, Huang H, et al. Clinical significance of germline copy number variation in susceptibility of human diseases[J]. J Gen Genomics, 2018, 45(1): 3-12. DOI: 10.1016/j.jgg.2018.01.001
Pös O, Radvanszky J, Buglyó G, et al. Copy number variation: Characteristics, evolutionary and pathological aspects[J]. Biomed J. doi:10.1016/j.bj.2021.02.003
Arlt M F, Wilson T E, Glover T W. Replication stress and mechanisms of CNV formation [J]. Curr Opin Genet Dev, 2012, 22(3): 204-10.
张慧莲, 陈鲤群. CNV及其与癌症间的关系[J] 中国细胞生物学学报, 2020, 42(6): 1082-1090. Doi:10.11844/cjcb.2020.06.0016
Lee J A, Carvalho CMB, Lupski JR. (2007). A DNA Replication Mechanism for Generating Nonrecurrent Rearrangements Associated with Genomic Disorders[J]. Cell, 131(7), 1235–1247. doi:10.1016/j.cell.2007.11.037
王秋权, 黄莎莎, 袁永一, 等. 基因拷贝数变异在遗传性耳聋研究中的进展[J]. 中华耳科学杂志, 2021, 19(2).
夏一芳, 潘继红, 常晓天. DNA拷贝数变化与肿瘤[J]. 国际肿瘤学杂志, 2012, 39(8): 563-566.
Rao J, Peng L, Liang X,et al. Performance of copy number variants detection based on whole-genome sequencing by DNBSEQ platforms[J]. BMC Bioinformatics, 2020, 21(1). doi:10.1186/s12859-020-03859-x
韦小妮, 严提珍, 韦德宁. 拷贝数变异的检测技术及其与人类疾病关系的研究进展[J]. 中国优生与遗传杂志, 2018, 26(5).
国家心血管病专家委员会血管外科专业委员会. 遗传性胸主动脉瘤/夹层基因检测及临床诊疗专家共识[J]. 中国循环杂志, 2019, 34(4): 319-325. DOI: 10.3969/j.issn.1000-3614.2019.04.003
中华医学会病理学分会,国家病理质控中心. BRCA1/2数据解读中国专家共识(2021版)[J]. 中华病理学杂志, 2021, 50(6): 565-571. DOI: 10.3760/cma.j.cn112151-20201027-00809
国家心血管病专家委员会血管外科专业委员会. 遗传性胸主动脉瘤/夹层基因检测及临床诊疗专家共识[J]. 中国循环杂志, 2019, 34(4): 319-325. DOI:10.3969/j.issn.1000-3614.2019.04.003
中国医师协会医学遗传医师分会, 中华医学会儿科学分会内分泌遗传代谢学组, 中国医师协会青春期医学专业委员会临床遗传学组,上海市医学会分子诊断专科分会. 全基因组测序在遗传病检测中的临床应用专家共识[J]. 中华儿科杂志, 2019, 57(6): 419-423.
林司杭, 周文斌. BRCA基因检测进展[J]. 中国医药科学, 2023,13(2).
陈晓丽, 上官少方, 谢华, 刘浩然, 刘维强, 安宇,沈亦平.结构性拷贝数增加的解读标准: 来自美国医学遗传学与基因组学学会(ACMG)和临床基因组资源中心(ClinGen)的建议[J]. 中华医学遗传学杂志, 2022, 39(1):1-10.




