单分子实时测序和纳米孔测序对血浆中细胞游离DNA大小与末端基序及组织来源的比较研究
【摘要】目的 最近使用单分子实时(SMRT)测序的研究显示,血浆中存在大量可分析的长细胞游离DNA(cfDNA),这种长cfDNA在妊娠和癌症方面的潜在临床应用已经得到证实,然而,不同的长读测序平台对长cfDNA的分析性能仍然未知。方法 使用不同大小超声处理的人和小鼠DNA的人工混合物评估了PacBio的SMRT测序和ONT的纳米孔测序的长度偏好,使用两个平台对不同妊娠期孕妇、乙肝携带者和肝癌患者的血浆cfDNA进行测序。结果 两个平台均显示出对更长DNA片段(1500bp vs 200bp)的测序偏好,PacBio显示出更强大的偏好(长片段5倍过表达vs ONT中2倍),PacBio中500bp cfDNA片段的百分比比ONT高约6倍,PacBio和ONT的cfDNA末端基序谱相似,但表现出平台依赖性,两个平台在单分子甲基化模式的组织来源分析上性能相当。结论 与纳米孔测序相比,SMRT测序产生的数据具有更高百分比的长cfDNA。然而,由于纳米孔测序的通量高得多,因此可以获得更多适合组织来源分析的长cfDNA片段。在分析cfDNA的大小和末端基序时,应了解所用测序平台的分析特性和可能的偏差。
血浆中细胞游离DNA(cfDNA)的分析已成为包括妊娠、癌症和移植在内的不同临床情况下的重要诊断工具。PacBio最近使用单分子实时(SMRT)测序的研究显示,孕妇和癌症患者血浆中存在大量可分析的长cfDNA,检测到最长的胎儿源性cfDNA和肿瘤源性cfDNA分别为23.6kb和13.6kb。研究表明,这种长cfDNA分子上一系列CpG位点的甲基化模式有可能揭示单个cfDNA分子的组织来源,单分子组织来源分析在单基因疾病的无创产前检测和肝细胞癌(HCC)患者的检测中具有潜在的临床应用价值。此外,在先兆子痫患者中也发现了与疾病相关的片段组学特征。例如,它与没有子痫前期的孕妇相比,患有子痫前期的孕妇表现出较低的长片段百分比和不同的4-mer末端基序特征。除了PacBio SMRT光信号检测测序之外,ONT的纳米孔测序技术通过电信号检测,理论上为评估长达50kb的长DNA分子提供了另一种选择,Cheng等先前使用早期版本的ONT分析了血浆中的cfDNA,其通量相对较低(每个流通池大约50000个读长[reads]),结果表明,来自不同个体的合并血浆样本中只有0.06-0.3的血浆DNA分子长度超过1kb。过去几年,纳米孔测序技术的快速发展极大地提高了通量和测序精度。然而,目前尚不清楚使用PacBio平台检测血浆中长cfDNA的最新发现是否可以通过ONT的最新技术实现。PacBio和ONT的两个长读测序平台的基本原理不同,Logsdon等人对这两种技术进行了全面的回顾。本文中将对这两种用于分析血浆中cfDNA的长读测序平台进行系统比较。
一、材料和方法
1. 研究设计:在本研究的第一部分,使用不同大小的人工合成的人类和小鼠基因组DNA比较了PacBio和ONT测序获得的DNA大小分布,将观察到的来自2个平台的长、短DNA片段的摩尔比与预期比率进行比较(图1)。在第二部分中,使用PacBio和ONT测序分析了来自不同妊娠期妇女,乙型肝炎病毒(HBV)携带者和HCC患者的血浆cfDNA样本。比较了两个平台的测序结果,包括大小和片段末端图谱以及基于cfDNA单分子甲基化模式的组织来源分析(图1)。
注:缩写:dNTPs,脱氧核苷酸三磷酸;ZMW,零模波导
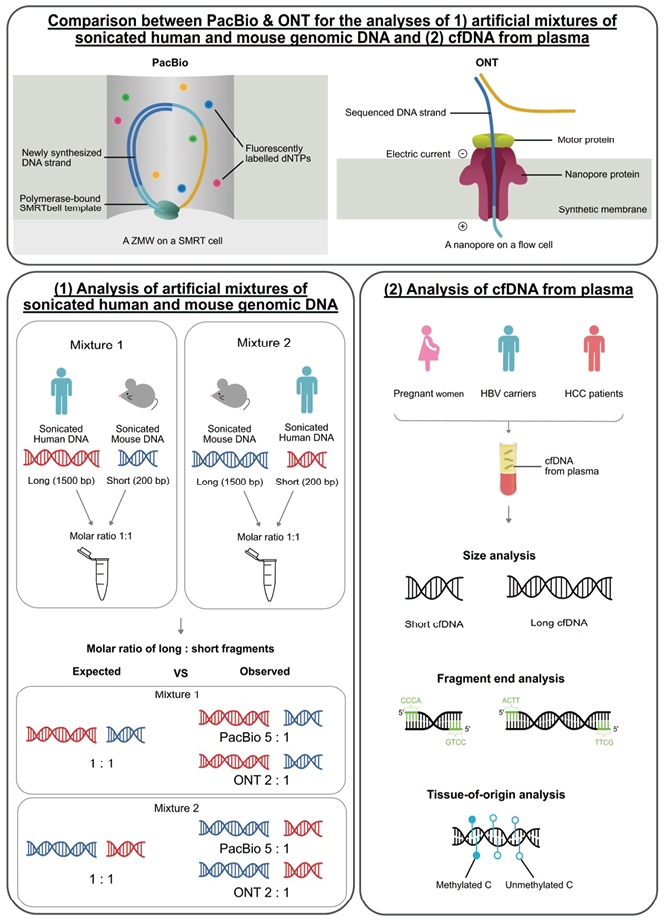
图1. 研究设计
2. 受试者来源与伦理:本研究得到了香港中文大学附属医院管理局新界东集临床研究伦理委员会的批准,在获得书面知情同意的情况下,从香港威尔斯亲王医院招募孕妇、慢性HBV感染患者(即HBV携带者)和HCC患者。所有涉及动物的实验程序均经香港中文大学动物实验伦理委员会批准,并按照《实验动物饲养管理和使用指南:第八版》进行。
3. 超声处理的人和鼠基因组DNA的人工混合物:分别从受试者和C57BL/6小鼠的血沉棕黄层中提取基因组DNA。根据制造商说明,使用S220聚焦超声波仪(Covaris)将人和小鼠基因组DNA超声处理成具有以下目标大小的片段:(1)1500bp的人DNA;(2)200bp的人DNA;(3)1500bp的小鼠DNA和(d)200bp的小鼠DNA。制备两种人和小鼠DNA的人工混合物:混合物1是1500bp人DNA和200bp小鼠DNA的等摩尔混合物,混合物2是1500bp小鼠DNA和200bp人DNA的等摩尔混合物。将每种混合物分成2份,分别制备成SMRT和纳米孔文库,对文库进行Sequel II(PacBio)和PromethION(ONT)测序。将测序读数与人类参考基因组(hg19)和小鼠参考基因组(mm10)进行比对。
4. cfDNA的PacBio测序:本研究中使用的部分血浆cfDNA SMRT测序数据,包括妊娠早期(n=7)、中期(n=8)和妊娠晚期(n=8)的数据,HBV携带者(n=6)和HCC患者(n=4),是从之前发表的数据集中获得,其余血浆cfDNA的SMRT测序数据为本研究新生成的数据,包括8例妊娠晚期和4例HCC患者,SMRTbell文库分别由4ml血浆中提取的cfDNA制备。
5. cfDNA的纳米孔文库制备和测序:PacBio数据集中45名患者的等分血浆样本用于生成纳米孔数据集。与PacBio使用的样本相比,这些样本没有经历额外的反复冻融。根据制造商的说明,使用牛津纳米孔技术连接测序(New England Biolabs)的NEB Next配套模块和连接测序试剂盒(SQK-LSK110),用3-4mL(平均体积:3.9mL)血浆的cfDNA进行纳米孔文库制备,清理步骤中使用AMPure XP磁珠,磁珠与样本比为1.8。短片段缓冲液保留了所有大小的DNA片段,用于接头连接步骤,将每个文库加载到PromethION流通池R 9.4.1上,并在PromethION设备上测序72小时。
6. 纳米孔测序数据的生物信息学处理:原始读取由集成到MinKNOW 4.3.12或5.1.5中的Guppy 5.0.16或6.1.5执行,使用Cutadapt 3.4进行接头序列剪切。对于每个文库,64.9%的碱基读长(reads)在5’端和3’端含接头序列,27.5%仅在5’端有接头序列、4.5%仅在3’端有接头序列,2.4%不含接头序列。使用minimap2 2.17进行接头序列剪切reads与人类参考基因组(hg19)比对,使用hg19是因为本研究中用于组织来源分析的肝组织甲基化图谱来自hg19的Roadmap表观基因组学计划。删除比对质量得分低于20的reads,对于每个样本,比对reads的中位数分别6.3%的hard-clipped reads(包含与参考基因组中不同位置比对的片段的嵌合读长)和51.6%的soft-clipped reads(在两端或任一端包含许多不能与参考基因组比对的碱基的读长)。为了从ONT数据中准确确定DNA片段的大小和末端,只将5'和3'末端都包含接头序列的reads与参考基因组比对,并且比对reads没有hard和soft clips的才用于片段大小和cfDNA末端序列的下游分析。
7. 碎片尺寸分析:对于SMRT测序分析的样本,每个DNA分子的大小是通过计算两侧发夹接头侧翼的测序核苷酸的数量,从读取的环形共有序列(CCS)确定的,对于用纳米孔测序分析的样本,测序的DNA片段的大小是由最外层的基因组坐标确定的。
8. 胎儿和母体特异性cfDNA片段的鉴定:使用iScan系统(Illumina)上的Infinium Omni2.5Exome-8 磁珠芯片对来自31例妊娠病例的配对母体和胎儿基因组DNA样本进行基因分型。PacBio和ONT的平均替换错误率(根据母亲和胎儿对同一等位基因纯合的单核苷酸多态性[SNP]基因座的所有读数中具有意外等位基因的读数比例估计)分别为0.8(范围:0.7%-1.0%)和3.4%(范围:2.4%-4.4%)。母亲为纯合子而胎儿为杂合子的SNP被认为是胎儿特异性等位基因的信息,反之亦然。ONT的高碱基替换错误被发现会影响胎儿和母体来源的cfDNA分化,如基于一个SNP信息鉴定胎儿和母体特异性片段时,胎儿和母体特异性片段之间大小差异无统计学意义。因此,基于ONT数据的2个SNP信息鉴定了胎儿和母体特异性片段。
9. 碎片末端分析:在末端核苷酸分析中,鉴定了测序DNA链5’端的第一个核苷酸。末端核苷酸百分比(即以A、C、G、T结尾的片段的百分比)是分别以A、C、G、T结尾的片段数除以总片段数。在末端基序分析中,识别测序DNA链5’端的第一个4核苷酸序列(4-mer基序),256个基序中每个基序的频率是通过以特定4-mer基序结尾的片段数量除以片段总数来计算的。对于SMRT测序,由于对双链DNA分子的Watson和Crick链进行了测序,因此将2条链的5’片段末端分别计为2个片段,而对于纳米孔测序,由于在1D测序中对单个DNA链(即Watson或Crick链)进行了测序,在片段末端分析中仅考虑测序链的5'片段末端。
10. 单分子组织来源分析:对于PacBio测序数据,使用基于卷积神经网络的模型(称为整体动力学模型)检测DNA分子上的5-甲基胞嘧啶。ONT测序数据使用基于隐马尔可夫模型的DNA修饰检测工具Nanopolish version 0.13.2检测5-甲基胞嘧啶。根据5-甲基胞嘧啶检测结果,确定每个血浆DNA分子的CpG甲基化模式,称为单分子甲基化模式。如前所述,对每个血浆DNA样本进行单分子组织来源分析。在母体血浆样本的分析中,将血浆DNA分子的单分子甲基化模式与血沉棕黄层和胎盘的参考甲基化谱进行了比较,甲基化谱分别基于94倍和75倍的高深度亚硫酸氢盐测序。如果血浆DNA分子包含至少5个信息丰富的CpG位点,则认为它们符合单分子组织来源分析的条件。血浆DNA分子的甲基化模式类似于胎盘的甲基化模式多于血沉棕黄层的甲基化模式,将被归类为胎盘源性。在分析来自HBV携带者和HCC患者的血浆样本时,血浆DNA的单分子甲基化模式分别与血沉棕黄层和肝脏的参考甲基化谱进行了75倍和36倍的比较,如果血浆DNA分子包含至少7个信息丰富的CpG位点,则认为它们有资格进行单分子组织来源分析。
二、研究结果
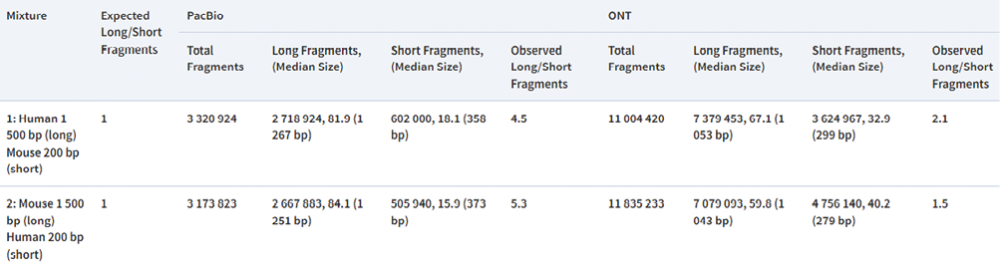
1. 使用超声后的人鼠DNA混合物评估PacBio和ONT测序长度偏好:对于每种混合物,预期的长、短DNA片段的比例为1:1,PacBio和ONT观察到的两种混合物的长、短DNA片段的比例分别约为5:1和2:1,这表明两种平台都显示出对长DNA片段的序列偏好,PacBio显示出比ONT更强的偏好(表1)。
表1. 使用人鼠DNA混合物进行的PacBio和ONT测序的长度偏好评估结果
2. PacBio和ONT的cfDNA测序数据:对于31份母体血浆cfDNA样本,分别从PacBio和ONT获得中位数为1,309,281(范围:89,586-3,406,954)的高质量CCSs和中位数为12,570,183(范围:755,935-40,128,142)的ONT数据。PacBio测序的所有高质量CCS数据均可直接用于下游测序长度和未端基序分析,但ONT数据需要额外的生物信息学处理,中位数为4,822,493(范围:208,457-19,003,253)的可用。
3. PacBio和ONT血浆DNA的大小分布:尽管从PacBio和ONT获得了长于500bp的cfDNA片段,但与ONT相比,PacBio测序的cfDNA片段要长得多(图2A)。例如,对于妊娠晚期母体血浆样本,PacBio长于500bp,1kb和3kb的片段百分比分别为39.3%,23.5%和2.78%,ONT分别为4.42%,0.74%和0.10%(图2A)。当使用PacBio和ONT对来自HBV携带者和HCC患者的血浆样本进行测序时,观察结果相似,其中与ONT相比,使用PacBio对更长的cfDNA片段进行测序。每三个月收集一次母体血浆样本的cfDNA片段,以更高的分辨率对cfDNA进行片段大小谱分析,发现与PacBio相比,ONT数据中的单核和双核体峰更高,超过400bp的寡核苷酸峰更低(图2,B和C)。在PacBio数据中明显的短cfDNA的特征性10bp周期峰在ONT数据中变得不那么明显(图2B),PacBio和ONT测序的最长cfDNA片段分别为42,139bp和60,423bp。在妊娠早期,中期和晚期,从ONT获得的>500bp的cfDNA片段的中位数百分比分别为4.30%,2.62%和5.37%;而从PacBio获得的>500bp片段的中位数百分比在3个月内分别为15.5%,16.1%和36.4%(图2D),从PacBio获得的>500bp的cfDNA片段的百分比显着高于从ONT获得的(P<0.001,Wilcoxon符号秩检验,图2D)。
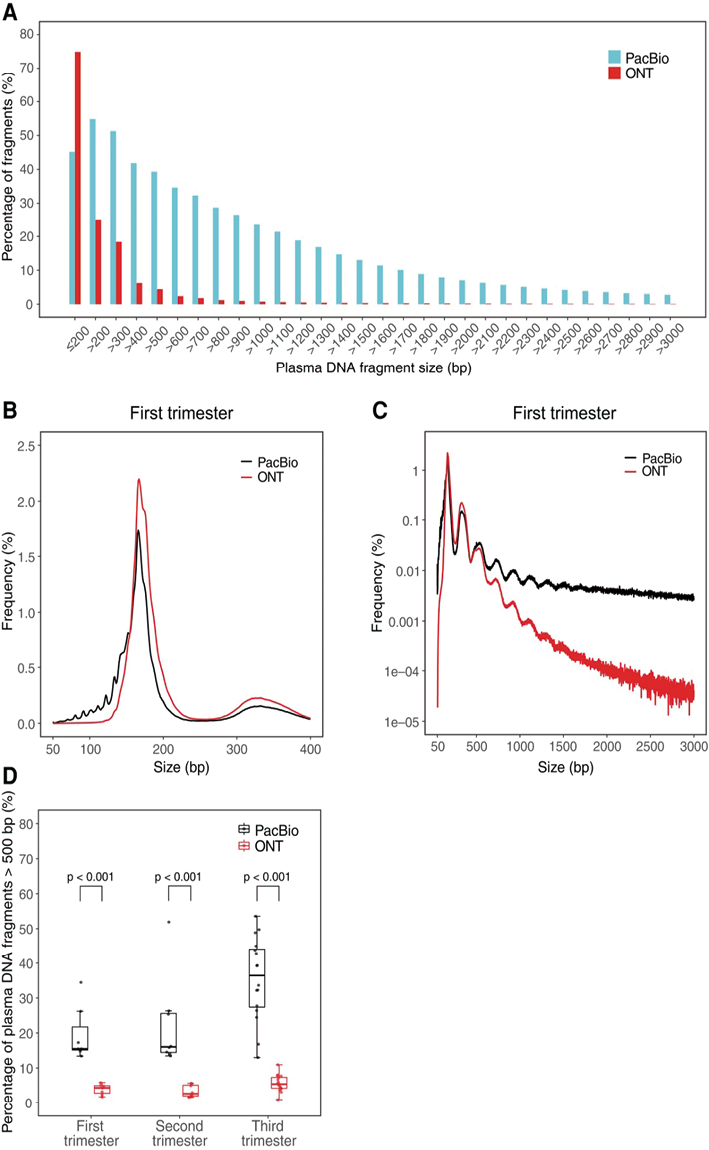
注:(A)PacBio和ONT对孕晚期孕妇血浆cfDNA样本进行测序,显示cfDNA片段低于和高于给定大小的百分比。用PacBio和ONT测序的孕早期母体血浆样本(n=7)cfDNA的大小分布;
(B)在x轴上绘制为50-400bp,在y轴上绘制为线性尺度;
(C)在x轴上绘制为50-3000bp,在y轴上绘制为对数尺度。
(D)PacBio和ONT对不同妊娠期孕妇血浆样本中长度超过500bp的cfDNA片段的百分比。
图2. PacBio和ONT测序分析母体血浆cfDNA的大小
关于胎儿和母体特异性cfDNA的大小分布,PacBio(图3,A和B)的长特异性cfDNA的比例比ONT(图3,C和D)高得多,PacBio和ONT胎儿特异性等位基因的最长cfDNA片段分别为23,635bp和13,989bp。
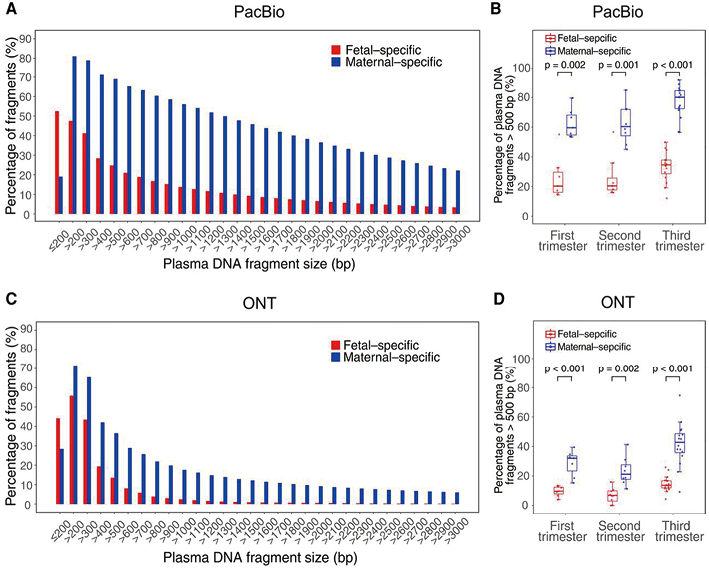
注:(A)从PacBio数据中测定的母体血浆中小于和大于给定大小的胎儿和母体特异性cfDNA片段的百分比;(B)箱形图显示了PacBio数据中测定的不同妊娠期母体血浆样本中长度超过500bp的胎儿和母体特异性cfDNA片段的百分比;(C)ONT数据中的母体血浆中低于或高于给定大小的胎儿和母体特异性cfDNA片段的百分比;(D)箱形图显示ONT数据的不同妊娠期的母体血浆样本中胎儿和母体特异性cfDNA片段长于500bp的百分比。
图3. PacBio和ONT测序的胎儿和母体特异性cfDNA的大小分布
4. PacBio和ONT的血浆DNA片段末端图谱:为了比较PacBio和ONT获得的cfDNA 5’端核苷酸谱,将每个妊娠期获得的血浆样本中的cfDNA片段汇总在一起。对于小于500bp的cfDNA片段,PacBio和ONT数据中C端片段最多,其次是PacBio中的G端、T端和A端片段(图4A)和ONT中的G端、A端、T端片段(图4B)。从500bp开始,PacBio和ONT数据中C端和T端片段的百分比逐渐下降,A端片段的百分比大幅上升,G端片段的百分比略有上升(图4)。从ONT获得的cfDNA 5’端核苷酸谱中,在1kb左右存在一些噪声,这可能是由于ONT数据中长度超过1kb的片段数量要少得多(图4B)。例如,通过ONT测序获得的7份孕早期母体血浆样本中>1kb的片段总数比PacBio获得的少6倍(即ONT获得的188,270个片段比PacBio获得的1,095,190个片段)。
当基于256个4-mer 5’端基序的频率进行分层聚类时,用PacBio和ONT测序的血浆cfDNA样本形成了不同的聚类(图4C)。通过汇总各个平台的数据比较PacBio和ONT之间的4-mer端基序排名,两者存在很强的正相关(Spearman相关,r=0.9,P<0.001)(图4D)。进一步检查PacBio中排名前10位但在ONT中排名11位或更低的基序,表明4-mer末端基序CAAA、CCAT、GAAA、GGAG和GGAA在ONT的结果中更多,而4-mer末端基序CCCT、CCAG、CCAA、GCCT和GCTT在PacBio的结果中更多。
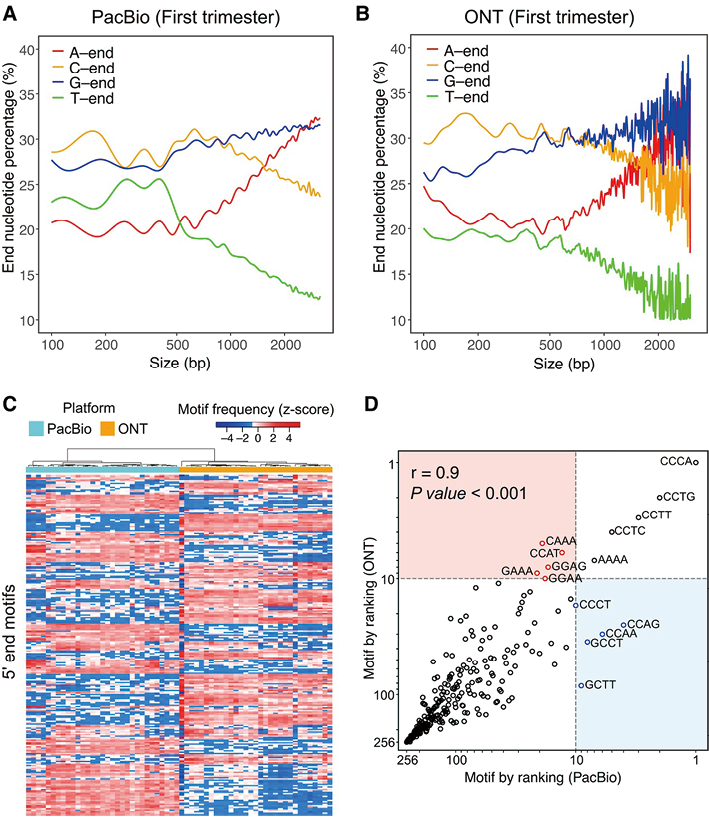
注:(A)和(B)在100bp到3kb的片段大小范围内,用(A)PacBio和(B)ONT测序的cfDNA片段的百分比,在5’端以A、C、G和T结尾(x轴以对数标尺绘制);(C)使用256个4-mer末端基序的频率对31个用PacBio和ONT测序的母体血浆cfDNA样本进行分层聚类分析,每列表示用PacBio或ONT测序的cfDNA样本(如第一行所示),从第二行开始,每行表示一种4-mer末端基序,根据标准分数(z分数),末端基序频率用一系列颜色梯度表示(即样本中低于或高于平均频率的SD数),颜色谱的红色末端表示末端基序的较高频率,颜色谱的蓝色末端表示末端基序的较低频率;(D)PacBio和ONT中256个4-mer末端基序的基序排名的相关性。每个圆圈表示一个4-mer基序。右下角的圆圈表示在PacBio中排名前10,但在ONT中排名11或以下的基序,左上角矩形内的圆圈表示在ONT中排名前10位但在PacBio中排名11或更低的。缩写:A,腺嘌呤;T,胸腺嘧啶;C,胞嘧啶;G,鸟嘌呤。
图4. PacBio和ONT测序的母体血浆中cfDNA的片段末端谱
5. 用PacBio和ONT分析血浆DNA单分子组织来源:为了评估基于甲基化模式的单分子ONT数据组织起源分析的性能,使用从SNP分析中鉴定出的491个胎儿特异性片段和13,888个母亲特异性片段,使用ONT数据进行的组织来源分析区分胎儿和母体cfDNA时,曲线下面积为0.86,这与先前报道的PacBio性能相当(0.88)(图5A)。当使用31个母体血浆DNA样本的PacBio和ONT数据进行组织来源分析时,样本符合组织来源分析条件的cfDNA片段中位数分别为27,987(IQR:19,096-37,878)和68,150(IQR:38,043-156,290),PacBio和ONT的合格cfDNA片段的中位数大小分别为667 bp(IQR:329-1,288bp)和462bp(IQR:337-728bp)。在合格的片段中,对于PacBio和ONT,样本被鉴定为胎盘来源的片段的中位数分别为4,357(IQR:2,819-9,733)和16,440(IQR:7,892-44,482)。与PacBio先前研究类似,使用ONT数据通过单分子组织来源分析确定的胎盘来源的cfDNA百分比与基于SNP的方法独立确定的胎儿DNA分数密切相关(Pearson相关,r=0.9,P<0.001)(图5B)。使用HBV携带者和HCC患者的血浆样本,进一步比较了PacBio和ONT对cfDNA的组织来源分析的性能,PacBio和ONT中样本中可用于组织来源分析的cfDNA片段的中位数分别为1,136(IQR:418-1,534)和4,221(IQR:2,523-11,902)。ONT单分子组织来起源分析数据确定的肝源cfDNA百分比与PacBio数据呈正相关(Pearson相关,r=0.99,P<0.001)(图5C)。对于每个样本,制定一个HCC甲基化评分的指标,用于区分患HCC和未患HCC的患者,发现,PacBio(P<0.001, Mann-Whitney U检验)和ONT数据(P=0.0026, Mann-Whitney U检验)中,HCC患者的HCC甲基化评分均显著高于HBV携带者(图5D)。使用无HCC的患者中高于平均HCC甲基化评分3SD的值作为临界值(0.77),实现了PacBio和ONT数据的75%灵敏度和100%特异性。
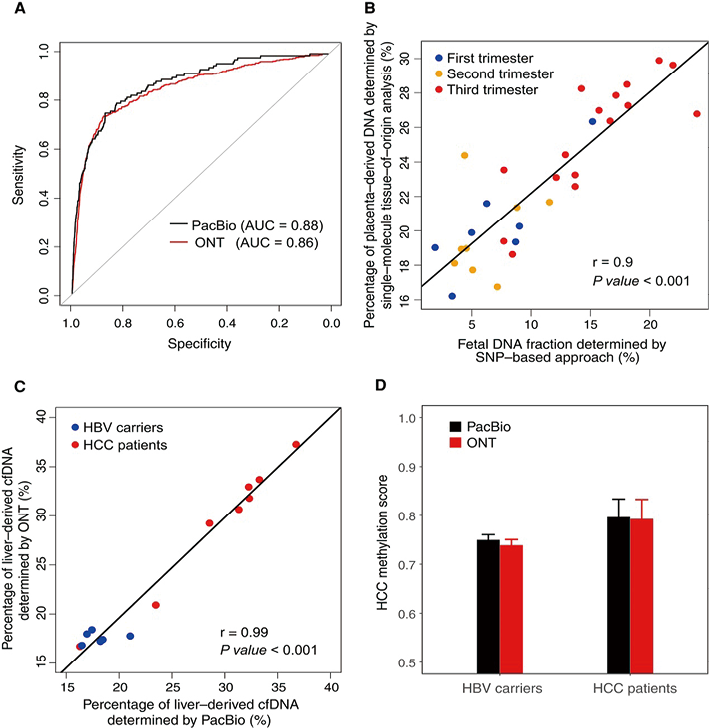
注:(A)ROC曲线显示,使用PacBio和ONT血浆cfDNA的单分子组织来源分析在区分胎儿和母体特异性cfDNA方面的性能;(B)在31个采用ONT测序的母体血浆DNA样本中,通过单分子组织来源分析确定的胎盘来源cfDNA百分比与基于SNP方法确定的胎儿DNA分数之间的相关性;(C)使用PacBio和ONT数据的单分子组织来源分析测定的HBV携带者和HCC患者血浆样本中肝源性cfDNA百分比之间的相关性;(D)使用PacBio和ONT数据计算的HBV携带者和HCC患者的HCC甲基化评分之间的比较,误差线代表1SD。
图5. PacBio和ONT对血浆cfDNA进行单分子组织来源分析
三、讨论与分析
在本研究中,比较了PacBio和ONT对血浆中长cfDNA的分析性能。一个惊人的发现是,与ONT相比,PacBio对更长的血浆cfDNA进行了测序。来自PacBio超过1kb的cfDNA片段百分比大约是ONT的30倍。从cfDNA的大小谱中注意到,在PacBio中很明显的短cfDNA特征性10 bp周期峰在ONT数据中几乎不可见,推测,由于短cfDNA通过纳米孔的快速和不稳定,ONT可能无法有效测序少于200个碱基的短接头连接片段(即连接到约80bp接头的小于150bp的cfDNA片段)。另一方面,PacBio对长片段序列的偏好可能部分是由于在磁珠清理步骤中,它与短片段相比,长片段的聚合酶结合复合物的回收率更高,和/或长片段到零模波导的加载效率更高,需要进一步的实验来验证这些观点。未来的研究需要使用基于非测序的方法进一步验证血浆中存在长cfDNA。ONT PromethION提供的每流细胞映射读数是PacBio Sequel II的10倍,尽管ONT测序长cfDNA片段的百分比较低,但ONT对每个样本进行单分子组织来源分析的cfDNA片段数量是PacBio的2至4倍。ONT PromethION在可扩展性方面具有额外的优势,允许同时测序多达48个流细胞,每Gb数据的估计成本大约是PacBio Sequel II的一半。与ONT reads相比,PacBio的CCS reads显示出更低的替换错误率(PacBio为0.8%,ONT为3.4%),使其成为需要准确变异识别的临床应用的更有利平台,例如,单基因疾病的无创产前检测和癌症相关单核苷酸变异的检测。
在优化分析工作流程的过程中,我们注意到ONT测序数据中的几个特征。首先,不包含接头或仅在一端包含接头的每个样本的碱基读数中位数为35.1%,碱基读数两端都没有接头序列可能是由于文库制备过程中2个接头中只有1个与DNA分子连接,测序DNA链上存在缺口,接头连接后DNA降解或原始读数两端的每个碱基序列质量低。此外,包含5'接头的读数多于包含3'接头的读数。这可能是因为马达蛋白连接到5'接头上,马达蛋白解开双链DNA并控制单链DNA通过纳米孔的易位,从而使DNA链得以测序,其次,51.6%的对齐读长中位数在两端或任意一端包含软剪辑(soft clips),soft clips的可能来自未修剪的接头序列、测序错误和真实序列变体。第三,我们注意到,在ONT数据中,每个样本的嵌合读长中位数为6.3%,这些嵌合读物可能代表真正的结构变体,接头连接之前进行cfDNA片段的钝末端连接,以及将串联的单独输入读取由basecaller转换为单个输出读取,这可能是由2个或多个接头连接的短cfDNA通过单个孔的快速易位引起的,是嵌合读取的主要贡献者,这是因为接头序列可以在这些读数的中间部分被识别,这些读数将那些可以与基因组不同部分比对的序列分开。虽然发生频率相对较低的hard-clipped读长对cfDNA分析的影响很小,但soft clips读长和接头修剪之前两端均无可识别的接头序列(影响近一半的读长)将对cfDNA的片段大小和末端分析产生直接而显著的影响。此外,从不同测序平台获得的cfDNA的大小和末端图谱存在明显差异,这些发现突出了ONT和PacBio在cfDNA测序中的一些重要分析特征,为解释从不同的长读测序技术获得的数据提供了见解。
总之,它与ONT测序相比,血浆cfDNA的PacBio测序产生具有更高百分比的长cfDNA数据。使用PacBio和ONT的数据进行基于单分子甲基化模式的cfDNA组织来源的分析性能类似,尽管PacBio生成的数据具有更高百分比的长cfDNA,但每个样本符合组织来源分析条件的cfDNA片段数量仅比ONT高出2-4倍。当使用ONT数据对cfDNA进行大小和末端基序分析时,需要实施额外的测序数据处理步骤,包括识别和修剪两端接头序列以及用soft clips去除读长,以确保准确确定片段大小和末端。希望这些观察结果可以作为新出现的长cfDNA诊断领域选择分析平台的初步参考。
翻译自《Clinical Chemistry》,Volume 69,Issue 2,February 2023




