微液滴数字PCR检测方法的建立与β-地中海贫血无创产前诊断临床应用评价
【摘要】目的 地中海贫血是遗传性血液疾病,也是迄今为止全球最常见的单基因疾病之一。β-地中海贫血在塞浦路斯的患病率特别高,IVSI-110 G>A(HBB:c.93-21G>A)致病性变异占总携带者的近79%。母体血浆中存在3%-20%的胎儿游离DNA(cffDNA),这一发现使β-地中海贫血等单基因疾病的无创产前诊断(NIPD)得以发展,从而避免了侵入性操作的风险。然而,NIPD的发展面临重大技术挑战,尚未达到临床应用水平。方法 本研究中应用微液滴数字PCR (ddPCR)结合相对突变剂量法建立了IVSI-110 G>A β-地中海贫血的NIPD检测方法。实现了ddPCR的优化流程以解决ddPCR检测的挑战,如不确定的雨区(rain)液滴,从而提高了检测的灵敏度和特异性。对父母双方均携带相同致病变异的40个母亲血浆样本(中位胎龄为10周)进行了评估。结果 正确分类33个样本,未确定分类6个样本,错误分类1个样本。检测具有97.06%的准确性(95% CI,82.46-99.68),100%的灵敏度(95% CI,76.84-100)和95%的特异性(95% CI,75.13-99.87),证明了其对母系、父系等位基因无创检测的有效性。结论 本研究建立了一种高效、简便、经济的ddPCR方法,可用于IVSI-110 G>A β-地中海贫血风险夫妇的胎儿基因型无创检测,使单基因疾病的NIPD更接近临床应用。
地中海贫血是遗传性血液病,也是目前最常见的单基因疾病之一。全球约有5%的人是无症状携带者,估计每年有超过33万名新生儿受到影响。β-地中海贫血在塞浦路斯的患病率特别高,携带者频率约为12%,其中IVSI-110 G>A(HBB:c.93-21G>A)β-珠蛋白致病性变异占总携带者的近79%,β-地中海贫血遵循常染色体隐性遗传,约76%的塞浦路斯夫妇进行产前诊断有IVSI-110 G>A致病变异的风险。目前,唯一可行的预防方法是侵入性产前诊断,但这会导致身体不适,并带来医源性流产的巨大风险。Lo等发现母体血液循环中存在胎儿游离DNA(cffDNA),这为非侵入性方法的发展提供了机会。无创产前诊断(Non-invasive prenatal diagnosis,NIPD)允许从简单的孕妇外周血样本中分析胎儿DNA,使其成为传统侵入性方法的无风险替代方法。母体血浆含有胎儿和母体游离DNA(cfDNA)的混合物,胎儿的占少数,平均占总cfDNA的10%,称为胎儿分数。cfDNA以短片段的形式存在,胎儿来源的DNA片段比母亲的短,主要为143bp,然而,胎儿基因组是作为一个整体表示。
cfDNA的所有这些特点造成了重大的技术挑战,阻碍了NIPD应用的发展。NIPD在已进入临床实践的单基因疾病中的应用仅限于检测来自母体缺失的等位基因,如父源性疾病。对于母源性单基因病,需要高灵敏度的定量方法在母体DNA序列相同的高背景下区分胎儿DNA序列。为此,多个研究团队尝试了不同的方式和方法,包括相对突变剂量(RMD)和相对单倍型剂量(RHDO)。下一代测序(NGS)是首选的方法:一种强大、高灵敏度的技术,能够检测基因组中的微小遗传变异。然而,NGS成本高且需要复杂的生物信息学分析,不太适合常规应用。微液滴数字PCR(ddPCR)是一种基于PCR的技术,能够对目标DNA分子进行绝对定量。它与NGS相比,具有较高的灵敏度和较低的成本,并且不需要复杂的样本制备和分析,该方法具有较高的精密度,可用于检测样本中罕见的靶DNA序列,为母体血浆中低含量的cffDNA检测提供了有利的方法学选择,在NIPD中具有良好的应用前景。已有一些研究者将ddPCR应用于单基因病的NIPD,并取得了较好的结果。尽管单基因病的NIPD取得了实质性进展,但尚未实现常规临床应用。一些用于单基因疾病的NIPD检测法已上市,但仅用于筛查目的,结果可能不可靠,需要有创性的随访。
在本研究中利用ddPCR开发和优化了用于检测cfDNA中最常见的塞浦路斯β-地中海贫血致病变异(IVSI-110 G>A)的NIPD检测方法,以覆盖塞浦路斯有β-地中海贫血风险的大部分孕妇。对于胎儿基因型的确定,采用基于等位基因比率和Z分数计算的相对变异剂量(RVD)方法。对40个来自父母均携带相同致病变异的β-地中海贫血风险夫妇的母体血浆样本进行该检测方法的评估。
一、研究对象与方法
1. 患者样本收集和伦理声明:从40名有IVSI-110 G>A新生儿β-地中海贫血风险的孕妇身上采集了约9ml的外周血样本。使用EDTA标准采血管(n=33)或细胞游离DNA BCT®管(Streck)(n=7)在妊娠的第8-13周之间(中位数=10)收集样本。只选择了单胎妊娠。还使用EDTA标准试管,从孕妇的配偶、夫妇的父母关于IVSI-110 G>A致病变异的所有3种基因型个体中采集了外周血样本。该研究得到塞浦路斯国家生物伦理委员会(ΕΕΒΚ/ΕΠ/2018/51)的批准,所有参与者均提供了书面知情同意,所有程序均符合赫尔辛基宣言。
2. 血浆分离:以2500g离心10min,从所有孕妇和3个IVSI-110 G>A携带者的EDTA或Streck采血管中分离血浆。保证采血后2小时内分离血浆,以防止母体细胞裂解和母体DNA释放。上清液在17,000g离心40min,以完全去除血小板和沉淀物,所得上清液收集到一次性使用1mL存储管,在-20℃下保存直至cfDNA提取,或在-80℃长时间保存。
3. DNA提取:使用QIAamp试剂盒从2mL血浆中提取游离DNA,并根据说明书用65μL缓冲液AVE洗脱,使用Puregene Blood Core Kit C或MagCore全血基因组DNA试剂盒提取基因组DNA(gDNA)。
4. 超声检查:将9份gDNA样本(7×GA、1×GG、1×AA)剪切成更小的片段,以模拟母体血浆中的胎儿cfDNA片段,用于ddPCR优化试验。使用Bioruptor Pico超声系统对样本进行超声处理,应用了40个循环,每个循环设置为3s开/30s关,通过重复的一系列循环试验确定,168-177bp的片段最适合于试验。为了观察超声处理后的碎片大小,Agilent 2200 TapeStation系统与高灵敏度D1000 ScreenTape和试剂一起使用。
5. 数字PCR:建立ddPCR检测IVSI-110 G>A致病性变异的方法。由于母体血浆中的cfDNA长度较小,因此设计引物长度小于100bp(93bp)的扩增子,FAM(荧光素酰胺)和HEX(六氯荧光素)荧光标记探针分别用于检测野生型(G)和突变型(A)等位基因。ddPCR使用QX200 AutoDG微液滴数字PCR系统,按照说明书进行,稍作修改。所有cfDNA样本均进行了6次重复分析,每次ddPCR运行包括4个母体cfDNA样本,在不同的ddPCR运行中分析样本以证明可重复性。通过QuantaSoft™分析软件(版本1.7.4.0917),手动设置阈值分配阳性或阴性液滴。后续分析中排除液滴总数少于10000个(即由于液滴生成错误)或1D和2D振幅图异常的情况。
6. RVD方法与分析策略:用QuantaSoft™软件对结果进行分析,确定胎儿的基因型。为此,我们将RVD方法与等位基因比率和z分数的计算相结合。RVD基于RMD方法的原理,即当母亲为杂合子时,该方法侧重于等位基因比率的计算。对于杂合子胎儿,两个等位基因完全平衡,因此等位基因比例率预计为1。在纯合子胎儿的情况下,等位基因比例率预计会向过表达的等位基因倾斜,因此为>1或<1。每个样本的等位基因比率被认为是其所有6次重复的平均值,由QuantaSoft™软件自动计算。根据与Perlado等人类似的原理,还使用RVD方法计算Z分数以确定胎儿基因型。更具体地说,Z分数是使用两个等位基因的浓度计算的,如下所示:
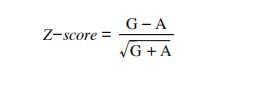
其中G和A分别为FAM标记的G等位基因和HEX标记的A等位基因的浓度(拷贝数/μL)。计算每次重复的z分数,样本的最终Z分数值为其全部6次重复的Z分数值的平均值。
7. 胎儿基因分型:为了确定胎儿基因型,建立了等位基因比率和Z分数的标准阈值。为此,首先分析的30份母体血浆样本的比率和Z分数,并将其分为3种可能的胎儿基因型。然后计算每个胎儿基因型的平均值(比率和Z分数),并添加了一个标准差(±1SD)以建立每个基因型的阈值。定义了四类:纯合GG,杂合GA,纯合AA(受影响)和灰色区域(不确定)(图1)。然后将这些阈值应用于其余10个母体血浆样本,根据样本的比率和Z评分值,应用RVD方法确定胎儿基因型。然后将NIPD结果与先前在实验室中进行的相应绒毛膜样本(CV)的侵入性产前诊断结果进行比较。
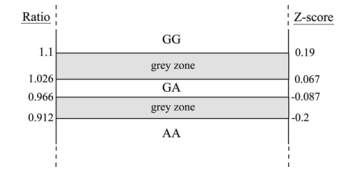
图1. 建立了等位基因比率和Z评分的阈值,以对样本的胎儿基因型进行分类。RVD方法对胎儿基因型进行了分类,共定义了四类。灰色区域是导致不确定测试结果的3种基因型阈值之间的空间
二、研究结果
1. 优化过程:ddPCR检测优化是该研究的主要部分,先前检测进行的试验表明,仅从gDNA和cfDNA杂合子样本中揭示的比率并不是最佳的,这两个等位基因不完全平衡。还发现存在称为雨区(rain)的液滴,位于中等荧光水平,不能清楚地分配阴性或阳性液滴簇,雨区液滴存在使得难以设置准确的阈值,导致等位基因拷贝数以及随后的等位基因比率和Z分数的计算不准确,优化过程的最终目的是在尽可能少的雨区液滴情况下,最大程度地清晰分离阴阳液滴簇。优化步骤需要在51-61℃的温度下进行梯度ddPCR,PCR扩增完成后(读取前)在4℃条件ddPCR平板孵育过夜,并将引物终浓度从450nmol/L增加到900nmol/L。PCR热曲线上的不同参数发生了变化,例如将每个PCR步骤的升温速率从2℃/s降低到1℃/s,将变性时间从30s增加到1min,并将PCR循环次数增加到45和50(图2)。优化策略是基于保存有价值的母体血浆样本(图2)。使用7个杂合子胎儿片段化gDNA样本,将其稀释至近似浓度的cfDNA(0.5ng/μL),进行条件改变的初步试验。然后,在3个杂合cfDNA血浆样本(IVSI-110 G>A携带者)上检测最优条件,并在母体cfDNA血浆样本上评估最终优化的检测方法。最终优化的方案包括:引入4℃平板孵育过夜,55℃退火/延伸,450nmol/L引物终浓度,1min变性,2℃/s升温速率和45个循环。引物浓度和上升速率与原始方案相同,因为改变的条件(900nmol/L和1℃/s)没有获益。

图2. ddPCR工作流程优化,工作流程表明了保存有价值的母体cfDNA样本所遵循的方法,以及优化过程中改变的条件
2. 母体血浆样本的评估:通过RVD方法评估所建立的优化ddPCR检测用于确定胎儿基因型。对来自高危妊娠的40份母亲血浆样本进行了评估,其中所有母亲都是IVSI-110 G>A致病变异的杂合子。计算每个样本的比率和Z评分,并根据RVD方法将其分为4类胎儿基因型之一。NIPD结果与胎儿相应CVS样本进行侵入性诊断结果进行比较。检测方法的优化是显而易见的,少量雨区液滴存在下实现了阴阳液滴簇之间更好的分离(图3),少量雨区液滴并不影响等位基因拷贝数的计算,因为使用3种不同的阈值(低,中和高)会产生相似的等位基因比率(表1)。因此,优化的ddPCR检测法可产生准确的阈值设置,从而产生最佳的等位基因比率和Z分数。最初,31个样本中成功确定了胎儿基因型,与通过侵入性产前诊断的基因型一致(表2),8个样本不确定(灰色区域),因此必须重复检测,8个中有7个样本的血浆足够重复ddPCR运行,重复分析中有2个成功确定了胎儿基因型,其余的仍然没有定论,40个样本中只有1个被错误分类,但在重复分析调查原因后,该样本被正确分类。在总体结果中,将这个样本列为“错误分类”,因为实际应用中不会选择它。因此,33个样本被正确分类,6个没有结论,1个被错误分类(图4)。总的来说,我们的检测在确定胎儿基因型(包括母系和父系遗传的胎儿等位基因)方面表现出97.06%的准确率(95%CI,82.46-99.68)、100%的敏感性(95%CI、76.84-100)和95%的特异性(95%CI,75.13-99.87)。
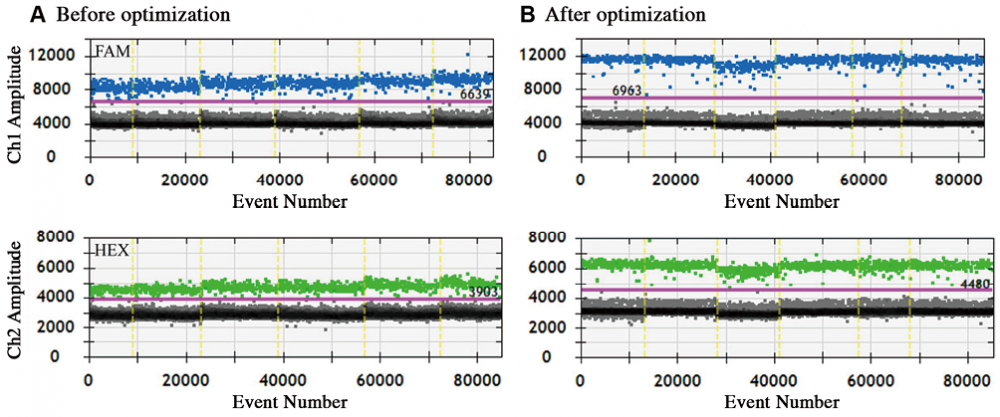
图3. 样本cfDNA266优化过程之前(A)和之后(B)的FAM和HEX荧光对比
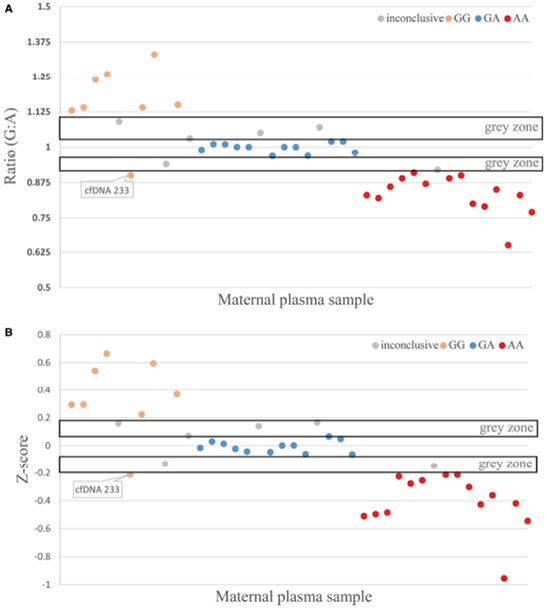
图4. 根据RVD方法分类的40个母体血浆样本的比率(A)和Z分数(B)的示意图。一个假阳性样本cfDNA 233。
表1. 来自6个母体血浆样本的3种不同阈值设置的等位基因比率结果
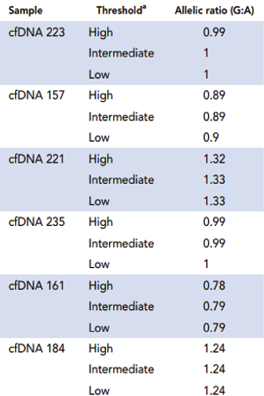
表2. 40个母体血浆样本的胎儿基因型a
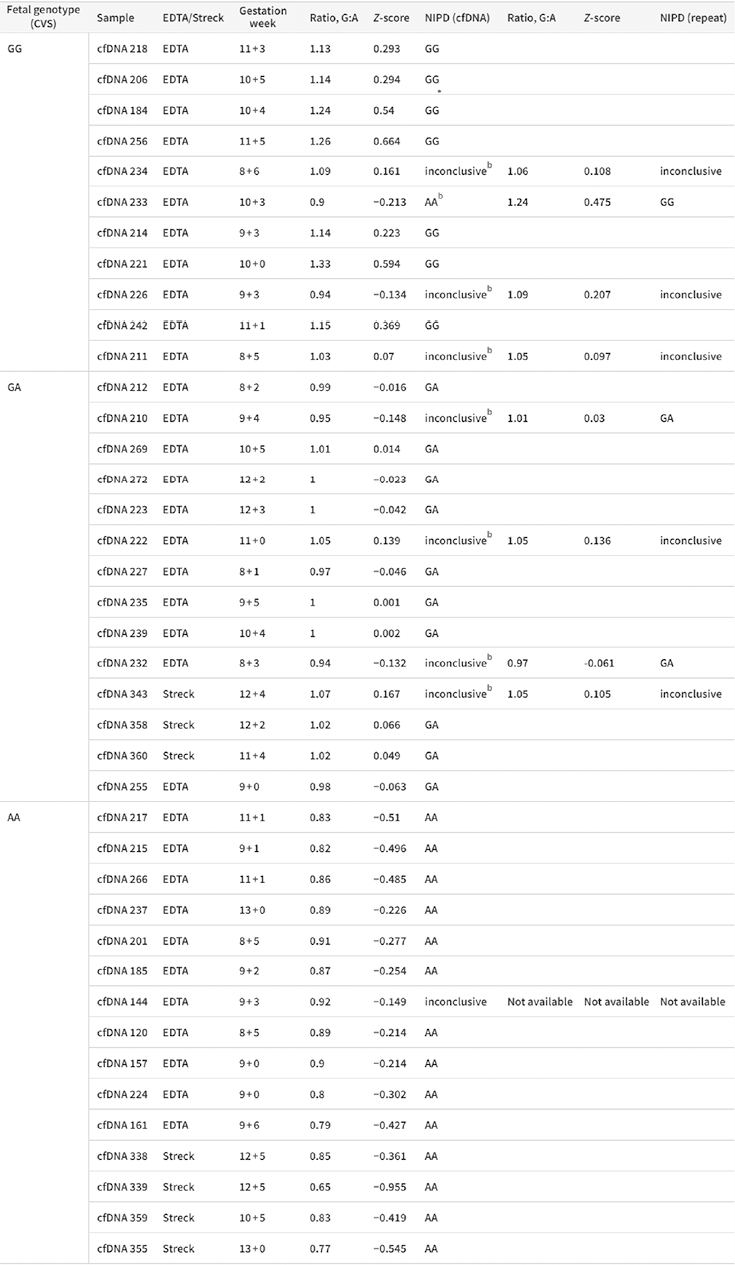
注:a 根据第一次ddPCR检测阐明的比率和Z评分阈值(图1),31个样本被正确诊断,8个样本不确定,1个样本被错误分类。b 在新的ddPCR检测中重复了7个不确定的样本和错误分类样本,其中2个不确定样本和错误归类的样本为正确的诊断
三、分析与讨论
单基因疾病(如β-地中海贫血)的产前诊断目前依赖于侵入性操作,在过去的几年中,许多研究报道成功开发了NIPD方法,但是,大多数这些研究都是基于仅确定父系遗传的胎儿等位基因,因此需要一种可以确定父母两个等位基因的方法,从而能应用于所有妊娠的方法。对于β-地中海贫血,几个小组利用NGS和ddPCR提高分析灵敏度来实现这一目标。尽管取得了进展,但单基因疾病的NIPD尚未进入临床应用,除了一些仅用于筛查目的且需要侵入性随访的疾病。本研究中,应用ddPCR结合RVD方法开发了检测父母等位基因的NIPD检测方法,使其适用于所有IVSI-110 G>A β-地中海贫血致病性变异风险的妊娠。经过一系列优化,建立了一种ddPCR方案,可成功从IVSI-110 G>A携带者母亲的cfDNA样本中确定胎儿的基因型。
通过初步试验,观察到存在无法明确区分阴阳性的雨区液滴,这导致难以设置区分两类液滴簇的阈值。雨区液滴可能是由液滴受损、非特异性扩增或液滴大小不规则引起的。由于NIPD需要高灵敏度和特异的检测方法来区分杂合子比例的细微变化,我们开始优化检测方法。为了最大限度地减少雨区液滴的存在,避免任何扩增误差和交叉反应并最大限度地提高液滴分离,我们通过改变ddPCR工作流程、PCR条件和试剂中的各种参数来提高检测性能。试验优化成功,我们设法增加了阳性和阴性液滴的分离并减少了雨区液滴的存在,少量雨区液滴的存在并不影响等位基因拷贝数的准确计算,因为阈值设置的广泛变化给出了相似的比率,从而得出了相同的胎儿基因型,这一发现非常重要,因为手动设置阈值不会影响临床环境中结果的准确性。使用RVD方法,在分析的40个cfDNA母体血浆样本中,有33个成功确定了胎儿基因型,不包括第二次检测的假阳性样本,这与之前常规产前诊断的相应CVS样本结果一致。8个样本在第一次检测中没有结论,其中7个在第二次ddPCR中重复检测,1个由于缺乏血浆样本而没有重复,重复的样本中有2个得出了正确的胎儿基因型,而其余的仍然没有结论。仅观察到1个假阳性结果,将未患病胎儿识别为患病胎儿,为了研究这种不一致是由于样本污染还是检测的特异性低,进行ddPCR重复确定正确的胎儿基因型,假阳性结果可能归因于样本处理的所有阶段可能发生的样本污染,如血浆分离、cfDNA提取或ddPCR平板制备。然而,实际应用中不会重复样本,因此在研究结果中它仍然是“错误分类”。
本研究是第一项基于NIPD分析开发的研究,该研究实现了ddPCR的优化过程,优化可提高其灵敏度和特异性,这对使用诸如cfDNA等具有挑战性的材料时至关重要。在此,我们提出了用于NIPD目的的ddPCR检测的优化流程,首先使用与cfDNA性质相似的稀释和碎片化杂合子gDNA样本进行优化,然后使用杂合子的cfDNA样本,最后对来自母体血浆样本的cfDNA进行检测条件评估,该方案旨在保存有价值的母体血浆样本。本研究也是第一项使用如此早期胎龄的样本进行的高成功率NIPD研究。NIPD检测的目的是在尽可能早的孕龄提供诊断,以便夫妇能够做出继续妊娠的知情选择。目前的NIPD检测使用中位胎龄为10(8-13)周的样本取得了97.06%的高度成功诊断率,这比传统的CVS产前诊断要早,早在妊娠第8周就成功对样本做出诊断。与之前的研究相比,通过分析低量血浆样本(2mL)实现了高准确性,需要1管9ml的外周血,以便在第一次运行没有结论的情况下进行额外的ddPCR试验。更重要的是,2个采血管之间的ddPCR检测结果没有差异,这可能由于血浆在采集后2h内足够早时间进行分离,以防止母体DNA释放到EDTA管中。
本研究结果表明,ddPCR是用于NIPD的一个很好的方法学选择,它提供了cfDNA的高度定量分析,对于计算等位基因比率和检测任何提示纯合胎儿的失衡至关重要。与常用的NGS技术相比,该技术周转时间短、工作流程简单、无需复杂的生物信息学分析,更适用于临床诊断。开发ddPCR检测的主要限制是需要预先已知变异,以及每个分析检测单个变异的能力。ddPCR提供多路复用选项,然而这将使检测法的开发变得复杂并可能降低每种变异的灵敏度,而这对NIPD检测至关重要。在检测中观察到一些无法确定胎儿基因型的样本,不确定的结果是检测的主要局限性,因为40个样本中有6个(15%)仍然不确定,可能是由于样本中胎儿来源比例低,不足以改变等位基因比率。由于我们还没有开发出一种排除低胎儿分数的计算方法,这给检测方法带来了另一个局限性,因为确认成功分离出胎儿cfDNA或估算出胎儿分数将提高结果的可信度,因此,为了提高可信度并减少潜在偏倚,对每个样本进行6次重复。对于纯合子胎儿,我们对结果的可靠性充满信心,因为等位基因比率的变化证实了胎儿DNA的存在,局限性在于杂合结果,无法确认胎儿cfDNA的存在,因为缺乏它也会导致杂合结果。尽管存在局限性,但通过对CVS样本的回顾性分析证实了检测的有效性。为了克服这些局限性,应该开发一种用于计算胎儿分数的检测方法,以确保胎儿分数的完整性。此外,可以对胎儿片段进行预扩增或富集,以确保样本中有足够的胎儿cfDNA。为避免错误分类,可将样本分别进行2次ddPCR试验以确认结果,如果不一致,可以进行另一次检查或进行有创分析,在少数不确定结果的情况下,这对夫妇可以选择侵入性产前诊断。此外,我们开发的ddPCR检测方法可以与另一种方法(如NGS)相结合,合并到一个管道中,用于确定胎儿基因型的综合NIPD策略,一种方法将用于确认另一种方法的结果,从而提高诊断准确性并将NIPD引入临床实践。
综上,本研究开发了一种高效、简单、经济的ddPCR检测方法,用于无创检测IVSI-110 G>A β-地中海贫血风险夫妇的胎儿基因型,适用于所有IVSI-110 G>A携带者女性的妊娠。该方法在分析中位胎龄为10周的40个母体血浆样本时显示出97%的准确性,100%的敏感性和95%的特异性,避免了对胎儿进行侵入性产前检测,并使NIPD更接近临床应用环境。
翻译自:《Clinical Chemistry》68:8(2022)




