方兴未艾的精准医疗
一、何谓“精准医疗”
2015年1月20日,时任美国总统巴拉克•奥巴马在国情咨文中首次提出了Precision Medicine Initiative (PMI) (国内翻译为“精准医疗计划”),目的在于使得美国大众能够更进一步的治疗诸如癌症、糖尿病等疾病,同时能够赋予美国人民能够获知为必需保持其自身和家庭健康的个性化信息。为了完成这一伟大计划,美国国立卫生研究院(NIH)的主任Dr. Francis Collins组建了精准医疗计划工作组(下文简称工作组),来实施这个数量超过一百万的志愿者受试人群队列招募及相关实验研究。
得益于美国一直领先的基因组研究、数据采集与储存、计算机分析、移动医疗应用等技术,才使得招募这一庞大队列及其研究成为可能。基于该工作组所鉴定出的许多具有价值的科学机会以及可采用的病例,可为精准医疗队列的招募提供许多有价值的信息。这些有用的信息包括,但不限于,1)通过整合环境暴露、遗传因素、基因与环境相互作用等信息对众多类型疾病的风险进行量化评估的开发;对常用治疗方案的有效性和安全性对于不同个体的差异进行验证;2)对鉴别人类常见疾病的发生发展具有风险增加或降低的生物标志物的发现;3)采用移动医疗技术将人类的活动、生理参数以及环境暴露等因素与其健康情况相关联;4)对杂合子缺失所引起功能性突变对于健康状况的影响进行确定;5)进行新的疾病分类与相互关系的开发;6)为参与者提供数据和信息使其有能力改善自己的健康状况;7)以及创建能够进行靶向治疗的平台。
(一)队列的招募
研究队列的组成将超过100万名志愿者,且工作组建议该队列会随时间而不断增加。与此同时,工作组推荐所有潜在的精准医疗队列参与者都必需同意分享其健康数据、提供生物标本、并且在之后的研究中能够被随访。当然,这样的队列必须能够反映美国人群的多样性。队列的入选条件主要有两种不同的方式,第一种是允许在美国生活的任何人都可以成为志愿者,这些直接的志愿者将签署知情同意书,同意接受随访、接受精准医疗计划的基本健康体检,并提供生物样本。这类志愿者可通过电子健康记录(EHR)数据或通过医疗服务商的初步体检向精准医疗计划提供其可公开的健康数据。另一种方式是与医疗健康服务机构合作,由这些机构招募受试者,完成基本的健康体检,收集生物样本,分享电子健康记录数据。在各方面通力配合的条件下,工作组预期精准医疗计划可在4年左右招募到至少100万名受试者。
(二)入选者的参与
工作组建议采用高度互动和主动的参与方式来进行队列数据的开发,除了在队列的计划和实施过程中提供反馈与输入,入选者也应该在精准医疗计划合监管委员会中发挥作用。工作组认为建立和保持相互信任是成功完成这一重大相互合作成功的重要基础。为了确保参与者与精准医疗计划之间的互动能够长期稳定的保持下去,工作组建议由一个中心实体来负责组织和管理相关的条款和相互交流。而精准医疗计划的知情同意书还可赋予参与者能够加入其它非精准医疗计划研究项目的权利。参与者也能够设定到底有多少个人信息是可以在整个精准医疗计划过程中可以进行交换的。为了能够对参与者团体和个体结果的开发与使用进行监管,工作组建议在监管结构中应该成立包含有参与者代表的下属委员会。
(三)数据
要想对精准医疗队列进行成功的开发,就必需要有经过验证和创新的方法和技术来对数据进行采集和管理。根据科学评估,精准医疗队列需要收集并处理类型多样化的数据,这些数据将组成核心数据组,从而使得能够在指定的研究中对亚队列进行队列范围的分析与鉴定。工作组建议,从全部队列参与者获得的初始核心数据组应该被集中采集和储存。在有可能的条件下,也可以将精准医疗队列的核心数据与其它可比较的核心数据进行比对。对数据的有效规划和管理是成功实施精准医疗计划的重中之重,工作组推荐采用与人类基因组计划类似的通用的数据模型,在可能的时候对从直接志愿者进行数据采集,尽管从临床调查获得的许多数据类型并不能简单地转化成已有的标准格式,或者只能在当时创建新的标准。因此最好的办法是在数据整理的科学诉求与对全部参与者的最高值进行标准化之间进行平衡。工作组建议在可能的时候,可采采用现有的数据标准和通用数据模型,同时也需要承认对于某些新生的模态,比如传感器技术,尚未出现相对应的标准。为了确保精准医疗队列的数据得以被安全和负责任的使用,工作组推荐根据数据的敏感性进行适当的数据获得许可控制,从公众可获得的总结性数据,到基于角色的对个体水平数据的数据获取。参与者是可以获得他们自己的精准医疗队列数据的,除非他们认为这样的获取会对他们产生潜在的伤害。总的来说,数据应该以去标示形式得以获取,并在安全的计算环境中对其进行分析从而提高数据的安全性和私密性。为了便于数据的获取、标准化以及与参与者的约定,工作者推荐了“轮毂和辐条“模型””,其具有一个协调中心,能够提供对于所需要协调数据、生物样本、参与者交流、以及科学研究工作之间的单点联络。对于数据储存和获取而言,工作组推荐采用杂合数据与分析架构,其能够同时实现对于核心数据的集中化数据储存,以及在有具体需求时在网络的节点能够保留对于额外数据的联合访问。这一杂合模型能够加速对更多研究需求的处理,并允许通过现有数据的通用数据模型以非地址的形式实现详细的数据获取请求。
(四)生物样本库
生物样本的采集对于精准医疗计划的成功实施是非常重要的。工作着认为有许多类型的生物样本都是需要被采集的,包括血液、微生物样本、指甲和头发,以及其它。在全国范围内最重要的样本首选是血液。工作组推荐每一个参与者都应该提供一份新的血液样本,在符合CLIA标准的条件下进行采集和处理,从而确保所有队列中的生物样本采集是具有质量控制和可比性的。所采集的样本应该送至集中的生物样本库,其能够对样本进行收集、处理、保存、恢复以及完成生化分析,和/或运输至其它分析实验室。生物样本库应该在队列启动之前就建立完毕。
(五)相策关政
精准医疗计划的实施将会对现有与研究、数据安全及隐私、电子健康技术的获取与交互操作等相关的法律、规章、制度等造成巨大的影响。这就需要在精准医疗计划中有一系列内部框架的政策来说明,参与者的加入要求,机构委员会对于知情同意的审批,信息的隐私、误用以及安全性,如何与研究人员进行数据和样本的共享,以及如何与参与者共享数据和研究结果等等内容。为了实现精准医疗计划能够反映美国人口多样性的目标,有一些人群,例如那些对最终决定有削弱作用的个体,则需要特别注意要将其收纳到队列中所涉及的伦理、物流、以及法律等相关问题。
(六)监管
涉及精准医疗计划的政府机构必需能够有效和及时地对相关机会做出咨询和审议。精准医疗计划是庞大且复杂的,涉及众多机构、单位、参与者、研究用案例以及必需的基础建设等等,因此需要快捷和创新的方法。工作组建议队列的招募应该由具有学术权威、职业素养以及组织支持的负责人来领衔,从而提供强有力的、值得信赖和有效的领导。工作组建议成立一个由一个小型执委会以及五个小组委员会组成指导委员会,五个小组委员会分别就结果与信息的反馈、数据、资源获取、生物样本以及安全性对指导委员会和主任进行汇报。工作组还建议成立一个独立的顾问委员会对精准医疗计划进行外部监督。NIH还需要确保能够与其他的联邦机构来一起对精准医疗计划进行协调,这些机构包括医疗保险和医疗补助服务中心(Centers for Medicare & Medicaid Services)、卫生资源和服务管理局(Health Resources and Services Administration)、食品药品管理局(FDA)、国家卫生信息技术协调员办公室(Office of the National Coordinator for Health Information Technology)、退伍军人事务部(Department of Veterans Affairs)以及国防部(Department of Defense)。最终,精准医疗计划队列的负责人是NIH的主任。
即使是在美国,从总统提出设想到拿出初步可操作的方案,时间已经从2015年1月到了9月,可见要想精准医学的难度确实很大。上述内容节选自《精准医疗计划队列项目-构建21世纪医学的研究基础》[1]。
我国在精准医疗领域也同样不甘落后,早在2015年3月中国科技部举办首届“国家精准医疗战略专家会议”上就启动了中国版“精准医疗计划”。经过长期调研与反复沟通之后,国家卫计委和科技部已在2015年底将“精准医疗计划”列入国家“十三五”计划重大科技专项。我国的精准医疗计划是应用现代遗传技术、分子影响技术、生物信息技术,结合患者生活环境和临床数据,实现精准的疾病分类与诊断,实施具有个性化的预防和治疗方案。2016年3月《科技部关于发布国家重点研发计划精准医学研究等重点专项2016年度项目申报指南的通知》(简称“国家指南”)公布,拉开了精准医疗重大专项科研行动的序幕。国家指南明确指出,精准医疗将是今年优先启动的重点专项之一,并正式进入实施阶段。本年度的科研专项涵盖八大目标,包括构建百万人以上的自然人群国家大型健康队列和重大疾病专病队列,建立生物医学大数据共享平台及大规模研发生物标志物、靶标、制剂的实验和分析技术体系,建设中国人群典型疾病精准医学临床方案的示范、应用和推广体系,推动一批精准治疗药物和分子检测技术产品进入国家医保目录等。
我国的精准医疗计划目前主要分为三个层次。
1、基础层次方面,基因测序是精准医疗的基础:无论是细胞治疗还是基因治疗,首先要通过基因测序诊断病情才能设计方案。在实施精准医疗方案过程中,需要大量的细胞和分子级别的检测。基因测序工具分为测序仪和试剂,医疗器械公司可以顺势介入测序设备生产领域。
2、中等层次方面,主要涉及细胞免疫治疗:通过对免疫细胞的功能强化和缺损修复,提高免疫细胞的战斗力。这种技术治疗癌症效果好,但操作难度大,对患者身体素质要求较高,难以大面积推广。
3、最高层次方面是基因编辑:癌症本质上是人体基因变异导致的细胞分裂失控。基因剪辑就是对患者癌变细胞的变异基因进行批量改造,使之成为正常细胞。
从目前的实施情况来看,无论是美国还是中国,都还是基础层次以基因测序为主在进行精准医疗的实践。
二、基因测序
虽然人类基因组计划(HGP)已经完成,但对于重大疾病,尤其是肿瘤、糖尿病、心血管疾病方法的探索并未因为HGP的成功而得到全方位的解释。而测序技术的不断深入,使得研究人员能够比以往更快、更便捷、更高通量的对基因相关的信息进行采集、分析与解读,如今的一套基因组数据动辄数十Gigabyte,跟以往的几十Kb,上百Mb的数据完全不可同日而语。从最初的Sanger测序技术,到现在最广泛的illumina测序技术,以及PacBio的三代测序技术,不同的技术已经在各自的领域受到了不可或缺的重视与应用,并且对许多的基本的诊断与治疗提供了巨大的帮助。

(一)乳腺癌
以下以乳腺癌为例简要介绍精准医疗的应用与实际效果。
乳腺癌是妇女中的高发癌症类型。美国影星安捷丽娜朱莉在经过基因检测之后,切除了乳腺以防止其乳腺癌和卵巢癌的发生,这一消息或许是基因检测对于大众的一次洗脑[2]。也正是从那时开始,基因检测才真正让广大临床医生和患者觉得越来越重要。NGS方法可通过对含有多种肿瘤细胞组织的DNA进行深层次解读,从而判断出每一种基因突变的正确比例,并与新开发出的分析算法相结合,进一步分辨出在肿瘤内部的不同克隆中,哪些基因组异常是在肿瘤细胞中普遍存在的,并具有类似的突变率[3]。在临床实践中,转移癌的类型往往跟原发癌的类型不同,也就是存在肿瘤的异质性,从基因组角度来看的话,在转移癌中的基因组突变通常与原发癌中的类似,且还包含有一些新的突变。且对于乳腺癌的单细胞拷贝数分析也证明了在转移癌中的肿瘤细胞继承了原发癌肿瘤细胞中的拷贝数变异,并产生一些新的拷贝数变异情况[4]。随着提取分离技术的不断进步,对循环肿瘤DNA(ctDNA)的基因组分析也变得更加深入和有效,与其他常规的血液生物标志物不同,ctDNA的假阳性率要低得多,因为肿瘤相关的体细胞突变仅仅发生在癌症或者癌症前的阶段。因此现在也有很多基于NGS和CGH的检测方法来测定ctDNA,以及采用PCR的方法对已知驱动基因进行热点突变的检测[5, 6]。
精准医疗更重要的应用于对不同患者用药的选择与监测。可用于乳腺癌的靶向药物已经有很多,但最重要的仍旧是对于Her-2阳性的曲妥珠(赫赛丁),在这个药物的开发过程中,也产生了一项称为“Herceptest”的伴随诊断,其能够有效的对于过表达Her2基因的患者具有明显的疗效并延长其生存期。经过在对于贝伐(bevacizumab)和依维莫司(everolimus)的研发过程中也进行了类似的开发,但并未发现能够用于伴随诊断的适用生物标志物,因此并不能对其疗效和生存期进行预测。为此表1中列示了一些最近研发的靶向药物及其临床试验阶段,其主要针对的是ER阳性、HER2阴性的乳腺癌患者。
在分子影响方面,Kurihara等研究人员也报道了采用正电子发射断层扫描(PET)来对体内生物标志物和药物作用效果进行评价的结果[7]。采用18F-氟脱氧葡萄糖(FDG),能够使得临床医生通过PET观察到患者体内的肿瘤位置,而采用一些特殊的示踪剂,还有可能够观察到某些药物分子、细胞以及生物学过程的表达和活性,从而获知这些改变时候能够对肿瘤有所影响,以及其对于这些治疗药物的反应情况。表2列示了一些目前可用于乳腺癌分子影像技术的一些示踪剂和靶点。
如图1所示为典型的Her-2阳性乳腺癌患者采用64Cu-DOTA-曲妥珠造影的结果。
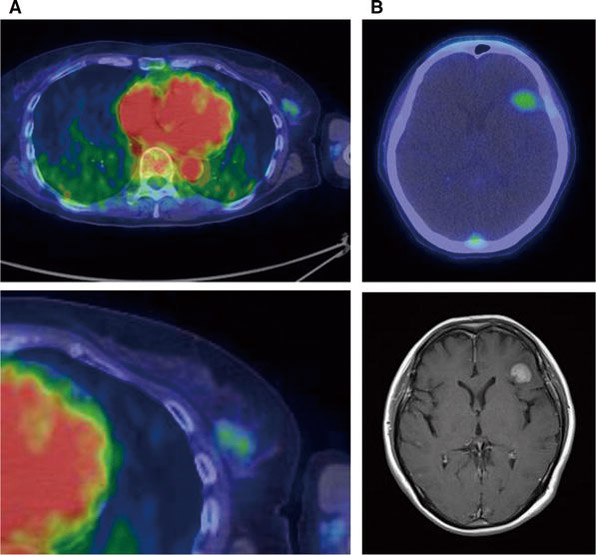
图1、a. 采用64Cu-DOTA-曲妥珠造影,可在患者的全身PET/CT看到Her-2阳性乳腺癌的原委损伤,与正常乳腺组织不同,可在肿瘤组织中发现更高的摄入。左上为轴向PET/CT融合影像,坐下为放大的融合影响(4x);b. 在Her-2阳性乳腺癌患者发现的转移性脑损伤(右上),这一高摄入也在MRI中得以确认(右下)。
除了影像学的辅助,当前对乳腺癌研究的热点也集中在其基因诊断与复发预测领域。因为通常很难采用常规的组织学检测来判断患者在接受化疗之后是否会复发。而采用多基因表达分析的方法却能够在这方面有所突破。表3为目前常见的多基因分析预测乳腺癌复发的检测[28]。
可以看出,虽然许多的资金和人员已经被投入到这样的研发过程中,但到目前为止,FDA批准的多基因检测乳腺癌复发预测项目也仅仅只有3项。在2016年8月新英格兰医学杂志的一篇报道中,研究人员招募了6693名早期乳腺癌女性患者,采用MammaPrint®对她们的基因组风险和临床风险进行了检测。具有低临床和基因组风险的患者没有接受化疗,而那些具有高临床和基因组风险的患者则接受化疗。而对于这两者风险不一致的患者,取风险较高的来决定是否采用化疗。这一研究的主要目的是评估,是否在未接受化疗、具有高风险临床特征和低风险基因表达谱的患者中,在95%的低致信区间范围内,其在没有远端转移条件下的5年生存率是否能够达到92%或者更高。结果表明,总共有1550名患者(23.2%)被认为是具有高临床风险和低基因组风险的。在第5年时,在那些没有接受化疗的患者中,未发生远端转移的生存率为94.7%。比接受化疗的生存率低1.5%。类似的情况也发生在ER阳性、Her-2阴性的患者人群中。由此可知,大约有46%的具有高临床风险的乳腺癌患者并不需要接受化疗(NCT00433589)[30]。
以上研究结果说明这些多基因检测确实能够为患者提供更适合、更准确的治疗手段。
(二)直肠癌
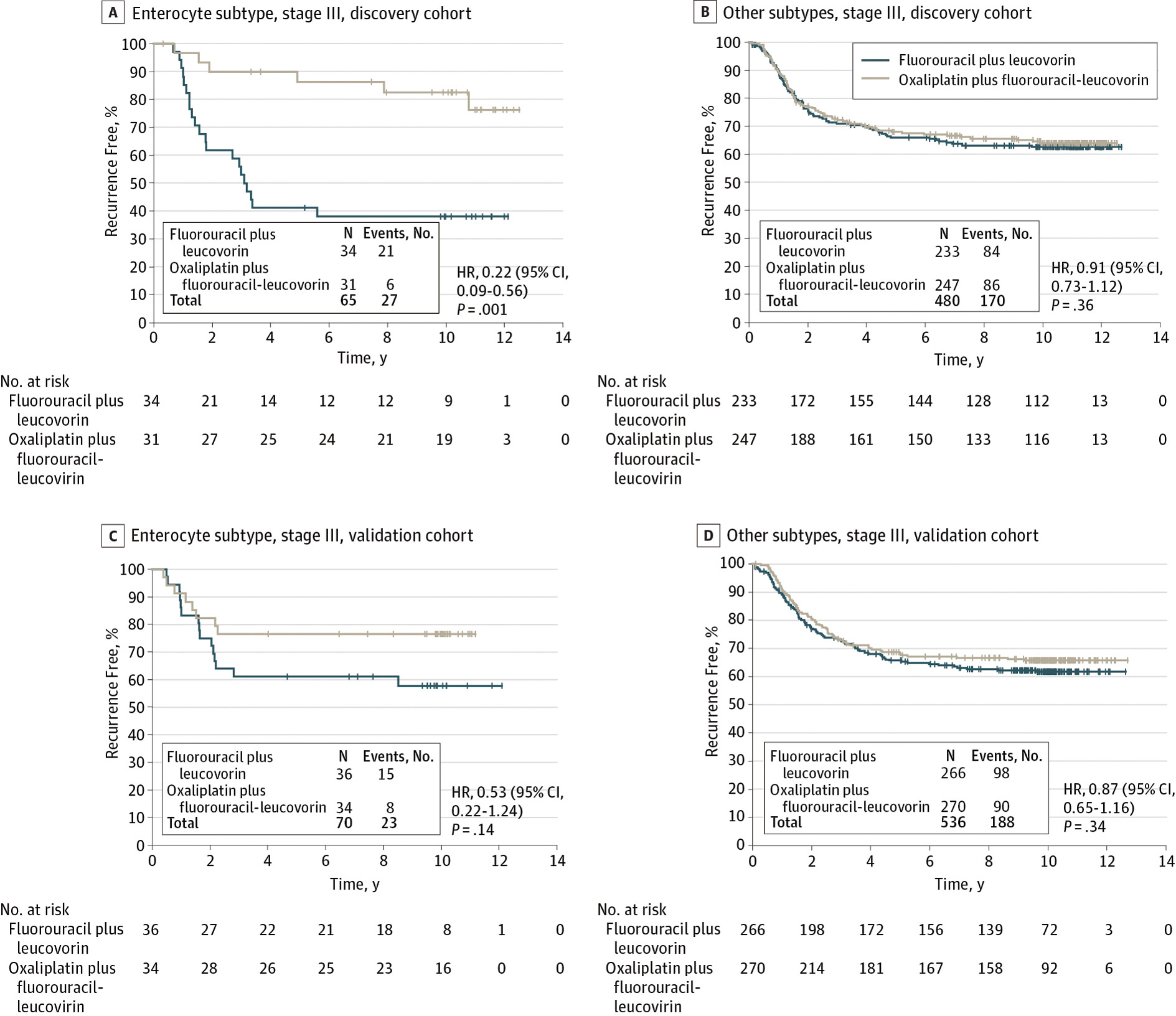
类似的情况也同样春现在直肠癌患者的人群中。根据现有的治疗指南,直肠癌患者是否需要进行辅助性化疗取决于其TNM分级。三级的患者首选考虑氟嘧啶和奥沙利铂治疗;二级患者则采用氟嘧啶单独给药或手术后不给药[38]。在2016年9月的JAMA oncology杂志中,来自于宾夕法尼亚NRG肿瘤中心的人员发表了采用295个基因以及6个阳性和8个阴性对照基因对患者人群进行检测,最终确定氟尿嘧啶、奥利沙伯和亚叶酸给药方式的研究报告。该研究指出,在2370名签署了知情同意的入组患者中,有1768名能够提供适合的肿瘤标本,另外39名患者则无法提供足够的RNA。剩余的1729名患者被分为发现组(848人)和验证组(881人)。通过对72个基因的表达情况进行分析,结果表明,在发现组中,具有肠细胞亚型的三级患者都表现出了对接受奥利沙伯给药的统计学显著性获益,而在验证组中,并未有此统计显著性获益。而类干细胞亚型则不能从奥利沙伯给药获益且预后很差。
从三级患者的无复发生存时间Kaplan-Meier图可以看出,在A和B图中,发现组中的肠细胞亚型能够在给药奥利沙伯后明显获益,而其他4个亚型的组合则没有明显表现。在C和D图中,验证组中的肠细胞亚型未能从给药奥利沙伯后明显获益,其他4个亚型的组合也是如此。(图2)
(三)肾细胞癌
肾细胞癌在世界范围内有逐渐升高的趋势,每年大约有超过300,000的新增病例和超过140,000的死亡病例,在全部成年人癌症中大约占比2.4%,在肾癌中约占90%。肾细胞癌起源于肾小管,也是具有异质性的癌症类型[39]。透明细胞肾细胞癌(clear cell renal cell carcinoma, ccRCC)约占所有病例的70-75%,乳头状肾细胞癌(papillary renal cell carcinoma, pRCC)约占10-15%。由于存在不同的预后和组织学差异,乳头状肾细胞癌又可分为I型和II型。还有一类是约占5%左右的色素肾细胞癌(chromophobe renal cell carcinoma, chRCC)[40]。采用NGS和GWAS方法可对肾细胞癌的遗传情况进行深入的了解。表4为已经验证过的对肾细胞癌具有倾向性的种系突变体。
同时也获知了在肾细胞癌中能够引起复发的体细胞异常以及治疗靶点。(表5)
这些信息分子水平的特征可以很好的对肾癌进行分类,并对了解其机理具有重要作用。所得的基因组学数据也可以为肾癌的诊断和治疗提供更适合的指导和候选,从而推动精准医疗的应用。
(四)糖尿病
糖尿病一直都是一项难以根治的代谢性疾病,虽然可以通过减肥手术来使得肥胖病人控制血糖,但并不是所有患者都能获益。采用大规模测序的方法可以了解是否哪些基因是能够对糖尿病的异常调控有功能的,从而能够判断是哪些患者是适合接受减肥手术的。最近刚有研究人员采用illumina的CardioMetaboChip对II型糖尿病患者进行了检测获得其遗传信息。表6为对手术诱导糖尿病缓解多基线参数的单变量分析。
通过关联其临床性状以及蛋白-蛋白相互作用,对所得SNP进行了关联分析,鉴定出8个糖尿病相关的基因,ABCA, ARHGEF12,CTNNBL1,GLI3,PROK2,RYBP,SMUG1 和STXBP5,其中三个基因是已知的,与胰岛素分泌、胰岛素敏感性和肥胖相关,但尚未了解其是否与减肥手术后的糖尿病恢复相关[45]。见下图。
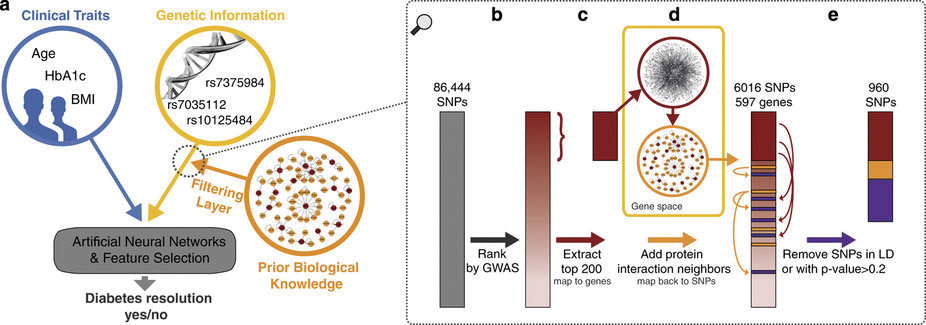
图3、b-e说明在采用生物学知识之前所获得的候选SNP数量,最终采用蛋白-蛋白相互作用数据,从GWAS SNP中获得了得分最高的200个。
这种结合测序与机器学习的方法将在精准医疗中具有越来越重要的地位[46, 47]。随着测序技术尤其是下一代测序技术(Next Generation Sequencing,NGS)技术的大量普及,单纯的科学研究成果已经不能够满足研究人员以及医疗行业从业人员的需求了,大家更希望这样的测序技术能够应用到临床领域,能够成为一项实实在在的检测项目,能够更好地服务于患者、同时也能够实现这项检测技术的商业价值。而要成为一项检测技术,就需要得到相关监管机构的认可。
2016年7月6日,FDA官网发布消息称,为支持总统先生提出的“精准医疗计划”,FDA于今日发布2份指南草案,当最终定稿之后,旨在为检测具有重要医学意义的患者基因组成份的测试实施灵活和高效的监管。
NGS能够扫描患者的DNA序列,从而检测到基因组变异,而这些变异有可能决定是否这名患者已经具有或者会具有罹患某项疾病的风险,并且为治疗决定提供信息帮助。鉴于现有的规范适合于对常规检测进行监管,而这些常规检测通常只需要测定与疾病或不适相关的少数几种物质,例如血糖或胆固醇水平;而新的测序技术却能够在同一时间测定数百万计的DNA变异体,因此有必要针对于这些新技术所进行的测试出台一些灵活的监管规范。
本次给出2份征求意见指南草案,第一份是《FDA对基于NGS用于诊断种系疾病的体外诊断(IVD)监管标准的使用 》(Use of Standards in FDA’s Regulatory Oversight of Next Generation Sequencing (NGS)-Based In Vitro Diagnostics (IVDs) Used for Diagnosing Germline Diseases),该草案对基于NGS技术检测罕见遗传疾病的设计、开发和验证提出了推荐,并强调了采用FDA认可的标准来给出分析性验证的重要性,即要说明某个检测在预测某个特定的遗传改变的存在与否时的表现是如何之好[48]。
第二份草案是《使用公共人类遗传变异数据库来支持基于NGS体外诊断(IVD)的临床验证》(Use of Public Human Genetic Variant Databases to Support Clinical Validity for Next Generation Sequencing (NGS)-Based In Vitro Diagnostics),该草案提供了一种方法,开发人员可采用该方法,根据从FDA认可的公共基因组数据库中所得到的临床证据来支持其检测结果的临床诉求,并且提供对于所述基因组检测结果具有正确临床解释的保证,这确实是一种更方面获得批准和同意的方法[49]。
FDA分管CDRH(Center for Devices and Radiological Health)的负责人MD. & JD. Jeffrey Shuren在面对媒体时指出,精准医疗只有在检测能够指导诊断和治疗时才真正有效,FDA的职责就是确保医生和患者能够信赖这些检测的准确性、可靠性和临床有效性。
NIH负责人Dr. Francis Collins也随即指出,这些草案是开发NGS检测的重要步骤,NIH非常高兴看到这些草案的公开,并且鼓励研发人员能够根据草案中提出的要求来进行开发,从而能够为那些真正需要的患者提供高质量的检测。
根据以往的惯例,FDA为公众提供了90天的评议期以对上述草案进行充分的讨论、探讨、以及质疑。相信很快就能看到FDA对基于NGS检测的最终监管文件。
而对于NGS的数据而言,如何将其庞大的磁盘占有变得更加高效一直都是研发人员的努力目标。最近,来自于加拿大英属哥伦比亚西蒙弗雷泽大学和英国惠康Sanger研究所的科研人员对高通量测序数据进行了研究,采用不同的压缩方式,使其文件大小能够明显降低,从而有助于更方便和快捷的存储以及计算,相关研究结果发表在Nature Method杂志[50]。
三、免疫治疗
除了靶向药物治疗和常规放化疗治疗等手段之外,免疫治疗一直都是癌症治疗领域的研究热点。
与基于高通量测序的靶向治疗相类似,肿瘤的免疫治疗也有几种主要的策略,疫苗,适应性细胞治疗、溶瘤病毒、单克隆抗体等[51, 52]。与免疫治疗相关的主要因素很多,主要包括诱导可持续临床应答的潜能如何,对常见药物抗药性的缺失,以及对自身免疫类似毒性物质的诱导等等。临床医生需要对这些问题都有详细的了解,并且对各种肿瘤免疫靶向治疗的临床应用烂熟于心,才能为患者提供更高质量的癌症治疗与关怀[53]。
肿瘤免疫治疗是基于免疫系统能够识别肿瘤相关的抗原,并能够介导针对于肿瘤细胞的高特异性细胞溶解效应。这个过程是在太过于复杂以至于到现在也没有能够完全理解。其实,早在1980年,就已经有若干的研究团队验证出了一些能够通过识别主要组织相容性复合体(MHC)从而被T淋巴细胞所识别的肿瘤抗原,在某些情况下,这些抗原可以使病毒蛋白,突变的去阻遏胚胎抗原,或者过表达的分化或者正常自身蛋白。已有研究揭示了肿瘤抗原是如何启动免疫应答的[54],在这样的循环中,抗原可通过一系列机理从肿瘤细胞中得以释放,包括,细胞内激酶、肿瘤细胞的先天性坏死、或者通过采用放疗、细胞毒性化疗、或者靶向治疗。除了抗原,濒死的细胞也可以释放诸多被称为危险相关分子模式(DAMPs)的因子,其是在细胞应激、缺氧、营养耗竭或者创伤时产生的。这些DAMPs分子可以结合细胞表面和细胞内的受体,包括类Toll受体,引发初级免疫应答[55]。而且,濒死肿瘤细胞和可溶性抗原还可以被专门的抗原提呈细胞,例如树突状细胞在肿瘤微环境中吞噬[56]。
要想获得较高的治疗效果,就需要使得未经处理的肿瘤细胞能够便显出免疫抑制或免疫逃避性质,从而防止宿主的免疫系统能够识别其肿瘤相关的抗原和其他危险信号[57, 58]。籍此,免疫治疗就能够提高对于肿瘤细胞的识别并诱导其免疫介导的破坏[58]。这些常见免疫治疗的机理如图4所示。
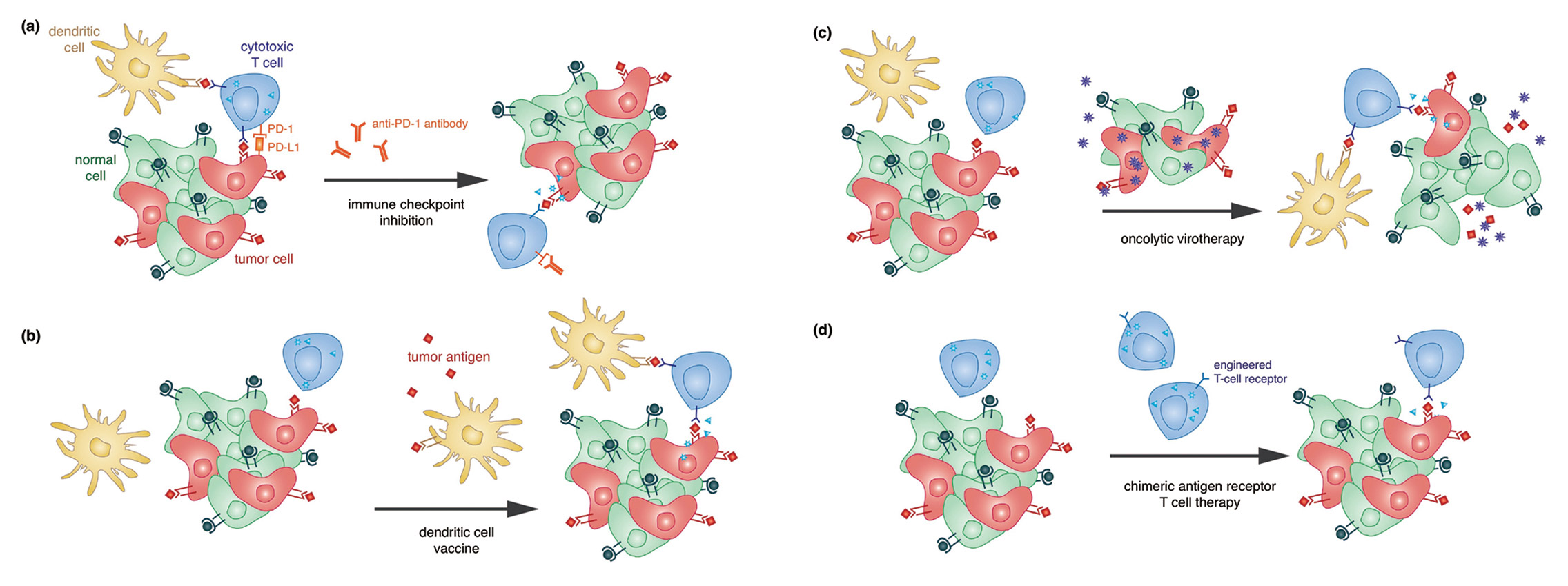
图4、常见免疫治疗的机理。(a)抗PD-1抗体与PD-L1在肿瘤微环境中的结合能够缓解该环境中的免疫抑制肿瘤信号,从而再次激活在免疫检查点阻断过程中的T细胞介导的抗肿瘤应答。(b)在体内或离体条件下将树突状细胞暴露于抗原提呈过程,可导致树突状细胞将这些多肽提呈至患者的T细胞,从而产生抗肿瘤应答。(c)在恶性细胞中选择性的复制溶瘤病毒可以导致一定程度上的肿瘤直接溶解,以及额外的通过招募免疫细胞而介导的抗肿瘤作用。(d)表达靶向肿瘤细胞受体的离体培养的工程化患者T细胞,在回输至患者体内时能够产生抗肿瘤细胞毒性作用。
肿瘤疫苗可通过刺激树突状细胞提呈肿瘤特异性抗原来实现,这些树突状细胞随后与宿主的淋巴细胞发生反应,从而介导细胞毒性作用[59]。第一个FDA批准的治疗性癌症疫苗-Sipuleucel-T(Provenge),就是通过将患者来源的树突状细胞进行离体培养,来提呈含有前列腺酸性磷酸酶(PAP)以及粒细胞巨噬细胞集落刺激因子(GM-CSF)的融合蛋白来实现的[52, 60, 61]。
适应性细胞治疗的工作原理引起了诸多主要公司的极大兴趣[62]。一旦能够准确地了解在肿瘤或者自身细胞上的靶向抗原,就可以把握T细胞受体(TCR)的信号转导通路,而遗传工程化修饰的细胞也就能够产生出治疗性T细胞,从而提高对于癌症的精准治疗[63]。这些工程化的TCRs或者嵌合抗原受体(CARs)能够引导杀伤性T细胞识别毒感染的细胞或特异的肿瘤细胞类群;而修饰的T细胞就在不引起大规模其他伤害的情况下破坏这些靶细胞。这样的治疗能够在采用某些生物或小分子治疗无效的血液癌症患者中完全缓解其肿瘤的发生与发展[64]。
在这些方法中,嵌合抗原受体(CAR)T细胞治疗在临床应用中表现出了相当的有效性。CD19是在正常B细胞和许多恶性B细胞上同时表达的B细胞特异性抗原。以CD19为靶向的第二代CART细胞治疗已经在学多临床试验中表现出了相当好的结果,在复发和不应期急性淋巴细胞白血病(ALL),慢性淋巴细胞白血病(CLL),滤泡性淋巴瘤(FL),弥散性大B细胞淋巴瘤(DLBCL),套细胞淋巴瘤(MCL)或多发性骨髓瘤(MM)中的全部缓解率可达到70-90%[65-70]。
有效的肿瘤免疫治疗策略可针对于促进癌症免疫循环并且封闭已有肿瘤的免疫抑制作用。具体的方法可包括增强树突状细胞成熟和T-细胞分化的细胞因子,能够防止T细胞无应答的检验点抑制剂,类Toll受体激动剂,能够诱导坏死肿瘤细胞死亡的溶瘤病毒,释放DAMPs,引发系统性抗肿瘤免疫,以及自然和设计为识别特异性肿瘤抗原的遗传工程化T细胞的适应性转移。
另外一种目前最常见的方法是采用靶向于免疫检查点的抗体,这样的抗体能够通过激活抗肿瘤的细胞毒性T淋巴细胞(CTLs)来杀死肿瘤细胞。抗CTL关联的抗原4(CTLA4)抗体是FDA批准的第一类免疫治疗药物抗体,可用于治疗黑素瘤并在进行其他类型肿瘤的临床试验。抗程序性细胞死亡蛋白-1及其配体(PD-1/PD-L1)抗体则能够阻断介导PD1与PD-L1结合的免疫抑制信号,从而成为当前最热门的抗肿瘤药物[71]。
PD-L1是在新千年的时候,由Dana-Faber癌症研究所的科研人员在正常细胞中发现的,他们发现PD-L1可通过结合T细胞共受体PD-1来发挥对T细胞的抑制作用,从而使得T细胞不去引起免疫系统的攻击[72]。不久之后,同一批科研人员又发现PD-L1不仅仅在正常细胞上存在,在某些特定的癌症细胞上也存在,于是他们就考虑是否可以采用某些PD-L1(或PD-L2)的抑制剂来启动免疫系统对于肿瘤细胞的攻击[73, 74]。
有多种实体瘤包括黑素瘤,RCC,NSCLC,甲状腺癌,卵巢癌和直肠癌等肿瘤类型都可以通过表达PD-L1来加入免疫防御从而产生免疫抑制性的肿瘤微环境同时避免T细胞溶解反应。肿瘤PD-L1过表达则会在多种肿瘤组织学中带来了较差的预后,使得这样的治疗性干预在免疫调节轴上具有很好的预期[75, 76]。
就像针对于Her2过表的曲妥珠一样,也有很多的研究是针对于Pd-L1的表达情况进行的,采用免疫组织化学的方法,对人类肿瘤标本中表达肿瘤PD-L1的研究结果如表7所示,根据不同的参数,过量表达PD-L1的肿瘤样本有多种,其表达情况也差异较大[77]。
因此可以看出,在不同类型组织中PD-L1的不同表达情况说明其并不是能够决定PD-1/PD-L1直接抑制的唯一因素[78]。为此,已经开发了一些对pd-1/PD-L1进行伴随诊断的试剂盒,如表8所示[77]。
作为一种负反馈调节系统,PD-1途径如果调控失当的话,也会对肌体造成很大伤害[79]。所以上述免疫组织化学的方法,也许并不是唯一能够测定PD-L1在细胞表面表达水平的方法。有研究人员推测在某些病人中有可能存在错配缺失的情况,因为在肿瘤中发现的体细胞突变有可能被患者自身的免疫系统所识别[80],而错配缺失的患者则有可能出现比正常体细胞突变高出10-100倍的可能性[81],而且,配对缺失的患者还含有明显的淋巴细胞侵润,这也和免疫反应的结果相一致[82]。而采用微卫星位点[83]检测也是另外一种方法来评估患者的PD-L1表达水平[84]。微卫星也称为具有相同碱基或序列的“短末端重复”(通常为10-60次),它们散在于基因组的编码区和非编码区,DNA聚合酶在这些区域非常容易出错,从而产生错配的DNA链,进而导致PD-L1表达水平的改变。无论是遗传性的还是随机性的,微卫星不稳定的癌症患者都会具有一些特殊的临床病例相关性,例如镜检时通常有肿瘤侵润的淋巴细胞,边界不清楚以及粘膜性分化;而微卫星稳定的患者则镜检时通常在侵润腺体中有坏死[85]。在2015年的NCCN指南中,也要求将具有5%或更高具有种系突变的患者进行微卫星检测,从而能够对适当人群进行用药前检测[84]。
但是我们也需要注意到,并不是每种癌症的临床试验都表现出对PD-1和PD-L1的利好消息,我们还需要进一步了解为什么在不同的癌症中抗PD-1/PD-L1抗体的效果差别是如此之大?还有哪些生物标志物能够被用于检测和诊断哪些患者适用于PD-1/PD-L1的药物?如何能够将抗PD-1/PD-L1抗体整合到现有的治疗方案中,尤其是在那些复发的病例里?是否还会存在其他表达PD-1的非T免疫细胞能够在抗PD-1/PD-L1治疗中发挥作用?
四、基因编辑
从2007年的诺贝尔奖授予了Capecchi、Evans和Smithies等三位科学家对于小鼠基因操作的工作之后,基因打靶/敲除就在学术界刮起了一阵旋风,似乎所有需要研究基因功能的试验都可以采用基因敲除技术了,基因敲除的小鼠更是供不应求,虽然其价格一直高高在上,但通过人工的方法能够实现预知基因的纯合子或杂合子,毕竟是可以说明很多生物学现象的。 当时采用的技术手段主要是DNA同源重组技术,其单位点的重组成功率一般在10-6~10-9之间。所以说,要想获得一个单位点得到修饰的小鼠,至少需要在10-6个胚胎干细胞中成功实现同源重组,然后再将这个干细胞,植入到小鼠的扩张囊胚中,最终才能获得嵌合体的基因敲除小鼠。要想获得种系稳定遗传的小鼠,还需要从若干嵌合体中进行筛选,工作量巨大,因此成本高也就是合情合理的了。但是这样的技术手段通常会存在低频率的自发同源重组,从而导致要想达到预期的同源重组并非一帆风顺,而要想解决这一难题,现在可以采用更新的基因组编辑技术了[85]。
HIV/AIDS一直都处在基因治疗和细胞治疗的前沿。常规的基因治疗方法通常都会涉及采用半随机整合病毒载体的方法向细胞中加入抗-HIV基因。而基于工程化核酸酶的新型基因组编辑技术则可以实现更精准的基因操作。基因组编辑可能出现的结果包括基因破坏、其最有可能被应用于CCR5共受体基因,或者是在靶向位点引入小的突变或者更大的整个基因读框的插入。使用锌指核酸酶来破坏CCR5是第一个在人体中进行的基因组编辑试验,并且保持的最高级别的临床条件,在T细胞核造血干细胞/祖细胞中,有7个已经完成和正在进行的临床试验[86]。
众所周知,HIV感染人体后,该病毒会将其本身的拷贝插入到细胞中,变为细胞的一部分遗传物质,在这样的情况下,整合病毒的表达是受抑制的,这就导致了病毒成为了潜伏状态,不会受到抗病毒药物治疗的影响。这些病毒将在体内保持相当长时间,甚至是终生。而一旦这些潜伏的病毒重新活化,且抗病毒药物治疗停止,这些病毒将会在很短时间内爆发。因此,HIV管理需要终生进行抗病毒药物治疗,却不能治愈患者[87]。尤其是在病毒感染的急性期,CD4+ T细胞的破坏将是不可逆的,许多患者终身不能在恢复到正常的CD4+ T细胞水平,及时抗病毒药物治疗能够有效地对病毒进行控制[88]。因此直到现在,绝大多数针对HIV的基因治疗方法还是在通过加入抗-HIV基因来保护CD4+T细胞免受感染。通过降低易感细胞的数量,这样的策略期望能够降低总病毒血症,而工程化细胞的选择性存活也能够增加随时间而上升的治疗效果。此外,直接保护CD4+T细胞能够重建免疫功能,使得机体能够还击感染的症状和病毒本身。
已经有几种抗-HIV基因以这样的方式进行了临床试验了,包括显性阴性病毒蛋白(Rev M10)和多肽(C46),病毒RNA诱饵(TAR和RRE),基于RNA方法采用核酶抑制宿主或病毒基因,反义RNA,以及RNA干扰技术[89-92]。这些技术都已用于临床实现,并创建了基因治疗的开始,但却都不能具有很高的效率。
近来的基因组编辑技术能够对此有所改进,从技术上来说,采用工程化的核酸酶在靶点座位引入DNA双链断裂,其随后的修复将导致不同的结果。如果是实现了非同源重组末端连接配对的话,其在断裂位点可以是随机的插入或者缺失,从而导致基因破坏。也有可能是,基于同源重组的更精准的配对途径,其能够拦截来自于导入的DNA同源模板,并拷贝其信息。这样的同源指导的修复能够促进在断裂位点的特定基因编辑/突变事件,或者允许位点特异性的加入较大的基因序列。可以使用不同的工程化核酸酶平台来产生最初的双链断裂,这些酶包括归巢内切核酸酶,锌指核酸酶(ZFNs),类转录激活子效应核酸酶(TALENs),以及成簇规律间隔短回文重复(CRISPR)/ Cas9 [93, 94]。
CCR5是大多数HIV毒株入侵时候使用的共受体,因此将其破坏是实现基因治疗的最简单选择[95]。CCR5△32等位基因含有一个32bp的缺失,其导致了一个不能在细胞表面表达的钝端蛋白。在纯合子人群中,这个等位基因可以在不引起严重健康后果的条件下抵抗HIV-1感染,并在HIV-1感染的杂合子人群中,其也能够延缓基本的进程[96]。然而,仅有一例成功案例使得这样方法并不能应用于更广泛的人群之中[97]。这样简单而单纯的失活一个基因只是个起点。
基于在T细胞的适应性转移经验,进行了第一例采用ZFNs来进行基因组工程化CCR5-阴性细胞的临床试验。经过验证的ZFN配对被引入CCR5-开放读框大约160bp的双键断裂位点,对应于该蛋白7个跨膜蛋白结构域中的第一个的位置[98]。这种方法最常见的处理结果是产生一个5bp的核苷酸插入,其大约能够占到全部修饰等位基因中的25%。这个5bp重复可引入2个框内终止子,从而封闭表达并可作为遗传标记来评估CCR5修饰的总体效率[99]。
第一个治疗HIV的人体基因组编辑临床试验实在2009年开始的(#NCT00842634),主要是为了评价修饰后同源CD4+T细胞在HIV-1感染人群中的安全性。除了安全性之外,随后的临床试验还进一步考虑了各种优化条件,如表9所示。
CCR5是大多数HIV毒株入侵时候使用的共受体,因此将其破坏是实现基因治疗的最简单选择[95]。CCR5△32等位基因含有一个32bp的缺失,其导致了一个不能在细胞表面表达的钝端蛋白。在纯合子人群中,这个等位基因可以在不引起严重健康后果的条件下抵抗HIV-1感染,并在HIV-1感染的杂合子人群中,其也能够延缓基本的进程[96]。然而,仅有一例成功案例使得这样方法并不能应用于更广泛的人群之中[97]。这样简单而单纯的失活一个基因只是个起点。
基于在T细胞的适应性转移经验,进行了第一例采用ZFNs来进行基因组工程化CCR5-阴性细胞的临床试验。经过验证的ZFN配对被引入CCR5-开放读框大约160bp的双键断裂位点,对应于该蛋白7个跨膜蛋白结构域中的第一个的位置[98]。这种方法最常见的处理结果是产生一个5bp的核苷酸插入,其大约能够占到全部修饰等位基因中的25%。这个5bp重复可引入2个框内终止子,从而封闭表达并可作为遗传标记来评估CCR5修饰的总体效率[99]。
第一个治疗HIV的人体基因组编辑临床试验实在2009年开始的(#NCT00842634),主要是为了评价修饰后同源CD4+T细胞在HIV-1感染人群中的安全性。除了安全性之外,随后的临床试验还进一步考虑了各种优化条件,如表9所示。
采用工程化的造血干细胞/祖细胞来替代CD4+T细胞能够提供更长的持续期,并对HIV-1敏感的CD4+T骨髓瘤细胞有更好的保护。这一方法也在小鼠中得到了很好的证明[100]。
除了ZFNs的方法,还有其他的方法也被开发用于T细胞和造血干细胞/祖细胞中CCR5的编辑,这些方法还包括TALENs[101],megaTALs[102],以及CRISPR / Cas9[103, 104]。CRISPR / Cas9的一个最大优势就是由许多的指导RNA能够被同时应用,从而激发出范围广大的基因破坏,从而诱导出比通过非同源末端产生的缺失更特异性的缺失[103]。然而,这样的多组分系统(需要核酸酶和指导RNA)会在临床开发过程中引入复杂性,而且向造血干细胞/祖细胞传送CRISPR/cas9的适合方法也在开发过程中[105]。可以预见,在今后还会有更多更适合的基因编辑方式来治疗HIV / AIDS。
今年6月21日,美国国立卫生研究院(NIH)顾问委员会批准了一项利用CRISPR-Cas9来帮助增强依赖于患者T细胞(一种免疫细胞)的癌症疗法实验项目。对于癌症而言,细胞疗法的前景良好,但绝大多数接受这种疗法的人罹患的都是会复发的病症。费城宾夕法尼亚大学生理学家、该项研究的负责人Edward Stadtmaue教授认为,基因编辑能够改善这类治疗方法,并弱化其对于癌症以及身体免疫系统的易受攻击性。首例临床试验规模很小,旨在测试CRISPR是否可在人体中安全使用,而不是它是否能有效地治疗癌症。该试验本身并没有预算,而是得到了今年4月由脸书网(Facebook)前总裁Sean Parker建立的2.5亿美元免疫治疗基金的支持。宾夕法尼亚大学将制作基因编辑细胞,并招募及治疗加利福尼亚州和得克萨斯州各中心地区的患者。
研究人员将从18名罹患各种癌症的患者体内取出T细胞,对它们进行3种CRISPR基因编辑。其中一种方式是插入一段基因形成工程化的蛋白,从而侦测到癌症细胞并指导T细胞靶向它们。第二种方式是去除可能干扰这一过程的自然T细胞蛋白。第三种方式是防御性的,将会去除识别T细胞的蛋白基因,阻止癌细胞让其丧失功能。接下来,研究人员会把这些经过编辑的细胞回输至患者体内[106]。
我国研究人员在这一领域也走在了世界领先行列。四川大学华西医院癌症学家卢铀研究团队计划开展临床试验,将CRISPR-Cas9用于肺癌患者的治疗,试验预计在2016年下半年开始,该临床试验已经在7月6日获得医院的伦理委员会批准。华西医院的试验将会招募恶性非小细胞肺癌患者,他们都是放化疗等常规治疗已经无效的患者。研究小组将从患者体内采集T淋巴细胞,然后用CRISPR-Cas9技术对这些细胞进行基因组编辑,灭活他们的PD-1基因,PD-1基因的表达让这些细胞失去攻击能力,关闭这种基因则是让这些细胞恢复识别和杀死癌细胞的作用。经过基因编辑关闭PD-1基因的工程化T淋巴细胞经过培养扩增,然后回输给患者。这些工程化T淋巴细胞返回到癌症组织从而实现攻击癌细胞的目的[107]。
就在上个月,卢铀团队首次将利用CRISPR-Cas9进行过基因编辑的细胞注射到了一名病人体内。截止到目前为止,这名病人表现得 “还不错”。这名病人已被安排第二次接受利用CRISPR-Cas9进行过基因编辑的免疫细胞注射,但是未给出具体的时间。这名病人与该项临床试验的9名其他的志愿者将被密切监控6个月,从而来确定这些经过基因编辑的细胞是否会导致任何不良影响。本刊也将对此继续关注和报道[108]。
综上,我们从高通量测序、免疫治疗和基因编辑三个方面对精准医疗进行了一点点粗浅的描述,希望能够抛砖引玉,让各位读者能够获得一些最新的信息及思路,从而在各自的工作组进一步激发想象力和创造力,争取将精准医疗早日应用的实际工作中去。
五、精准医疗的不同声音
(一)精准医疗的赞同之声
1、癌症的精准治疗
癌症的本质是多基因的遗传疾病,随着肿瘤发展,癌症细胞仍然在不断地进行分裂和增殖积累突变,有高度的异质性,其基因组具有不稳定性。近年来我们意识到癌症的分类按照简单的组织部位是不够的,药物的单一治疗效果也不理想(有效率仅有20%)。比起传统的病理报告,基因组测序信息能提供更加精准有效的分型诊断。要理解癌症,我们首先要识别那些导致癌症风险的异常基因和蛋白质,才能更好地进行精确的癌症诊断和开发针对性疗法。目前NCI开展多项临床研究,目标是使癌症的分子表征成为准确的诊断和治疗的临床标准;鉴定和发展可匹配至肿瘤分子特点的疗法,成功地控制疾病。同时还开展基因组学和癌症生物学,免疫学和免疫治疗,癌症成像等方面的研究来配合。
随着人口老龄化的进程加快,癌症已是全球地区主要的死亡原因。研究人员已发现,许多分子病变是驱动癌症的诱因,这表明每种癌症都有自己的基因印记、肿瘤标记物以及不同的变异类型。虽然癌症主要是由日常生活中基因损伤积累所导致的,但可遗传性基因变异通常会增加患癌风险。这种对致癌机制的新理解已影响了在药物和抗体设计过程中,对癌症风险、分类诊断以及治疗策略的评估。许多靶向疗法已经或正在研发,它们中有些已为民众带来益处,有些则效果显着。此外,最新的癌症免疫疗法也产生了一些积极的反响,有迹象表明肿瘤标记物能成为预测癌症的显着因子[109]。
这些研究让我们努力提高癌症预测、预防、诊断和治疗的紧迫和前瞻性。然而,实现这一承诺则需要我们在诸多方面付出努力。大量地分析癌症基因组数据,有助于我们对癌症的深入理解以及发现分子诊断的其他工具。为加快在成人和儿童患者中采用新疗法,我们也需要探索更多新颖的方法来设计临床试验。
精准医疗将加大科研进步,从而提升对疾病风险评估、疾病机制把握以及许多疾病最佳治疗方案的预测,这对扩大精准医疗在健康和卫生保健等诸多领域带来益处。
2、精准医疗可以改善国民健康
精准医疗可以改善国民健康。第一,通过实施精准医疗对个人和公共健康的护理和疾病预防进行补充,从而改善国民健康水平。第二,遗传靶向方法确实能让国民健康受益。第三,最重要的一点,精准医疗不能限制于基因、药物和疾病。技术的进步和大数据能让精准医疗迈向精准公共医疗的新时代,跨越个人个体化医疗的时代。通过使用新方法和新技术检测疾病、病原体,改善国民生活习惯和易感性,实施新政策和针对性程序改善健康水平,精准医疗可提高国民疾病预防能力,提高健康水平,缩小健康差距。精准公共健康的一个潜在重要应用是利用基因组学研究和控制传染病,病原体全基因组测序在临床和公共卫生微生物学方面正在快速进步。此外基因组学有望成为其他经济部门的中心如环境、农业、动物健康、生物技术和替代能源等。
很明显我们正处于精准医疗和公共卫生发展的十字路口。有许多方法可以让精准医疗加强与医学和公共健康的协作处理公共健康问题,缩小医疗水平差距。目前精准医疗的侧重点包括为癌症和其他疾病的个体化治疗开发新型药物。更进一步,医疗专家需要更加强调精准医疗与公共健康的生物、社会和环境决定因素相结合,为个人和公共健康开发精准的疾病干预措施[110]。
3、精准医疗引发传统医疗方法的革命
通过在正确的时间提供正确的治疗,医疗卫生活动会变得更加准确和有效,同时为患者提供更高质量的护理。精准医疗目前存在的显著挑战是如何在医疗卫生行业中大规模实施,目前面临的问题有:生成的证据不足,数据分享和基础设施的不足,延缓了临床护理和研究采用基因组信息的进程,精准医疗目前的成本较高,目前还没有得到更多患者和医师的认可和信任。
积累精准医疗受益性的证据,促进医师、医疗卫生组织和患者采用精准医疗,获得监管部门批准,完善精准医疗的报销策略,建立用于评估的稳固的循证框架,获得精准医疗对预后有益的证据。加速临床证据整合和评估,加强精准医疗对人体健康和疾病复杂性的认识将有助于获得大量详细数据并将其纳入综合知识网络。促进分子指导意见纳入护理活动,在日常医疗卫生护理中使用基因组和其他信息,是将精准医疗整合到医疗卫生体系中的关键,需要考虑患者基因组序列基本信息,进行患者护理并采用适当的医疗方法。开发以创新为导向的回报和监管框架,以创造的价值为基础,回报诊断试验的开发者付出的努力,否则这会对新型试验和疗法的转化产生负面作用,合理的回报体系对开发支持精准医疗的循证和经济框架十分必要。强化公众的参与,加强公众的信任水平。公众的参与和信任是精准医疗的研究的重中之重。有效的参与是证明精准医疗对个体和团体发挥应有价值的必要条件。为了取得成功,精准医疗需要研究者、临床团体和公众的不断合作。
有效地实施这些方法需要加强个体和团体的参与[111]。信任是精准医疗研究的基础,也是加速精准医疗服务推广,获得个体和团体采用的基础。除此之外还需要利益相关者和公众、个人、学术组织、政府之间的协调与合作,改变发展媒介通过协调精准医疗政策议题,支持精准医疗研究、数据获取、整合和在医疗卫生行业中的应用,同时提高从业者和公众的教育水平。
(二)精准医疗的质疑之声
1、肿瘤精准治疗的局限性
目前肿瘤精准治疗的实际临床益处是有限的,在研究计划、临床研究、分子靶向药物、肿瘤进化和肿瘤内异质性及成本等方面存在局限性。研究资助是有限的,集中于精准医疗研究可能会剥夺相关资源用于其它有价值的研究途径(免疫治疗例外)。多种因素可能导致目前个临床评估的成果有限,包括临床试验中和临床试验以外获得的有限的靶向药物,以及技术问题。分子靶向药物主要有两个局限性,大多数靶向药物只是部分抑制信号通路,而且很多药物毒性太大不能联合用药。癌症精准治疗核心问题在于尽管肿瘤内异质性存在,是否有任何治疗策略能够治愈癌症或使之长期缓解[112]。精准医疗的应用需要很高的成本,上市的治疗癌症的新药价格不断上升,根据异常通路选择多种分子靶向药物来治疗肿瘤非常昂贵。
我们并不是建议完全放弃个体化医疗,而是建议进行一些小规模的,经过精心设计的合作研究,来试图解决存在的局限性。同时,应该给病人一个明确的信息:肿瘤的个体化治疗还没有显示出实质性的疗效,还处于临床验证阶段。
2、精准医疗时代的不确定性
精准医疗能不能引领一个诊断和预后确定性的时代呢?结果可能恰恰相反。与我们目前医师和患者正在使用的工具相比,制定治疗方法的新工具将需要能更好地适应不确定性,并能更好的计算和解析可能性。
肿瘤治疗一直被成为是“近期加强精准医疗影响的明确选择”。精准医疗时代的到来并不意味着我们对完全由试验确立的治疗决策的分子机制有十足的把握。但是通常在医学实践中,当结果出现时,我们很可能离确定的答案还很远。
值得注意的是让结果在统计上和临床上可解析很重要,连续的变异被对半分为“高”或“低”—换句话说,因为可解析性牺牲了精密度。“根据每个患者的不同特点制定针对性的治疗方案”这一想法本身就存在值得深究的问题[113],将成百上千或成千上万的患者聚集在一起就为了检索可再现的结论?
不同的基因表达产物可能导致不同的风险分级,随着技术变化和数据成熟它们都应该会被改进,随着时间的推移分类和建议也会发生变化。
未来我们可能面对令人困惑的可能性——基于遗传种系筛查评估疾病风险,一旦被诊断,就能确定由“组学”和其他分析指导的预后和治疗方案。评估这些可能性、根据这些可能性来确定数据显示方法、风险量化方法,我们很大程度上缺乏对不确定性之间联系的了解,这也是我们努力的方向。多数情况下,最佳的建议都不明显并经常取决于对成熟数据的初步估计,同时开发“组学”分析工具。我们迫切需要发明一些方法来帮助患者获取大量复杂的信息进而有助于他们在不断增多的大量选项和权衡中做出选择。这些方法应该也有助于我们的同行们回答这个古老的问题“医生,你会怎么做?”
3、精准医疗不太可能改善国民健康
精准医疗可能不能改善国民健康涉及3个基本问题。第一,疾病的发病机理尤其是超级复杂的发病机理不容易得到明确,大量的证据已经证明特异基因多样性和环境的关系。在大多情况下,遗传相关性的作用与行为和社会因素的作用相反,行为和社会因素的作用效果较大而遗传相关性的作用却较小。第二,人们对精准医疗的主要希望是鉴别疾病的预测因素从而有助于指导干预措施的使用。这种方法已经证明对一些疾病有效,尤其是癌症,但是不见得对大部分其他复杂疾病有效。现在已经鉴别出的相关性远远少于那些没有被鉴别出的相关性,所以不能提供足够的证据来改善一些个体对疾病的预测。第三,精准医疗假设的潜在受益性(准确和有意义的风险预测)是可以避免一些国民性疾病或通过对大量国民进行准确的风险预测预先阻止疾病发生,这将改变他们的行为习惯,降低个人风险。尽管这些方法似乎是可信的,但是现有数据表明个体在得知他们已经成为高危人群前是不太可能改变生活习惯的。
六、未来发展趋势
(一)精准医疗与预测模型
精准医疗定制健康护理,为每个患者制定独立制定医疗决策[114]。有时,精准医疗能鉴别单一变异如基因或生物标志物,成功地区别疗法受益和受害的患者。但是在许多情况下,研究的结果与多个变量有关。这样的话,统计学风险预测模型能评估最有可能对治疗干预有所启示,因此有助于辅助医疗决策的制定。尤其是,这些模型能同时将多个患者的特点集合到简单的“风险预测评分”中,这个评分能提供个性化预后概率来确定患者是否需要治疗。
随机临床试验在形成新知识时非常重要。但是大部分的案例中源于临床试验发现的丰富数据在达到主要研究目标时使用却很有限。不仅如此,方法通常会应用平均试验结果到更个性化的方法中,却很少能超过简单的亚组分析。未来,预测模型会变得更加常见,潜在影响临床试验和精准医疗之间的联系。研究者在设计未来的试验时应该合理地计划数据采集促进预测模型的构建,并将这种方法应用在初级统计分析计划中。
在没有发挥其全部潜力前,预测模型的目标不能随着其发表而终止。在EHR的时代,临床决策辅助可以自动进行概率评估,并将这些评估结果提供给床旁临床医师。但是准确的临床预测仅仅刚刚开始;概率评估需要可控并与明确的决策相关联。基于不同的评估风险和受益组合,临床医师需要明确的护理管理策略菜单。很有必要继续加强实践者教育,患者才能更好地理解这些模型的受益性和局限性。概率评估的解析也是一项挑战。理解优点、弱点并验证用于开发和估计这些评估的方法会更加复杂。
(二)精准医疗与公共卫生发展
未来主要的挑战是如何利用来自多水平简化分子标志物(基因组学及各种组学)的新信息评估宏观风险因素(行为习惯、环境、政策等),实现对健康决定因素的更加深入的理解。精准公共健康依靠与人群数据相关的证据,根据亚种群、细化分类的人群、地点、时间,利用获得数据和新技术预测预后。尽管数以百万计的生物学数据取自个体,但是国民水平的干预措施会影响家庭、营养学和贫困等获得资源的因素,较高的教育水平使个体的受益水平也比个人干预措施的效果要好。实际上,将国民水平的干预措施与为高危群体特别设计的干预措施相结合才是有效提高国民健康水平、缩小健康差距的有效方法。
(三)精准医疗与大数据、机器学习和临床医学
数据自身并没有意义,为了让这些数据变得有意义,必须分析、解析和使用这些数据。因此,算法而不是数据集将发挥革命性作用[115]。
机器学习是按照住院医师模式处理问题:从数据中学习规则。这一活动以患者水平的观察开始,算法根据大量变量而变化,寻求组合,来可靠地预测预后。机器学习的闪光点在于它能处理海量的预测因素—有时性能非常卓越,更多地处理预测因素而不是进行观察——并通过非线性和高度交互的方式将其组合起来。这一能力可以让我们使用新型数据,这些数据量和数据复杂性在先前的分析中都是不可想象的。机器学习将显著改善健康专家的预后评估能力,将取代很多放射师和解剖病理学家的工作,将改善诊断准确度。
临床医学需要医生处理大量的数据,从宏观生理学和行为到实验室和成像研究的“组学”数据。管理这一复杂性的能力通常让一位好医生不眠不休。机器学习将成为临床医师真正了解其患者的不可或缺的工具。由于患者环境和医疗技术越来越复杂,机器学习的作用也会增大,随着这一趋势,临床医学将受到挑战。在其他行业中,这一挑战将塑造成功者和失败者,但是在医学中这决定着患者的生死。就算法而言,我们乐观地看到,活着并且医疗记录完善的患者就是最大的赢家,机器学习将在未来改变医疗科学。
(四)超越癌症治疗
精准医疗临床试验在癌症领域的应用已经为它在其他疾病领域的应用打下基础。神经病学领域的发病基础的复杂性和癌症类似但问题更多,神经病学已经确认一些导致疾病的突变,阻止或抵消这种突变可以治愈患者。例如已经有实验室鉴别出许多和肌萎缩侧索硬化症(ALS)有关的遗传变异体,但是他们不知道这些突变引起疾病的机理。癫痫是一种复杂的疾病,已经有药物针对KCNT1基因的突变来治疗癫痫综合症,但是癫痫患者通常是很小的很虚弱的小孩子。在面对精准医疗时必须现实和冷静,距离精准医疗真正发挥巨大的作用还很遥远,积跬步而至千里是研究者必须忍受的过程。精准医疗会对医学做出杰出的贡献,这一点我们从不怀疑,但是需要花费大量的时间和不懈的努力。
随着人类对癌症认识的不断加深,精准医疗的必要性与迫切性已经得到了医疗界和各国政府的广泛重视。未来:表遗传学方向将继续揭示甲基化是如何调节基因表达,加深我们对癌症的理解。新组学(蛋白质组学和代谢组学等)可以揭示癌症生物学的其他层面,对进一步理解癌症生物学和药物开发有重要作用。临床试验数据整合和标准化将为精准治疗的下一步发展奠定基础。未来的方向将是生物学,病理学,计算机等多学科的协作,开发新的分子诊断测试,进一步收集患者测序数据并使其方便共享和搜索,发展基础研究到临床数据的整合网络,建立新的数据统计和风险预测模型等。随着各项配套技术的日趋成熟与完善,精准治疗将在癌症治疗中发挥越来越重要的作用。
参考文献
1.Kathy Hudson, et al., The Precision Medicine Initiative Cohort Program-Building a Research Foundation for 21st Century Medicine, in Precision Medicine Initiative(PMI)Working Group Report to the Advisory Committee to the Director, NIH2015, NIH.
2.Jolie, A., My Medical Choice, in New York Times2013: New York.
3.Sato, F., S. Saji, and M. Toi, Genomic tumor evolution of breast cancer. Breast Cancer, 2016. 23(1): p. 4-11.
4.Navin, N., et al., Tumour evolution inferred by single-cell sequencing. Nature, 2011. 472(7341): p. 90-94.
5.Leary, R.J., et al., Detection of Chromosomal Alterations in the Circulation of Cancer Patients with Whole-Genome Sequencing. Science Translational Medicine, 2012. 4(162): p. 162ra154-162ra154.
6.Murtaza, M., et al., Non-invasive analysis of acquired resistance to cancer therapy by sequencing of plasma DNA. Nature, 2013. 497(7447): p. 108-112.
7.Kurihara, H., et al., Molecular imaging using PET for breast cancer. Breast Cancer, 2016. 23(1): p. 24-32.
8.Kalinyak, J.E., et al., Breast cancer detection using high-resolution breast PET compared to whole-body PET or PET/CT. European Journal of Nuclear Medicine and Molecular Imaging, 2014. 41(2): p. 260-275.
9.Pace, L., et al., Comparison of whole-body PET/CT and PET/MRI in breast cancer patients: Lesion detection and quantitation of 18F-deoxyglucose uptake in lesions and in normal organ tissues. European Journal of Radiology. 83(2): p. 289-296.
10. Salskov, A., et al., FLT: Measuring Tumor Cell Proliferation In Vivo With Positron Emission Tomography and 3′-Deoxy-3′-[<sup>18</sup>F]Fluorothymidine. Seminars in Nuclear Medicine. 37(6): p. 429-439.
11. Pio, B.S., et al., Usefulness of 3′-[F-18]Fluoro-3′-deoxythymidine with Positron Emission Tomography in Predicting Breast Cancer Response to Therapy. Molecular Imaging and Biology, 2006. 8(1): p. 36-42.
12. Gemignani, M.L., et al., Feasibility and Predictability of Perioperative PET and Estrogen Receptor Ligand in Patients with Invasive Breast Cancer. Journal of Nuclear Medicine, 2013. 54(10): p. 1697-1702.
13. Inoue, T., et al., Positron emission tomography using [18F]fluorotamoxifen to evaluate therapeutic responses in patients with breast cancer: preliminary study. Cancer Biother Radiopharm, 1996. 11(4): p. 235-45.
14. Verhagen, A., et al., Preclinical evaluation of a positron emitting progestin ([<sup>18</sup>F]fluoro-16α-methyl-19-norprogesterone) for imaging progesterone receptor positive tumours with positron emission tomography. Cancer Letters. 59(2): p. 125-132.
15. Dehdashti, F., et al., Assessment of 21-[18F]Huoro-16α-Ethyl-19-Norprogesterone as a Positron-Emitting Radiopharmaceutical for the Detection of Progestin Receptors in Human Breast Carcinomas. Journal of Nuclear Medicine, 1991. 32(8): p. 1532-1537.
16. Lee, J.H., et al., Development of [F-18]Fluorine-Substituted Tanaproget as a Progesterone Receptor Imaging Agent for Positron Emission Tomography. Bioconjugate Chemistry, 2010. 21(6): p. 1096-1104.
17. Dijkers, E.C., et al., Biodistribution of 89Zr-trastuzumab and PET Imaging of HER2-Positive Lesions in Patients With Metastatic Breast Cancer. Clinical Pharmacology & Therapeutics, 2010. 87(5): p. 586-592.
18. Tamura, K., et al., 64Cu-DOTA-Trastuzumab PET Imaging in Patients with HER2-Positive Breast Cancer. Journal of Nuclear Medicine, 2013. 54(11): p. 1869-1875.
19. Smith-Jones, P.M., et al., Imaging the pharmacodynamics of HER2 degradation in response to Hsp90 inhibitors. Nat Biotech, 2004. 22(6): p. 701-706.
20. Makris, N.E., et al., PET/CT-Derived Whole-Body and Bone Marrow Dosimetry of 89Zr-Cetuximab. Journal of Nuclear Medicine, 2015. 56(2): p. 249-254.
21. Bhattacharyya, S., et al., Zirconium-89 labeled panitumumab: a potential immuno-PET probe for HER1-expressing carcinomas. Nuclear Medicine and Biology. 40(4): p. 451-457.
22. Wang, H., et al., Assessment of 11C-labeled-4-N-(3-bromoanilino)-6, 7-dimethoxyquinazoline as a positron emission tomography agent to monitor epidermal growth factor receptor expression. Cancer Science, 2007. 98(9): p. 1413-1416.
23. Heskamp, S., et al., ImmunoSPECT and ImmunoPET of IGF-1R Expression with the Radiolabeled Antibody R1507 in a Triple-Negative Breast Cancer Model. Journal of Nuclear Medicine, 2010. 51(10): p. 1565-1572.
24. Wang, H., et al., A new PET tracer specific for vascular endothelial growth factor receptor 2. European Journal of Nuclear Medicine and Molecular Imaging, 2007. 34(12): p. 2001-2010.
25. Gaykema, S.B.M., et al., 89Zr-Bevacizumab PET Imaging in Primary Breast Cancer. Journal of Nuclear Medicine, 2013. 54(7): p. 1014-1018.
26. Chen, X., et al., MicroPET and Autoradiographic Imaging of Breast Cancer αv-Integrin Expression Using 18F- and 64Cu-Labeled RGD Peptide. Bioconjugate Chemistry, 2004. 15(1): p. 41-49.
27. Beer, A.J., et al., Patterns of αvβ3 Expression in Primary and Metastatic Human Breast Cancer as Shown by 18F-Galacto-RGD PET. Journal of Nuclear Medicine, 2008. 49(2): p. 255-259.
28. Naoi, Y. and S. Noguchi, Multi-gene classifiers for prediction of recurrence in breast cancer patients. Breast Cancer, 2016. 23(1): p. 12-18.
29. Sapino, A., et al., MammaPrint Molecular Diagnostics on Formalin-Fixed, Paraffin-Embedded Tissue. The Journal of Molecular Diagnostics. 16(2): p. 190-197.
30. Cardoso, F., et al., 70-Gene Signature as an Aid to Treatment Decisions in Early-Stage Breast Cancer. New England Journal of Medicine, 2016. 375(8): p. 717-729.
31. Carlson, J.J. and J.A. Roth, The impact of the Oncotype Dx breast cancer assay in clinical practice: a systematic review and meta-analysis. Breast Cancer Research and Treatment, 2013. 141(1): p. 13-22.
32. Zhang, Y., et al., Breast Cancer Index Identifies Early-Stage Estrogen Receptor-Positive Breast Cancer Patients at Risk for Early- and Late-Distant Recurrence. Clinical Cancer Research, 2013. 19(15): p. 4196-4205.
33. Naoi, Y., et al., High genomic grade index associated with poor prognosis for lymph node-negative and estrogen receptor-positive breast cancers and with good response to chemotherapy. Cancer, 2011. 117(3): p. 472-479.
34. Filipits, M., et al., The PAM50 Risk-of-Recurrence Score Predicts Risk for Late Distant Recurrence after Endocrine Therapy in Postmenopausal Women with Endocrine-Responsive Early Breast Cancer. Clinical Cancer Research, 2014. 20(5): p. 1298-1305.
35. Sestak, I., et al., Factors Predicting Late Recurrence for Estrogen Receptor-Positive Breast Cancer. Journal of the National Cancer Institute, 2013. 105(19): p. 1504-1511.
36. Dubsky, P., et al., The EndoPredict score provides prognostic information on late distant metastases in ER+/HER2- breast cancer patients. Br J Cancer, 2013. 109(12): p. 2959-2964.
37. Naoi, Y., et al., Comparison of efficacy of 95-gene and 21-gene classifier(Oncotype DX)for prediction of recurrence in ER-positive and node-negative breast cancer patients. Breast Cancer Research and Treatment, 2013. 140(2): p. 299-306.
38. Grothey, A. and D.J. Sargent, Adjuvant therapy for colon cancer: Small steps toward precision medicine. JAMA Oncology, 2016. 2(9): p. 1133-1134.
39. Riazalhosseini, Y. and M. Lathrop, Precision medicine from the renal cancer genome. Nat Rev Nephrol, 2016. 12(11): p. 655-666.
40. Shuch, B., et al., Understanding Pathologic Variants of Renal Cell Carcinoma: Distilling Therapeutic Opportunities from Biologic Complexity. European Urology, 2015. 67(1): p. 85-97.
41. Henrion, M., et al., Common variation at 2q22.3 (ZEB2) influences the risk of renal cancer. Human Molecular Genetics, 2013. 22(10): p. 2128-2128.
42. Bertolotto, C., et al., A SUMOylation-defective MITF germline mutation predisposes to melanoma and renal carcinoma. Nature, 2011. 480(7375): p. 94-98.
43. Henrion, M.Y.R., et al., Common Variation at 1q24.1 (ALDH9A1) Is a Potential Risk Factor for Renal Cancer. PLOS ONE, 2015. 10(3): p. e0122589.
44. Schodel, J., et al., Common genetic variants at the 11q13.3 renal cancer susceptibility locus influence binding of HIF to an enhancer of cyclin D1 expression. Nat Genet, 2012. 44(4): p. 420-425.
45. Pedersen, H.K., et al., Ranking factors involved in diabetes remission after bariatric surgery using machine-learning integrating clinical and genomic biomarkers. Npj Genomic Medicine, 2016. 1: p. 16035.
46. Darcy, A.M., A.K. Louie, and L. Roberts, MAchine learning and the profession of medicine. JAMA, 2016. 315(6): p. 551-552.
47. Obermeyer, Z. and E.J. Emanuel, Predicting the Future - Big Data, Machine Learning, and Clinical Medicine. N Engl J Med, 2016. 375(13): p. 1216-9.
48. FDA, Use of Standards in FDA Regulatory Oversight of Next Generation Sequencing (NGS)-Based In Vitro Diagnostics (IVDs) Used for Diagnosing Germline Diseases, FDA, Editor, FDA.
49. FDA, Use of Public Human Genetic Variant Databases to Support Clinical Validity for Next Generation Sequencing (NGS)-Based In Vitro Diagnostics, FDA, Editor 2016.
50. Numanagic, I., et al., Comparison of high-throughput sequencing data compression tools. Nat Meth, 2016. advance online publication.
51. Topalian, S.L., G.J. Weiner, and D.M. Pardoll, Cancer Immunotherapy Comes of Age. Journal of Clinical Oncology, 2011. 29(36): p. 4828-4836.
52. Miao, D. and E.M. Van Allen, Genomic determinants of cancer immunotherapy. Current Opinion in Immunology, 2016. 41: p. 32-38.
53. Kaufman, H.L., Precision Immunology: The Promise of Immunotherapy for the Treatment of Cancer. Journal of Clinical Oncology, 2015. 33(12): p. 1315-1317.
54. Chen, Daniel S. and I. Mellman, Oncology Meets Immunology: The Cancer-Immunity Cycle. Immunity, 2013. 39(1): p. 1-10.
55. Kato, J. and C.I. Svensson, Chapter Nine - Role of Extracellular Damage-Associated Molecular Pattern Molecules (DAMPs) as Mediators of Persistent Pain, in Progress in Molecular Biology and Translational Science, J.P. Theodore and D. Gregory, Editors. 2015, Academic Press. p. 251-279.
56. Spel, L., et al., Antitumor immune responses mediated by dendritic cells: How signals derived from dying cancer cells drive antigen cross-presentation. OncoImmunology, 2013. 2(11): p. e26403.
57. Schreiber, R.D., L.J. Old, and M.J. Smyth, Cancer Immunoediting: Integrating Immunity’s Roles in Cancer Suppression and Promotion. Science, 2011. 331(6024): p. 1565-1570.
58. Politi, K. and R.S. Herbst, Lung Cancer in the Era of Precision Medicine. Clinical Cancer Research, 2015. 21(10): p. 2213-2220.
59. Melero, I., et al., Therapeutic vaccines for cancer: an overview of clinical trials. Nat Rev Clin Oncol, 2014. 11(9): p. 509-524.
60. Arlen, P.M. and L.V. Wood, Prostate cancer vaccines: moving therapeutic vaccination forward in the post-Provenge era. Expert Rev Vaccines, 2012. 11(3): p. 287-302.
61. Di Lorenzo, G., M. Ferro, and C. Buonerba, Sipuleucel-T (Provenge(R)) for castration-resistant prostate cancer. BJU Int, 2012. 110(2 Pt 2): p. E99-104.
62. Fischbach, M.A., J.A. Bluestone, and W.A. Lim, Cell-Based Therapeutics: The Next Pillar of Medicine. Science Translational Medicine, 2013. 5(179): p. 179ps7-179ps7.
63. Bluestone, J.A. and Q.Z. Tang, Immunotherapy: Making the case for precision medicine. Science Translational Medicine, 2015. 7(280).
64. June, C.H., S.R. Riddell, and T.N. Schumacher, Adoptive cellular therapy: A race to the finish line. Science Translational Medicine, 2015. 7(280): p. 280ps7-280ps7.
65. Grupp, S.A., et al., Chimeric Antigen Receptor-Modified T Cells for Acute Lymphoid Leukemia. New England Journal of Medicine, 2013. 368(16): p. 1509-1518.
66. Lee, D.W., et al., T cells expressing CD19 chimeric antigen receptors for acute lymphoblastic leukaemia in children and young adults: a phase 1 dose-escalation trial. The Lancet. 385(9967): p. 517-528.
67. Maude, S.L., et al., Chimeric Antigen Receptor T Cells for Sustained Remissions in Leukemia. New England Journal of Medicine, 2014. 371(16): p. 1507-1517.
68. Davila, M.L., et al., Efficacy and Toxicity Management of 19-28z CAR T Cell Therapy in B Cell Acute Lymphoblastic Leukemia. Science Translational Medicine, 2014. 6(224): p. 224ra25-224ra25.
69. Porter, D.L., et al., Chimeric Antigen Receptor-Modified T Cells in Chronic Lymphoid Leukemia. New England Journal of Medicine, 2011. 365(8): p. 725-733.
70. Jackson, H.J., S. Rafiq, and R.J. Brentjens, Driving CAR T-cells forward. Nat Rev Clin Oncol, 2016. 13(6): p. 370-383.
71. Deng, X. and Y. Nakamura, Cancer Precision Medicine: From Cancer Screening to Drug Selection and Personalized Immunotherapy. Trends in Pharmacological Sciences.
72. Eppihimer, M.J., et al., Expression and regulation of the PD-L1 immunoinhibitory molecule on microvascular endothelial cells. Microcirculation, 2002. 9(2): p. 133-45.
73. Ishida, M., et al., Differential expression of PD-L1 and PD-L2, ligands for an inhibitory receptor PD-1, in the cells of lymphohematopoietic tissues. Immunol Lett, 2002. 84(1): p. 57-62.
74. Liang, S.C., et al., Regulation of PD-1, PD-L1, and PD-L2 expression during normal and autoimmune responses. Eur J Immunol, 2003. 33(10): p. 2706-16.
75. Hino, R., et al., Tumor cell expression of programmed cell death-1 ligand 1 is a prognostic factor for malignant melanoma. Cancer, 2010. 116(7): p. 1757-1766.
76. Rosell, R. and R. Palmero, PD-L1 expression associated with better response to EGFR tyrosine kinase inhibitors. Cancer Biol Med, 2015. 12(2): p. 71-3.
77. Patel, S.P. and R. Kurzrock, PD-L1 Expression as a Predictive Biomarker in Cancer Immunotherapy. Molecular Cancer Therapeutics, 2015. 14(4): p. 847-856.
78. D’Angelo, S.P., et al., Prevalence of tumor-infiltrating lymphocytes and PD-L1 expression in the soft tissue sarcoma microenvironment. Human Pathology. 46(3): p. 357-365.
79. Nishimura, H., et al., Autoimmune Dilated Cardiomyopathy in PD-1 Receptor-Deficient Mice. Science, 2001. 291(5502): p. 319-322.
80. Segal, N.H., et al., Epitope Landscape in Breast and Colorectal Cancer. Cancer Res, 2008. 68(3): p. 889-892.
81. Comprehensive molecular characterization of human colon and rectal cancer. Nature, 2012. 487(7407): p. 330-337.
82. Young, J., et al., Features of Colorectal Cancers with High-Level Microsatellite Instability Occurring in Familial and Sporadic Settings: Parallel Pathways of Tumorigenesis. The American Journal of Pathology, 2001. 159(6): p. 2107-2116.
83. Le, D.T., et al., PD-1 Blockade in Tumors with Mismatch-Repair Deficiency. New England Journal of Medicine, 2015. 372(26): p. 2509-2520.
84. Dudley, J.C., et al., Microsatellite Instability as a Biomarker for PD-1 Blockade. Clinical Cancer Research, 2016. 22(4): p. 813-820.
85. Weiss, M.J. and C.G. Mullighan, Welcoming a new age for gene therapy in hematology. Blood, 2016. 127(21): p. 2523-2524.
86. Wang, C.X. and P.M. Cannon, The clinical applications of genome editing in HIV. Blood, 2016. 127(21): p. 2546-2552.
87. Luo, R., M.J. Piovoso, and R. Zurakowski, Quantitative analysis of viral persistence and transient viral load rebound from HIV clinical data. Conference proceedings : ... Annual International Conference of the IEEE Engineering in Medicine and Biology Society. IEEE Engineering in Medicine and Biology Society. Conference, 2011. 2011: p. 3585-3588.
88. Kelley, C.F., et al., Incomplete Peripheral CD4+ Cell Count Restoration in HIV-Infected Patients Receiving Long-Term Antiretroviral Treatment. Clinical Infectious Diseases, 2009. 48(6): p. 787-794.
89. Morgan, R.A., et al., Preferential survival of CD4+ T lymphocytes engineered with anti-human immunodeficiency virus (HIV) genes in HIV-infected individuals. Hum Gene Ther, 2005. 16(9): p. 1065-74.
90. Li, M.-J., et al., Long-Term Inhibition of HIV-1 Infection in Primary Hematopoietic Cells by Lentiviral Vector Delivery of a Triple Combination of Anti-HIV shRNA, Anti-CCR5 Ribozyme, and a Nucleolar-Localizing TAR Decoy. Mol Ther, 2005. 12(5): p. 900-909.
91. Podsakoff, G.M., et al., Selective survival of peripheral blood lymphocytes in children with HIV-1 following delivery of an anti-HIV gene to bone marrow CD34+ cells. Mol Ther, 2005. 12(1): p. 77-86.
92. Levine, B.L., et al., Gene transfer in humans using a conditionally replicating lentiviral vector. Proceedings of the National Academy of Sciences, 2006. 103(46): p. 17372-17377.
93. Belfort, M. and R.J. Roberts, Homing endonucleases: keeping the house in order. Nucleic Acids Research, 1997. 25(17): p. 3379-3388.
94. Gaj, T., C.A. Gersbach, and C.F. Barbas, III, ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31(7): p. 397-405.
95. Hoffmann, C., The epidemiology of HIV coreceptor tropism. Eur J Med Res, 2007. 12(9): p. 385-90.
96. Liu, R., et al., Homozygous Defect in HIV-1 Coreceptor Accounts for Resistance of Some Multiply-Exposed Individuals to HIV-1 Infection. Cell. 86(3): p. 367-377.
97. Hütter , G., et al., Long-Term Control of HIV by CCR5 Delta32/Delta32 Stem-Cell Transplantation. New England Journal of Medicine, 2009. 360(7): p. 692-698.
98. Perez, E.E., et al., Establishment of HIV-1 resistance in CD4+ T cells by genome editing using zinc-finger nucleases. Nat Biotech, 2008. 26(7): p. 808-816.
99. Holt, N., et al., Zinc finger nuclease-mediated CCR5 knockout hematopoietic stem cell transplantation controls HIV-1 in vivo. Nature biotechnology, 2010. 28(8): p. 839-847.
100. Wang, J., et al., Homology-driven genome editing in hematopoietic stem and progenitor cells using ZFN mRNA and AAV6 donors. Nat Biotech, 2015. 33(12): p. 1256-1263.
101. Mussolino, C., et al., TALENs facilitate targeted genome editing in human cells with high specificity and low cytotoxicity. Nucleic Acids Research, 2014. 42(10): p. 6762-6773.
102. Sather, B.D., et al., Efficient modification of <em>CCR5</em> in primary human hematopoietic cells using a megaTAL nuclease and AAV donor template. Science Translational Medicine, 2015. 7(307): p. 307ra156-307ra156.
103. Mandal, Pankaj K., et al., Efficient Ablation of Genes in Human Hematopoietic Stem and Effector Cells using CRISPR/Cas9. Cell Stem Cell. 15(5): p. 643-652.
104. Li, C., et al., Inhibition of HIV-1 infection of primary CD4+ T-cells by gene editing of CCR5 using adenovirus-delivered CRISPR/Cas9. Journal of General Virology, 2015. 96(8): p. 2381-2393.
105. Rahdar, M., et al., Synthetic CRISPR RNA-Cas9-guided genome editing in human cells. Proceedings of the National Academy of Sciences of the United States of America, 2015. 112(51): p. E7110-E7117.
106. Reardon, S., First CRISPR clinical trial gets green light from US panel. Nature, 2016.
107. Cyranoski, D., Chinese scientists to pioneer first human CRISPR trial. Nature, 2016. 535(7613): p. 476-477.
108. Cyranoski, D., CRISPR gene-editing tested in a person for the first time. Nature, 2016. 539(7630).
109. Collins FS, Varmus H. A New Initiative on Precision Medicine. The New England Journal of Medicine. 2015;372.
110. Khoury MJ, Galea S. Will PrecisionMedicine Improve Population Health? Jama the Journal of the American Medical Association. 2016;316:2.
111. Dzau VJ, Ginsburg GS. Realizing the Full Potential of PrecisionMedicine in Health and Health Care. Jama the Journal of the American Medical Association. 2016;316.
112. Tannock IF, Hickman JA. Limits to Personalized Cancer Medicine. The New England Journal o f Medicine. 2016;375.
113. Hunter DJ. Uncertainty in the Era of Precision Medicine. The New England Journal of Medicine. 2016;375.
114. Pencina MJ, Peterson ED. Moving FromClinical Trials to PrecisionMedicine. Jama the Journal of the American Medical Association. 2016;315.
115. Obermeyer Z, Emanuel EJ. Predicting the Future — Big Data, Machine Learning, and Clinical Medicine. The New England Journal of Medicine. 2016;375.




