基于ARMS定量肺腺癌患者 EGFR突变的新型试验
摘 要
表皮生长因子受体(EGFR)突变对非小细胞肺癌(NSCLC)患者使用酪氨酸激酶抑制剂(TKI)治疗疗效的预测具有重要意义。然而,临床医师缺少一种灵敏且便捷的方法来定量EGFR突变的丰度。本研究介绍了一种新的方法,即扩增阻滞突变检测系统(ARMS-PLUS),来对EGFR 外显子19缺失(19Del、L858R和T790M突变)进行定量分析。本研究从77例肺腺癌患者中采集了福尔马林固定石蜡包埋的标本。利用ARMS-Plus进行EGFR突变的DNA提取和分析。然后将ARMS-Plus与常规ARMS聚合酶链反应(ARMS-PCR)以及微液滴数字PCR(ddPCR)的性能进行比较。结果表明ARMS-Plus和ddPCR之间EGFR突变检测的一致率为98.7%(76/77,Kappa=0.9739)。分别在23例和12例患者中检出了19Del和L858R突变。在评估19Del突变体丰度时,ARMS-Plus和ddPCR的结果之间存在显著差异(P=0.0002);但是,这两种技术在评估L858R突变体的丰度时,却没有这种差异(P=0.7334)。与ddPCR相比,L858R突变体丰度的ARMS-Plus结果是一致的(R2=0.8081)。这些结果表明,在鉴别EGFR突变时,ARMS-Plus的灵敏度和特异性与ddPCR相似。在定量分析中,L858R突变体丰度的评估结果与ddPCR的结果呈正相关。因此,ARMS-Plus可以成为一种可靠、经济且可以量化EGFR突变的替代方法,从而有助于肺腺癌患者的治疗决策的制定。
简 介
世界范围内肺癌是导致癌症相关死亡事件的主要原因,每年导致超过100万的死亡事件。非小细胞肺癌(NSCLC)占到总肺癌病例数的85%。NSCLC的预后很差,5年存活率低于20%。表皮生长因子受体(EGFR)是NSCLC经常表达的一种横跨膜酪氨酸激酶。EGFR的活化包括癌症细胞的扩散和增殖,因此EGFR是个体化疗法有前景的目标。近年来,EGFR酪氨酸激酶抑制剂(TKI)如吉非替尼、厄洛替尼和阿法替尼已经用于NSCLC的一线治疗。通过有选择地与受体激酶域的ATP结合位点相结合,TKI阻断EGFR的活化及其下游信号传导,如ST3、MAP激酶(MAPK)和AKT信号。尽管NSCLC患者使用EGFR-TKI获得了快速且激动人心的肿瘤抑制效果,但是最初使用吉非替尼治疗的临床研究(2005年)中,仅有9%—19%的未经选择的患者对该疗法产生反应。随后,研究者报告称EGFR突变状态可能是EGFR-TKI治疗效果的关键预测因素。外显子19缺失(19Del)和L858R碱基置换突变是最常见的EGFR突变,这两种突变占到总EGFR突变的90%。一些研究揭示,出现EGFR突变的患者在使用EGFR-TKI时预后较好。2项II期临床试验确认了这一现象,证明存在EGFR突变的NSCLC患者对吉非替尼的反应率为75%—78%。但是在使用第一代EGFR-TKI治疗后,第二种突变的出现导致治疗的耐药性。T790M突变是第一代EGFR-TKI耐药的主要原因,大约占到获得性耐药的70%。报告称T790M突变与非小细胞肺癌患者EGFR-TKI治疗的无进展生存率(PFS)有关。相对EGFR突变体丰度也可能是预测EGFR-TKI敏感性的关键因素。根据Zhou等人的报告,在经过吉非替尼治疗后,EGFR突变丰度较高的晚期NSCLC患者的PFS显著较长。在另一项研究中,Zhao等人证明,EGFR突变的丰度可能是另一个预测因素,可能有助于筛选那些对主要TKI产生耐药性并可以通过再次使用EGFR-TKI获益的患者。由于上述EGFR突变对于确定NSCLC患者的治疗策略至关重要,因此迫切需要一种可靠的方法来鉴别和测量EGFR突变体的丰度。
在过去的十年中,直接DNA测序是最常用的EGFR突变检测方法。尽管该方法普及性较高,但灵敏度相对较低。直接DNA测序的检测限约为20%,这可能导致较高的假阴性结果。更重要的是,进行直接的DNA测序需要很长的处理时间,并可能延误临床决策。同时,大量灵敏度更高的方法已经被用于EGFR突变检测。扩增阻滞突变检测系统(ARMS)是一种基于等位基因特异性聚合酶链反应的检测方法。报告称其检测限在1%左右,比直接DNA测序灵敏得多。微液滴数字PCR(ddPCR)是另一种检测EGFR突变的新方法。ddPCR基于DNA单分子的区室化和扩增,具有很高的灵敏度。因此,现在普遍认为ddPCR是最灵敏的突变检测和定量方法。然而,这种方法需要专门的设备,而且在劳动力和成本方面比较昂贵。本文中,我们将介绍一种基于ARM的改进型分析方法——即ARM-Plus,基于实时PCR平台对EGFR突变进行分析。本研究采用ARMS-Plus和ddPCR方法对提取的FFPE DNA样本进行分析,目的是比较两种方法的性能,探讨使用ARMS-Plus测量EGFR突变体丰度的可行性。
材料和方法
样品采集和处理
收集2013-2015年来自广东省中医院患者的FFPE组织样品。所有患者均签署书面知情同意书。本研究方案经广东省中医院机构审查委员会批准。病理学家对FFPE组织标本进行显微解剖并用苏木精和伊红染色法进行评估。使用QIAamp®FFPE组织试剂盒提取基因组DNA(中国上海Qiagen)。收集FFPE提取的DNA样本,然后进行下列试验。
ARMS-Plus
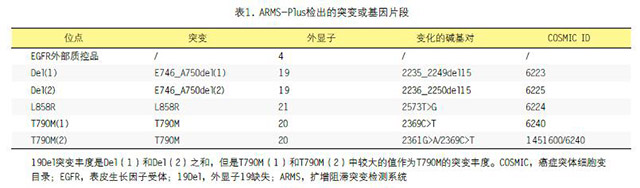
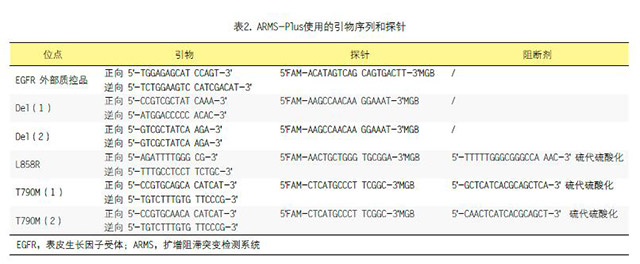
根据制造商的说明书,分离的DNA样本由人类EGFR基因突变定量检测试剂盒处理(荧光素qPCR;上海GenoSaber生物技术有限公司。这是一种配备外部质控品可以检测外显子4中4种不同EGFR突变来监测样品质量的定量PCR试剂盒(表1)。简而言之,添加5µl样本到预先混合45µl反应混合物中,使用Applied Biosystems®7500实时-PCR系统(USA、MA沃尔瑟姆赛默飞世尔科技)执行PCR。PCR扩增条件如下:在95℃热启动4分钟;在95℃10秒执行50个周期;在61℃进行30秒荧光读数(FAM)。引物和探针序列见表2。通过两种不同的反应检测到了2种碱基对的改变从而发现了19Del突变。我们还发现,2361位点的单核苷酸多态性(SNP)可能影响PCR特异性,因此通过两种反应进行T790M试验来提高检测率。突变体丰度被定义为整个DNA样品中存在的突变体拷贝数的百分比。为了计算拷贝数,在试剂盒中提供了连续稀释的质粒作为标准样品生成标准化曲线。从理论上讲,突变体丰度的计算方程如下:
突变体丰度 = 观测到的突变体拷贝数 / 观测到的野生型拷贝数
ddPCR
根据前述内容执行ddPCR。简单地说,微液滴发生器应用探针、引物、模板DNA和2X液滴SuperMix(Bio-Rad Laboratories, Hercules,CA,USA)的混合物。然后,生成的油-水混合物被转移到96孔的PCR板,热封并放置在C1000Touch热循环器(Bio - Rad Laboratories)中用于PCR扩增。热循环条件如下:95℃热启动10分钟;94℃30秒40个循环,62℃1分钟;98℃运行10分钟。使用QX-200微液滴阅读器(Bio-Rad laboratories)读取PCR产物,使用Quantilife软件分析数据。使用Quantilife软件根据泊松统计量计算出突变体和野生型DNA的拷贝数。
ARMS-PCR
采用ADx EGFR突变检测试剂盒(中国厦门Amoy Diagnostics)重新检测分离的DNA样本,该试剂盒获得了中国食品药品监督管理局批准,可以在中国主要地区进行临床使用。该试剂盒涵盖来自exon 18到21的29个EGFR突变热点。根据制造商的协议使用MX3000P实时PCR系统(美国加州圣克拉拉安捷伦科技有限公司Stratagene)执行该试验。
统计分析
通过Kappa试验,分析了ARMS-Plus和ddPCR在鉴别EGFR突变状态中的一致率。通过威克森测试对样本进行检验确定ARM-Plus和ddPCR之间的EGFR突变是否存在统计学差异。P<0.05被认为有统计学意义。采用线性回归分析方法,研究ARMS-Plus和ddPCR EGFR突变丰度结果的相关性。使用SAS9.2(SAS Institute,Inc. Cary,NC,USA)和GraphPad Prism 5(GraphPad Software,Inc.,La Jolla,CA,USA)进行统计分析和图形生成。
结 果
ARMS-Plus检测效率的评估
以1%和5%的比例将突变细胞系H1650(19Del)和H1975(L858R和T790 M)加入EGFR野生型细胞系A549内(2×103的H1650和1×104的H1975细胞分别加入2×105的A549细胞内)。用QIAamp DNA FFPE组织试剂盒(Qiagen)从细胞混合物中提取DNA。由ARMS - Plus和ddPCR(每种细胞混合物3次重复)分析最终等位基因频率。结果表明,ARMS-Plus可以稳定检测所有突变(图1)。一般来说,ddPCR检出的突变体丰度高于ARMS-Plus检出的突变体丰度。
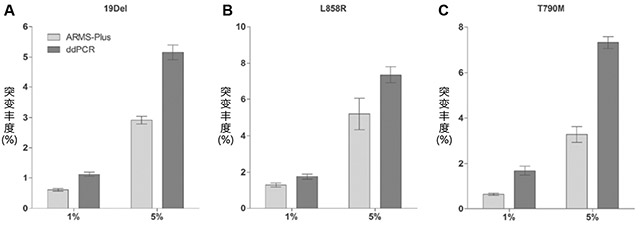
图1. 细胞混合物中ARMS-Plus和ddPCR进行的EGFR突变检测。体1%和5%的比率将突变细胞系添加到EGFR野生型细胞系内。随后检测突变丰度。ARMS-Plus和ddPCR可以稳定检测(A)19Del、(B)L858R、(C)T790M突变。数据表示为平均值加减标准标准误差。EGFR,表皮生长因子受体;ARMS,扩增阻滞突变检测系统;ddPCR,微液滴数字聚合酶链反应;19Del,外显子19缺失。
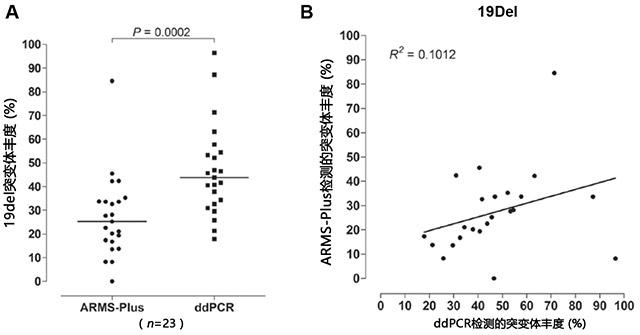
图2. 成对样品中19Del突变的定量分析。(A)威尔科克森秩和检验和(B)线性回归分析评估的19Del突变体丰度,ARMS-Plus和ddPCR比较。19Del,外显子19缺失;ARMS,扩增阻滞突变检测系统;ddPCR,微液滴数字聚合酶链反应。
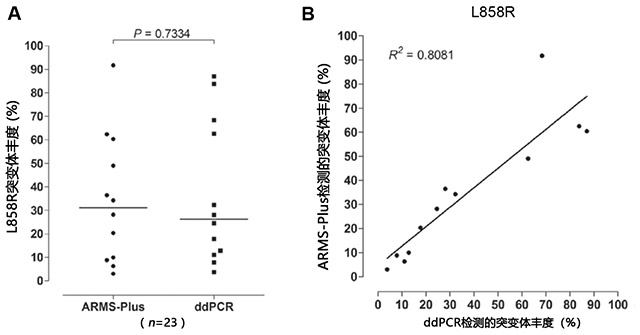
图3. 成对样品中L858R突变的定量分析。(A)威尔科克森秩和检验和(B)线性回归分析评估的L858R突变体丰度,ARMS-Plus和ddPCR比较。19Del,外显子19缺失;ARMS,扩增阻滞突变检测系统;ddPCR,微液滴数字聚合酶链反应。
患者登记
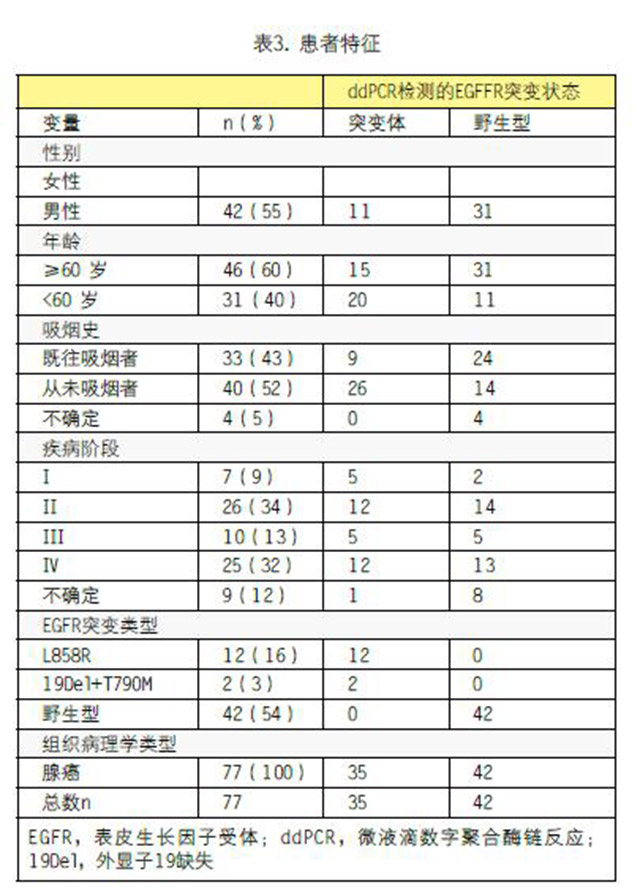
该研究采集了77名肺腺癌患者的FFPE肿瘤样品(表3)。在这些患者中,45%(35/77)为女性,中值年龄为62岁(37-91岁),52%从不吸烟。近一半的患者(45%,35/77)被诊断为癌症晚期(III-IV期)。只有4名患者在样本采集前接受过EGFR-TKI治疗。
EGFR突变状态的确定
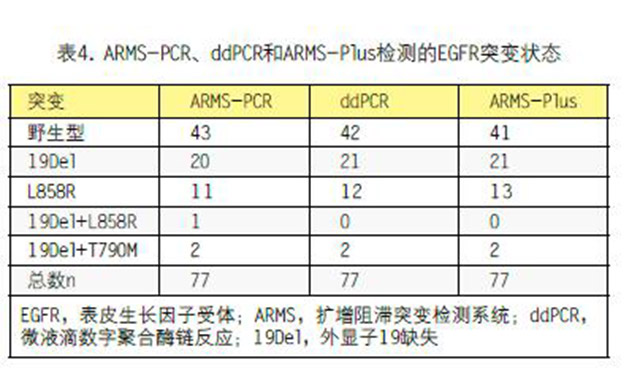
执行ARMS-Plus分析EGFR突变状态。随后使用ddPCR和ARMS-PCR重新检测该结果(表4)。
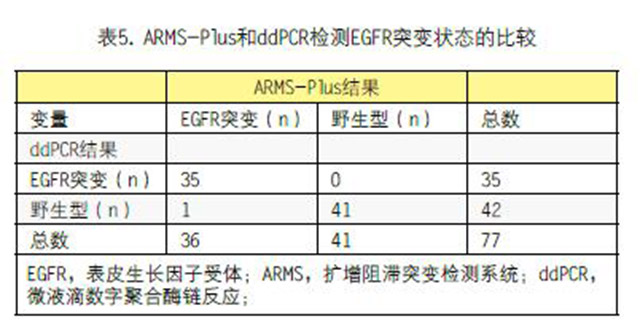
使用ddPCR评估的77份配对样本中,35份(45.5%)为突变阳性,其他42份样本为突变阴性。对于ARMS-Plus结果,只有一份样本显示与ddPCR结果不一致。一个ARMS-Plus检测为L585R弱阳性,丰度为0.5%的结果,ddPCR检测为阴性(表5)。总的来说,ARMS-Plus和ddPCR检测EGFR突变的结果的相关性为98.7%(76/77),两种方法鉴别的EGFR突变状态结果高度一致(Kappa=0.9739,95%CI:0.923-1)。
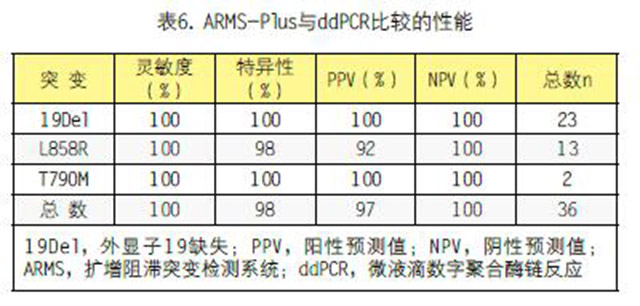
为了评估ARMS-Plus识别EGFR突变的实用性,我们评估了该试验与ddPCR相比的灵敏度和特异性(表5)。对于19Del和T790M突变,ARMS-Plus结果与ddPCR 100%匹配。通过ARMS检出,但ddPCR未检出的只有一例L858R突变。EGFR突变的总体检测灵敏度和特异度分别为100%和98%,证明ARMS-Plus在鉴别EGFR突变方面很有前景。
ARMS-PCR结果与ARMS-Plus和ddPCR相近。仅有1个ARMS-Plus和ddPCR确定为19Del阳性的病例被ARMS-PCR分为阴性,1个ARMS-Plus和ddPCR确定为19Del阴性的病例被ARMS-PCR分为阳性。总而言之,这些数据表明ARMS-Plus、ddPCR和ARMS-PCR在检测FFPE DNA样品内的EGFR突变方面具有一致性。
EGFR突变的定量分析
在77份样本中,ARMS-Plus和ddPCR鉴别出有23份样品存在19Del突变体。结果显示ARMS-Plus(中值=25.26%)和ddPCR(中值=43.76%)检测的19Del丰度存在显著的差异(威尔科克森秩和检验,P=0.0002,图2A)。由ARMS-Plus检测到的19Del突变丰度与基于ddPCR期望丰度不一致(线性回归分析,R2=0.1012,图2B)。
对于L858R突变,ARMS-Plus和ddPCR鉴定出77份样本中有12份存在L858R突变。
ARMS-Plus(中值=31.18%)和ddPCR(26.27%)检测的L858R突变丰度没有观测到显著的差异(威尔科克森秩和检验,P=0.7334,图3A)。由ARMS-Plus检测到的L858R突变丰度与基于ddPCR的期望丰度高度一致(线性回归分析,R2=0.8081,图3B)。
T790M突变丰度的数据未被分析,仅有2份样品被鉴定为T790M突变(两个患者均为初始EGFR-TKI治疗)。然而,ARMS-Plus检测到突变丰度与ddPCR检测的丰度(RMS-Plus和ddPCR检测分别为病例1:29.21%和43.17%;病例2:11.18和16.42%)大体一致。
讨 论
自从EGFR-TKI在过去10年间获得批准以来,大量的研究一直在探索EGFR突变在预测TKI治疗灵敏度方面的临床意义。目前来说显而易见EGFR突变与TKI疗法的疗效有关。建议所有NSCLC患者都应该诊断后进行EGFR突变检测来节约成本,以避免因错误的治疗方案产生不良影响。最近的临床研究显示,第三代EGFR-TKI奥希替尼能够克服T790M的耐药性,与第一代EGFR-TKI相比,首次治疗时,该药物对发生EGFR突变的晚期NSCLC的治疗效果显著。因此,一种可以识别出具有“重新出现的”T790M突变患者的灵敏试验对于选择能因一线奥希替尼治疗获益的患者非常有利。
由于NSCLC被发现时通常已经处于晚期,少量诊断性活检标本是突变检测的主要来源。由于之前的形态学和免疫表型分析,这些标本的DNA数量通常非常有限。因此,需要高度敏感的试验检测EGFR突变。目前,普遍认为ddPCR是最灵敏的评估低突变率的突变的方法。它利用一种水油乳剂微液滴系统将DNA样品分离成数千个纳米级的液滴。这种分离使得在单个样本内能够测量数千个独立的扩增事件,大幅提高了分析的灵敏度。2014年,Zhang等人的报告称ddPCR检测EGFR突变具有优势。结果表明,ddPCR能够检测和定量最低为0.12%~2.73%的EGFR突变。尽管ddPCR的灵敏度很高,但其昂贵的成本和对专门设备的要求极大地阻碍了它的临床应用。
本研究中,我们以ddPCR为标准解析了ARMS-Plus在鉴别和定量EGFR突变方面的能力。ARMS-Plus是一种基于AMRS的检测方法,可对4种不同的EGFR突变进行定量,其中包括在两个不同位点的19Del以及L858R和T790M突变。虽然ARMS-Plus分析了有限的突变位点,但它们代表大多数突变,因此在指导EGFR-TKI治疗的选择方面具有较好的临床意义。此外,ARMS-Plus的引物和探针针对EGFR突变检测进行了优化,每种定量PCR的扩增子缩短到50—80bp。这些特征能够对FFPE肿瘤样本中提取的高度分散的DNA分子进行精确分析。为了针对性定量EGFR突变,在ARMS-Plus PCR反应使用了一种3'端磷酸化的阻断引物。通过对突变位点补足野生型DNA,这些引物阻断了非特异性延伸,并提高了定量的准确性。此外,可以使用实时PCR系统方便地运行ARMS-Plus。ARMS-Plus的这些特点使其可以临床实践中快速、可靠和经济有效地进行分子分析。
通过细胞实验评价了ARMS-Plus检测效率。使用ARMS-Plus和ddPCR试验稳定地检测了源于得到充分研究的细胞系(H1650和H1975)的EGFR突变。总的来说,ddPCR检出的突变体丰度要高于ARMS-Plus。这可能是由于ddPCR中野生型模板的非特异性扩增。
为了探讨ARMS-Plus灵敏度,我们分别用ARMS—PCR、ddPCR和ARMS-Plus总计分析了77个FFPE肿瘤样本的EGFR突变的状态。病理学家在测试前进行显微解剖以去除FFPE样本中可能会影响突变的定量的正常组织。ddPCR(35/77)发现的所有突变都与ARMS-Plus的结果一致,仅有一例丰度为0.5%的弱阳性L858R EGFR突变除外(ARMS-Plus检出而ddPCR未检出),总体一致性率为98.7%(Kappa=0.9739)。这些结果表明,ARMS-Plus是一种很有前景的高灵敏度EGFR突变检测试验。
在EGFR突变的定量分析中,不同类型的突变的结果也不同。L858R突变中,ARMS-Plus和ddPCR检测的丰度没有显著差异(P= 0.7334),结果显示出明显的正相关(R2=0.8081)。但是,ARMS- Plus和ddPCR检测19Del突变的结果存在显著的差异(P= 0.0002)。ARMS-Plus检测的19Del突变丰度与基于ddPCR方法的期望丰度不一致(R2=0.1012)。ddPCR检测19Del突变的双相探针覆盖区域明显大于ARMS-Plus (ddPCR:K749-S752和A755-D761区域,ARMS-Plus:e746_a750区域)。这或许可以解释两种方法检测19Del突变体丰度出现差异的原因,其中ddPCR的结果明显高于ARMS-Plus(中值=43.76% vs.25.26%)。需要更大的样本容量的后续研究来阐明ARMS-Plus评估19Del突变的丰度的性能。对于T790M突变,由于只鉴别了2份样品,因此没有执行分析来评估其与ddPCR和ARMS-Plus的相关性。
我们的研究存在一些局限性。首先,FFPE样本只能代表一种独特的肿瘤细胞克隆,不能反映所有肿瘤的遗传多样性。EGFR突变检测的高灵敏度分子筛查对于EGFR-TKI治疗而言存在过分宽泛的风险。因此,结合液体活检是更好的决策制定选择。事实上,ddPCR已经被证明在检测血浆样本中的EGFR突变方面具有足够的灵敏度。此外,还需要进一步研究探讨液体活检中ARMS-Plus的性能。其次,本研究的样本量较小,无法评估ARMS-Plus定量T790M突变的性能。第三,虽然进行了显微解剖来确保每个样本中>80%的肿瘤含量,但肿瘤含量的变化可能导致定量的不准确。
总而言之,ARMS-Plus提供了一种与ddPCR相比具有类似灵敏度和特异性的可靠且经济有效的定量测量工具,并可以用于肺腺癌患者的EGFR突变的评估并指导治疗方案选择。
本期“技术导航”栏目编译的文章仅供内部参考,版权归原作者及原刊物所有,作者如有问题,请致电本刊编辑部。




