下一代测序技术信息学 临床应用面临的挑战和策略
下一代测序技术(Next-generation sequencing,NGS)使用大规模并行测序平台已经极大地改变了基因组医学的模式。NGS系统具备的高通量能力导致测序数据呈指数积累,已经超出了我们当前全面管理和解释基因组信息的技术能力。测序费用的大幅度降低以及引入经济有效的台式测序仪进实验室引发了个体化用药的巨大需求,将离散的NGS数据整合进临床领域。尽管NGS平台仍处于技术成熟阶段,但受临床医生、病理学家、分子生物学家、科学家和临床管理人员的推动,该技术已经逐步渗入临床分子实验室。甚至连患者也意识到高通量基因组分析将以较低的价格为许多变异提供答案。考虑到当前临床信息学基础设施,NGS的迅速应用有很明显的操作不均衡性。因为这种颠覆性技术不可否认是通向临床基因组医学新时代的大门,必须在NGS生物信息学和临床信息学运行缝隙之间建立来桥梁,为下一代分子诊断提供长期可持续发展的环境。
Gullapalli及其同事于2012年发表了两篇同行评议的文章,描述了当代NGS技术并指出了一些相关的信息学问题在临床实施中的挑战。也简单地讨论了关于NGS分析的当代技术,如虚拟化技术和云计算。但是,近年不断获得新进展的NGS技术也为NGS数据管理提出了全新的挑战。而且分析学和云计算有了显著的进步,成为高通量基因组学的宝贵资源。在最近出版的“Next-Generation DNA Sequencing Informatics”一书中,作者对NGS技术做了全面的介绍。当然该书并没有涉及NGS在临床应用的操作。最近,下一代测序:临床检测II标准化的信息学工作组发表了临床NGS信息学的原则和指南,详细描述了NGS的设计、优化、确认和应用生物信息学传递途径检测种系序列变异的相关信息。但是该指南并没有涉及实验室工作流程和支持生物信息学应用的信息学基础设施。
因此,需要有帮助病理学信息学家在临床实验室中处理NGS的指导。本综述不仅概述了测序技术的现状,也就实验室工作流程信息学的各个方面和有关NGS分子检测的部署和维护的数据管理挑战做了详细的讨论。同时还基于已发表的科学数据和作者在处理其机构内问题的个人经验总结了当前某些瓶颈的解决办法和未来发展的范围。
一、测序技术:演变及现状
Frederick Sanger于1977年引入DNA测序技术,他描述了链终止法复制单链DNA片段(500-1000个碱基)的核苷酸序列。为了复制原始DNA序列,Sanger使用DNA多聚酶、一个短的寡核苷酸引物、延伸链和终止链核苷酸、化学修饰的核苷酸碱基、聚丙烯酰胺凝胶和示踪标记。随后在测序化学和方法学上的改进,如整合荧光标记的双脱氧核苷酸给终止序列染色(链终止法)、耐热DNA聚合酶催化的循环反应以及激光检测法——提高检测的灵敏度。电泳分离后复制的DNA片段产生一系列荧光信号峰,这些荧光信号峰包括了色谱中的目标核苷酸序列和跟踪文件,其转换成FASTA或FASTQ的文件格式,后者包括了碱基质量评分(Phred质量评分)。下一步需要将这些片段与参考基因组比对以定义基因组来源和确定变异。Sanger法是准确确定DNA序列的基准,差错率低于1/10000碱基。
下一代测序平台能从靶基因库中合成许多重叠的短DNA片段(50-400个碱基),通过在空间中将其分隔成数组然后平行复制。同时监控整合进新复制DNA链的核苷酸。通过这种方式,多次合成靶DNA。每个碱基被分析的最低次数是整合进重叠片段的次数,也就是覆盖范围。虽然使用NGS法合成单条DNA链比Sanger法(1/10000碱基)有更高的差错率(1/1000碱基),但是其覆盖范围广加上对序列变异有最低阈值要求,这使得与Sanger测序相比有更低的差错风险。正是这种在文本(序列)中保存分离信息和被测基因组的信息冗余的能力构成了该技术的高通量特点并赋予了其对生成的数据有空前的生物学洞察力。类似于过去30年半导体计算机芯片的发展,NGS也减少了基因组测序所花费的时间和费用,从而为促进基因组调查,为临床诊断应用提供了充足的保真性和分辨能力。
实验室台式测序仪的发展使得引入NGS技术到临床诊断中成为可能。罗氏454 GS Junior和Illumina MiSeq模型都是将其自己的旗舰全基因组测序仪微型化。相反,Thermo-Fisher Scientific是通过获得Ion Torrent技术而进入这一市场。这一技术结合了平行测序的各个方面,包括乳液PCR,但补充了一个金属氧化物半导体芯片来检测氢离子(电子信号)。这种方法不再需要化学发光染色、系列光学影像获取、机动的照像装置和巨大的储存空间以进行分析前文件的处理。DNA条码是独一无二的短核苷酸序列,其整合进了目的DNA片段中。这些DNA条码有固定的长度,且必需对待分析的靶DNA有唯一的识别能力。使用这些条码可以在一次测序反应中感应出多个样品,使得NGS成为一项经济有效的技术。
当前NGS工作流程的主要局限在于DNA片段库或扩增子库的准备过程烦锁费时。工程和自动化的改进得以降低人力、时间和该过程的复杂性从而满足临床对准确且快速的结果的需求。因此,认识到新兴的第三代测序技术通过直接检测DNA分子绕开了这些要求是十分重要的。Pacific Biosystems(门洛帕克,加利福尼亚)开发了一个在一个纳米光子室合成DNA链从而直接测序的平台,实时检测原始序列的复制情况。Oxford Nanopore(牛津,英国)开发了一个系统,能够检测DNA链中的单个DNA分子。虽然单分子检测系统的准确性尚不能与使用模板库的NGS匹配,但是阅读长链DNA及直接检测DNA的表观遗传学修饰的能力是这些方法重要的改进。
二、处理NGS数据的生物信息学工作流程的一般模式
最受欢迎的NGS技术是用于得到成千上万的相对短的序列读长(50-400个碱基对),与指定的基因组区域(靶序列)有许多重叠,或在全基因组中扩展(全基因组测序)。测序中读长真正形成的靶基因组的部分表示一次测序提供的覆盖程度。生物信息学流水线(图1)指的是一系列复杂且计算昂贵的数据分析过程,源于从未加工的NGS信号输出到信号处理和与参考基因组比对的一系列基因转变。典型的生物信息学流水线始于专门针对某一平台的算法从主要的荧光、电学发光或电流信号得到的连续的碱基识别。给每个预期的核苷酸碱基指定一个质量评分(Phred样的评分或Q评分),反应该碱基识别正确的统计学置信程度。该过程产生的测序读长以几种形式之一的文件形式储存(FASTQ, XSEQ, 未比对BAM或FASTA),有或者没有碱基质量评分信息。因为是某个平台专有的,因此这些质量评分与其它不同的测序系统间不可比。
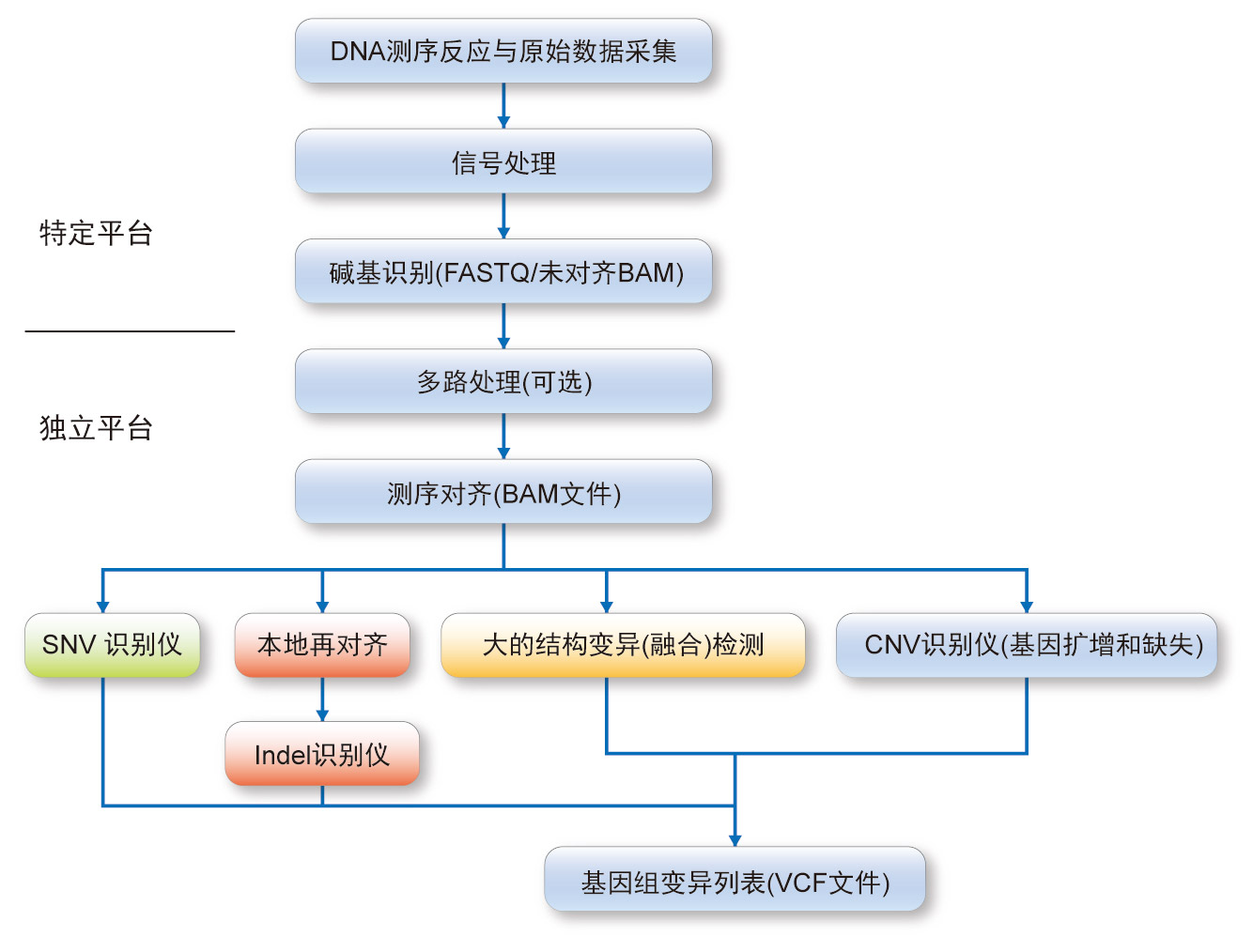
随后的分析涉及到对测序数据(一般是FASTQ)进行质量控制(QC)检查以评估读长分布、质量评分、GC含量、过表达、序列和k-mer含量。这些检查的目的是确定生成的序列是否有质量不佳的指示(如,该区域的GC碱基含量是否比预期的高很多或低很多)。FastQC是其中一个可用于该过程的工具。其它步骤,如适配器和质量不佳的序列的修饰,可能需要依赖于QC结果和流水线结构。QC检查阶段是位于在FASTQ文件中将重叠的读长与参考人类基因组(如GRCh37)比对之后的。好的比对算法能够克服重复序列的模糊和测序错误。某些对齐程序(开源和专用的)在性能规范上有差异并影响检测序列变异或特定基因组改变的能力,如短的插入/缺失(Indels)和在的结构变异。
在SAM/BAM文件中的对齐读长是多种应用的基础,如检测许多核苷酸变异(SNVs)、短Indels、大的结构改变、拷贝数变化和基因融合。当前存在许多开源和商业化应用,从变异识别仪到提供从对齐到寻找变异的整条流水线。一般使用本地测序对齐、复制标志和调整的碱基质量评分阈以提高变异识别仪的辨识力。但是,结构变异体和拷贝数量改变需要不同的处理流水线,该类流水线正处于发展和检测的阶段。建议使用一连串的算法在最大范围捕获变异体,然后基于二次确认后的最低共识阈值进行选择。这种方法可用于发现新变异体,但应校准并优化识别仪的灵敏度以对已建立的变异体进行快速扫描。测序变异体列表通常以许多形式呈现,如变异体识别形式(VCF)、基因组VCF(gVCF)和一般特征的格式(GFF3)。美国疾病控制和预防中心与许多合作伙伴正致力于VCF文件标准临床版本的工作。NGS生成的这3种文件类型(FASTA/FASTQ、SAM/BAM 和 VCF),VCF占据的存储容量最小,最容易读取。因此,VCF可能是最理想的用于在NGS仪器、实验室信息系统(LIS)和电子病历(EHR)间传递和储存基因组数据的文件。
三、临床NGS信息学工作流程
生物信息学流水线的一级和二级分析是NGS检测必不可少的一部分。数据分析算法与完善的测序硬件和化学一致需要继续改进。配置和优化的生物信息学流水线的桌面服务器试图使用户体验更加天衣无缝。诸如实验室的其它检测,一项新的试验引入到临床实验室需要与已存在的信息学设备有良好的互操作性。但是,临床信息学领域被台式NGS测序引入临床实验室强势攻占,没有足够的时间发展信息学策略以适应大量复杂的NGS数据,并将其无缝整合进现存的临床信息设备(如LIS和EHR)。置身于NGS技术的飞跃发展和NGS测序系统生成人类可读变异体文件的喜悦中时,很少有人注意到实验室工程流程的下游部分,如与现存信息系统的互操作性、患者和样品信息与测序数据的整合、报告生成、数据储存和传递以及质量保证(QA)。这些NGS测序运行的上游及下游元素对于在临床中成功建立以NGS为基础的检测是非常重要的。临床信息学与NGS技术间的鸿沟部分是由于现存的LIS和HER无法输入、传递和储存NGS数据,包括最小的VCF文件。但是,当前的鸿沟也有部分是由于NGS平台无法使用标准电子信息处理过程(如health level 7[HL7],卫生信息交换标准)来安全地接受、使用和传递卫生数据。
四、NGS临床信息学工作流程:关键元素、瓶颈
和实施策略
在实验室进行以NGS为基础的分子检测的临床信息学工作流程与标准实验室工作流程有许多相似之处。图2展示了临床环境下的信息流原理图。以下的讨论集中于与NGS检测有关关于实验室工作流程检验前、检验中和检验后组分的数据管理。
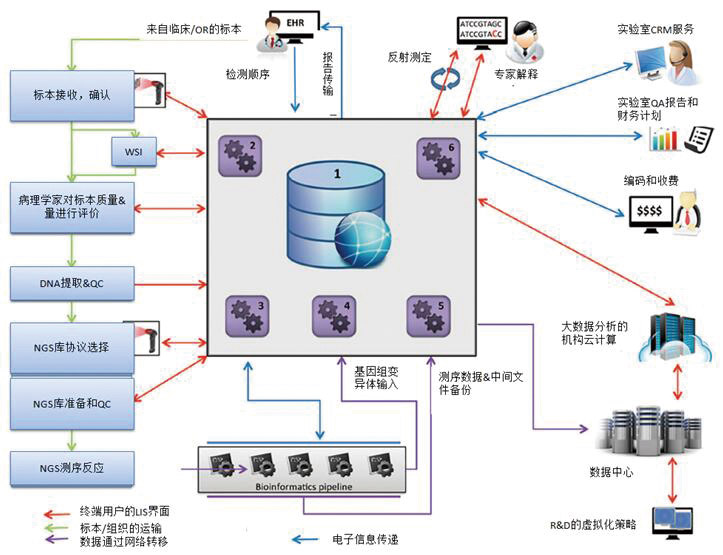
图2 NGS临床信息学工作流程
1.数据采集
在检测周期中,数据采取是指从实验室工作流程的其它结点获取信息并将其与样品或患者的其它数据点整合的过程。对于传统的临床实验室检测,大部分数据获取与检测的分析前阶段有关(如申请录入、样品接收等)。但是,NGS检测详细而复杂的流程在这一过程产生了一些有价值有QC和解释性数据,从而准确持续地获取这些数据点。
(1)申请接受和标本登记——与其它检测一样,临床NGS检测始于申请过程,伴随检测标本的接收。申请可从HER(内部或外部)、外部LIS和纸质申请单处获得。纸质申请单的效率最低。在计算机的医生申请录入的布署中,检测申请尤其需要改进。大型分子实验室,除了接收医生检测申请外,也处理许多其它客户端实验室的申请,可能使用或不使用电子申请过程。基于网络的客户门户网站管理检测申请在某些新的实验室信息管理系统(LIMS)中越来越受欢迎。临床决策支持算法也可以检测和改变提供者和实验室工作人员的申请减少不必要的重复检测申请。
(2)标本条形码——与之前讨论的DNA条码不同,物理条形码在检测过程中可以准确识别和追踪标本。与一维条形码相比,二维条形码在临床和部分分子实验室中有更好的效果。手写标签、登记和标本追踪非常费时而且很可能出现识别错误和进行错误的检测。使用物理条形码很好地解决这一问题。物理条码促进临床实验室工作流程的自动化。二维条形码系统可以在一小标签中整合更多信息,可能更适用于分子实验室使用的小的瓶子、管和流动池。在整个检测过程和报告过程中都可以使用条形码,尤其是当LIS和检测仪器支持的时候。支持NGS检测流程的多点数据获取要求可极大地促进条形码的使用。
(3)工作流程管理和样品追踪——一个设计良好紧密监控的实验室工作流程保证了检测结果的有效性和准确性。与其它临床实验室相似,NGS检测的应用也要求有信息学设备支持的工作流程以密切监控这一过程。某些流程结点可产生数据点到监控系统,如申请接受、标本登记、样品评价、核酸提取、库准备、芯片/流动池加载、测序反应、生物信息学分析、解释、报告和QA文件化。流程中的每个结点都是潜在的误差来源,这种误差可能传播到下游的流程中。因此,高度可靠的流程管理和追踪系统对临床NGS检测的成功应用是非常重要的。虽然流程追踪办法可从NGS仪器厂商处获得,但他们的范围局限于测序反应步骤和相关的分析。上游样品和患者信息管理、下游变异体的解释、说明和报告没有包括在内。理想的监控策略应该是获得实时数据。当然,这对监控系统和仪器间的互操作性有很高的要求。电子审核跟踪也是流程管理中非常重要的一部分,以促进故障排除。值得高兴的是,NGS测序仪器及其分析服务允许多个水平的数据通信,而不像传统的仪器如常规PCR仪。
(4)工作流程QC——对复杂NGS检测工作流程的监控和QC是分子实验室提供这类检测的重要一步。由于流程中有许多结点,因此优化这一过程以在繁忙的实验室中实现高效是非常重要的。过程改进方法,如精益和六西格玛,是优化实验室工作流程的有用工具,而且已经在其它实验室解剖设备和临床病理上展现出其有效性。尤其在分子实验室设备中,在亨利福特医院,精益过程用于改进分子诊断实验室的服务质量。在梅约诊所,精益过程也应用于微生物实验室以改进整体实验室性能并获得更高的成本效率。NGS检测工作流程比传统的分子检测更加复杂,为了利用这些过程改进工具,系统的离散的数据的获取是很重要的。
2.数据确认
应当注意罕见的系统性误差可能出现的原始信号输出中。这些误差可归因于样品和库准备以及每个平台的测序化学。已经有证据证明不同的DNA聚合酶可在G-C含量和测序长度引入系统偏倚。Illumina测序平台在三个一组的碱基识别中可能出现误读,可能与发夹结构有关。罗氏454和Ion Torrent测序平台也有碱基识别错误,随着均聚物的长度而增加,可能与不正确的流识别有关。Illumina测序仪产生更多替代识读现象,而罗氏454和Ion Torrent测序仪对单个碱基缺失有一定偏倚,尤其是在三核苷酸重复和均聚物中。但是,应该认识到平台提供者不断致力于改进其检测的质量和性能。
上述误差形式可归因于NGS中的“湿”和“干”测序成分。“湿”包括了一些过程,如标本处理和储存、核苷酸提取、扩增、库准备和测序读长的生成。“干”指的是计算机和生物信息学分析。为了监控潜在误差,有必要检测每个步骤的QC度量。生物信息学算法包括碱基识别、读长对齐和变异体识别生成的评分。这些质量评分反应了每个过程的准确性,并对生成的结果的总体质量有影响。因此,为了保证检测结果的可靠性,每个NGS运行的质量评分都应记录下来并进行分析。
除了监控NGS运行的质量外,在数据传输、电子信息发送和数据储存过程也需要进行数据确认。使用数字校验和就是一个例子。校验和是由某一给定数据源计算的一种小型的数据值,可用于传输和储存过程中确认数据文件的完整性。NGS检测时创建出各种分析和结果文件后,每个文件会分配一个校验值。当这些文件从实验室服务器移动或复制到集成数据中心或在实验室间分享时,就会在已接收的文件中重复追溯校验值并与进行比对以确认数据的完整性。
NGS测序仪确认数据输出的其中一个挑战是不同厂商间质量评分系统的异质性。一般每个厂商特异的NGS流水线的Q评分和QC度量是以算法为基础的。优化这些算法可提高变异体检测的准确性和敏感性。因此,NGS QC度量的阈值在不同的NGS平台间是不可比的。这在平台间确认过程、实验室间交换和能力验证结果不一致时提出了一个挑战。直至2015年,尚未出现一款软件能进行不同质量评分间的比对。
3.数据分析
如上所述,数据分析是NGS检测的核心。这一过程一般包括一些生物信息学步骤以将原始测序数据转换成合适的对齐序列,从而产生一系列变异识别。进行这些工作的硬件要求比较特殊,需要中央处理器(CPU)、低延时和空间足够大的记忆内存。有趣的是,除了原始测序数据外,流程中的许多结点(非测序数据)在背景中占据大量的数据。这类数据包括检测申请、预处理和核苷酸提取信息、整个检测循环中生成的QC度量、重复分析记录、检测结果、病理学说明、半结构化和非结构化的病理报告以及收费信息。NGS数据的解释说明是一个大数据的问题。虽然现在没有对大数据的共识或定量性定义,但是各种不同的定义的核心概念包括3种V(数据容量、多种数据形式和数据累积速度)。分析时经常需要选择性数据储存要求(如NoSQL数据库)和强大的数据处理功能以获得有意义的视角。传统计算机系统无法管理这种大型数据,这是美国国家标准与技术研究院(NIST)对大数据定义的潜在概念。Oracle的定义强调了大数据的非结构化特点,而Intel的定义则更关注数据容量。
除了在无数网络数据库解释变异这一挑战外,另一个问题是基于不断发展的认识和临床患者结局重新解释序列变异体。基于当前的研究,每天都有新知识不断涌现。因此,对某一疾病基因组变化的临床解释随着新的生物学观点而不断改变。到2015年为止,也没有对基因组变异体重新解释的要求的规程或建议。
挖掘大数据提取知识具有相当大的潜在效益。对于分子实验室,已经有针对大数据分析的良好设计。例如,在分子实验室中提取检测数据并进行一年或半年的回顾是一项相当艰巨的任务。这一任务通过包括了繁重的手工工作、产生一些电子表格而且相当费时。这类过程中使用电子表格和Access数据库是十分耗时的,而且经常遇到数据模型问题。虽然它某种程度上简化了数据提取过程,但是在大容量检测环境下的可提取性并不高。同样地,在实验室中实时回顾QA/QC活动、分析检测申请和进行成效分析也是一个很大的挑战。大型实验室和健康机构寻求商业智能系统,商业智能系统一般有合适的配置,可以储存有用的知识分析大量数据。由于大部分LIS没有这种能力,实验室可能希望购买第三方系统以进行这类分析。因此,厂商应致力于下一代LIS系统的开发。
用户关系管理是当代实验室运行的重要组分。用户关系管理不仅是及时发送高质量的分子病理报告,还在于实验室提供给患者和医疗服务人员的一系列服务。比如提供检测细节、检测前告知标本要求、有专门人员回答与检测相关的问题、提供分子检测和结果解释的专家咨询、不特征检测质量的前提下尽可能缩短周转时间、提供检测前和检测后的基因学咨询、危急值和报告延迟通知等。用户关系管理对分子实验室的公众形象是很重要的。实时和高级的分析解法可提供要求的报告。稳健的决策支持系统可促进用户关系管理,反过来,这是以实时数据分析为基础的。
4.数据报告
NGS检测结果的临床解释和报告是一个复杂的过程,在不同的实验室和机构间有明显差别。变异体解释和报告的复杂性源于NGS检测的变异体非常丰富,许多超过了当前我们对分子病理学的理解。NGS检测中发现的全新的基因组改变需要广泛的文献回顾、变异体数据回顾、基因型与表型的关系和临床试验回顾。解释的细节,包括分析工具和算法,超过了本次综述的范围。此外,这一部分讨论将聚集于报告NGS数据和工具的临床信息学对这一过程的促进作用。
基因组数据解释和报告生成是紧密联系的过程,因此需要提供信息学策略以这些任务联系起来。当前,变异体解释使用许多软件检索释文和基因组数据。这些步骤有一部分是人工进行的。随后的报告生成,也主要是人工处理过程,耗时且容易出现错误。为了避免这一问题,应进一步发展LIS以处理原始基因组数据和合成可读性报告。当复杂报告需要从LIS传送到HER时,另一个问题就出现了。直至2015年,几乎没有什么信息学策略能处理NGS检测结果在LIS和EMR间的互通性。
5.互操作性
互操作性指的是不同的信息系统(如LIS、EHR)和软件(如仪器固件、中间件)间互相通信和交换数据的能力。对NGS而言互操作性是个重要的问题,因为大部分文库制备装置、测序仪器和软件并不是为临床网络环境而设计的。实现“即插即用”互操作性的挑战取决于不断增长的技术改进和软件升级。理想情况下,分子信息系统和相关的设备应被设计用于支持句法的互操作性,如使用HL7实现LIS和EHR的交流(图3)。
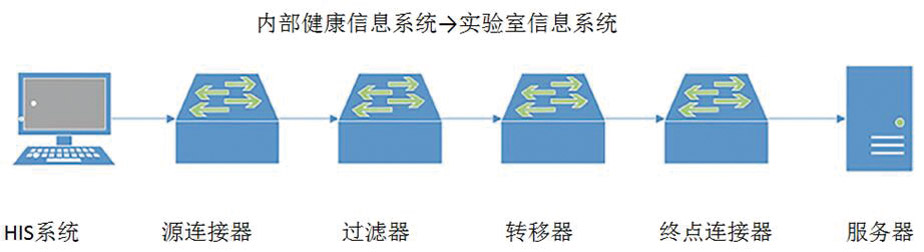
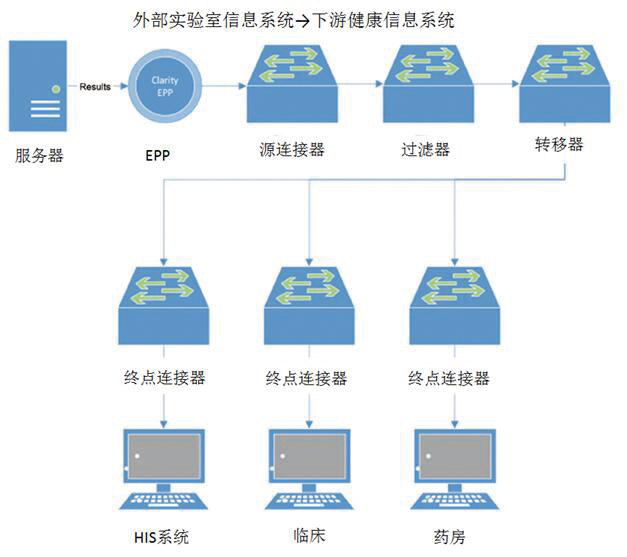
图3 实验室信息系统与健康信息系统的交流
HL7是电子卫生(健康)信息传递最常用的信息标准,电子健康信息包括临床实验室数据、医院与实验室间的信息系统。虽然HL7是临床和解剖病理数据传递的一个约定俗成的标准,但是它尚未完全优化用于处理特定等级的信息,如分子和数字图像数据。HL7临床基因组工作组在这一领域已取得一定进展,但是大部分仍有待进一步研究。为了处理复杂数据的呈现问题,2005发布了HL7v3版本。这个版本使用可扩展标本语言(XML)和XML模式定义(XSD)表示健康信息数据。虽然比HL7的第二个版本更冗长,但是它是计算机可读的,而且允许呈现复杂和分层数据。XML使用网络服务可用于表示和分享基因组数据。HL7临床基因组工作组正致力于为离散型基因组数据在不同的机构和临床信息系统的呈现和传递建立标准。
JavaScript 对象表示法(JSON)是一种新的很受欢迎的信息格式。这种格式尤其适合于用表述性状态转移(REST)界面来呈现和报告基因组数据。
五、数据管理
1.数据储存
数据大小随着基因组测序范围的扩大而增加。大部分临床分子实验室并没有相应的基础设施以有效安全地储存这样庞大的数据。虽然储存成本越来越便宜,但是与克莱德法则一致,新测序数据的产生速度更快。
某些台式测序仪与7-11GB的分析服务器相连,这对于某些小到中型的基因谱检测已经足够。便携式外部存储器是临床实验室常用的一种媒介,可用于备份庞大的检测结果数据。云储存,是当前实现大型NGS数据管理最合适的方式。
尽管数据存储越来越便宜而高效,但是其能力是有限的。NGS会生成大量文件类型,实验室需要对这些文件类型进行分类、储存。此外,生物信息学组分包括原始数据处理和碱基识别是相对稳定的,很少需要重复。相反,中间文件(如BAM,FASTQ)比原始数据文件相对来说小一些。同时还应考虑储存时间长度。与中间数据和下游结果文件不同,原始数据一旦存档,在一段时间后应清除。匹兹堡大学医学中心的分子和基因组病理实验室,原始数据文件在3个月后会自动清除[55]。
重要数据的意外损失会损害机构的生意和发展。因此,在设置NGS检测的前期准备工作中,实验室应在IT支持人员的帮助下制定备份计划和灾难恢复计划,以确保灾难性事件发生时复原相关数据。
2.数据的安全性和完整性
在临床NGS检测中使用云计算技术的挑战之一在于数据隐私。所有临床NGS都必须取得美国临床实验室改进法案的执照,因此必须满足针对分子基因检测和NGS检测的特定要求。在美国,健康保险流通与责任法案(HIPAA)制定了关于电子健康信息的安全性的相关规定,所有相关机构必须服从。根据经济和临床健康信息技术法案(HITECH)和随后的HIPAA公共规则,要求所有相关机构报告受保护的健康信息的违背情况,这不仅对患者有负面影响,也会造成相关机构的名誉和收入损害。基因组数据面临的这种独特的挑战是因为大型基因组数据(如全基因组)可以准确地实现个体识别,尤其是在当今这种互相连接的,信息丰富的,有高能量测序系统的环境中。作为概念验证,Gymrek及其同事演示了从一个公开的基因组资源库和基因家系数据库中提取个人识别信息的能力。为了处理这一问题,遗传信息不歧视法案(GINA)于2008年开始执行,阻止保险公司和雇主基于遗传信息对某些个体的歧视。
虽然云计算为临床NGS检测提供了最完整的数据管理策略,但如上所述,它在健康信息保护方面面临着具大的挑战。首要问题围绕在安全、控制和责任上。许多大型云服务器提供者(CSP)的数据中心有最先进的物理和电子安全性,这样的数据中心比机构自己的数据中心好。但是,将这种技术应用于临床基因组和健康护理数据仍是比较新的。因此,许多用户不愿意为了无法预料的安全性问题而使用CSP。此外,CSP一般在全球的多个地理位置上有多个数据中心。这甚至比“云堆叠”更复杂,因为多个CSP提供不同层面的支持。因为国家健康护理政策和数据隐私规则在跨边界时是不适用的,所以云端的临床NGS数据可能会违反某一地区的隐私规程。
为了提高用户满意度,CSP正在寻求符合国家和国际认可的安全和审计标准,如ISO/IEC 27001、 SOC1/SSAE 16/ISAE 3402、HIPAA、国防部信息安全认证和认可过程(DIACAP)、联邦信息安全现代法案(FISMA)和服从安全问责(CSA)认证。强烈建议相关组织或个人在做知情决策以前应寻求云计算技术评论和理解CSP技术、数据声明和提供者。CSP服务的可靠性、运行中断和数据遗失策略,以及服务中止时的数据删除策略都是应当考虑的重要内容。
六、信息技术基础设施
1.网络(数据通信)
对大规模基因组数据异地归档的网络宽带要求是一个常见的信息学瓶颈问题。许多健康护理系统的网络宽带在10-100Mbps之间,这对于大型数据而言并不是最优的。高宽带网络设施使用光纤技术,速度可达到10Gbps,促进高通量NGS数据在异地间进行转移。这种网络基础设施比较昂贵。其它可选择的策略包括在非高峰时监控预定的备份也是合理的。值得注意的是不论使用哪种基础设施,数据转移时使用合适的数据安全工具是非常重要的。
2.计算的要求
不论是否整合了病案元数据的基因组数据都是复杂且庞大的,需要高通量的处理过程。因此,合适的计算机资源是临床NGS检测必不可少的一部分。在临床分子实验室中,持续技术改进和规章制度间的协调如果没有稳健的IT基础设施的话是很难实现的。庆幸的是,针对大数据管理的新型计算机技术的应用已经为高通量基因组的临床实验室的全面应用提供了坚实的基础。
3.集群计算
云计算的核心在于在网络环境中管理多重现成计算机硬件以实现资源紧密型数据处理,其效率和能力大于任何一个工作站。云中的每台独立的计算机通常视为一个节点。例如,紧密型过程的计算机处理,如测序对齐,可被主节点分成多个小的平行的过程,并分配给云内的每个独立的从属节点。所有分配的工作都完成以后,每个独立分析的结果将整合起来并通过主节点返回给用户。云计算最大的优势之一在于可扩展性,在不需要改变已经存在的基础设施的情况下通过增加更多节点而提高计算机能力。Hadoop和MapReduce是使用专门设计的文件系统和简单的程序模型促进大数据分析容错的常见架构。其分析优势在于减少大型数据集在云间的转移。
4.网格和云计算
网格计算通过在一个广泛的地理区域使用网络计算机和云而提供增强的联合的计算能力。同时也引入了计算资源配置这一概念,处理多种数据分析工作从而使计算资源得到最优化。NIST将云计算定义为“使无处不在的便捷的按需网络访问与共享的可配置的计算资源(如网络、服务器、储存、应用和服务)相连接的模型,能通过最少的管理工作或服务提供者交互实现快速配置和发布”。云计算支持不同的计算要求。服务一般基于终端用户对软件(SaaS)、平台(PaaS)或基础设施(IaaS)的要求而分层(图4)。例如,厂商可以在云端配置一个LIMS,并通过SaaS提供给用户。下一代测序检测要求,如数据储存和分析,通过使用云服务可以得到最好的解决。关于基因组数据的管理,云计算具有许多优势,如按需可扩展性、负载平衡和动态资源分配、虚拟化对策、多重操作系统部署、可靠的数据备份和资源共享。
云计算可部署成以下4种描述的模型之一:公共云、私有云、混合云和社区云(图5)。私有云是为服务封闭用户群体的。大型学术机构经常使用这种模型(机构云),其存在于机构防火墙内部。相反,公共云是所有用户都可获得的,通常需要交纳使用费用。混合云模型中,数据中心位于机构防火墙内部,但为机构外的用户提供一个亚服务云。社区云的架构与公共云类似,但是,其服务限于受商业合同或协议约束的组或社区的用户。
生物医学研究领域和学术生物信息学的调查发现NGS技术是云计算的早期采用者。最近,生物信息学策略不断发展,已经广泛运用云架构用于NGS数据的分析。
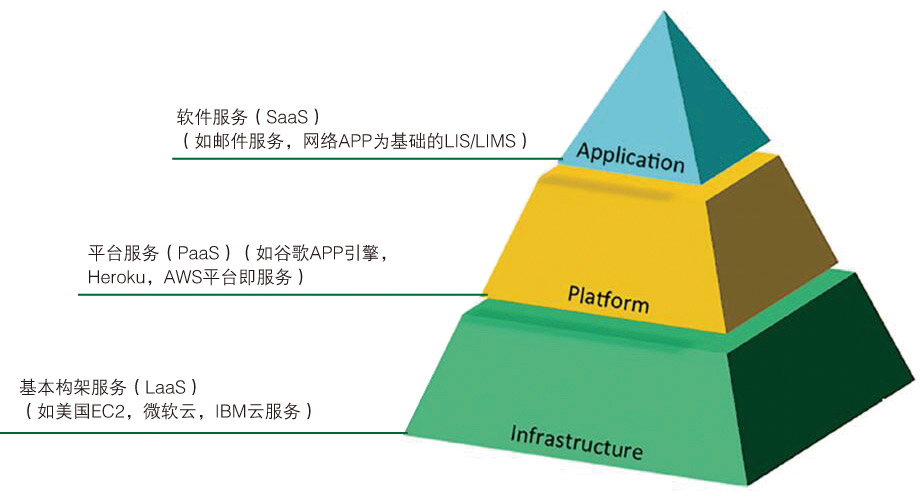
图4 分层布置的云服务
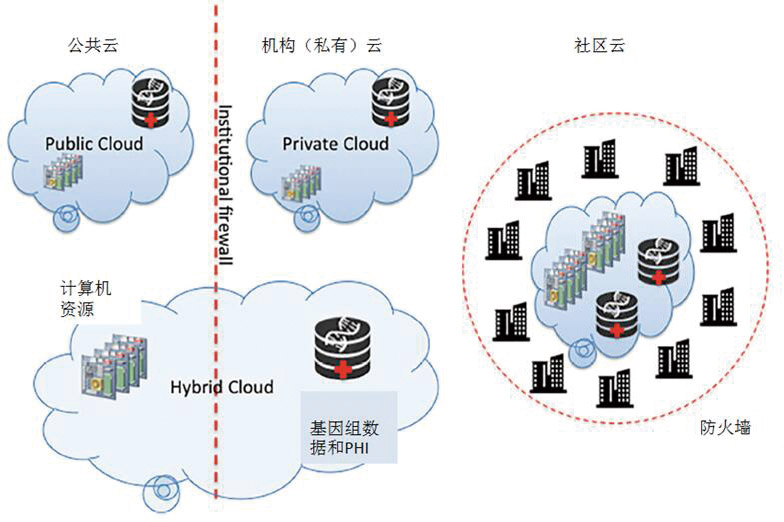
图5 云部署模型
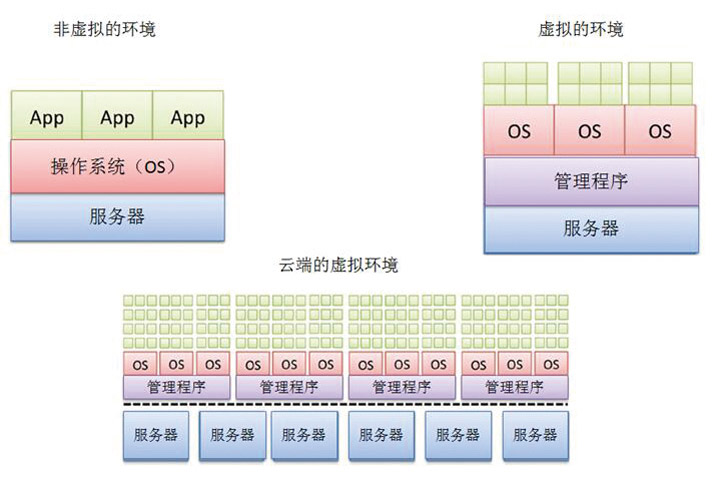
图6 虚拟化
5.虚拟化
虚拟化是一种可以从底层硬件中提取OS的软件技术,通常用一个管理程序,它可以对服务器硬件进行有效使用和管理(图6)。该技术是云计算的核心之一,使得云服务更加划算、更加环保。完善的软件系统可辅助云端进行硬件资源使用和虚拟计算机的管理。随着技术的改进,可在OS以上的多个水平实现虚拟化。最大程度地的简化了云计算机服务的供给,而且用户可以通过网络自己安装虚拟环境而不需要服务提供者的帮助。通过从CSP上建立一个大型机器或一组虚拟化机器(虚拟集群)可以在几分钟内提供NGS分析流水线。这种虚拟化计算环境可在用户间共享,培养一个基因组研究的协同合作的环境。
七、分子的LIS及其未来前景
前面的内容中,我们回顾了NGS检测在临床分子实验室中独一无二的环境和要求。NGS检测的某些方面要求使用统一的信息管理系统(IMS)以确保实验室工作流程的可靠性、安全性和有效性。NGS检测的IMS具有一些特点,许多在当前的商品化LIS/LIMS产品中无法实现。传统的LIMS是设计用于管理主要的实验室工作流程,如标本追踪、库存管理、检测菜单管理和协议设计工具,和支持有限的报告功能。而LIS,提供更多综合报告的能力(包括传真报告)、与其它电子信息系统的互通(如EHR,其它LIS/LIMS)、检测顺序管理和收费信息管理。实验室进行NGS检测时,需要满足数据安全、收费、正式报告和与其它临床系统的互通等额外要求,这要求IMS同时具备LIMS和LIS的特点。这种独特的需要证明了分子LIS(M-LIS)的概念,处理综合的基因组和健康信息数据。
当前临床实验室设置的主要挑战是需要一个动态的工作流程,与实验室NGS检测的增加和标本量的扩大相协调。在这个动态环境中实验室QA是很重要的。同时质量保证活动也依赖于实验室进行的合适的数据分析。实验室工作流程的改变和测序技术的持续发展,要求实验室信息学要求的可塑性。M-LIS厂商应基于实验室的动态需要不断改进,和/或使得开放的软件架构能允许实验室进行自定义修改。
八、结论
NGS引入到临床分子实验室模糊了生物信息学和临床信息学间的界限。以NGS为基础的检测的精准医学的使用在医学领域得到了广泛的应用。随着该领域的发展,信息学挑战源源不断地出现。包括新的分析流水线、图像管理系统、患者隐私保护和实验室管理。下一代测序技术及相关数据,如果管理得当的话,将重新定义医学实践。为了保持临床分子诊断的快速发展,智能计算机技术的应用是非常关键的。在不久的将来,计算机病理学家将通过大数据和现代计算机的常规工作为患者提供诊断和治疗服务。
参考文献
1. Gullapalli RR, Lyons-Weiler M, Petrosko P, Dhir R, Becich MJ, LaFramboise
WA. Clinical integration of next-generation sequencing technology. Clin Lab Med. 2012;32(4):585-599.
2. Gullapalli RR, Desai KV, Santana-Santos L, Kant JA, Becich MJ. Next generation sequencing in clinical medicine: Challenges and lessons for pathology and biomedical informatics. J Pathol Inform. 2012;3:40.
3. Brown SM, ed. Next-Generation DNA Sequencing Informatics. 2nd ed. New York, NY: CHS Press; 2015.
4. Gargis AS, Kalman L, Bick DP, et al. Good laboratory practice for clinical next-generation sequencing informatics pipelines. Nat Biotechnol. 2015;33(7): 689-693.
5. Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. 1977;74(12):5463-5467.
6. Marsh M, Tu O, Dolnik V, et al. High-throughput DNA sequencing on a capillary array electrophoresis system. J Capillary Electrophor. 1997;4(2):83-89.
7. McBride LJ, Koepf SM, Gibbs RA, et al. Automated DNA sequencing methods involving polymerase chain reaction. Clin Chem. 1989;35(11):2196-2201.
8. Prober JM, Trainor GL, Dam RJ, et al. A system for rapid DNA sequencing with fluorescent chain-terminating dideoxynucleotides. Science. 1987;238(4825):336-341.
9. Mardis ER. Next-generation sequencing platforms. Annu Rev Anal Chem (Palo Alto Calif). 2013;6:287-303.
10. Shendure J, Ji H. Next-generation DNA sequencing. Nat Biotechnol. 2008;26(10):1135-1145.
11. Head SR, Komori HK, LaMere SA, et al. Library construction for nextgeneration sequencing: overviews and challenges. Biotechniques. 2014;56(2):61-64, 66, 68, passim.
12. Metzker ML. Sequencing technologies - the next generation. Nat Rev Genet. 2010;11(1):31-46.
13. Harismendy O, Ng PC, Strausberg RL, et al. Evaluation of next generation sequencing platforms for population targeted sequencing studies. Genome Biol. 2009;10(3):R32.
14. Foquet M, Samiee KT, Kong X, et al. Improved fabrication of zero-mode waveguides for single-molecule detection. J Appl Physics. 2008;103(34301):1-9.
15. Clarke J, Wu HC, Jayasinghe L, Patel A, Reid S, Bayley H. Continuous base identification for single-molecule nanopore DNA sequencing. Nat Nanotechnol. 2009;4(4):265-270.
16. Andrews S. FastQC: a quality control tool for high throughput sequence data. 2010. http://www.bioinformatics.babraham.ac.uk/projects/fastqc/. Accessed November 1, 2015.
17. Pabinger S, Dander A, Fischer M, et al. A survey of tools for variant analysis of next-generation genome sequencing data. Brief Bioinform. 2014;15(2):256-278.
18. Shang J, Zhu F, Vongsangnak W, Tang Y, Zhang W, Shen B. Evaluation and comparison of multiple aligners for next-generation sequencing data analysis. Biomed Res Int. 2014;2014:309650.
19. Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 2009;25(14):1754-1760.
20. Li H, Durbin R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 2010;26(5):589-595.
21. Langmead B, Trapnell C, Pop M, Salzberg SL. Ultrafast and memoryefficient alignment of short DNA sequences to the human genome. Genome Biol.
2009;10(3):R25.
22. Li R, Yu C, Li Y, et al. SOAP2: an improved ultrafast tool for short read alignment. Bioinformatics. 2009;25(15):1966-1967.
23. Li H, Ruan J, Durbin R. Mapping short DNA sequencing reads and calling variants using mapping quality scores. Genome Res. 2008;18(11):1851-1858.
24. Novocraft Technologies Sdn Bhd. Novoalign: powerful tool designed for mapping of short reads onto a reference genome from Illumina, Ion Torrent, and 454 NGS platforms. http://www.novocraft.com/products/novoalign/. Accessed November 5, 2015.
25. Xin H, Lee D, Hormozdiari F, Yedkar S, Mutlu O, Alkan C. Accelerating read mapping with FastHASH. BMC Genomics. 2013;14(suppl 1):S13.
26. McKenna A, Hanna M, Banks E, et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 2010;20(9):1297-1303.
27. Koboldt DC, Zhang Q, Larson DE, et al. VarScan 2: somatic mutation and copy number alteration discovery in cancer by exome sequencing. Genome Res. 2012;22(3):568-576.
28. Evani US, Challis D, Yu J, et al. Atlas2 Cloud: a framework for personal genome analysis in the cloud. BMC Genomics. 2012;13(suppl 6):S19.
29. Cibulskis K, Lawrence MS, Carter SL, et al. Sensitive detection of somatic point mutations in impure and heterogeneous cancer samples. Nat Biotechnol. 2013;31(3):213-219.
30. Spencer DH, Tyagi M, Vallania F, et al. Performance of common analysis methods for detecting low-frequency single nucleotide variants in targeted next generation sequence data. J Mol Diagn. 2014;16(1):75-88.
31. Grasso C, Butler T, Rhodes K, et al. Assessing copy number alterations in targeted, amplicon-based next-generation sequencing data. J Mol Diagn. 2015;17(1):53-63.
32. Liu B, Morrison CD, Johnson CS, et al. Computational methods for detecting copy number variations in cancer genome using next generation sequencing: principles and challenges. Oncotarget. 2013;4(11):1868-1881.
33. Snyder ML, Carter A, Jenkins K, Fantz CR. Patient misidentifications caused by errors in standard bar code technology. Clin Chem. 2010;56(10):1554-1560.
34. Condel JL, Sharbaugh DT, Raab SS. Error-free pathology: applying lean production methods to anatomic pathology. Clin Lab Med. 2004;24(4):865-899.
35. Riebling N, Tria L. Six sigma project reduces analytical errors in an automated lab. MLO Med Lab Obs. 2005;37(6):20, 22-23.
36. Schweikhart SA, Dembe AE. The applicability of Lean and Six Sigma techniques to clinical and translational research. J Investig Med. 2009;57(7):748-755.
37. Elder BL. Six Sigma in the microbiology laboratory. Clin Microbiol Newsletter. 2008;30(19):143-147.
38. Cankovic M, Varney RC, Whiteley L, et al. The Henry Ford production system: LEAN process redesign improves service in the molecular diagnostic laboratory: a paper from the 2008 William Beaumont hospital symposium on molecular pathology. J Mol Diagn. 2009;11(5):390-399.
39. Mitchell PS, Mandrekar JN, Yao JD. Adoption of lean principles in a highvolume molecular diagnostic microbiology laboratory. J Clin Microbiol. 2014; 52(7):2689-2693.
40. Nakamura K, Oshima T, Morimoto T, et al. Sequence-specific error profile of Illumina sequencers. Nucleic Acids Res. 2011;39(13):e90.
41. Bragg LM, Stone G, Butler MK, Hugenholtz P, Tyson GW. Shining a light on dark sequencing: characterising errors in Ion Torrent PGM data. PLoS Comput Biol. 2013;9(4):e1003031.
42. Boland JF, Chung CC, Roberson D, et al. The new sequencer on the block: comparison of Life Technology’s Proton sequencer to an Illumina HiSeq for whole-exome sequencing. Hum Genet. 2013;132(10):1153-1163.
43. Ward JS, Baker A. Undefined by data: a survey of big data definitions. arXiv:1309.5821. 2013.
44. Health Level 7 International. HL7 version 2 implementation guide: clinical genomics; fully LOINC-qualified genetic variation model (US realm). 2013. http://www.hl7.org/implement/standards/product_brief.cfm?product_id¼23. Accessed
November 1, 2015.
45. Deckard J, McDonald CJ, Vreeman DJ. Supporting interoperability of genetic data with LOINC. J Am Med Inform Assoc. 2015;22(3):621-627.
46. Chute CG, Ullman-Cullere M, Wood GM, Lin SM, He M, Pathak J. Some experiences and opportunities for big data in translational research. Genet Med.
2013;15(10):802-809.
47. Izzo M, Mortola F, Arnulfo G, Fato MM, Varesio L. A digital repository with an extensible data model for biobanking and genomic analysis management. BMC Genomics. 2014;15(suppl 3):S3.
48. Lindenbaum P, Redon R. mod_bio: Apache modules for next-generation sequencing data. Bioinformatics. 2015;31(1):112-113.
49. Yates A, Beal K, Keenan S, et al. The Ensembl REST API: Ensembl data for any language. Bioinformatics. 2015;31(1):143-145.
50. Louis DN, Feldman M, Carter AB, et al. Computational pathology: a path ahead. Arch Pathol Lab Med. 2016;140(1):41-50.
51. Hartzler A, McCarty CA, Rasmussen LV, et al. Stakeholder engagement: a key component of integrating genomic information into electronic health records. Genet Med. 2013;15(10):792-801.
52. McGuire AL, Basford M, Dressler LG, et al. Ethical and practical challenges of sharing data from genome-wide association studies: the eMERGE Consortium experience. Genome Res. 2011;21(7):1001-1007.
53. Walter C. Kryder’s law. Sci Am. 2005;293(2):32-33.
54. Stein LD. The case for cloud computing in genome informatics. Genome Biol. 2010;11(5):207.
55. Roy S, Mitchell R, Burdelski G, et al. Informatics infrastructure requirements for clinical next generation sequencing: challenges and solutions. abstracts: pathology informatics 2014. J Pathol Inform. 2014;5:S13.
56. Aziz N, Zhao Q, Bry L, et al. College of American Pathologists’ laboratory standards for next-generation sequencing clinical tests. Arch Pathol Lab Med. 2015;139(4):481-493.
57. New York State Department of Health. ‘‘Next Generation’’ Sequencing (NGS) guidelines for somatic genetic variant detection. March 2015. http://www.wadsworth.org/labcert/TestApproval/forms/NextGenSeq_ONCO_Guidelines.pdf. Accessed December 15, 2015.
58. The Health Insurance Portability and Accountability Act of 1996 (HIPAA). Pub L No. 104-1911996.
59. American Recovery and Reinvestment Act of 2009 (ARRA): Division A: Title XIII—Health Information Technology, and Division B: Title IV—Medicare and Medicaid Health Information Technology; Miscellaneous Medicare Provisions (collectively known as the Health Information Technology for Economic and Clinical Health or HITECH Act). Pub L No. 111-52009.
60. Modifications to the HIPAA Privacy, Security, Enforcement, and Breach Notification Rules Under the Health Information Technology for Economic and Clinical Health Act and the Genetic Information Nondiscrimination Act; Other Modifications to the HIPAA Rules; Final Rule (HIPAA Omnibus Rule). Vol 45. CFR Parts 160 and 1642013.
61. Rodriguez LL, Brooks LD, Greenberg JH, Green ED. Research ethics: the complexities of genomic identifiability. Science. 2013;339(6117):275-276.
62. Gymrek M, McGuire AL, Golan D, Halperin E, Erlich Y. Identifying personal genomes by surname inference. Science. 2013;339(6117):321-324.
63. Regulations Under the Genetic Information Nondiscrimination Act of 2008; Final Rule 2010;75(216). 29 CFR Part 1635. http://www.gpo.gov/fdsys/pkg/FR-2010-11-09/pdf/2010-28011.pdf. Accessed November 1, 2015.
64. Dove ES, Joly Y, Tasse AM, et al. Genomic cloud computing: legal and ethical points to consider. Eur J Hum Genet. 2015;23:1271-1278.
65. Dean J, Ghemawat S. MapReduce: Simplified data processing on large clusters in OSDI’04. Paper presented at: Sixth Symposium on Operating System Design and Implementation; December 5, 2004; San Francisco, CA.
66. Mohammed EA, Far BH, Naugler C. Applications of the MapReduce programming framework to clinical big data analysis: current landscape and future trends. BioData Min. 2014;7:22.
67. Mell P, Grance T. The NIST Definition of Cloud Computing. Gaithersburg, MD: National Institute of Standards and Technology; 2011. Special publication 800-145.
68. Heath AP, Greenway M, Powell R, et al. Bionimbus: a cloud for managing, analyzing and sharing large genomics datasets. J Am Med Inform Assoc. 2014; 21(6):969-975.
69. Angiuoli SV, Matalka M, Gussman A, et al. CloVR: a virtual machine for automated and portable sequence analysis from the desktop using cloud computing. BMC Bioinformatics. 2011;12:356.
70. Li J, Doyle MA, Saeed I, et al. Bioinformatics pipelines for targeted resequencing and whole-exome sequencing of human and mouse genomes: a virtual appliance approach for instant deployment. PLoS One. 2014;9(4):e95217.
71. Karczewski KJ, Fernald GH, Martin AR, Snyder M, Tatonetti NP, Dudley JT. STORMSeq: an open-source, user-friendly pipeline for processing personal genomics data in the cloud. PLoS One. 2014;9(1):e84860.




