晚期肺癌患者化疗有效性 和毒性的实时监测
摘 要
背景:实体瘤疗效评价标准(RECIST)和常见不良反应事件评价标准(CTCAE)被用于评估晚期肺癌患者的化疗效果和毒性。然而,目前尚无可以实时、同步评价化疗结果的指标。我们的目的是使用一种新型同步方法,来评价晚期肺癌患者化疗后的肿瘤反应性和毒性。
结果:我们纳入了316例接受顺铂治疗的晚期肺癌患者并对其随访3年。在每个化疗周期开始前后收集患者血浆样本。我们定量检测血浆总DNA含量和APC/RASSF1A基因的甲基化水平,并对以下四个参数进行了评估:化疗前的甲基化水平(meth0h),化疗后24小时的甲基化水平(meth24h),化疗前的血浆总DNA含量(DNA0h)以及化疗后24小时的血浆总DNA含量(DNA24h)。用至少一种基因的meth24h>meth0h预测肿瘤细胞对顺铂的反应性时,预测正确率为82.4%。另外,DNA24h/DNA0h≤2的患者化疗的副反应(毒性)较轻。因此,meth24h>meth0h和DNA24h/DNA0h≤2被视为肿瘤反应性更好和不良事件更少的评判标准,二者联合使用的预测正确率可达84.7%。
结论:血浆总DNA和血浆APC/RASSF1A甲基化的联合、定量检测,是评价晚期肺癌患者化疗效果的实时、同步、快速的监测指标,可以作为制定化疗效果评价指南时的参考或补充。
关键词:高甲基化,晚期肺癌,循环DNA,APC,RASSF1A
背 景
晚期肺癌患者由于在诊断时已发生转移,因此常常需要化学治疗法。但是,肺癌仍旧具有高死亡率和低5年生存率,其原因可能是对化疗方案不敏感以及缺乏有效手段来监测化疗效果[1-9]。若干年前曾有学者提出了实时凋亡监测的观念,其主导思想是认为有必要寻找一种实时监测化疗效果的手段[10]。临床常用RECIST指南和肿瘤标志物来评估化疗效果。但是这些手段提供有效信息的用时较长,因此并不是十分敏感的指标。所以,目前缺乏能够快速评价化疗效果的指标。
总循环DNA来源于坏死/凋亡的正常或肿瘤细胞。在正常人,总循环DNA含量处于低水平,主要来自少量凋亡的血液有核细胞[11]。一旦正常细胞和肿瘤细胞被化疗药物破坏,总循环DNA会明显升高,其中正常细胞来源的循环DNA可以反映化疗的副反应。由于基因高甲基化是肿瘤细胞的特异性[12-15],因此肿瘤细胞的大量破坏会导致基因甲基化水平的升高。
众多研究都报道了抑癌基因甲基化与肿瘤患者预后的关系。但是,很少有研究关注可同时监测化疗后肿瘤反应和毒副反应的指标。我们的研究主要是将APC/RASSF1A甲基化和总血浆DNA检测相结合,为化疗有效性和毒性的评估提供新的策略。
材料与方法
1、细胞培养、MTT检测及培养上清收集
1.5×104 cells/100μl的人肺腺癌A549细胞系接种于96孔板。培养24小时后,每孔加入顺铂,使其终浓度分别为5 mg/ml、0.5mg/ml和0.05mg/ml。对照孔不加顺铂。收集加药前以及加药后6、12、24、48和72小时的培养上清。收集完上清后每孔加20μl MTT(5mg/ml)再培养4小时。再加入10% HCL-SDS(100μl)后,上机比色(570nm)。计算细胞抑制率:[(对照孔吸光度-试验孔吸光度)/对照孔吸光度]×100%。
2、肺癌裸鼠模型、血液和组织标本收集
24只裸鼠用于该部分实验。8只为正常对照,其中死亡2只。另外16只后肢注射2×106个A549细胞来建立肺癌模型。25天后,当所成瘤直径超过5mm,16只荷瘤鼠随机分为两组,连续3天给予瘤内注射:8只注射顺铂+生理盐水,另8只仅注射生理盐水。注射后24、48和72小时,收集全血和癌组织。全血在4℃两步法离心(3,000rpm,10min和16,000rpm,10min)后得到血浆[19]。癌组织制片并HE染色后镜下观察。
3、晚期肺癌患者
收集2007年10月-2009年6月在南京医科大学第一附属医院肿瘤科住院的IIIb和IV期肺癌患者。所有患者随访至2010年11月。入选标准:(1)组织病理学证实为肺癌;(2)根据AJCC分期新诊断的患者;(3)年龄在18-80岁;(4)有转移并无法行手术者;(5)病灶可测量大小;(6)ECOG评分为0-2;(7)预测的生存时间>3月;(8)未曾化疗;(9)铂类化疗方案至少进行2个疗程。排除标准:(1)其他肿瘤病史;(2)接受过手术、化疗或放疗;(3)拒绝多次抽血者。所有患者知情同意。该研究得到南京医科大学第一附属医院伦理委员会批准。216人纳入training study,另100人纳入validation study。
4、化疗有效性和毒性评估
所有患者随访了至少4个疗程。化疗方案为IP、DC或GP。首次化疗后50-60天,临床依据RECIST1.1指南判断化疗的有效性。主要评估内容为:有效反应率(ERR)、肿瘤标志物(首次化疗后2月的结果)及总生存期(OS)。有效反应包括完全缓解(CR)和部分缓解(PR)。非有效反应为疾病稳定(SD)和疾病进展(PD)。肿瘤标志物包括CEA、NSE和CY21-1。毒性评估主要包括CTCAE指南中15个常见不良事件。
5、培养上清和血浆中DNA提取
根据BilaTest DNA Kit试剂盒说明书,提取200μl培养上清或血浆中DNA。
6、总血浆DNA检测
双重实时定量PCR法检测总血浆DNA浓度。
7、APC /RASSF1A启动子甲基化水平检测
双重qMSP法检测两个抑癌基因甲基化水平。1.5×105,1.5×104,1.5×103和1.5×102copies/ml的H460细胞为阳性对照,建立标准曲线。CpGenome DNA modification Kit修饰50μl DNA后进行qMSP扩增。50μl反应体系如下:模板DNA 5μl,5×buffer10μl,400μM dNTP,2mM MgCl2,200nM引物,170nM探针及2U Takara Ex Taq HS酶。ABI7500PCR扩增仪进行扩增:95℃ 1min、95℃ 5s、60℃ 34s、55个循环。同一患者化疗前后标本同批次检测。
8、统计分析
统计软件为Stata9.1和SPSS13.0。体外实验的甲基化数据用Two-way ANOVA tests。裸鼠实验的甲基化数据用Fisher’s exact test。患者化疗前后甲基化的数据用Wilcoxon matched-pairs signed-rank test。组间比较用Kruskal-Wallis signed-rank test。生存曲线的统计比较用Kaplan-Meier法和log-rank test。P<0.05为有显著差异。
结 果
1、体外培养及裸鼠实验提示APC /RASSF1A甲基化升高的峰值在顺铂用药后24小时
为了解A549细胞中的APC或RASSF1A基因启动子的甲基化情况,我们首先检测了该细胞系与阳性对照细胞系H460细胞的甲基化状态。APC和RASSF1A的Ct值分别是28.5和29.7(H460)以及28.2和29.6(A549),表明A549细胞中的APC和RASSF1A甲基化为阳性。
用不同浓度的顺铂处理A549细胞6、12、24、48和72小时后,收集培养上清。MTT检测提示A549细胞对5mg/ml的顺铂最敏感(图1A)。在此浓度下,APC和RASSF1A甲基化水平升高的峰值均在用药后24小时(图1B-C)。
为了更好地理解经过顺铂治疗后这两种基因的甲基化状态变化,我们使用了荷瘤裸鼠模型。荷瘤裸鼠血浆中的APC或 RASSF1A甲基化率在注射药物后24小时最高(图1D-E)。病理切片及HE染色显示:顺铂治疗组大多数肿瘤细胞死亡,而2组生理盐水处理的对照组的肿瘤细胞仍在增殖(图1F-G)。
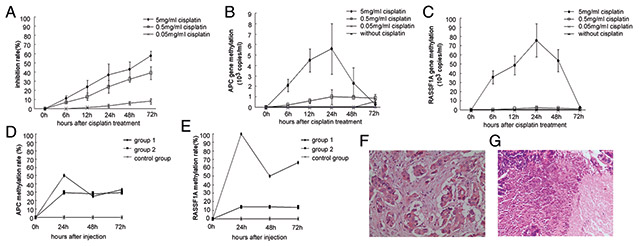
图1. A549细胞或荷瘤裸鼠给予顺铂后24小时甲基化水平升高。A. MTT检测表明A549细胞对5mg/ml顺铂敏感。B. A549细胞的APC甲基化水平在给予顺铂后24小时达到峰值。C. A549细胞的RASSF1A甲基化水平在给予顺铂后24小时达到峰值。D-E. 注射NS和顺铂的荷瘤裸鼠在注射后24小时APC和eRASSF1A的甲基化率最高。F. 注射NS的荷瘤裸鼠的肿瘤组织病理切片。组织
中的大多数肿瘤细胞在注射后72小时仍存活(×200)。G. 顺铂处理组裸鼠的肿瘤组织病理切片。组织中的肿瘤细胞在注射后72小时几乎全部死亡(×200)。
2、铂类化疗后APC /RASSF1A甲基化水平升高与肿瘤有效反应相关
化疗前,APC/RASSF1A甲基化率分别为:腺癌48.9% (46/94),鳞癌50.0%(28/56),其他类型肺癌39.4%(26/66)。
两个疗程后,123个患者至少有一个基因的甲基化水平升高,即化疗后24小时的甲基化水平高于化疗前(meth24h>meth0h)。ERR为75.6%(93/123)。123名患者中,83.5%(101/121)的患者其CEA下降;64.7%(77/119)的患者NSE下降;85.6%(95/111)的患者CY21-1下降。而另外93名患者,两个基因均无甲基化水平的升高(meth24h≤ meth0h)。ERR仅为8.6%(8/93),明显低于有甲基化升高的患者。93名患者肿瘤标志物CEA, NSE及CY21-1的下降率分别为12.9%(12/93)、21.2%(18/85)、15.4%(10/65)(图2A)。因此,以meth24h>meth0h判断肿瘤对铂类药物的反应时,其正确预测率为82.4% [(93+37+48)/216]。同时,OS曲线也显示,化疗后任一基因甲基化升高的患者具有更长的生存时间(图2B-D)。
这些结果提示:铂类化疗后任一基因甲基化水平升高(meth24h>meth0h)与肿瘤细胞对药物反应性良好相关。3年随访获得的大量临床数据也证实了这一观点(图2E-H)。
3、铂类化疗后总血浆DNA水平升高与不良事件分级相关用第一次化疗前总血浆DNA(DNA0h)和用药后24小时的总血浆DNA(DNA24h)后来评估药物的毒性反应。
216名患者中,132例的DNA24h小于/等于DNA0h的2倍(DNA24h/DNA0h≤2),另外84例的DNA24h大于DNA0h的2倍(DNA24h/DNA0h>2)(图3A)。不良事件,特别是肝毒性、神经毒性和胃肠道反应,主要发生在DNA24h/DNA0h>2的患者。DNA24h/DNA0h>2的患者其不良事件发生率高于DNA24h/DNA0h≤2的患者,尤其是1级以上的不良事件发生率在DNA24h/DNA0h>2和DNA24h/DNA0h≤2的患者中均有显著差异(13.18% vs. 6.77%,P<0.05)(图3B)。
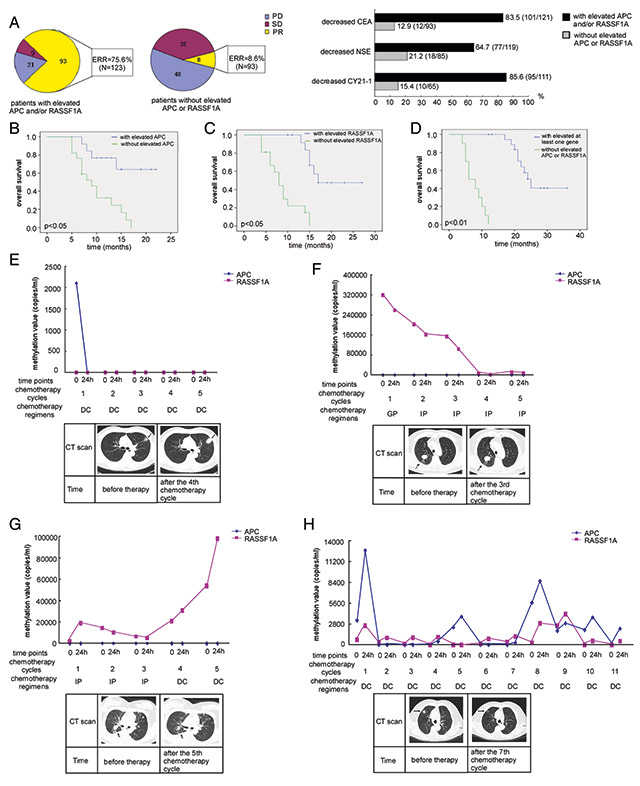
图2. 晚期肺癌患者经顺铂化疗后甲基化水平升高与肿瘤反应性良好相关。A. 在APC和/或RASSF1A甲基化水平升高的患者中,有效缓解率(ERR)更高;大多数患者的肿瘤标志物水平下降。B. APC甲基化水平的OS曲线(中值OS,18个月 vs. 9个月,P<0.05)。C. RASSF1A甲基化水平的OS曲线(中值OS,20个月 vs. 8个月,P<0.05)。D. 至少一种基因的甲基化水平D的OS曲线(中值OS,25个月 vs. 6个月,P<0.01)。E-H. 四位代表性患者的甲基化状态和CT扫描图像。E-F. 两位患者分别在诊断后4和5个月内死亡。G-H. 另外两位患者在本研究期间仍然存活。
4、APC/RASSF1A甲基化和总血浆DNA联合检测与临床判断疾病结果具有较高的一致性
2个疗程后,meth24h>meth0h、meth24h≤meth0h、DNA24h/DNA0h>2、DNA24h/DNA0h≤2的患者分别为123、93、67、149例。根据甲基化及总血浆DNA检测结果,我们人为地划分成四个象限(图4A):I象限即meth24h>meth0h并且DNA24h/DNA0h>2;II象限即meth24h≤meth0h并且DNA24h/DNA0h>2;III象限即meth24h≤meth0h并且DNA24h/DNA0h≤2;IV象限即meth24h>meth0h并且DNA24h/DNA0h≤2。I-IV象限分别有47、26、67、76例患者。在I象限中,临床判断为肿瘤反应好同时副反应较重的患者有38例,甲基化及DNA联合指标与临床判断的符合率为80.9%(38/47)。同理,在II-IV象限中,联合指标与临床判断符合率分别为84.6%(22/26)、85.1%(57/67)及86.8% (66/76)。因此,用联合指标判断化疗后反应的总正确率为84.7% [(22+57+38+66)/216]。
5、甲基化和总血浆DNA联合指标的进一步验证
我们也另收集了100例患者用于验证该联合指标。54例临床判断为CR/PR及轻微不良反应,其中85.2%(46/54)的患者为meth24h>meth0h及DNA24h/DNA0h≤2。28例患者临床为SD/PD及中/重度不良反应,其中82.1%(23/28)为meth24h≤meth0h及DNA24h/DNA0h>2。10例患者临床为CR/PR及中/重度不良反应,其中80%(8/10)为meth24h>meth0h及DNA24h/DNA0h>2。最后8例患者临床为SD/PD及轻度不良反应,其中75%(6/8)为meth24h≤meth0h及DNA24h/DNA0h≤2(图4B-D)。这些结果证实:化疗后,meth24h>meth0h和DNA24h/DNA0h≤2的患者具有良好的肿瘤反应和较少的不良事件。
讨 论
化疗是晚期肺癌的首选治疗手段。但是肿瘤对化疗药物的反应性千差万别。有实验证实采用个体化的化疗方案比标准化方案更能使患者受益[16,17]。但是,现有的检测手段不能及时、敏感地给临床医生提供化疗敏感性方面的信息。
细胞死亡后其DNA会释放入血,峰值在24小时[19,20]。本研究中肺癌细胞系及荷瘤裸鼠实验均说明血浆APC/RASSF1A甲基化水平峰值也在用药后24小时。
众多研究已证实肺癌中APC/RASSF1A具有较高的甲基化率[21-28],故本研究也选用这两个基因。我们自创立双重qMSP方法检测APC/RASSF1A的甲基化水平。该方法的特点是可以同时检测两个基因的甲基化。也有研究认为APC/RASSF1A在不同病理类型的肺癌中具有不同的甲基化率[29-33],其原因可能是由于检测的启动子位点有所不同。另外,我们同时检测APC/RASSF1A两个基因,并以至少一个甲基化阳性即作为阳性,就是消除了“单一基因在不同病理类型中有不同甲基化率”的弊端。当然,我们也比较了本研究和另外5个研究中的APC/RASSF1A甲基化率[32,34-37],其结果显示本研究的标本选择无明显偏倚。因此我们的甲基化检测结果是可靠的。
综上,我们最终选择化疗后24小时的APC/RASSF1A甲基化定量水平来评估肿瘤对化疗药物的反应性。根据实验结果,我们提出“meth24h>meth0h”是肿瘤对铂类药物敏感的指标。细胞对药物敏感时,则会有大量细胞被杀伤,引起循环DNA明显升高。如果升高的DNA主要来源于肿瘤细胞,则说明肿瘤对药物反应性好,就像位于第IV象限的那种情况。如果升高的DNA主要来源于正常细胞,那么大量正常细胞的死亡势必引发严重的毒副反应,就像位于第II象限的情况。
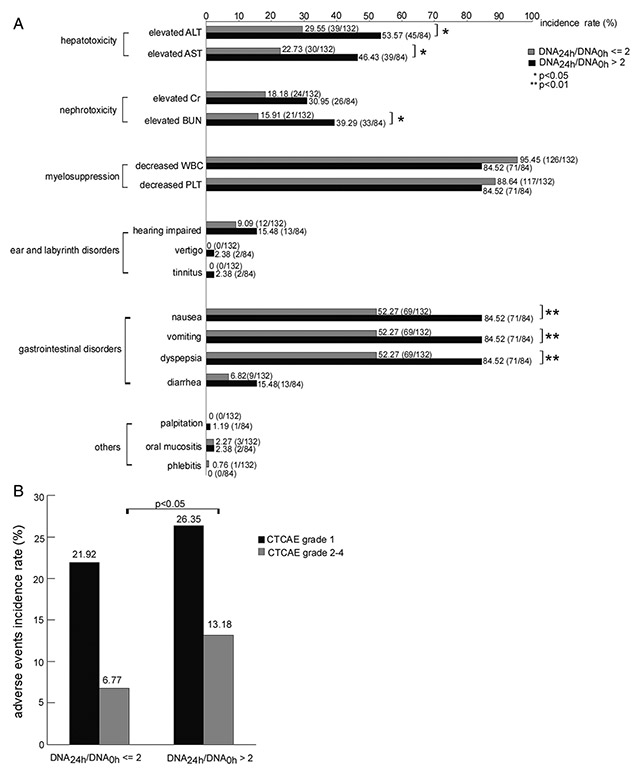
图2. 晚期肺癌患者经顺铂化疗后甲基化水平升高与肿瘤反应性良好相关。
A. 在APC和/或RASSF1A甲基化水平升高的患者中,有效缓解率(ERR)更高;大多数患者的肿瘤标志物水平下降。B. APC甲基化水平的OS曲线(中值OS,18个月 vs. 9个月,P<0.05)。C. RASSF1A甲基化水平的OS曲线(中值OS,20个月 vs. 8个月,P<0.05)。D. 至少一种基因的甲基化水平D的OS曲线(中值OS,25个月 vs. 6个月,P<0.01)。E-H. 四位代表性患者的甲基化状态和CT扫描图像。E-F. 两位患者分别在诊断后4和5个月内死亡。G-H. 另外两位患者在本研究期间仍然存活。
图3. 晚期肺癌患者患者经顺铂化疗后总血浆DNA升高与不良事件等级相关。A. 肝脏毒性、肾脏毒性和胃肠道症状,主要发生于84例DNA24h/DNA0h>2的患者。B. 1级以上的不良事件发生率,DNA24h/DNA0h>2组显著高于DNA24h/DNA0h≤2组(13.18% vs. 6.77%,P<0.05)。
另外,化疗有效时患者肿瘤病灶会发生相应的改变,但是在患者的症状和体征上却不会及时的反映出来。因此在出现明显症状和体征表现之前,82.4%的患者可利用APC/RASSF1A甲基化检测来准确预测化疗效果。所以,化疗后24小时检测甲基化是比CT和肿瘤标志物更早的一个预测化疗效果指标。
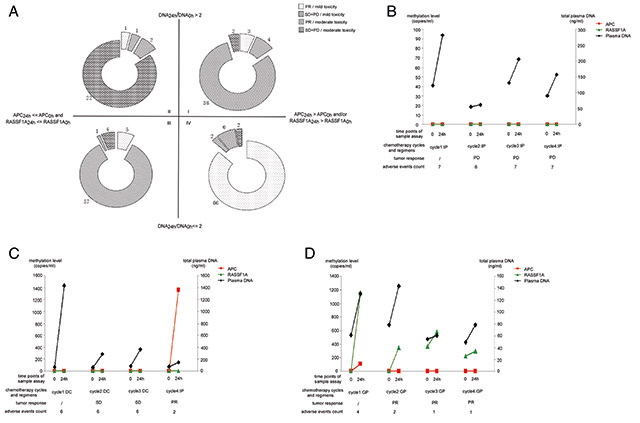
图4. 晚期肺癌患者的APC/RASSF1A甲基化和总血浆DNA的联合检测结果与临床判断一致。A. 不同甲基化状态和血浆DNA水平的患者具有不同的肿瘤反应性和不良事件等级。第一象限至第四象限的一致率分别为80.9%、84.6%、85.1%和86.8%。B-D. 代表性的三位患者的甲基化状态、血浆DNA水平和临床信息。B. 一位男性患者在诊断后5个月内死亡。C. 一位女性患者在随
访结束时已存活36个月。D. 一位患者在随访结束时存活近24个月。
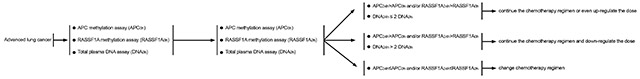
图5. APC/RASSF1A甲基化和血浆总DNA联合用于评估化疗效果和毒性的调整方案
我们也研究了总血浆DNA对化疗毒副反应的监测,并得出结论:不良事件常发生于化疗后DNA升的特别高(DNA24h/DNA0h>2)的患者。化疗后,结合甲基化和总DNA情况,我们提出3种模式:
(1)任一基因甲基化水平升高同时DNA24h/DNA0h≤2。这种模式说明患者对化疗药物敏感而且无严重毒副反应。因此,在甲基化和总DNA监测下,甚至可适当提高药物剂量从而获得更佳化疗效果。
(2)任一基因甲基化水平升高但是DNA24h/DNA0h>2。这种模式中,虽然肿瘤对药物反应性较好,但是其毒副反应也较严重。此时临床医生有必要重新考量药物剂量并进行适当调整(当然是下调)。
(3)两基因甲基化均无升高。此时无论总DNA如何改变,肿瘤对药物根本不敏感,说明该化疗方案不是最佳的,需要立即更换化疗方案。
根据以上所有实验结果,我们提供了一个新的评估肺癌化疗有效性和毒性的策略(图5),利用这个评估体系,临床医生可以针对患者个体及时进行化疗肺癌的调整。
结 论
定量检测APC/RASSF1A甲基化和总血浆DNA可快速同步地预测肺癌化疗有效性和毒性。特别是化疗后24小时甲基化检测,对于评估肿瘤对化疗药物的敏感性方面,快于RECIST指南所依据的影像学数据。另外,血浆DNA甲基化和总DNA定量检测是无创的检查手段,极大方便了临床多次采集标本和随访。我们提出的新评估体系可作为肺癌化疗效果评价的一个重要参考或补充。
参考文献
【1】Mascaux C, Peled N, Garg K, Kato Y, Wynes MW, Hirsch FR: Early detection and screening of lung cancer. Expert Rev Mol Diagn 2010, 10: 799-815.
【2】Punnoose EA, Atwal S, Liu W, Raja R, Fine BM, Hughes BG, Hicks RJ, Hampton GM, Amler LC, Pirzkall A, Lackner MR:Evaluation of circulating tumor cells and circulating tumor DNA in non-small cell lung cancer: association with clinical endpoints in a phase II clinical trial of pertuzumab and erlotinib. Clin Cancer Res 2012, 18: 2391-2401.
【3】Molina JR, Yang P, Cassivi SD, Schild SE, Adjei AA:Non-small cell lung cancer: epidemiology, risk factors, treatment, and survivorship. Mayo Clin Proc 2008, 83: 584-594.
【4】Parkin DM, Bray F, Ferlay J, Pisani P:Global cancer statistics, 2002. CA Cancer J Clin 2005, 55: 74-108.
【5】Sato M, Shames DS, Gazdar AF, Minna JD:A translational view of the molecular pathogenesis of lung cancer. J Thorac Oncol 2007, 2: 327-343.
【6】Ramirez JL, Rosell R, Taron M, Sanchez-Ronco M, Alberola V, de Las Peñas R, Sanchez JM, Moran T, Camps C, Massuti B, Sanchez JJ, Salazar F, Catot S:14-3-3σmethylation in pretreatment serum circulating DNA of cisplatin-plus-gemcitabine-treated advanced non-small-cell lung cancer patients predicts survival:The Spanish Lung Cancer Group. J Clin Oncol 2005, 23: 9105-9112.
【7】Allingham-Hawkins D, Lea A, Levine S: ERCC1 expression analysis to guide therapy in non-small cell lung cancer. PLoS Curr 2010, 2: RRN1202.
【8】Kenneth J. O' Byrne, Martin P. Barr, Steven G. Gray: The role of epigenetics in resistance to cisplatin chemotherapy in lung cancer. Cancers 2011, 3: 1426-1453.
Koomen JM, Haura EB, Bepler G, Sutphen R, Remily-Wood ER, Benson K, Hussein M, Hazlehurst LA, Yeatman TJ, Hildreth LT, Sellers TA, Jacobsen PB, Fenstermacher DA, Dalton WS:Proteomic contributions to personalized cancer care. Mol Cell Proteomics 2008, 7: 1780-1794.
【9】Jain KK: Textbook of Personalized Medicine. New York: Springer 2009, 186.
【10】Gormally E, Caboux E, Vineis P, Hainaut P: Circulating free DNA in plasma or serum as biomarker of carcinogenesis: Practical aspects and biological significance. Mutat Res 2007, 635: 105-117.
【11】Kamat AA, Baldwin M, Urbauer D, Dang D, Han LY, Godwin A, Karlan BY, Simpson JL, Gershenson DM, Coleman RL, Bischoff FZ, Sood AK: Plasma cell-free DNA in ovarian cancer:an independent prognostic biomarker. Cancer 2010, 116: 1918-1925.
【12】Kumar S, Guleria R, Singh V, Bharti AC, Mohan A, Das BC: Plasma DNA level in predicting therapeutic efficacy in advanced non-small cell lung cancer. Eur Respir J 2010, 36: 885-892.
【13】Liggett T, Melnikov A, Yi QL, Replogle C, Brand R, Kaul K, Talamonti M, Abrams RA, Levenson V: Differential methylation of cell-free circulating DNA among patients with pancreatic cancer versus chronic pancreatitis. Cancer 2010, 116: 1674-1680.
【14】Vlassov VV, Laktionov PP,Rykova EY: Circulating nucleic acids as a potential source for cancer biomarkers. Curr Mol Med 2010, 10: 142-165.
【15】Sequist LV, Martins RG, Spigel D, Grunberg SM, Spira A, Jänne PA, Joshi VA, McCollum D, Evans TL, Muzikansky A, Kuhlmann GL, Han M, Goldberg JS, Settleman J, Iafrate AJ, Engelman JA, Haber DA, Johnson BE, Lynch TJ: First-line gefitinib in patients with advanced non-small-cell lung cancer harboring somatic EGFR mutations. J Clin Oncol 2008, 26: 2442-2449.
【16】Gamelin E, Delva R, Jacob J, Merrouche Y, Raoul JL, Pezet D, Dorval E, Piot G, Morel A, Boisdron-Celle M: Individual fluorouracil dose adjustment based on pharmacokinetic follow-up compared with conventional dosage: Results of a multicenter randomized trial of patients with etastatic colorectal cancer. J Clin Oncol 2008, 26: 2099-2105.
【17】Shepherd FA: Molecular selection of patients for first-line treatment of advanced non-small-cell lung cancer with epidermal growth factor inhibitors: not quite ready for prime time. J Clin Oncol 2008, 26: 2426-2427.
【18】Lo YM, Tein MS, Lau TK, Haines CJ, Leung TN, Poon PM, Wainscoat JS, Johnson PJ, Chang AM, Hjelm NM: Quantitative analysis of fetal DNA in maternal plasma and serum: implications for noninvasive prenatal diagnosis. Am J Hum Genet 1998, 62: 768-775.
【19】Jahr S, Hentze H, Englisch S, Hardt D, Fackelmayer FO, Hesch RD, Knippers R: DNA fragments in the blood plasma of cancer patients: Quantitations and evidence for their origin from apoptotic and necrotic cells. Cancer Res 2001, 61: 1659-1665.
【20】Hsu HS, Chen TP, Hung CH, Wen CK, Lin RK, Lee HC, Wang YC:Characterization of a multiple epigenetic marker panel for lung cancer detection and risk assessment in plasma. Cancer 2007, 110: 2019-2026.
【21】Rosell R, Cecere F, Santarpia M, Reguart N, Taron M:Predicting the outcome of chemotherapy for lung cancer. Curr Opin Pharmacol 2006, 6: 323-331.
【22】Brock MV, Hooker CM, Ota-Machida E, Han Y, Guo M, Ames S, Glöckner S, Piantadosi S, Gabrielson E, Pridham G, Pelosky K, Belinsky SA, Yang SC, Baylin SB, Herman JG:DNA methylation markers and early recurrence in stage I lung cancer. N Engl J Med 2008, 358: 1118-1128.
【23】Gormally E, Caboux E, Vineis P, Hainaut P: Circulating free DNA in plasma or serum as biomarker of carcinogenesis: practical aspects and biological significance. Mutat Res 2007, 635: 105-117.
【24】Toyota M, Suzuki H, Yamashita T, Hirata K, Imai K, Tokino T, Shinomura Y: Cancer epigenomics: implications of DNA methylation in personalized cancer therapy. Cancer Sci 2009, 100: 787-791.
【25】Ramírez JL, Salazar MF, Gupta J, Sánchez JM, Taron M, Sanchez-Ronco M, Alberola V, De las Peñas R: Methylation patterns and chemosensitivity in NSCLC. Adv Exp Med Biol 2006, 587: 195-209.
【26】Fischer JR, Ohnmacht U, Rieger N, Zemaitis M, Stoffregen C, Manegold C, Lahm H: Prognostic significance of RASSF1A promoter methylation on survival of non-small cell lung cancer patients treated with gemcitabine. Lung Cancer 2007, 56: 115-123.
【27】Buckingham L, Penfield Faber L, Kim A, Liptay M, Barger C, Basu S, Fidler M, Walters K, Bonomi P, Coon J. PTEN, RASSF1 and DAPK site-specific hypermethylation and outcome in surgically treated stage I and II nonsmall cell lung cancer patients. Int J Cancer 2010, 126: 1630-1639.
【28】Guo S, Tan L, Pu W, Wu J, Xu K, Wu J, Li Q, Ma Y, Xu J, Jin L, Wang J: Quantitative assessment of the diagnostic role of APC promoter methylation in non-small cell lung cancer. Clin Epigenetics 2014, 24; 6(1): 5.
【29】Pesek M, Kopeckova M, Benesova L, Meszarosova A, Mukensnabl P, Bruha F, Minarik M: Clinical significance of hypermethylation status in NSCLC: evaluation of a 30-gene panel in patients with advanced disease. Anticancer Res 2011, 31: 4647-4652.
【30】Hawes SE, Stern JE, Feng Q, Wiens LW, Rasey JS, Lu H, Kiviat NB, Vesselle H: DNA hypermethylation of tumors from non-small cell lung cancer (NSCLC) patients is associated with genderand histologic type. Lung Cancer 2010, 69: 172-179.
【31】Gao L, Xie E, Yu T, Chen D, Zhang L, Zhang B, Wang F, Xu J, Huang P, Liu X, Fang B, Pan S:Methylated APC and RASSF1A in multiple specimens contribute to the differential diagnosis of patients with undetermined solitary pulmonary nodules. J Thorac Dis 2015, 7: 422-432.
【32】Pan SY, Xie EF, Shu YQ, Gao L, Zhang LX, Chen D, Chen JB, Zhao WJ, Mu Y, Zhang JN: Methylation quantification of adenomatous polyposis coli (APC) gene promoter in plasma of lung cancer patients. Ai Zheng 2009, 28: 384-389.
【33】Usadel H, Brabender J, Danenberg KD, Jeronimo C, Harden S, Engles J, Danenberg PV, Yang S, Sidransky D: Quantitative adenomatous polyposis coli promoter methylation analysis in tumor tissue, serum, and plasma DNA of patients with lung cancer. Cancer Res 2002, 62: 371-375.
【34】Rykova EY, Skvortsova TE, Laktionov PP, Tamkovich SN, Bryzgunova OE, Starikov AV, Kuznetsova NP, Kolomiets SA, Sevostianova NV, Vlassov VV: Investigation of tumor-derived extracellular DNA in blood of cancer patients by methylation-specific PCR. Nucleosides Nucleotides Nucleic Acids 2004, 23: 855-859.
【35】Begum S, Brait M, Dasgupta S, Ostrow KL, Zahurak M, Carvalho AL, Califano JA, Goodman SN, Westra WH, Hoque MO, Sidransky D: An epigenetic marker panel for detection of lung cancer using cell-free serum DNA. Clin Cancer Res 2011, 17: 4494-4503.
【36】Zhang Y, Wang R, Song H, Huang G, Yi J, Zheng Y, Wang J, Chen L: Methylation of multiple genes as a candidate biomarker in non-small cell lung cancer. Cancer Lett 2011, 303: 21-28.




