mNGS耐药基因检测分析
在人类历史上,传染病一直是发病率和死亡率的主要原因,抗生素除了直接治疗传染病外,还可以进行侵入性手术、器官移植和癌症化疗等治疗,为人类疾病的控制做出了巨大贡献。但现代抗生素的广泛使用丰富了环境中微生物的耐药性机制,致使抗生素耐药性基因(ARGs)相继出现并迅速传播,时至今日,抗生素耐药生物(ARGs)已成为全球公共卫生关注的一个重要问题,世界卫生组织将抗生素耐药性描述为当今传染病面临的最大挑战,无论是国内生产总值高还是低的国家都面临同样的威胁。一项来自欧盟的数据分析估计,2015年有超过60万例感染和3.3万人死于耐药微生物(ARGs),与2007年的类似估计相比,这一数字增长了近2.5倍。因此为了对抗全球抗生素耐药性的威胁,快速确定AROs和ARGs的特征、监控它们在空间和时间上的分布、识别新的ARGs和抗性途径显得格外重要。
临床上AROs的鉴定通常采用培养的方法,在这种方法中,微生物菌落被分离出来,通过表型特征如形态学、大小和色素沉着等方法来进行鉴定菌株,并通过纸片扩散法、抗生素梯度稀释的方法来确定菌株抗生素敏感性,以确定最低抑制浓度。
基于培养的方法来分离鉴定AROs,技术成熟,阳性预测价值高,可对分离株进行表征,分离出的菌株可进一步进行全基因组(WGS)测序,能够准确地确定分离株的亲缘关系和耐药情况,并且用来追溯ARO菌株的爆发。澳大利亚最近的一项研究使用WGS和基于培养的方法来跟踪一种新出现的病原体,即含有抗万古霉素的粪便肠球菌[1];荷兰某医院和几个没有病源关联的医疗保健中心(HCCs)内出现同序列型肺炎克雷伯菌感染病例,借助WGS技术鉴定含NDM-1耐药基因质粒从肠杆菌跨种转移至肺炎克雷伯菌,溯因院内感染暴发[2]。
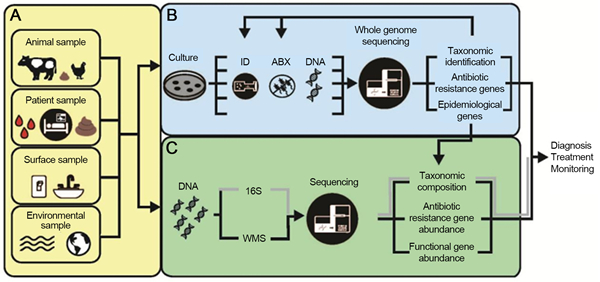
图1. 利用全基因组测序(WGS)和宏基因组学(mNGS)分析生物和环境样品中ARO和ARG
但分离培养的方法操作繁琐、费时、费力,研究限制在少数感兴趣的基因或易于培养的生物体上,从而遗漏复杂环境中无法培养微生物群落中出现的AROs及ARGs。但ARGs可以从环境微生物传播到人类病原体,因此有必要对环境微生物和人类病原体进行大范围调查,那么从患者或者环境中筛选大量和多种分离物将是一个冗长且昂贵的过程。基于宏基因组的第二代高通量测序技术(mNGS),作为一种不需培养的新型检测技术,在一次测序反应中即可获得细菌整个基因组的耐药、毒力等相关基因信息,同时还可对目的基因、基因定位、基因环境、质粒相关序列等开展全面研究,这将有助于揭示病原体毒力及耐药机制,对追溯环境中耐药生物、临床治疗及指导用药均具有重要意义。在一项河流抗生素耐药基因研究中,基于高通量测序技术对河流沉积物中ARGs的广谱谱线进行了表征,并通过对河流沉积物和原始生态位(遥远的南极土壤和深海沉积物)之间的ARGs元基因组图谱的比较研究,确定了人类活动对ARGs存在的贡献[3]。
随着宏基因组测序的成本逐年下降,宏基因组测序使用的范围越来越广泛,也越来越多的用于鉴定ARGs和AROs。鉴定ARGs的一种常见策略是将测序的数据通过比对后使用已知或假定的ARGs的核苷酸或氨基酸序列比对到数据库中的已知ARGs或标记基因上。然而,虽然更加严谨细致的实验流程被逐渐开发[5],也出现了众多的比对工具(BLAST[6] or Bowtie2[7])以及大量的公共数据库比如综合抗菌素耐药性数据库(CARD)、抗生素耐药基因在线(ARGO)、抗生素耐药基因数据库(ARDB)、抗菌药物肽数据库(APD3)、抗菌药物多肽收集(CAMP)、抗菌活性及多肽结构数据库(DBAASP)等,但是从mNGS技术生成的大量结果中提取精准有用的信息仍然充满了未知和挑战。比如大量序列与先前测序的基因或生物体没有明显的相似性,如果没有已知的参考序列,耐药基因就不能很好的从宏基因组数据中识别出来。
即便如果,mNGS仍然是现在耐药基因鉴定,预测未来抗生素耐药的进化的有效手段,并将使参与耐药基因转移的基因元件的进一步研究成为可能。宏基因组学将帮助我们了解不同种类的抗生素对共生菌群和致病菌群的组成和功能的影响。作为直接检测方法的一种综合,mNGS并不是要取代培养、抗原检测和PCR等临床微生物方法,间接过程如病毒血清学测试将继续发挥诊断感染的关键作用,培养和表型敏感性测试,仍然将是研究工作的有用手段。监测和对抗AROs和ARGs的未来在于成功地将基于培养的方法、基因组学、宏基因组分析相结合并优化,以快速识别、表征和跟踪新出现和演变的病原体。
参考文献:
Douglas, A.P. et al. Utilizing genomic analyses to investigate the first outbreak of vanAvancomycin-resistant Enterococcus in Australia with emergence of daptomycin nonsusceptibility. J Med Microbiol, (2019)
Bosch, T., Lutgens, S. P., Hermans, et al. An outbreak of NDM-1 producing Klebsiella pneumoniae in a Dutch hospital with interspecies transfer of the resistance plasmid and unexpected occurrence in unrelated healthcare centers. Journal of Clinical Microbiology, (2017).
Chen, H., Bai, X., Jing, L., Chen, R. & Teng, Y. Characterization of antibiotic resistance genes inthe sediments of an urban river revealed by comparative metagenomics analysis. Sci TotalEnviron 653, 1513-21 (2019)
Casas V ,Maloy S . Genomic and Metagenomic Approaches for Predicting Pathogen Evolution[J]. Microbiology Spectrum, 2014, 2(1):OH.
M. S. Zinter, M. Y. Mayday, K. K. Ryckman, L. L. Jelliffe-Pawlowski, J. L. DeRisi, Towards precision quantification of contamination in metagenomic sequencing experiments. Microbiome7, 62 (2019).
Altschul, S.F., Gish, W., Miller, W., Myers, E.W. & Lipman, D.J. Basic local alignment searchtool. J Mol Biol 215, 403-10 (1990).
Langmead, B. &Salzberg, S.L. Fast gapped-read alignment with Bowtie 2. Nat Methods 9,357-9 (2012).




