逆转录活性的热稳定核酸聚合酶在肿瘤和微生物诊断中的应用前景

作者:张俊华 李贺鑫 肖飞
单位:北京医院病理科
[摘要] RNA检测已成为一种常见的肿瘤和病原微生物诊断与鉴定的检测方法,具有拷贝数多、特异性强、灵敏度高等优点,对疾病的早期诊断具有重要意义。逆转录聚合酶链式反应(RT-PCR)是一种常用的RNA检测技术,由于传统的逆转录酶不耐高温,目前常见的RT-PCR一般为两步法,首先逆转录酶在较低温度下将RNA逆转录为DNA,然后DNA聚合酶以DNA为模板进行高温扩增反应。因此,通过寻找与改造具有逆转录活性的热稳定核酸酶,将两步法RT-PCR发展成一步法单酶RT-PCR,从而简化反应体系与步骤,缩短反应时间,提高检测效率。本文主要从传统逆转录酶在PCR中的应用,耐高温逆转录酶的实现策略,以及热稳定逆转录-核酸聚合酶的研究进展进行综述,并对这种耐高温逆转录-核酸聚合酶在肿瘤和微生物诊断中的应用前景进行了探讨。
[关键词] 热稳定逆转录酶;核酸聚合酶;逆转录聚合酶链式反应;RNA检测;肿瘤与微生物诊断
核酸聚合酶是一类专门生物催化合成脱氧核糖核酸(DNA)和核糖核酸(RNA)的酶。根据合成产物的不同,它可以分为DNA聚合酶与RNA聚合酶两大类,前者以DNA或RNA为模板,以四种脱氧核苷三磷酸为底物,完成5′→3′的DNA合成[1];后者以DNA或RNA为模板,以四种核苷三磷酸为底物,完成5′→3′的RNA合成[2]。RNA聚合酶的催化方式与DNA聚合酶相似,但不具有校正作用的外切核酸酶活性,聚合反应也不需引物。新的研究发现,RNA聚合酶可以主动选择正确的底物防止错误掺入[3]。核酸聚合酶不仅参与DNA或RNA的复制,还在核酸损伤修复过程中发挥重要作用,对于维持基因组完整性和遗传物质的稳定性有重要意义[4]。自大肠杆菌DNA聚合酶I在上世纪五十年代被首次发现后[5],就有很多研究者对各类核酸聚合酶的结构与功能进行了深入的研究,本文就具有逆转录活性的热稳定核酸聚合酶的生物学研究进展与临床诊断应用前景进行综述。
一、DNA聚合酶和逆转录酶在PCR中的应用
聚合酶链式反应(Polymerase chain reaction, PCR)于1985年被美国科学家Kary Mullis首次提出,它是一种生物体外用于放大扩增特定DNA片段的分子生物学技术,用于模拟生物体内的核酸复制[6]。PCR的原理为首先使DNA在高温时(如95℃)变性成单链,待温度冷却至较低温度时(如60℃)引物与其中一条单链按碱基互补配对的原则结合,再将温度升高至大多数DNA聚合酶的最适反应温度(如72℃),使退火引物在DNA聚合酶的作用下沿着5′→3′方向延伸。这种热变性-复性-延伸的过程为一个PCR循环,PCR就是在合适条件下这种循环的不断重复[7, 8]。经过方法学的不断改进,PCR从定性的分析发展到定量的测定,扩增的核酸片段长度可长至几十kb,技术已有十几种之多。例如将PCR与逆转录酶结合成为逆转录PCR,将PCR与抗体结合成为免疫PCR,利用不等量的一对引物产生大量单链DNA的不对称PCR,扩增已知序列旁侧DNA的反向PCR等[9]。
到目前为止,PCR技术在临床诊断以及微生物鉴定分析中有着非常重要的地位。其检测原理为使用某物种或特定生物体内某组织或细胞的特异性引物对核酸模板进行PCR扩增,通过检测扩增产物中是否出现特异性条带来确定阳性与否[10]。例如,多重PCR(Multiple PCR)使用二对以上的引物同时扩增出多个核酸片段,根据对扩增出的特异性条带进行谱带大小与序列比对分析,多种病原微生物可被同时检测,某些遗传病或肿瘤基因也可被分型鉴定[11]。因此,不断改进的PCR技术经济简便,其高效性与系统性为临床提供了更多准确的诊断信息。
在PCR体系中,最为核心的一个成分则是具有扩增核酸活性的聚合酶。目前的研究报道,大多数嗜热古菌DNA聚合酶具有较好的热稳定性与保真性,可以很好的耐受95℃的DNA变性温度,保证PCR每一个反应循环中的扩增效率[12]。此外,也有少数DNA聚合酶只能在较低温度下进行反应,例如来源于枯草芽孢杆菌噬菌体的Phi29 DNA聚合酶,可以在30℃进行滚环复制和多重置换扩增反应,但存在非特异扩增的问题[13]。总的来说,目前大多数的PCR技术使用的是具有热稳定性的DNA聚合酶。
但是,当检测的样本为RNA模板时,一般先要使用逆转录酶(Reverse transcriptase, RT)将RNA逆转录为DNA,再通过DNA聚合酶的作用进行PCR扩增。在逆转录一步中,首先逆转录酶以RNA为模板以5′→3′方向合成与RNA互补的一条DNA(cDNA)链,然后逆转录酶发挥RNase H活性,将逆转录出的DNA与模板RNA形成的杂交分子中的RNA水解掉,最后逆转录酶以余下的这条单链DNA为模板,以dNTP为底物,合成另一条互补DNA[14-16]。逆转录酶不具有3′→5′外切酶活性的校正功能,因此,由逆转录酶催化合成的DNA出错率比较高[17]。此外,有些逆转录酶还具有DNA内切酶活性,这可能与病毒基因整合到宿主细胞染色体DNA中有关[18]。逆转录酶的发现对于遗传工程技术起了很大的推动作用,目前它已成为一种重要的工具酶。
目前商品化的逆转录酶主要有从禽类成髓细胞瘤病毒纯化到的禽类成髓细胞病毒(Avian myeloblastosis virus,AMV)逆转录酶和从克隆有莫洛尼氏鼠白血病病毒逆转录酶基因的大肠杆菌中分离到的莫洛尼鼠白血病病毒(Moloney murine leukemia virus,MMLV)逆转录酶[19, 20]。然而,这些逆转录酶有个共同的特点,即不能耐受普通PCR过程中的高温,由此对于逆转录PCR来说,只能先在较低的温度下进行第一步逆转录过程,等逆转录完成后,高温加热使逆转录酶失活,接着再依靠DNA聚合酶进行第二步DNA扩增反应[21]。鉴于不耐高温这一特点,传统使用的逆转录酶仍具有不可忽视的弱点,进而推迟反应至少30分钟以影响PCR的时效性。因此,对耐高温逆转录酶的追求成为PCR改进工作中的一大探索问题。
二、耐高温逆转录酶的实现策略
为了能实现逆转录PCR中逆转录酶的耐高温性,科学家们致力于寻找、开发一种能替代两种局限性的双酶(逆转录酶和DNA聚合酶)的单酶,该酶同时具有逆转录和DNA聚合活性,既能在高温下将RNA逆转录生成cDNA模板,又具有足够的热稳定性,可以承受反复的热循环过程直至接近沸腾温度。这种含有单酶的逆转录PCR(Reverse transcription-polymerase chain reaction,RT-PCR)体系配制简单,可以明显缩短实验结果等待的时间,在反应效率方面比双酶体系更具优势。目前研究发现有一些耐高温的DNA聚合酶在高温下也具有逆转录RNA的活性。例如,从温泉中的嗜热病毒元基因组中筛选出一种热稳定的A家族聚合酶(被鉴定为PyroPhage Pol),它可以在不需要锰离子的高温条件下就表现出自身的逆转录酶活性,从而在单酶RT-PCR和RNA模板的等温扩增和检测应用中具有潜力[22, 23]。然而,PyroPhage Pol的持续性不足限制了其作为一步RT-PCR检测中核心酶试剂的用途。
因此,除了直接对耐高温逆转录酶的寻找之外,科学家们进行更多的实验是对已有的耐高温DNA聚合酶进行基因工程改造,以希望改造之后的酶本身能获得新的逆转录酶活性。某些源自嗜热菌属细菌的DNA聚合酶可通过诱变或在反应缓冲液中添加锰离子而被诱导出逆转录能力。例如来源于Thermotoga petrophila K4的DNA聚合酶K4PolI具有3′→5′外切酶活性的热稳定DNA聚合酶活性,但不能以RNA为模板进行复制。通过计算机模拟聚合酶的三级结构得到它与模板DNA的结合特征,再以RNA为模板,对一些参与模板互作的氨基酸残基进行定点突变,最终成功筛选到了具有逆转录酶活性的DNA聚合酶突变体,但对应的3′→5′核酸外切酶活性也随之消失[24, 25]。由于RNA聚合酶通常缺乏3′→5′核酸外切酶结构域,因此在整个逆转录酶家族中也不存在校对活性,从而导致低保真逆转录的复制行为。与逆转录酶相比,DNA聚合酶家族已发展出精巧的校对机制,可在基因组复制过程中提高DNA的合成保真度。因此,科学家们在不断纠错酶复制的过程中直接定向进化DNA聚合酶以得到具有逆转录活性的高保真酶。例如从B家族DNA聚合酶进化出的逆转录异聚合酶(Reverse transcription xenopolymerases,RTX)[26]。
三、热稳定逆转录-核酸聚合酶的改造实例
1. RTX的产生:为了确定逆转录酶和DNA聚合酶之间的进化鸿沟原因,研究者们通过一种改进的定向进化策略,即Reverse transcription-compartmentalized self-replication(RT-CSR),将一种B家族DNA聚合酶直接定向进化成逆转录异聚合酶(RTX)[27]。研究表明,古菌B家族DNA聚合酶(polB)因其单体、持续合成性以及具有热稳定和外切校对功能而被选择为RTX定向进化的对象[28]。首先,制备该家族中KOD聚合酶变体随机文库(每个基因进行一个或两个突变),使用低严格性RT-CSR(引物中添加10个RNA残基)启动随机文库的进化。随着聚合酶的富集,通过逐步将RNA碱基添加到引物中来逐渐提高RT-CSR的严格性。到第18轮RT-CSR时,引物完全由RNA组成,此时在乳化液PCR(Emulsion PCR)中可进行176个RNA碱基的逆转录且每个热循环中保持指数扩增(Fig. 1)[26]。
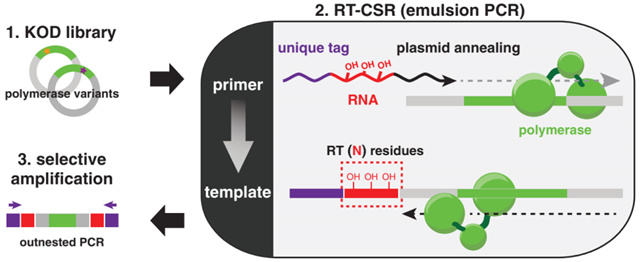
图1. Framework for the directed evolution of hyperthermostable RT using RT-CSR.
对筛选到的聚合酶分析得出,包含37个位点突变的突变体B11具有较好的RT活性,可逆转录至少500个碱基对。然而,测序和活性实验证实了B11失去了校对活性。接下来,研究者们试图设计具有最少突变位点却可以恢复核酸外切酶校对能力的聚合酶。通过测序分析和对模板结合界面的计算机建模,最终几种具有良好RT突变的聚合酶的模型设计被合成出来,并分别进行了活性测试。结果显示,表现最佳的RTX包含的突变位点数少于B11中发现的突变位点数的一半,但这并不会影响RTX对RNA的逆转录及后续DNA聚合效率,可对长度超过5kb的RNA进行RT-PCR。RTX中的突变也未影响亲本KOD聚合酶的特性。测序分析表明,MMLV的逆转录错误率在1.1×10-4~4.8×10-4,而RTX的错误率在3.5×10-5~3.7×10-5,比MMLV的错误率低3~10倍[26]。这表明RTX在逆转录中的校对能力使PCR具有更高的保真度。因此,RTX能够实现高温下的单酶RT-PCR,具有简化实验流程与提高转录精度的优点,从而可以被引入到RNA测序平台以实现复杂RNA样品的高通量和高保真测序。
2. M160-nuc的产生:前文所述的A家族聚合酶PyroPhage Pol(也称为3173)是从嗜热病毒元基因组的筛选中得到的,但其逆转录活性不足以支撑一步RT-PCR。研究者们利用天然病毒多样性作为酶定向进化的基础,对PyroPhage Pol进行改造以使其作为RT-PCR中的核心试剂。后续通过蛋白质工程进一步完善了酶系统的功能,并提供了一种基于镁离子的单酶RT-PCR方法,该方法与双酶系统相比,具有反应体系易于配制、反应时间快和灵敏度高等优势[29]。
首先,研究者们选择了包括3173在内的六个已有直系同源物(3173、4B9、1608、488、1440和967)进行活性检测,3173和1608在RT-PCR中表现出一定的耐热性。在此基础上,采用基因改组(Gene shuffling)的方法生成多样化的变体文库。将3173、1608和967三种聚合酶的序列进行比对,选定八个大约等间隔的交换位点,其中含有至少七个密码子(即21个核苷酸)的保守区域。运用这八个位点的引物,分别从三个DNA模板库中随机扩增出九个DNA片段,生成重叠的DNA序列池,然后在预先定义的交换位点处DNA片段被随机混合并重新组装成完整的嵌合序列(Fig. 2)[29]。通过这种方法,可以估算出获得文库的理论大小(39),并且可以保留天然基因片段的顺序,最大程度地减少假组装的产生。
接着,使用RT-PCR中的Cq值(代表每个反应管内的荧光信号到达设定的域值时所经历的循环数)对大约400个突变体进行功能筛选,得到了六个保留高热稳定性并具有升高的逆转录酶活性的新型变体,即M66、M160、M180、M295、M384和M392。其中,M160的Cq值比3173降低了5.2个循环,是最显著改善逆转录功能的一个突变体。于是,为了能够更加广泛地用于实时RT-PCR(Real-time RT-PCR),研究者们将Taq DNA聚合酶的5′→3′核酸酶结构域与高性能聚合酶突变体M160进行基因融合,可记作M160-nuc,命名为Magma DNA聚合酶。实验表明,该融合酶可与基于探针(如TaqMan® probes)的检测方法兼容,它不需要单独的逆转录过程就可以进入热循环程序,Cq值比M160低。与标准的不耐热逆转录方法相比,Magma DNA聚合酶扩增531nt片段的Cq值比Taq pol/MMLV RNase H(-)的低约5.5个周期,这些结果表明Magma DNA聚合酶在反转录时的效率之高。此外,Magma DNA聚合酶能够高度敏感地检测结构化的RNA靶点,在高温下松弛RNA中的二级结构,以确保引物顺利延伸通过该区域[29]。由此,M160-nuc不仅能够实现高温下的单酶RT-PCR,而且具备水解探针能力,使检测的活性和灵敏度均得到了提高。
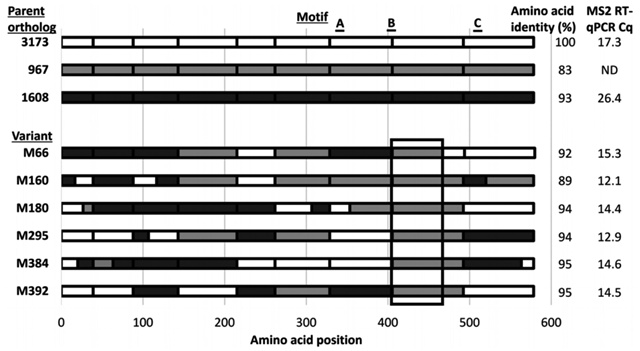
图2. Gene shuffling and screening.
四、热稳定逆转录-核酸聚合酶的应用前景
近些年来,高通量测序技术通过检测生物体肿瘤的全部基因突变图谱,提升了人类对肿瘤基因组和肿瘤发生发展中细胞内机制的理解[30, 31]。除了基因组DNA检测,RNA与肿瘤的发生发展也密切相关,这包括mRNA、miRNA(micro RNA)、lncRNA(long non-coding RNA)和circle RNA等[32-35]。研究报道,在不同的肿瘤组织中可检测到相关RNA的不同水平的异常表达[36, 37]。与DNA检测相比,RNA检测在肿瘤早期诊断、疾病进展及预后判断具有明显的优势。首先,某些情况下DNA发生杂合突变,也就是只有一条链发生了突变,但转录后的RNA不一定发生突变,从而不会导致生物体出现明显的病征。其次,一般RNA的拷贝数比DNA多,更易于检测。相比于DNA,RNA的转录表达具有较高的组织特异性,这提高了肿瘤检测的敏感性和特异性。
除了肿瘤检测,RNA检测目前也广泛应用于微生物的检测中。基于高通量测序平台的方法包括对微生物DNA与RNA的联合检测,精度高,但价格相对昂贵,对数据分析的要求较高。此外,在已知待测微生物核酸序列的条件下,通过设计特异性引物或探针,对微生物中的特异性RNA片段进行RT-PCR检测,通过扩增曲线即可进行结果判读。值得注意的是,一些致病微生物(如流感病毒、冠状病毒等RNA病毒)的遗传物质是RNA而非DNA,通过对其进行RNA特异检测可以直接鉴定出该微生物的种类。这些检测方法为临床诊断提供了重要的应用价值。
目前发展出的RNA检测方法很多,主要有高通量测序技术、Northern印迹技术、miRNA基因芯片法、qRT-PCR(Quantitative real-time PCR)法等[38-40]。这些检测平台各有优缺点,研究者可根据自己的需求和条件选择最合适的方法。其中,qRT-PCR法由于线性范围广、灵敏度和特异性高以及便于普通实验室开展,成为了最为常见的检测方法之一[40]。与传统逆转录酶相比,被改良的热稳定逆转录-核酸聚合酶给qRT-PCR带来了诸多优点:反应体系更简单、反应时间更短、逆转录RNA效率更高、灵敏度更高、成本更低。这些优点使得RNA检测在肿瘤与微生物传染性疾病的快速筛查与诊断方面更加经济快捷,具有更大的临床应用前景。
参考文献
Johansson E, N Dixon. Replicative DNA polymerases. Cold Spring Harb Perspect Biol[J], 2013, 5(6): a012799.
Sutherland C, KS Murakami. An Introduction to the Structure and Function of the Catalytic Core Enzyme of Escherichia coli RNA Polymerase. EcoSal Plus[J], 2018, 8(1): 10.1128.
Gamba P, N Zenkin. Transcription fidelity and its roles in the cell. Curr Opin Microbiol[J], 2018, 42: 13-18.
Mercadante AA, SS Mohiuddin, Biochemistry, Replication and Transcription, in StatPearls. 2020, StatPearls Publishing Copyright© 2020, StatPearls Publishing LLC.: Treasure Island (FL).
Kornberg SR. Adenosine triphosphate synthesis from polyphosphate by an enzyme from Escherichia coli. Biochim Biophys Acta[J], 1957, 26(2): 294-300.
Mullis K, F Faloona, S Scharf, et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb Symp Quant Biol[J], 1986, 51 Pt 1: 263-273.
Mullis KB, FA Faloona. Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods Enzymol[J], 1987, 155: 335-350.
Saiki RK, DH Gelfand, S Stoffel, et al. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science[J], 1988, 239(4839): 487-491.
Orlando C, P Pinzani, M Pazzagli. Developments in quantitative PCR. Clin Chem Lab Med[J], 1998, 36(5): 255-269.
Buchheidt D, M Reinwald, WK Hofmann, et al. Evaluating the use of PCR for diagnosing invasive aspergillosis. Expert Rev Mol Diagn[J], 2017, 17(6): 603-610.
Jeon HK, KH Kim, WM Sohn, et al. Differential Diagnosis of Human Sparganosis Using Multiplex PCR. Korean J Parasitol[J], 2018, 56(3): 295-300.
Zhang L, M Kang, J Xu, et al. Archaeal DNA polymerases in biotechnology. Appl Microbiol Biotechnol[J], 2015, 99(16): 6585-6597.
练杜娟, 仇建萍, 张平静, et al. Phi29 DNA聚合酶最新应用研究进展. 药物生物技术[J], 2016, 02: 150-154.
Mustafin RN, EK Khusnutdinova. The Role of Reverse Transcriptase in the Origin of Life. Biochemistry (Mosc)[J], 2019, 84(8): 870-883.
Moelling K, F Broecker. The reverse transcriptase-RNase H: from viruses to antiviral defense. Ann N Y Acad Sci[J], 2015, 1341: 126-135.
Menéndez-Arias L, A Sebastián-Martín, M Álvarez. Viral reverse transcriptases. Virus Res[J], 2017, 234: 153-176.
Schönbrunner NJ, EH Fiss, O Budker, et al. Chimeric thermostable DNA polymerases with reverse transcriptase and attenuated 3'-5' exonuclease activity. Biochemistry[J], 2006, 45(42): 12786-12795.
Pyatkov KI, IR Arkhipova, NV Malkova, et al. Reverse transcriptase and endonuclease activities encoded by Penelope-like retroelements. Proc Natl Acad Sci U S A[J], 2004, 101(41): 14719-14724.
Houts GE, M Miyagi, C Ellis, et al. Reverse transcriptase from avian myeloblastosis virus. J Virol[J], 1979, 29(2): 517-522.
Katano Y, T Hisayoshi, I Kuze, et al. Expression of moloney murine leukemia virus reverse transcriptase in a cell-free protein expression system. Biotechnol Lett[J], 2016, 38(7): 1203-1211.
Bachman J. Reverse-transcription PCR (RT-PCR). Methods Enzymol[J], 2013, 530: 67-74.
Schoenfeld T, M Liles, KE Wommack, et al. Functional viral metagenomics and the next generation of molecular tools. Trends Microbiol[J], 2010, 18(1): 20-29.
Moser MJ, RA DiFrancesco, K Gowda, et al. Thermostable DNA polymerase from a viral metagenome is a potent RT-PCR enzyme. PLoS One[J], 2012, 7(6): e38371.
Sano S, Y Yamada, T Shinkawa, et al. Mutations to create thermostable reverse transcriptase with bacterial family A DNA polymerase from Thermotoga petrophila K4. J Biosci Bioeng[J], 2012, 113(3): 315-321.
Okano H, M Baba, K Kawato, et al. High sensitive RNA detection by one-step RT-PCR using the genetically engineered variant of DNA polymerase with reverse transcriptase activity from hyperthermophilies. J Biosci Bioeng[J], 2018, 125(3): 275-281.
Ellefson JW, J Gollihar, R Shroff, et al. Synthetic evolutionary origin of a proofreading reverse transcriptase. Science[J], 2016, 352(6293): 1590-1593.
Ghadessy FJ, JL Ong, P Holliger. Directed evolution of polymerase function by compartmentalized self-replication. Proc Natl Acad Sci U S A[J], 2001, 98(8): 4552-4557.
Jozwiakowski SK, BA Connolly. A modified family-B archaeal DNA polymerase with reverse transcriptase activity. Chembiochem[J], 2011, 12(1): 35-37.
Heller RC, S Chung, K Crissy, et al. Engineering of a thermostable viral polymerase using metagenome-derived diversity for highly sensitive and specific RT-PCR. Nucleic Acids Res[J], 2019, 47(7): 3619-3630.
Castle JC, S Kreiter, J Diekmann, et al. Exploiting the mutanome for tumor vaccination. Cancer Res[J], 2012, 72(5): 1081-1091.
郑晓, 鞠景芳, 卢斌峰, et al. 肿瘤基因突变组学在肿瘤免疫治疗中的意义. 中国肿瘤生物治疗杂志[J], 2015, 22(6): 794-798.
Chan JJ, Y Tay. Noncoding RNA:RNA Regulatory Networks in Cancer. Int J Mol Sci[J], 2018, 19(5): 10.3390.
Acunzo M, G Romano, D Wernicke, et al. MicroRNA and cancer--a brief overview. Adv Biol Regul[J], 2015, 57: 1-9.
Yang G, X Lu, L Yuan. LncRNA: a link between RNA and cancer. Biochim Biophys Acta[J], 2014, 1839(11): 1097-1109.
Fanale D, S Taverna, A Russo, et al. Circular RNA in Exosomes. Adv Exp Med Biol[J], 2018, 1087: 109-117.
Bhan A, M Soleimani, SS Mandal. Long Noncoding RNA and Cancer: A New Paradigm. Cancer Res[J], 2017, 77(15): 3965-3981.
Galamb O, BK Barták, A Kalmár, et al. Diagnostic and prognostic potential of tissue and circulating long non-coding RNAs in colorectal tumors. World J Gastroenterol[J], 2019, 25(34): 5026-5048.
Haque A, J Engel, SA Teichmann, et al. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. 2017, 9(1): 75.
Urbanek MO, AU Nawrocka, WJ Krzyzosiak. Small RNA Detection by in Situ Hybridization Methods. Int J Mol Sci[J], 2015, 16(6): 13259-13286.
Narrandes S, W Xu. Gene Expression Detection Assay for Cancer Clinical Use. J Cancer[J], 2018, 9(13): 2249-2265.





