数字PCR在COVID-19疫情中的应用
作者:樊东东 陈芊如
【摘要】2019年12月出现了一种新型的冠状病毒,该病毒具备极高的传 染性和隐匿性,这使其在全球范围内迅速扩散,迄今已经有约1.5亿人被 感染,超过300万人丧失生命。在疫情防控、感染确诊和临床治疗中, 以PCR为代表的分子诊断技术发挥了巨大的作用,特别是在疫情初筛 阶段,更是发挥了保障社会正常运转的作用。在PCR领域,数字PCR (dPCR)是一种新兴的PCR检测技术,具备绝对定量、灵敏度高、特 异性好、抗干扰能力强等诸多优点。本文首先介绍了基于微液滴技术的 dPCR的技术原理、检测的过程、以及微液滴技术赋予dPCR的优势。基于此,本文回顾了dPCR在新冠疫情中的研究进展。比如,基于绝对定量 的能力可以研究样本类型中新冠病毒的含量、评估样本制备方法对病毒检 出率的影响、进行新冠新药的临床评估;基于高灵敏性可用于低丰度病毒 样本的检测、用于检测灰区的甄别等。随着研发的深入,dPCR的应用前 景比较广阔。
2019年12月爆发了一种由新型冠状病毒(SARS-CoV-2)引起的传染病[1],该疾病已经在全球范围内持续流行了18个月之久。SARS-CoV-2与中东 呼吸综合征冠状病毒(MERS-CoV)、蝙蝠SARS样冠状病毒(SARS-like bat CoV)和严重急性呼吸道综合征冠状病毒(SARS-CoV)同属乙型冠状病毒支 系,基因组分析发现,SARS-CoV-2与SARS-like bat CoV最为相近[2]。SARSCoV-2基因组编码29个蛋白,其中结构蛋白由4个结构基因编码,包括尖峰 (S)、包膜(E)、膜(M)和核盖体(N)基因;而非结构蛋白主要由 ORF1ab基因编码[2]。由此病原体引发的传染病被命名为新型冠状病毒肺炎 (COVID-19),COVID-19疫情影响全世界,截至2021年4月29日,约1.5亿人 染病、超过300万人死亡(https://coronavirus.jhu.edu/map.html)。 COVID-19的传染性极强,及时、准确的诊断对于治疗和疫情防控十分 重要。当前主要以病毒RNA检测作为确诊COVID-19的方法,临床上应用最多 的技术为实时荧光逆转录PCR(RT-qPCR)[3]。其他一 些核酸检测技术也被开发用于SARS-CoV-2的检测, 包括数字PCR(dPCR)、环介导逆转录等温扩增技术 (RT-LAMP)、转录介导扩增技术(TMA)等[4]。其 中,dPCR是继实时荧光定量PCR(qPCR)之后新发展 起来的一种核酸定量分析技术,dPCR具备高灵敏度、 高精度和绝对定量等独特优势,在新冠疫情中被用于 病毒的检测、定量、病情监测、新药评估等多种研 究。本文将对dPCR的技术原理和优势进行介绍,回顾 dPCR在新冠疫情中的应用,并尝试为dPCR在新冠疫情 与相关临床应用中的改进和发展方向提出见解。
一、数字PCR技术
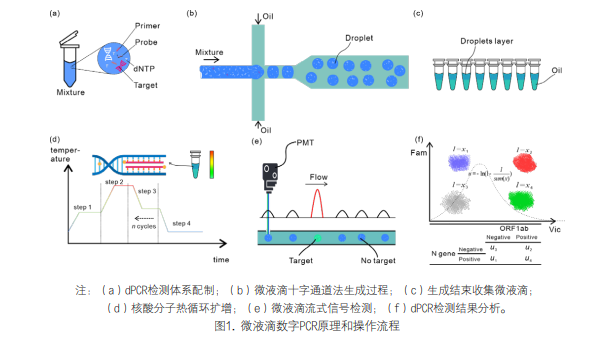
1. 数字PCR技术原理:dPCR的原理是将PCR的反 应体系离散为众多微小单元,然后通过对微小单元内 反应结果的检测反映整个体系的原始模板拷贝数目。 通常来说,dPCR包括微液滴式和微孔式,微液滴dPCR 采用基于微流控的液滴技术离散整个反应单元[5];微 孔式dPCR主要采用微纳米加工技术将一片基材加工成 包含数万个微反应室以实现反应单元的离散[6]。 以微液滴dPCR的SARS-CoV-2检测为例,图1展示 了dPCR的技术原理和检测程序。首先是配制含有靶标 检测引物和探针的混合液,通常使用特异性的TaqMan 探针或者是非特异性的染色法标记技术,在SARSCoV-2检测中多使用TaqMan法,以N基因和ORF1ab基因 作为靶点。然后采用微流控芯片生成微液滴,液滴直 径通常在90-120μm,微液滴生成有十字法和T型通 道法等,目前采用最多的是十字法,见图1b。生成的 液滴存在三种状态:不含靶标核酸分子、含有单个靶 标核酸分子、含有多个靶标核酸分子,在靶标分子丰 度不高的情况下,液滴处于以上三种状态的概率遵从 泊松分布。液滴后续被放置到热循环仪上进行扩增, 这一步骤和普通PCR过程基本一致。经过扩增之后, 原包含有靶标核酸分子的液滴内部的核酸量将达到百 亿量级,靶标分子在扩增的过程中和荧光基团结合, 使其能接受光信号的激发并释放荧光信号。之后,液 滴将逐个通过光学检测位,不包含靶标分子的液滴的 被激发光信号较弱,包含有荧光靶标分子的被激发 光信号较强,见图1e。光信号通过一些例如放大、聚 焦、过滤等的处理操作后被光电倍增管(PMT)转换 为电信号,每一个液滴对应一个或多个信号峰值,峰 值的高低和频段反映了液滴的状态。最终液滴的统计 信息根据泊松分布转化为原始靶标分子的浓度,图1f 展示了双重靶标dPCR的分析结果,图中散点的位置代 表了携带不同靶标的液滴。
2. 数字PCR优势:绝对定量:qPCR通过计算扩增 过程的荧光曲线的Ct值,并配合参考品同时扩增建立 的标准曲线,对待测样本进行定量,而荧光曲线往往 容易受到扩增效率、抑制剂、样本质量、系统误差等 因素影响。与qPCR不同,dPCR将待测靶分子分散在数 万甚至数百万反应单元中,并根据每个反应单元扩增 反应终点的荧光强度进行阴阳性划分。dPCR的拷贝数 计算依据阴阳性反应单元的数量和统计学模型,可对 靶分子的数量实现精准的绝对定量,不容易受干扰, 也不依赖于参考品。因此,dPCR能够提供更可靠和准 确的定量结果。Hindson等人比较了dPCR与qPCR定量 miRNA的能力,结果显示dPCR能够提供比qPCR更精确和 更高重复性的定量结果[7]。
更高的灵敏度:dPCR技术对整个反应体系进行万 倍以上的离散,由于背景模板被大量的反应单元所分 割和稀释,在含有目标模板的反应单元中,背景模板 的数量呈数量级减少(比如将PCR体系分割为10000个 反应单元,则每个反应单元内的背景模板的数量被减 少4个数量级),因此PCR扩增时目标模板受到的非 特异性干扰也呈数量级地减少,从而保证目标模板的 特异扩增。因此dPCR能提供更高的灵敏度,使得在大 量背景模板的干扰下对稀有靶标检测变得可能。如在 BRAF V600E稀有突变的检测中,dPCR和qPCR分别能够检 测到0.001%和约1%的突变比例[8]。
更好的稳定性:较大数量级的离散使得原先在反 应体系中存在的一些抑制剂也被分散至各个微反应单 元中,此举大大减少了每个反应单元内的抑制剂总 量,使得dPCR对抑制剂的抗性增强。在一些病毒(如 巨细胞病毒)检测体系中,即便抑制剂可以影响PCR的 扩增,但是影响程度相对qPCR较小,并且受影响的液 滴也仍然可以通过比对其荧光信号强度与阳性信号阈 值进行正确判定[9, 10]。
更高的精度:dPCR的定量原理决定了其比qPCR具 有更高的定量精度。在qPCR中,由于荧光信号在每个 循环中都会加倍,因此一次检测通常只能解析两倍的 拷贝数差异;在dPCR中,通过将反应体系进行离散以 及检测扩增反应后每个反应单元的荧光信号,理论上 可以达到定量单个分子的效果。可以通过将反应体系 分割为更多反应单元来提高精度。例如在人类巨细胞 病毒的定量检测研究中,结果显示dPCR的定量变异系 数小于qPCR的,在10000拷贝/mL和1000拷贝/mL的水平 dPCR的变异系数较qPCR的分别减少4倍和1.5倍[11]。
二、 数字PCR在COVID-19 疫情中的应用
1. 高灵敏度新冠核酸检测:RT-qPCR虽然是目前 COVID-19诊断的金标准,但其被报道存在假阴性率高 的问题[12, 13]。假阴性结果可能由多种因素造成,包 括引物和探针靶区域突变、样本中的病毒量不足、采 样过程不规范、运输不当和样本中存在扩增抑制剂等[14]。由于dPCR技术具有高灵敏度和较好的抑制剂抗性 的特性,其应用于检测SARS-CoV-2时能够有效的减少 假阴性结果。研究人员比较了基于dPCR和RT-qPCR的 SARS-CoV-2检测方法的最低检测限(LoD),结果表明 dPCR的方法具有更低的LoD和更高的灵敏度[15-17]。在临 床样本的检测中,dPCR也表现出比RT-qPCR具有更高 的灵敏度,部分RT-qPCR检测为疑似或阴性的COVID-19 样本,dPCR都成功检测出阳性[15, 17-21]。在Suo等人[15]的 研究中,26例COVID-19确诊患者的样本被dPCR成功检 出,而这些样本的RT-qPCR检测结果均为阴性。此外, Falzone等人[16]的研究也显示dPCR对扩增抑制剂的作用 较不敏感。由此说明,dPCR在低病毒载量样本的检测 中具有优势,能够作为RT-qPCR的补充。
2. 临床样本类型病毒载量评估:COVID-19临床 样本的取样部位和类型很多,包括鼻拭子、咽拭子、 痰液、血液、尿液、粪便、支气管肺泡灌洗液等等。 选择合适的临床样本有助于进行准确的临床诊断,对 疾病防控和治疗有重大意义。因此,有必要评估各个 类型的样本的病毒载量,尽可能选择病毒载量最多的 样本以提高检出率。Yu等[18]使用dPCR定量鼻拭子、咽 拭子、痰液、血液、尿液中的病毒载量,结果发现痰 液中具有最高的病毒载量,鼻拭子和咽拭子次之,提 示了以痰液作为检测样本能够提高阳性检出率。但是 鼻、咽拭子的样本都需要专业的医护人员获取,采样 便利性不及唾液,因此不断有研究人员验证唾液检测 病毒的可靠性。Sui等[22]对35个无症状但核酸阳性的 病人采取了183份样本,类型包括唾液、痰液、鼻咽 拭子和口腔拭子,检测结果显示唾液的阳性检出率 仅仅比鼻咽拭子略低一点,高于其他类型的样本, 并且相对其他样本,能够提供更低的假阴性率。Li等[23]采用数字PCR对鼻咽拭子、肛周拭子、唾液等不同 类型的样本进行了定量检测,结果显示鼻咽拭子能够 提供最高的检出率,而肛周拭子仅次于鼻咽拭子,检 出率为24.3%,这为新冠病毒检测的样本类型提供了 一种新的可能。以上研究表明dPCR的绝对定量能力在 COVID-19临床样本评估、病毒定量中发挥重要作用。 3. 样本制备方法评估:为了保护医学实验室人 员免受感染,大多数实验室在检测前会对临床样本进 行灭活处理。然而,灭活处理可能会对检测带来影 响,导致假阴性结果,因此有必要明确样本前处理对 检测结果的具体影响。Chen等人[24]使用dPCR定量3种 灭活方法处理前后样本中的病毒量,结果显示灭活处 理减少了可检到的病毒量,并可能因此导致假阴性结果,其中热灭活的样本中病毒拷贝数大幅下降,使用 TRIzol试剂对RNA拷贝数的影响最小。dPCR在核酸样 本灭活处理的评估中发挥了其绝对定量的优势,同样的方法能够用于其他影响因素的研究。
4. 病情进展动态监测:在临床管理与治疗中需 要对患者的病情进展进行监测,其中病毒载量通常与 病情的严重程度相关。Yu等人[18]利用dPCR定量患者不 同时期的样本中的病毒载量,发现在早期和进展期的 病毒载量明显高于恢复期的病毒载量,提示动态监测 样本中的病毒载量有助于评估疾病的进展。Szwebel 等[25]发现SARS-CoV-2核酸病毒血症可以作为监测新 冠抗IL6受体治疗过程的特异性标志物,因为该疗法 效果个体差异性较大,因此可以通过dPCR检测这一标 志物来筛选符合条件的患者。这类研究说明dPCR高灵 敏度与精确定量的特性使其能够胜任患者感染病毒 后,病情发展、治疗等过程中病毒载量变化的监测。 5. 核酸参考品制备:在SARS-CoV-2的核酸检 测中,核酸参考品被用于评估检测方法的性能、制 作RT-qPCR定量标准曲线以及作为阳性对照。van Kasteren等人[26]使用dPCR定量病毒RNA拷贝数并对样 本进行系列稀释,以系列稀释液评估不同RT-qPCR试 剂盒的检测灵敏度。Fung等人[27]通过dPCR定量病毒 核酸,制备系列稀释液,以此测定七种SARS-CoV-2 检测方法的检测下限。此外,在Hu等人[28]的研究使 用dPCR标定的系列参考品评估单、双、三靶标的新冠 qPCR体系,结论进一步得到了采用经dPCR定值的57分 临床样本的验证。dPCR绝对定量的优势使其能够满足 核酸参考品制备的需求,即对核酸进行精确的定量。 对于中检院和国家计量院制定新冠国家核酸参考品来 说,dPCR是一个必不可少的工具,并且发挥了重要作 用;对于从事新冠检测的企业制备核酸企业参考品, 也是同样的重要。
6. 环境中SARS-CoV-2监测:COVID-19的传染力强,了解病毒的传播方式与环境中病毒的分布, 有助于指导疫情防控策略的制定。由于从不同环境 中得到的样本质量不一,这就对病毒的检测提出了 高灵敏度及强抗抑制剂干扰性的要求。Liu等人[29]通 过dPCR定量COVID-19爆发期间武汉两家医院不同区域气溶胶中的病毒RNA,隔离病房和通风的病房中检 测到的病毒RNA浓度非常低,但在患者使用的洗手 间和某些医务人员区域检测到较高浓度,结果显示 SARS-CoV-2具有通过气溶胶传播的潜力。Lv等人[30]利用RT-qPCR和dPCR检测样品运输和接收设施、测试 仪器、个人防护设备等实验室相关物体表面上残留的 病毒。RT-qPCR检测结果均为阴性,而dPCR在61个样 品中检测出13个SARS-CoV-2阳性。SARS-CoV-2核酸 密度最高的区域是操作员的外手套,其余操作员戴着 手套的手直接或间接接触的物体也检测出病毒,表明 手接触是导致实验室环境污染的主要传播途径。基于 dPCR能够灵敏检测与定量环境中的病毒RNA浓度,由 此为防疫措施与消毒程序提供参考依据。
7. SARS-CoV-2突变检测:RNA病毒的突变率非常高,高突变率与毒力调节和进化能力相关,病毒 突变研究对于评估病毒耐药性、了解免疫逃逸和发病 机理相关的机制至关重要。通过基因测序和比对能够 获得病毒变异的基因组信息,但要在实验室样品与临 床样品中检测出已知的SARS-CoV-2遗传变异则需要灵 敏度更高的技术。Wong等人[31]开发了一种基于dPCR的 检测方法来检测Vero-E6传播的分离株、类器官、实验 感染的仓鼠和COVID-19患者样本中的PRRA位点或位点 上游缺失变异。他们发现PRRA缺失的蝙蝠状冠状病毒 变异体可以在COVID-19患者中自然存在,而且仍然可 以传播,但这些变异在自然感染中的频率较低,表明 它们的竞争性不如插入PRRA的野生型。dPCR对模板进 行稀释和分割,使其能够更加精准、灵敏捕捉是否存 在突变型核酸,适合用于已知的SARS-CoV-2遗传变异 检测,特别是对于一些低频突变。
8. 抗SARS-CoV-2新药的评价:在抗病毒药物 的研发过程中,病毒载量的变化是评估药物有效性的 重要指标。例如,在remdesivir针对成人重症COVID-19 患者的一项研究中,Wang等人[32]对受试者的呼吸道样 本的病毒载量进行检测以明确药物是否能够加快病毒 载量下降或降低病毒检出率;在arbidol和lopinavir/ ritonavir治疗药物的比较研究中,Zhu等人[33]通过病 毒载量的变化来评价药物的效果。这两个研究都使用 RT-qPCR来测量病毒载量的变化,其中Wang等人通过 标准曲线确定病毒载量,Zhu等人仅使用Ct值来表现病 毒载量的变化。由于dPCR能够实现不依赖标准曲线的 绝对定量,而且能够检测到拷贝数的微小差异,笔者 团队辅助多家药厂采用dPCR进行新冠新药的临床评价 (尚未发表),认为dPCR比RT-qPCR更适合用于评价抗 SARS-CoV-2药物的有效性,能够给出更具说服力和准 确的评价结果。
三、 总结和展望
dPCR将核酸模板稀释、分配到大量独立的微反应单元中进行扩增反应,不仅可以对样本进行绝对定量,针对低丰度的样本,还能提供更加灵敏和特异的性能,对扩增抑制剂的抗性更强。本文综述了dPCR 在COVID-19疫情监测、防控和临床治疗等过程中的应用,如低拷贝病毒样本检测、临床样本病毒载量定量、参考品标定、环境中病毒浓度定量、病毒突变检测等,这些应用也恰恰是其绝对定量、灵敏度、特异 性高等优势的体现。
虽然dPCR的优势与临床应用前景已在许多研究中 得到确认,但想要进一步在临床广泛应用,甚至在各 级医疗单位包括疫区与基层医院推广使用,dPCR仍面 临一些挑战。首先是dPCR的动态范围受限于离散液滴 数的限制需进一步提高,通常能做到的上限一般在 106 量级。其次,商用dPCR系统目前的检测通量仍然有 限,无法充分满足COVID-19普筛的需求,需要进一步 提升样本检测通量。再者,dPCR的临床实验室标准仍 不成熟,需要制定国家或行业统一标准,推出商品化 的质控品;最后是dPCR设备成本较高,目前可选择的 商用dPCR平台有限,并且推出的临床可用的试剂盒数 量较少,这些都需要行业的共同推进和努力。总之, 随着研究和行业的持续深入,未来dPCR在实验室或医 院中的应用前景,非常值得我们共同期待。
-
致谢:感谢北京市科学技术委员会科技计划 (Z201100005420025)和清华大学春风基金滚动支持 课题(2020Z99CFG010)对本研究的基金支持。本研 究成果发表于《View》期刊2021年第2期(Applications of digital PCR in COVID-19 pandemic: https://doing. org/10.1002/VIW.20200082),感谢《View》编辑部 许可将英文原稿翻译成中文并根据研究进展更新后发表于 本期刊。
参考文献
C. Huang, Y. Wang, X. Li, L. Ren, J. Zhao, Y. Hu, L. Zhang, G. Fan, J. Xu, X. Gu, Z. Cheng, T. Yu, J. Xia, Y. Wei, W. Wu, X. Xie, W. Yin, H. Li, M. Liu, Y. Xiao, H. Gao, L. Guo, J. Xie, G. Wang, R. Jiang, Z. Gao, Q. Jin, J. Wang and B. Cao, The Lancet, 2020, 395, 497-506. -
A. Wu, Y. Peng, B. Huang, X. Ding, X. Wang, P. Niu, J. Meng, Z. Zhu, Z. Zhang, J. Wang, J. Sheng, L. Quan, Z. Xia, W. Tan, G. Cheng and T. Jiang, Cell Host Microbe, 2020, 27, 325- 328.
-
Y. H. Jin, L. Cai, Z. S. Cheng, H. Cheng, T. Deng, Y. P. Fan, C. Fang, D. Huang, L. Q. Huang, Q. Huang, Y. Han, B. Hu, F. Hu, B. H. Li, Y. R. Li, K. Liang, L. K. Lin, L. S. Luo, J. Ma, L. L. Ma, Z. Y. Peng, Y. B. Pan, Z. Y. Pan, X. Q. Ren, H. M. Sun, Y. Wang, Y. Y. Wang, H. Weng, C. J. Wei, D. F. Wu, J. Xia, Y. Xiong, H. B. Xu, X. M. Yao, Y. F. Yuan, T. S. Ye, X. C.
-
Zhang, Y. W. Zhang, Y. G. Zhang, H. M. Zhang, Y. Zhao, M. J. Zhao, H. Zi, X. T. Zeng, Y. Y. Wang, X. H. Wang, f. t. Z. H. o. W. U. N. C. Management, E.-B. M. C. o. C. I. E. Research Team, M. Promotive Association for and C. Health, Military Medical Research, 2020, 7, 4.
-
L. J. Carter, L. V. Garner, J. W. Smoot, Y. Li, Q. Zhou, C. J. Saveson, J. M. Sasso, A. C. Gregg, D. J. Soares, T. R. Beskid, S. R. Jervey and C. Liu, ACS Central Science, 2020, 6, 591-605.
-
B. J. Hindson, K. D. Ness, D. A. Masquelier, P. Belgrader, N. J. Heredia, A. J. Makarewicz, I. J. Bright, M. Y. Lucero, A. L. Hiddessen and T. C. Legler, Analytical Chemistry, 2011, 83, 8604-8610.
-
M. Laig, C. Fekete and N. Majumdar, Quantitative RealTime PCR, Springer, 2020, pp. 209-231. C. M. Hindson, J. R. Chevillet, H. A. Briggs, E. N. Gallichotte, I. K. Ruf, B. J. Hindson, R. L. Vessella and M. Tewari, Nature Methods, 2013, 10, 1003-1005. B. J. Hindson, K. D. Ness, D. A. Masquelier, P. Belgrader, N. J. Heredia, A. J. Makarewicz, I. J. Bright, M. Y. Lucero, A. L. Hiddessen, T. C. Legler, T. K. Kitano, M. R. Hodel, J. F. Petersen, P. W. Wyatt, E. R. Steenblock, P. H. Shah, L. J. Bousse, C. B. Troup, J. C. Mellen, D. K. Wittmann, N. G. Erndt, T. H. Cauley, R. T. Koehler, A. P. So, S. Dube, K. A. Rose, L. Montesclaros, S. Wang, D. P. Stumbo, S. P. Hodges, S. Romine, F. P. Milanovich, H. E. White, J. F. Regan, G. A. Karlin-Neumann, C. M. Hindson, S. Saxonov and B. W. Colston, Analytical Chemistry, 2011, 83, 8604-8610. T. C. Dingle, R. H. Sedlak, L. Cook and K. R. Jerome, Clinical Chemistry, 2013, 59, 1670-1672.
-
R. H. Sedlak, J. Kuypers and K. R. Jerome, Diagnostic Microbiology and Infectious Disease, 2014, 80, 285-286. R. Hall Sedlak and K. R. Jerome, E x p e r t R e v i e w o f Molecular Diagnostics, 2014, 14, 501-507.
-
Y. Li, L. Yao, J. Li, L. Chen, Y. Song, Z. Cai and C. Yang, J Med Virol, 2020, 92, 903-908.
-
J. Wu, J. Liu, X. Zhao, C. Liu, W. Wang, D. Wang, W. Xu, C. Zhang, J. Yu, B. Jiang, H. Cao and L. Li, Clinical Infectious Diseases, 2020, 71, 706-712.
-
A. Tahamtan and A. Ardebili, Expert Review of Molecular Diagnostics, 2020, 20, 453-454.
-
T. Suo, X. Liu, J. Feng, M. Guo, W. Hu, D. Guo, H. Ullah, Y. Yang, Q. Zhang, X. Wang, M. Sajid, Z. Huang, L. Deng, T. Chen, F. Liu, K. Xu, Y. Liu, Q. Zhang, Y. Liu, Y. Xiong, G. Chen, K. Lan and Y. Chen, Emerging Microbes & Infections, 2020, 9, 1259-1268.
-
L. Falzone, N. Musso, G. Gattuso, D. Bongiorno, C. I. Palermo, G. Scalia, M. Libra and S. Stefani, International Journal Of Molecular Medicine, 2020, 46, 957-964. R. Lu, J. Wang, M. Li, Y. Wang, J. Dong and W. Cai, medRxiv, 2020, DOI: 10.1101/2020.03.24.20042689, 2020.2003.2024.20042689.
-
F. Yu, L. Yan, N. Wang, S. Yang, L. Wang, Y. Tang, G. Gao, S. Wang, C. Ma, R. Xie, F. Wang, C. Tan, L. Zhu, Y. Guo and F. Zhang, Clinical Infectious Diseases, 2020, DOI: 10.1093/ cid/ciaa345.
-
L. Dong, J. Zhou, C. Niu, Q. Wang, Y. Pan, S. Sheng, X. Wang, Y. Zhang, J. Yang, M. Liu, Y. Zhao, X. Zhang, T. Zhu, T. Peng, J. Xie, Y. Gao, D. Wang, Y. Zhao, X. Dai and X. Fang, medRxiv, 2020, DOI: 10.1101/2020.03.14.20036129, 2020.2003.2014.20036129.
-
Y. Dang, N. Liu, C. Tan, Y. Feng, X. Yuan, D. Fan, Y. Peng, R. Jin, Y. Guo and J. Lou, Clinica Chimica Acta, 2020, DOI: 10.1016/j.cca.2020.08.033.
-
C. Alteri, V. Cento, M. Antonello, L. Colagrossi, M. Merli, N. Ughi, S. Renica, E. Matarazzo, F. Di Ruscio, L. Tartaglione, J. Colombo, C. Grimaldi, S. Carta, A. Nava, V. Costabile, C. Baiguera, D. Campisi, D. Fanti, C. Vismara, R. Fumagalli, F. Scaglione, O. M. Epis, M. Puoti and C. F. Perno, PLoS One, 2020, 15, e0236311.
-
Z. Sui, Y. Zhang, S. Tu, J. Xie, W. Huang, T. Peng, L. Dong, J. Yang, Y. Ouyang and S. Liu, Journal of Infection, 2021, 82, e38-e40.
-
L. Li, C. Tan, J. Zeng, C. Luo, S. Hu, Y. Peng, W. Li, Z. Xie, Y. Ling and X. Zhang, Journal of Translational Medicine, 2021, 19, 1-8.
-
H. Chen, R. Wu, Y. Xing, Q. Du, Z. Xue, Y. Xi, Y. Yang, Y. Deng, Y. Han, K. Li, Y. Luan, Y. Zhang, X. Wei, T. Yu, H. Li, L. Zhu, S. Su, H. Lian, L. Lu, C. Tan, H. Zheng, B. Chen, P. Yu, Y. Guo and C. Ma, Journal of Clinical Microbiology, 2020, DOI: 10.1128/JCM.00958-20.
-
T.-A. Szwebel, D. Veyer, N. Robillard, D. Eshagh, E. Canoui, T. Bruneau, A. Contejean, C. Azoulay, T. Serrano and T. Hueso, Stem Cell Reviews and Reports, 2021, 1-4. P. B. van Kasteren, B. van der Veer, S. van den Brink, L. Wijsman, J. de Jonge, A. van den Brandt, R. Molenkamp, C. Reusken and A. Meijer, Journal of Clinical Virology, 2020, 128, 104412.
-
B. Fung, A. Gopez, V. Servellita, S. Arevalo, C. Ho, A. Deucher, E. Thornborrow, C. Chiu and S. Miller, Journal of Clinical Microbiology, 2020, DOI: 10.1128/JCM.01535- 20.
-
X. Hu, L. Zhu, Y. Luo, Q. Zhao, C. Tan, X. Chen, H. Zhang, X. Hu, L. Lu and Y. Xiao, Clinica Chimica Acta, 2020, 511, 143-148.
-
Y. Liu, Z. Ning, Y. Chen, M. Guo, Y. Liu, N. K. Gali, L. Sun, Y. Duan, J. Cai, D. Westerdahl, X. Liu, K. Xu, K. F. Ho, H. Kan, Q. Fu and K. Lan, Nature, 2020, 582, 557-560. J. Lv, J. Yang, J. Xue, P. Zhu, L. Liu and S. Li, Sci Total Environ, 2020, 742, 140370.
-
Y. C. Wong, S. Y. Lau, K. K. Wang To, B. W. Y. Mok, X. Li, P. Wang, S. Deng, K. F. Woo, Z. Du, C. Li, J. Zhou, J. F. Woo Chan, K. Y. Yuen, H. Chen and Z. Chen, Clinical Infectious Diseases, 2020, DOI: 10.1093/cid/ciaa953.
-
Y. Wang, D. Zhang, G. Du, R. Du, J. Zhao, Y. Jin, S. Fu, L. Gao, Z. Cheng, Q. Lu, Y. Hu, G. Luo, K. Wang, Y. Lu, H. Li, S. Wang, S. Ruan, C. Yang, C. Mei, Y. Wang, D. Ding, F. Wu, X. Tang, X. Ye, Y. Ye, B. Liu, J. Yang, W. Yin, A. Wang, G. Fan, F. Zhou, Z. Liu, X. Gu, J. Xu, L. Shang, Y. Zhang, L. Cao, T. Guo, Y. Wan, H. Qin, Y. Jiang, T. Jaki, F. G. Hayden, P. W. Horby, B. Cao and C. Wang, The Lancet, 2020, 395, 1569-1578.
-
Z. Zhu, Z. Lu, T. Xu, C. Chen, G. Yang, T. Zha, J. Lu and Y. Xue, Journal of Infection, 2020, 81, e21-e23.





