弯曲菌的流行病学特征与临床微生物检测方法

鲁辛辛,教授,博士生导师,首都医科大学附属北京同仁医院检验科主任医师。从事病原微生物检验、教学及科研工作。主持多项NSFC及科技部重大专项。第九、第十届中华医学会检验分会委员,第十三届北京医学会检验分会副主任委员。

刘畅,主管检验师,北京市密云区医院检验科。从事临床微生物学技术工作9年,主要负责病原微生物的鉴定、细菌耐药检测、临床耐药监测及临床咨询工作。
弯曲菌(Campylobacter spp.)广泛存在于食用动物和水果蔬菜中,主要通过食物链途径传播给人类,是食源性腹泻中最常见的细菌病原体之一。全球估计有弯曲菌感染病例9,600万/年,弯曲菌可引起轻到重度不等的腹泻,还可引起牙周病、颈部脓肿、菌血症、脓毒症、结肠癌及格林-巴利综合征等严重疾病。从人类分离出的弯曲菌除有较强的致病性外,对抗菌药物也表现出较高的耐药性。在我国,由于未将其纳入常规腹泻病原菌检测范围,临床对弯曲菌的认识存在较大缺口,同时因缺乏完善的食源性疾病监测网,各地区流行病学数据的完整性和连续性不尽人意。本文从弯曲菌的生物学、流行病学特点以及致病性特征入手,重点介绍病原学检验方法及检测流程,以期提高弯曲菌的检出率。
一、弯曲菌的生物学特征
1963年Sebald和VeÂron首次提出弯曲菌[1],直到1972年科学家们认为某些弯曲菌是食源性疾病的重要病因[2]。研究表明,导致人类腹泻的弯曲菌仅需摄入500-800cfu[3],也有报告100cfu或更少剂量的弯曲菌即可引起感染[4]。弯曲菌作为重要的人畜共患病原体,目前有35个种,其中5个种进一步分为11个亚种,空肠弯曲菌的基因组约为1.64Mb。弯曲菌导致的人类疾病中90%由空肠弯曲菌(C. jejuni)引起,其余大部分归属结肠弯曲菌(C. coli)[5]。虽然乌普萨拉弯曲菌(C. upsaliensis)在20多年前被认为是重要的新兴胃肠道病原体,且从粪便中分离该菌的频率高于C. coli[6],但由于它对选择性培养基中的抗生素敏感,大多数临床实验室培养阴性[1]。随着分子生物学技术的进一步发展,将对新弯曲菌,如简明弯曲菌(C. concisus)、乌普萨拉弯曲菌和解脲弯曲菌(C. ureolyticus)有了更多了解[7]。
弯曲菌属于弯曲菌科、弯曲菌目、ε-变形菌纲,是一类革兰阴性、呈杆状,螺旋形,有单鞭毛或双鞭毛,大小约为0.2-0.8x0.5-5μm的一类可培养细菌[8]。最适生长温度为42℃,最佳生长环境为5% O2、10% CO2和85% N2的微需氧环境[9]。在培养基上生长缓慢,通常需要48-72h[10]。当暴露于低温、氧气、酸等不利条件下,弯曲菌可保持存活,但处于不可培养(VBNC)状态[11]。与其他人畜共患病原体相比具有苛刻的生长要求[12]。尽管弯曲菌对人类有致病性,但在许多食用动物的肠道中可无症状繁殖[13],因此,宿主因素可能是人类发病的重要原因[14]。空肠弯曲菌和结肠弯曲菌可在鸟类、羊、牛和猪等多种动物宿主的消化道中定植,在许多国家,鸡是感染人类的主要来源[15]。蔬菜、水果和新鲜农产品也被列为是引起感染的危险因素,可能与使用有机肥料相关[16]。
二、弯曲菌的致病性
人类弯曲菌感染主要引起急性、自限性肠炎,一般可持续5-7d。主要临床表现有腹痛、水样或血性腹泻、头痛、发热及寒战,大便中可见大量白细胞和红细胞[17-19]。同时,弯曲菌可导致牙周病、颈部脓肿、菌血症、脓毒症、结肠癌、脑膜炎、反应性关节炎以及包括格林-巴利综合征等免疫性疾病[20]。尽管弯曲菌是世界范围内食源性腹泻常见原因之一,但定植的确切分子机制、感染途径和发病机制尚未完全阐明[21]。
1. 关于弯曲菌肠内定植和毒力的研究有鞭毛介导的运动、细菌对肠粘膜的粘附、侵袭能力和产生毒素的能力等[22]。重要的毒力决定因素包括CadF(37-kDa 的纤连蛋白结合外膜蛋白),负责弯曲菌与纤连蛋白的粘附。FlpA也是一种纤连蛋白结合蛋白,可与CadF同时发挥作用。III型蛋白质分泌系统(T3SS)将毒力因子注入分泌到宿主细胞中,这种基于鞭毛的T3SS由 FlhA、FlhB、FliO、FliP、FliQ和FliR组成。在从鞭毛分泌的各种因子中,有FlaC、CiaC和CiaI,它们是定植、入侵和细胞内存活所必需的。最具特征的毒素是使细胞膨胀毒素,它由三个亚基CdtA、CdtB、CdtC组成,CtdA和CdtC负责传递活性亚基 CdtB,它以脱氧核糖核酸酶的形式进入宿主细胞核,导致细胞周期停滞和死亡。Konkel等认为感染途径可确定为三个基本步骤[23]:首先,弯曲菌通过粘蛋白降解酶、鞭毛、粘附素等共同作用粘附到粘液层,并侵入小肠粘膜上皮的单层肠上皮细胞和固有层,在上皮细胞内通过细胞易位和产生毒素导致人类产生白细胞介素;其次,树突状细胞、巨噬细胞和中性粒细胞趋化到感染部位,从而诱导肠道炎症,使肠绒毛坏死;之后,肠上皮的损伤导致功能丧失,在炎症的作用下,电解质释放到肠腔,最后导致严重腹泻。
2. 关于肠外感染机制,有研究显示弯曲菌对上皮细胞的粘附伴随着强烈的促炎免疫反应[24, 25],弯曲菌的脂寡糖(LOS)外层核心结构与人类神经节苷脂(GM)的结构相似,因此,针对LOS结构表位产生的抗体不仅与弯曲菌LOS结构结合,而且还与GM结合,导致神经细胞的免疫损伤而发生格林-巴利综合征[26-29]。除了已知的毒力因子基因外,其他功能组,如跨膜蛋白、细菌形状决定因素、调节系统、能量系统、呼吸、铁摄取、蛋白质合成/修饰/分泌基因上调以支撑弯曲菌在细胞内存活的研究,需要进行基因失活、表型和转录组学分析,以提高对弯曲菌发病机制的理解[30]。
三、弯曲菌的流行病学特征
在全球范围内,弯曲菌病的发病率因国家和地区不同而出现较大差异,其真实发病率在很大程度上是未知的,特别是在发展中国家[31]。
在美国,食源性疾病主动监测网(FoodNet)对由弯曲菌、环孢子虫、李斯特菌、沙门菌、产志贺毒素大肠埃希菌、志贺菌、弧菌及小肠结肠耶尔森菌均开展主动监测。2019年美国食源性疾病主动监测网的10个监测点显示,弯曲菌平均发病率为19.5/10万,感染9,731人,住院1,988人,与2016-2018年相比,2019年弯曲菌的发病率显著增加[32]。2020年监测显示,在COVID-19大流行期间,弯曲菌发病率自2007年以来首次下降,发病率为14.4/10万。为防止SARS-CoV-2传播而实施的广泛的公共卫生干预措施可能与病例减少有关[33]。
在欧盟,弯曲菌是最常报告的食源性病原微生物,每年的病例数估计约为900万。自2008年以来,欧盟通过欧洲疾病监测系统(TESSy)发布感染数据[34]。2019年TESSy数据显示欧盟成员国弯曲菌的平均发病率为54.0/10万[35]。2020年由于英国脱欧和COVID-19流行病的影响,2020年欧盟确诊人数为120,946例,平均发病率为40.3/10万,较2019年减少了25.4%[36]。
在澳大利亚,估计每年有410万人感染食源性疾病,产生的费用约12亿美元[37]。2000年澳大利亚政府卫生部建立了用于调查食源性疾病的流行病学监测网站(OzFoodNet),也是澳大利亚传染病控制的最高机构。在澳大利亚,最新可获得的食源性弯曲菌数据来自2016年,人类弯曲菌病平均发病率为146.9/10万,确诊病例总数为24,171例,较2011-2015年的5年平均确诊病例数上升了33%[38]。
在低收入和中等收入国家,由于缺乏完善的食源性疾病监测体系、对弯曲菌腹泻认识不足以及弯曲菌特殊的检测方法所限[39],弯曲菌的流行情况基本缺失[40]。印度东北部2014-2016年间的一项研究发现,因腹泻住院的5岁以下儿童中,弯曲菌总检出率为10.1%,其中空肠弯曲菌占80.5%[41]。孟加拉国一家医院2019年6月至2020年6月间住院腹泻患者中,弯曲菌的总检出率为31.5%,其中包括空肠弯曲菌占69.2%,结肠弯曲菌占30.8%,0-5岁年龄组患者检出率最高达52%[42]。中国仅部分地区开展了弯曲菌监测,且检出率差异较大[43]。2018年,在北京对13家食源性疾病哨点医院门诊全年腹泻患者的2021例粪便标本进行监测,弯曲菌检出率为8.66%[44]。2018年9-11月,深圳市南山区某三甲医院对腹泻患者进行监测,弯曲菌检出率为12%[45]。上海崇明区2015-2019年监测点肠道门诊的弯曲菌检出率为2.1%[46]。然而,大部分腹泻病原谱研究中缺少弯曲菌相关流行数据[47-49]。本实验室数据显示北京地区2018-2019年PCR阳性率为8.1%(168/2,062),qPCR阳性标本进行双孔滤膜培养法培养,阳性率为61.9%[50]。
四、弯曲菌的耐药性
虽然大多数弯曲菌腹泻为自限性,无需抗菌药物治疗,但在高烧、血便、症状持续超过1w、妊娠、感染HIV和其他免疫功能低下时应予抗生素治疗[51]。随着临床和畜牧业抗生素的广泛使用,弯曲菌已形成了多种耐药机制,特别是喹诺酮类药物[52]。美国NARMS监测网站数据显示,在空肠弯曲菌的临床分离株中,环丙沙星耐药率从1997-1999年的17%增加到2015-2017年的27%,大环内酯类的耐药性目前保持稳定,2017年空肠弯曲菌对大环内酯类阿奇霉素耐药率为3%。日本大阪在2008-2014年监测显示,结肠弯曲菌对环丙沙星耐药率达59.5%,红霉素的耐药率为4.8%[53]。在比利时,2006-2015年空肠弯曲菌对环丙沙星的耐药率为56.3%,红霉素2%[54]。1994-2010年,北京空肠弯曲菌耐药性研究发现,喹诺酮耐药率从50%上升至93.1%,红霉素的耐药率为2%[55]。
鉴于弯曲菌对喹诺酮类抗菌药物耐药严重,WHO于2017年将耐氟喹诺酮弯曲菌列入全球高度优先开发新抗生素疗法的清单[56]。虽然大环内酯类抗生素是目前治疗人类感染弯曲菌的首选[57],但孟加拉国最近的一项研究表明,空肠弯曲菌对红霉素的耐药率已达45.8%,结肠弯曲菌的耐药率高达62.5%[42]。中国北京2017-2018年数据显示空肠弯曲菌对红霉素的耐药率为9%,结肠弯曲菌对红霉素的耐药率高达44.4%[58],大环内酯类作为抗弯曲菌一线治疗药物在发展中国家并不乐观。本实验室研究数据显示,北京地区2018-2019年空肠弯曲菌和结肠弯曲菌对环丙沙星耐药率达94.0%和100.0%,对红霉素敏感率为94.0%和66.7%[50]。
五、弯曲菌的耐药机制
关于弯曲菌耐药机制的一些研究显示,大环内酯类通过与核糖体结合并改变其构象来抑制蛋白质合成。最常见的耐药机制是23SrRNA突变和通过erm(B)基因甲基化修饰靶位点。氟喹诺酮类药物的结合靶点是DNA促旋酶,以抑制DNA合成而使细菌死亡。耐药是因喹诺酮结合靶位点被修饰,即形成促旋酶A(gyrA)喹诺酮耐药决定区,这是氟喹诺酮类药物在弯曲菌中的主要耐药机制。弯曲杆菌对四环素的耐药性为中度至高度,通常由tet(O)基因介导,该基因常见于pTet质粒上,但也存在于基因组岛上。β-内酰胺类抗生素通过与青霉素结合蛋白结合,并在细胞壁合成过程中破坏肽聚糖交联而发挥作用。通过β-内酰胺酶blaOXA-61产生的耐药性在空肠弯曲菌和结肠弯曲菌中广泛存在。弯曲菌的多药外排泵CmeABC可协同对β-内酰胺类以及四环素类、大环内酯类和氟喹诺酮类的耐药[59]。
六、实验室检查方法
包含两大类,即培养法和不依赖培养的检测方法[56],针对目前使用较多的技术手段介绍如下:
1. 培养方法:弯曲菌生长缓慢,有正常粪便菌群存在的情况下很难分离[60]。传统培养法是先将便标本进行增菌,然后转种于选择性培养基上,在42℃的微需氧环境中孵育48-72h[20]。临床常用的选择性培养基有:改良炭头孢哌酮脱氧胆酸盐琼脂(modified cefoperazone charcoal deoxycholate agar,mCCDA)、Campy-Line琼脂、Karmali琼脂、木炭选择性培养基(charcoal selective medium,CSM)和头孢哌酮,万古霉素,两性霉素B(Campy cefoperazone, vancomycin, amphotericin B,CVA)琼脂等[61-63]。培养基之间的主要区别是抑制杂菌的抗生素类型和浓度不同[54]。一项研究对4种选择性培养基进行性能评估,认为它们分离弯曲菌的效果相当,没有显著差异[64]。膜过滤培养法已证明可提高弯曲菌检出率,但是这种方法并没有得到广泛应用[65, 66]。虽然培养法是实验室主要选择,但由于存活但不可培养(VBNC)状态细菌的存在,以及肠杆菌科其他细菌的耐药性严重,弯曲菌极易被漏检,造成假阴性结果。本实验室数据证实,中国CDC监制的双孔滤膜法在临床上获得了较好的应用数据[50]。菌落鉴定简单快速的方法是MALDI-TOF质谱,基本解决了弯曲菌的鉴别问题。鉴定明确菌落可通过MH血平板置CO2培养箱48h进行AST(抗菌药物敏感试验),判断标准采用CLSI M45。KB纸片法的药敏结果对临床有良好的指导作用。

图1. 空肠弯曲菌,双孔滤膜CCDA42℃ 72h
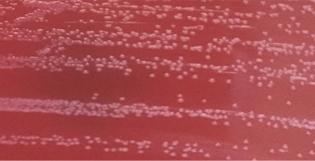
图2. 胎儿弯曲菌血平板42℃,微需氧72h
2. 不依赖培养的检验方法:不依赖培养的检验方法(culture-independent diagnostic tests,CIDT)越来越多的用于弯曲菌检测,主要包括粪便抗原和PCR核酸检测[67]。初期研究认为抗原法与培养法具有相当或更好的特异性和敏感性[68, 69],但近期的实验室评估和临床应用研究显示,对抗原检测灵敏性和阳性预测值提出了质疑[70-72]。美国一项多中心研究,对2,767份粪便标本对商品化弯曲菌检测方法比较,包括粪便抗原、4种选择培养基以及PCR方法,结果显示,粪便抗原检测的敏感性为79.6%至87.6%,特异性为95.9%至99.5%,阳性预测值为41.3%至84.3%。虽然抗原法使用方便,但特异性和阳性预测值变化较大,临床对粪便弯曲菌抗原检测的价值仍存在争议[64]。
PCR检测则显示出更高的灵敏性和特异性,被认为是一种可靠的诊断工具,尤其是实时荧光多重定量PCR(mPCR)[66]。目前FDA批准的包括弯曲菌检测的多重试剂盒有:Prodesse ProGrastro SSCS、xTAG® GPP、BD Max™肠道细菌检测、Verigene、FilmArray。因发展中国家的检验能力有限,弯曲菌感染严重被低估,专家建议将PCR作为首选方法[73]。弯曲菌早期核酸检测的目标是16SrRNA基因可变区,可区分空肠弯曲菌、结肠弯曲菌和拉里弯曲菌。近年开发了针对其他基因的qPCR方法,如编码鞭毛蛋白(flaA/flaB)、热休克蛋白(hsp60)、马尿酸酶(hipO)、致死细胞膨胀毒素(cdt)和硝酸还原酶(nap)的基因。为鉴别细菌是否具有活性,开发了预富集回收活细胞、靶向RNA以及化学法(单叠氮化乙锭和单叠氮化丙锭等化合物)穿透死细胞膜联合DNA检测等。ddPCR具有更好细菌拷贝数定量和敏感性[22]。2017年国内也开发了mPCR检测常见弯曲菌的商用产品。
基于序列的分型方法,包括多位点序列分型 (MLST)、DNA微阵列和全基因组测序,提高了对弯曲菌流行株遗传进化及致病性的认识。MLST是基于七个高度保守的管家基因进行PCR扩增,对扩增片段进行测序,比较核苷酸序列,建立标准系统发育进化树的过程。MLST的优点是使用国际标准化的命名法,数据明确、高度可信并可复现。DNA微阵列法可 对弯曲菌的基因组进行分型,还可检测额外基因组元件、耐药性和毒力基因等,缺点是针对已知靶标和已知细菌,难以识别新发菌株,也无法区分基于MLST法的高度克隆株。下一代基因组测序(NGS)是研究毒力、耐药性和遗传进化更为强大的工具。通过全基因组NGS可发现基因组重排和重组,进一步分析物种进化特征,提高菌株追溯和环境监测的精确度。检验流程见图3。
注:qPCR:实时荧光定量聚合酶链反应;NARMS:美国国家肠道细菌耐药监测中心;CLSI:美国临床与实验室标准化学院
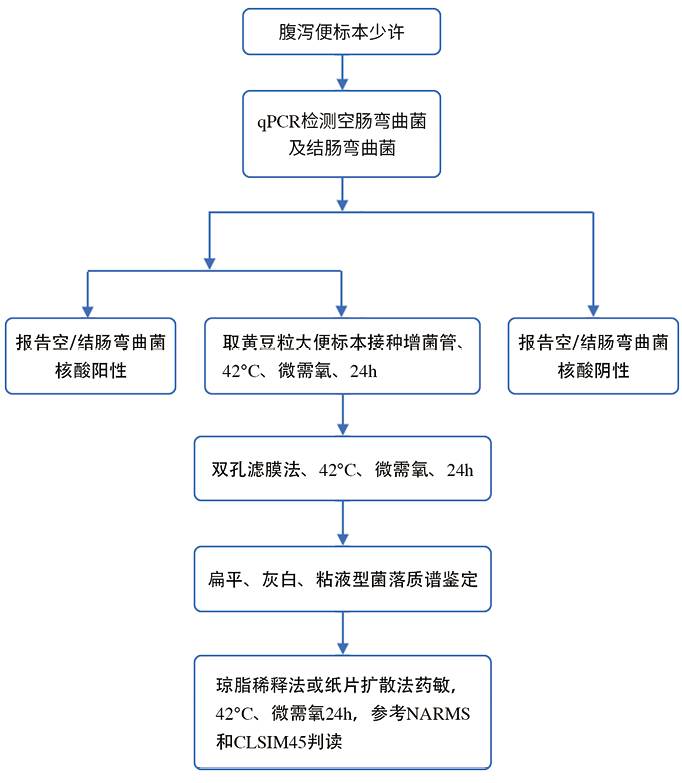
图3. 弯曲菌的实验室常规检验流程图
七、结论与分析
弯曲菌不仅可引起急性腹泻,还可导致严重的慢性疾病,首要问题是提高认识。由于弯曲菌的高度适应性和活跃的遗传进化特点,使弯曲菌在环境中的适应环境能力较强,并可通过基因水平转移获得耐药性和毒力因子。因此,需要更广泛的研究以明确毒株与宿主、食物链与流行之间的关系。临床实验室应建立合理的检测流程,使用快速、低成本的检测方法是保障弯曲菌检测、监测及追踪溯源的有效途径。
参考文献
On S L. Taxonomy of Campylobacter, Arcobacter, Helicobacter and related bacteria: current status, future prospects and immediate concerns[J]. Symp Ser Soc Appl Microbiol, 2001, (30): 1S-15S .doi: 10.1046/j.1365-2672.2001.01349.x.
Dekeyser P, Gossuin-Detrain M, Butzler JP, et al.Acute enteritis due to related vibrio: first positive stool cultures[J]. J Infect Dis, 1972, 125(4): 390-392. doi: 10.1093/infdis/125.4.390.
Black RE, Levine MM, Clements ML, et al. Experimental Campylobacter jejuni infection in humans[J]. J Infect Dis, 1988, 157(3): 472-479.doi: 10.1093/infdis/157.3.472.
Tribble DR, Baqar S, Scott DA, et al. Assessment of the duration of protection in Campylobacter jejuni experimental infection in humans[J]. Infect Immun, 2010, 78(4): 1750-1759. doi: 10.1128/IAI.01021-09.
Gillespie IA, O'Brien SJ, Frost JA, et al. Campylobacter Sentinel Surveillance Scheme Collaborators. A case-case comparison of Campylobacter coli and Campylobacter jejuni infection: a tool for generating hypotheses[J]. Emerg Infect Dis, 2002, 8(9): 937-942. doi: 10.3201/eid0809.010817.
Bourke B, Chan V L, Sherman P. Campylobacter upsaliensis: waiting in the wings[J]. Clinical microbiology reviews, 1998, 11(3): 440-449. doi: 10.1128/CMR.11.3.440.
Costa D, Iraola G. Pathogenomics of Emerging Campylobacter Species[J]. Clinical microbiology reviews, 2019, 32(4): e00072-18. doi: 10.1128/CMR.00072-18.
Hansson I, Sandberg M, Habib I,et al.Knowledge gaps in control of Campylobacter for prevention of campylobacteriosis[J]. Transboundary and emerging diseases, 2018, 65 (Suppl 1):30-48. doi: 10.1111/tbed.12870.
Garénaux A, Jugiau F, Rama F, et al. Survival of Campylobacter jejuni strains from different origins under oxidative stress conditions: effect of temperature[J]. Curr Microbiol, 2008, 56(4): 293-297. doi: 10.1007/s00284-007-9082-8.
Kaakoush NO, Castaño-Rodríguez N, Mitchell HM, et al. Global Epidemiology of Campylobacter Infection[J]. Clinical microbiology reviews, 2015, 28(3): 687-720. doi: 10.1128/CMR.00006-15.
Portner D C, Sleuchner R G K, Murray B S. Optimising the viability during storage of freeze-dried cell preparations of Campylobacter jejuni[J]. Cryobiology, 2007, 54(3): 265-270. doi: 10.1016/j.cryobiol.2007.03.002.
Park S F. The physiology of Campylobacter species and its relevance to their role as foodborne pathogens[J]. Int J Food Microbiol, 2002, 74(3): 177-188. doi: 10.1016/s0168-1605(01)00678-x.
Man S M. The clinical importance of emerging Campylobacter species[J]. Nat Rev Gastroenterol Hepatol, 2011, 8(12): 669-685. doi: 10.1038/nrgastro.2011.191.
Dasti JI, Tareen AM, Lugert R, et al. Campylobacter jejuni: a brief overview on pathogenicity-associated factors and disease-mediating mechanisms[J]. Int J Med Microbiol, 2010, 300(4): 205-211. doi: 10.1016/j.ijmm.2009.07.002.
Enany S, Piccirillo A, Elhadidy M, et al. Editorial: The Role of Environmental Reservoirs in Campylobacter-Mediated Infection[J]. Front Cell Infect Microbiol, 2021, 11:773436. doi: 10.3389/fcimb.2021.773436.
Mohammadpour H, Berizi E, Hosseinzadeh S, et al. The prevalence of Campylobacter spp. in vegetables, fruits, and fresh produce: a systematic review and meta-analysis[J].Gut Pathog, 2018, 10:41. doi: 10.1186/s13099-018-0269-2.
Black RE, Levine MM, Clements ML, et al. Experimental Campylobacter jejuni infection in humans[J]. J Infect Dis, 1988, 157(3): 472-479. doi: 10.1093/infdis/157.3.472.
van Spreeuwel JP, Duursma GC, Meijer CJ, et al.Campylobacter colitis: histological immunohistochemical and ultrastructural findings[J]. Gut, 1985, 26(9): 945-951. doi: 10.1136/gut.26.9.945.
Perkins D J, Newstead G L. Campylobacter jejuni enterocolitis causing peritonitis, ileitis and intestinal obstruction[J]. Aust N Z J Surg, 1994, 64(1): 55-58. doi: 10.1111/j.1445-2197.1994.tb02137.x.
FacciolàA, Riso R, Avventuroso E, et al. Campylobacter: from microbiology to prevention[J]. J Prev Med Hyg, 2017, 58(2): 79-92.
Kreling V, Falcone FH, Kehrenberg C, et al. Campylobacter spp.: Pathogenicity factors and prevention methods-new molecular targets for innovative antivirulence drugs? [J]Appl Microbiol Biotechnol, 2020, 104(24): 10409-10436. doi: 10.1007/s00253-020-10974-5.
Soto-Beltrá N M, Lee BG, Amézquita-López BA, et al. Overview of methodologies for the culturing, recovery and detection of Campylobacter[J]. Int J Environ Health Res. 2022 Feb 16:1-17. doi: 10.1080/09603123.2022.2029366.
Konkel ME, Monteville MR, Rivera-Amill V, et al.The pathogenesis of Campylobacter jejuni-mediated enteritis[J] . Curr Issues Intest Microbiol, 2001, 2(2): 55-71.
Aguilar C, Jiménez-Marín Á, Martins RP, et al. Interaction between Campylobacter and intestinal epithelial cells leads to a different proinflammatory response in human and porcine host[J]. Vet Immunol Immunopathol, 2014, 162(1-2): 14-23. doi: 10.1016/j.vetimm.2014.09.003.
Hickey TE, Baqar S, Bourgeois AL, et al.Campylobacter jejuni-stimulated secretion of interleukin-8 by INT407 cells[J]. Infect Immun, 1999, 67(1): 88-93. doi: 10.1128/IAI.67.1.88-93.1999.
Endtz HP, Ang CW, van Den Braak N, et al. Molecular characterization of Campylobacter jejuni from patients with Guillain-Barré and Miller Fisher syndromes[J]. Journal of clinical microbiology, 2000, 38(6): 2297-2301. doi: 10.1128/JCM.38.6.2297-2301.2000.
Mccarthy N, Giesecke J. Incidence of Guillain-Barré syndrome following infection with Campylobacter jejuni[J]. Am J Epidemiol, 2001, 153(6): 610-614. doi: 10.1093/aje/153.6.610.
Godschalk PC, Kuijf ML, Li J, et al.Structural characterization of Campylobacter jejuni lipooligosaccharide outer cores associated with Guillain-Barre and Miller Fisher syndromes[J]. Infect Immun, 2007, 75(3): 1245-1254. doi: 10.1128/IAI.00872-06.
Houliston RS, Vinogradov E, Dzieciatkowska M, et al.Lipooligosaccharide of Campylobacter jejuni: similarity with multiple types of mammalian glycans beyond gangliosides[J]. J Biol Chem, 2011, 286(14): 12361-12370. doi: 10.1074/jbc.M110.181750.
Bolton DJ. Campylobacter virulence and survival factors[J]. Food Microbiol. 2015 Jun;48:99-108. doi: 10.1016/j.fm.2014.11.017.
World Health Organization. The global view of campylobacteriosis: report of an expert consultation, Utrecht, Netherlands[R]. Geneva: World Health Organization, 2012-7-9
Tack DM, Ray L, Griffin PM, et al.Preliminary Incidence and Trends of Infections with Pathogens Transmitted Commonly Through Food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 2016-2019[J]. MMWR Morb Mortal Wkly Rep, 2020, 69(17): 509-514. doi: 10.15585/mmwr.mm6917a1.
Ray LC, Collins JP, Griffin PM, et al.Decreased Incidence of Infections Caused by Pathogens Transmitted Commonly Through Food During the COVID-19 Pandemic - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 2017-2020[J]. MMWR Morb Mortal Wkly Rep, 2021, 70(38): 1332-1336. doi: 10.15585/mmwr.mm7038a4.
Lake IR, Colón-González FJ, Takkinen J, et al. Exploring seasonality across Europe using The European Surveillance System (TESSy), 2008 to 2016[J].Euro Surveill, 2019, 24(13) :1800028. doi: 10.2807/1560-7917.ES.2019.24.13.180028.
European Food Safety Authority; European Centre for Disease Prevention and Control. The European Union One Health 2019 Zoonoses Report[J]. EFSA J, 2021, 19(2): 06406. doi: 10.2903/j.efsa.2021.6406.
European Food Safety Authority; European Centre for Disease Prevention and Control. The European Union One Health 2020 Zoonoses Report[J]. EFSA J. 2021;19(12):6971. doi: 10.2903/j.efsa.2021.6953
Kirk M, Ford L, Glass K, et al.Foodborne illness, Australia, circa 2000 and circa 2010[J]. Emerg Infect Dis, 2014, 20(11): 1857-1864. doi: 10.3201/eid2011.131315.
Bell R, Draper A, Fearnley E, et al.OzFoodNet Working Group.Monitoring the incidence and causes of disease potentially transmitted by food in Australia: Annual report of the OzFoodNet network, 2016[J]. Commun Dis Intell (2018), 2021,30(9):45. doi: 10.33321/cdi.2021.45.52.
Asuming-Bediako N, Parry-Hanson Kunadu A, Abraham S, et al.Campylobacter at the Human-Food Interface: The African Perspective[J]. Pathogens, 2019, 8(2): 87. doi: 10.3390/pathogens8020087.
Igwaran A, Okoh A I. Human campylobacteriosis: A public health concern of global importance[J]. Heliyon, 2019, 5(11): e02814. doi: 10.1016/j.heliyon.2019.e02814.
Borkakoty B, Jakharia A, Sarmah MD, et al. Prevalence of enteritis in children under 5 years hospitalised for diarrhoea in two cities of Northeast India[J]. Indian J Med Microbiol, 2020, 38(1): 32-36. doi: 10.4103/ijmm.IJMM_19_498.
Rahman MA, Paul PR, Hoque N, et al.Prevalence and Antimicrobial Resistance of Campylobacter Species in Diarrheal Patients in Mymensingh, Bangladesh[J]. BioMed research international, 2021, 3(8):9229485. doi: 10.1155/2021/9229485.
Liang H, Wen Z, Li Y, et al. Comparison of the Filtration Culture and Multiple Real-Time PCR Examination for spp[J]. From Stool Specimens in Diarrheal Patients . Frontiers in microbiology, 2018, 9(12):2995. doi: 10.3389/fmicb.2018.02995.
马晓晨, 王同瑜, 王超, 等. 2018年北京市门诊腹泻病例弯曲菌流行特征[J].首都公共卫生, 2019, 13(6):283-285. doi:10.16760/j.cnki.sdggws.2019.06.017.
刘敏, 鞠长燕, 马艳萍, 等. 深圳市南山区腹泻患者弯曲菌感染及病原学特征分析[J].疾病监测, 2020, (1): 5. doi:10.3784/j.issn.1003-9961.2020.01.006
陆焕, 张蓓洁, 王力, 等. 上海市崇明区2015-2019年成人腹泻病病原学监测结果分析[J].海峡预防医学杂志, 2021, 27(3): 3.
宫霄欢, 吴寰宇, 陈洪友, 等. 上海市2014-2017年成年人感染性腹泻门诊病例的季节分布及病原学特征分析[J].中华流行病学杂志, 2019, 040(008): 889-894. doi:10.3760/cma.j.issn.0254-6450.2019.08.004.
李勇, 强鑫华, 周丽华, 等. 浙江湖州地区2016—2018年食源性腹泻患者病原菌检出情况调查分析[J]. 中国乡村医药, 2020, 27(6): 2. doi:10.3969/j.issn.1006-5180.2020.06.034.
王维, 王林. 2018~2020年西安地区儿童食源性腹泻病病原菌分布特征和耐药性及鼠伤寒沙门菌聚类分析[J].现代检验医学杂志, 2021, 36(4): 5. doi:10.3969/j.issn.1671-7414.2021.04.011.
刘畅, 杨静, 赵建玉,等.食源性腹泻弯曲菌快速鉴定方法评估及耐药性检测[J].中华检验医学杂志,2021, 44(9):6.doi:10.3760/cma.j.cn114452-20210126-00069
Kaakoush NO, Castaño-Rodríguez N, Mitchell HM, et al. Global Epidemiology of Campylobacter Infection[J]. Clinical microbiology reviews, 2015, 28(3): 687-720. doi: 10.1128/CMR.00006-15.
Dai L, Sahin O, Grover M, et al. New and alternative strategies for the prevention, control, and treatment of antibiotic-resistant Campylobacter[J]. Translational research : the journal of laboratory and clinical medicine, 2020, 223:76-88. doi: 10.1016/j.trsl.2020.04.009.
Asakura H, Sakata J, Nakamura H, et al. Phylogenetic Diversity and Antimicrobial Resistance of Campylobacter coli from Humans and Animals in Japan[J]. Microbes Environ, 2019, 34(2): 146-154. doi: 10.1264/jsme2.ME18115.
Elhadidy M, Ali MM, El-Shibiny A, et al. Antimicrobial resistance patterns and molecular resistance markers of Campylobacter jejuni isolates from human diarrheal cases[J]. PLoS One, 2020, 15(1): e0227833. doi: 10.1371/journal.pone.0227833.
Zhou J, Zhang M, Yang W, et al. A seventeen-year observation of the antimicrobial susceptibility of clinical Campylobacter jejuni and the molecular mechanisms of erythromycin-resistant isolates in Beijing, China[J]. International Journal of Infectious Diseases, 2016, 42(C):28-33. doi: 10.1016/j.ijid.2015.11.005.
Hansson I, Sandberg M, Habib I, et al. Knowledge gaps in control of Campylobacter for prevention of campylobacteriosis[J]. Transboundary and emerging diseases, 2018, 65 (Suppl 1):30-48. doi: 10.1111/tbed.12870.
Whitehouse C A, Zhao S, Tate H. Antimicrobial Resistance in Campylobacter Species: Mechanisms and Genomic Epidemiology[J].Adv Appl Microbiol, 2018, 103(12):1-47. doi: 10.1016/bs.aambs.2018.01.001.
Zhang P, Zhang X, Liu Y, et al. Multilocus Sequence Types and Antimicrobial Resistance of Campylobacter jejuni and C. coli Isolates of Human Patients From Beijing, China, 2017-2018[J]. Frontiers in microbiology, 2020, 11:554784. doi: 10.3389/fmicb.2020.554784.
Whitehouse C A , Zhao S , Tate H . Antimicrobial Resistance in Campylobacter Species: Mechanisms and Genomic Epidemiology[J]. Advances in applied microbiology, 2018, 103. doi: 10.1016/bs.aambs.2018.01.001.
Buss JE, Cresse M, Doyle S, et al. Campylobacter culture fails to correctly detect Campylobacter in 30% of positive patient stool specimens compared to non-cultural methods[J]. Eur J Clin Microbiol Infect Dis. 2019,38(6):1087-1093. doi: 10.1007/s10096-019-03499-x.
Bolton F J, Robertson L. A selective medium for isolating Campylobacter jejuni/coli[J]. J Clin Pathol, 1982, 35(4): 462-467. doi: 10.1136/jcp.35.4.462.
Line J E. Development of a selective differential agar for isolation and enumeration of Campylobacter spp[J]. J Food Prot, 2001, 64(11): 1711-1715. doi: 10.4315/0362-028x-64.11.1711.
Karmali MA, Simor AE, Roscoe M, et al. evaluation of a blood-free, charcoal-based, selective medium for the isolation of Campylobacter organisms from feces[J]. Journal of clinical microbiology, 1986, 23(3): 456-459. doi: 10.1128/jcm.23.3.456-459.1986.
Fitzgerald C, Patrick M, Gonzalez A, et al.Multicenter evaluation of Clinical Diagnostic Methods for Detection and Isolation of Campylobacter spp. from Stool[J]. Journal of clinical microbiology, 2016, 54(5): 1209-1215. doi: 10.1128/JCM.01925-15.
Lastovica A J, LE Roux E. Efficient isolation of campylobacteria from stools[J]. Journal of clinical microbiology, 2000, 38(7): 2798-2799. doi: 10.1128/JCM.38.7.2798-2799.2000.
Valledor S, Valledor I, Gil-Rodríguez MC, et al. Comparison of several Real-Time PCR Kits versus a Culture-dependent Algorithm to Identify Enteropathogens in Stool Samples[J]. Sci Rep, 2020, 10(1): 4301. doi: 10.1038/s41598-020-61202-z.
Mcadam A J. Unforeseen Consequences: Culture-Independent Diagnostic Tests and Epidemiologic Tracking of Foodborne Pathogens[J]. Journal of clinical microbiology, 2017, 55(7): 1978-1979. doi: 10.1128/JCM.00678-17.
Granato PA, Chen L, Holiday I, et al. Comparison of premier CAMPY enzyme immunoassay (EIA), ProSpecT Campylobacter EIA, and ImmunoCard STAT! CAMPY tests with culture for laboratory diagnosis of Campylobacter enteric infections[J]. Journal of clinical microbiology, 2010, 48(11): 4022-4027. doi: 10.1128/JCM.00486-10.
Bessède E, Delcamp A, Sifré E, et al. New methods for detection of campylobacters in stool samples in comparison to culture[J]. Journal of clinical microbiology, 2011, 49(3): 941-944. doi: 10.1128/JCM.01489-10.
Giltner CL, Saeki S, Bobenchik AM, et al. Rapid detection of Campylobacter antigen by enzyme immunoassay leads to increased positivity rates[J]. Journal of clinical microbiology, 2013, 51(2): 618-620. doi: 10.1128/JCM.02565-12.
Floch P, Goret J, Bessède E, et al. evaluation of the positive predictive value of a rapid Immunochromatographic test to detect Campylobacter in stools[J]. Gut Pathog, 2012, 4(1): 17. doi: 10.1186/1757-4749-4-17.
Myers AL, Jackson MA, Selvarangan R. False-positive results of Campylobacter rapid antigen testing[J]. Pediatr Infect Dis J, 2011, 30(6): 542. doi: 10.1097/INF.0b013e31821524db.
Platts-Mills JA, Liu J, Gratz J, et al. Detection of Campylobacter in stool and determination of significance by culture, enzyme immunoassay, and PCR in developing countries[J]. Journal of clinical microbiology, 2014, 52(4): 1074-1080. doi: 10.1128/JCM.02935-13.




