外膜孔蛋白在革兰阴性菌耐药中的作用机制研究及展望

赵虎,医学博士,主任技师。现就职于复旦大学附属华东医院,长期从事临床微生物学和分子生物学方面的研究工作,尤其是细菌耐药机制、分子检测和质谱方面的研究,主持国家863项目等科研基金20余项,发表SCI论文50余篇,编著专著6部,获得国家发明专利8项,上海医学科技2等奖1项。编写相关专业指南和专家共识多部。

刘宜昕,博士,现就职于浙江大学医学院附属第一医院;主要研究领域:幽门螺杆菌耐药机制,发表SCI论文6篇。
【摘要】抗菌药物发挥抑菌作用取决于其在细菌胞内达到抑制各自靶标临界浓度的能力。革兰阴性(G-)菌复杂的双层膜构成的渗透屏障严重影响病原菌对抗菌药物的易感性。G-菌的外膜孔蛋白(Porin)直接参与外膜的通透性调控,是介导抗生素跨膜运输的重要通道。本文重点介绍了Porin的结构、表达和开-闭状态的调控在影响抗菌药物通透性方面的最新进展,并总结了Porin在临床抗菌药物耐药性中的重要贡献。这些研究进展有助于我们进一步了解与Porin相关的细菌耐药机制,推动设计和改良具有更大渗透量的抗菌药物分子。
【关键词】Porin;G-病原菌;抗菌药物渗透;抗菌药物耐药
世界卫生组织已确认G-病原体中的抗菌药物耐药性对人类健康构成了紧迫威胁,因此迫切需要了解抗菌药物摄取和在细胞内积累的分子基础,以更好地探究G-菌的耐药机制。G-菌的外膜(outer membrane,OM)由位于内叶的磷脂(phospholipids,PL)和位于外叶的脂多糖(lipopolysaccharides,LPS)组成[2],这种不对称的OM结构将周质与外部环境隔开,所形成的低渗透屏障抑制细菌在低剂量抗菌药物下获得足够的抗菌药物效果[1]。抗菌药物通过外膜进入细菌细胞的关键机制包括具有β-桶状孔隙结构的孔蛋白(Porin)介导的亲水性抗菌药物的一般扩散和脂质介导的疏水性抗药物的渗透途径[3]。鉴于Porin在抗菌药物转运中的核心作用,其是影响多种细菌耐药性的关键因素[4],当前的挑战之一是有效解释外膜Porin介导的抗菌药物渗透性的分子基础。本文综述了Porin的结构特点、表达和开-闭状态的调控及其在抗菌药物耐药性中的作用,旨在进一步理解和探索与Porin相关的G-菌耐药的分子机制。
一、Porin的一般结构
模式菌大肠埃希菌(Escherichia coli)可产生3种经典的三聚体Porin,即OmpF、OmpC和PhoE。基于对这些Porin的研究构成了我们目前了解许多其他Porin的基础,因此它们(及其同源物)被称为“经典孔蛋白”[5],能够允许分子量小于600Da分子的非特异性扩散。OmpF和OmpC同源物存在于肠杆菌科的其他相关成员中,包括产气克雷伯菌(Klebsiella aerogenes)中的Omp35和Omp36、阴沟肠杆菌(Enterobacter cloacae)中的OmpEc35和OmpEc36,以及肺炎克雷伯菌(Klebsiella pneumoniae)中的OmpK35和OmpK36[6]。
外膜Porin由排列成β-桶状的8~24条β链组成,通常被组织为三聚体,由桶的疏水表面和细胞外环L2提供单体间的接触,该环锁定在相邻单体的凹槽中,并产生许多极性相互作用(图1a、b)[7]。目前为止,没有强有力的证据表明三聚体内部存在协同性,Porin单体可能作为独立的亚单位发挥作用,三聚体排列可能只是为了赋予额外的稳定性。从垂直于膜的横截面来看,通道呈沙漏状,最窄的部分称为“小孔”或“收缩区”。对于大肠埃希菌OmpF和OmpC,收缩区的形状大致为圆形,直径分别为6.5-7Å和5.5-6Å。收缩区域由长度为~35个残基的细胞外环L3构成,该环向内折叠,形成一个狭窄的孔(图1a),如果没有这种环,产生的~15Å×23Å的巨大孔隙将严重损害外膜的不透水性[7]。
L3环的另一个重要作用是在收缩区域产生强电场(横向),这是由桶壁上的一排带正电残基(大肠埃希菌OmpC中的Lys16、Arg37、Arg74和Arg124)和L3环上指向收缩区域的主链羰基形成的,这些残基为位于对面收缩区(图1c-e)的带负电残基(OmpC中的Asp105和Glu109)。这种电场在外膜介导的传输方面有两个重要作用,首先它使孔内的水分子定向,使得疏水性小分子渗透到收缩区域[8],其次电场对极性小分子的渗透也有直接影响[9]。
注:(a-b)大肠埃希菌OmpC三聚体的胞外(a)和侧面(b)视图(每个Porin单体的孔用星号表示)。(c-d)OmpC单体的胞外(c)和板条侧(d)视图。(e)OmpC的横截面显示通道的内部漏斗状形状。着色部分为静电点位。
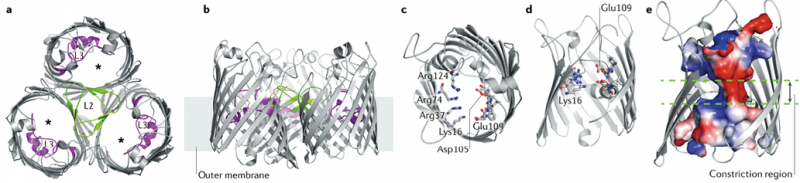
图1. 肠杆菌Porin结构[7]
二、Porin的抗菌药物特异性
除了上述的一般“经典孔蛋白”,其他Porin可以根据其结构(单体或三聚体)和底物特异性进行分类,例如糖分子的特异性扩散通道LamB、核苷转运蛋白Tsx、转运金属和维生素的TonB依赖性转运蛋白FhuA和BtuB等[7]。由此我们需要知道,一般Porin的底物是否有特异性以及这些通道是否具有与其底物真正的结合位点[10]。
早期的电生理学研究描述了包括抗菌药物在内的各种小分子存在下大肠埃希菌OmpF的瞬时电阻,这表明在收缩区域附近存在弱结合位点[11],且3种抗菌药物(氨苄西林、羧苄青霉素和厄他培南)与大肠埃希菌OmpF的共晶结构已被报道,3者结合的位置非常不同:厄他培南结合在细胞外前庭,氨苄青霉素结合在收缩区的细胞外侧,而羧苄青霉素结合在周质前庭[12]。然而,在大肠埃希菌中,潜在的氨苄青霉素和羧苄青霉素结合位点中的极性或带电残基的单核苷酸突变引起了最小抑菌浓度(minimum inhibitory concentration,MIC)的小幅度降低。这些结果除了提示破坏一个抗菌药物结合位点可能会增加易感性,还可能解释了观察到的抗菌药物敏感性的改变是由通道静电的局部变化引起的,这与Porin静电特性的细微变化会影响抗菌药物渗透的观察结果一致[13]。因此,就特异性而言,经典Porin和底物特异性通道(如LamB和核苷通道Tsx)之间存在明显差异,对于这些通道,约毫摩尔浓度足以占据晶体结构中的底物结合位点[14]。尽管如此,最近越来越多的证据表明,经典Porin在一定程度上也具有选择性,并且允许某些化合物比其他物质更容易通过[9, 15]。
三、Porin表达的调控
1. 包膜应激反应:G-菌具有针对细胞膜和细胞壁的独特包膜应激反应(envelopestress responses,ESR)。在这里,我们主要关注通过调节Porin表达和其他Porin调节因子(EnvZ/OmpR、Cpx和σE)来影响抗菌药物耐药性的ESR。porin基因表达的调节涉及多种效应蛋白和调节级联[16-21],其中包括:①双组分系统EnvZ/OmpR:环境渗透压响应系统;②双组分系统CpxAR(Cpx):位于内膜的传感器检测信号并传送到细胞质调节器;③应激反应可替代sigma因子σE:它可以重定向部分或全部的RNA聚合酶以激活转录。其中,应激反应可替代sigma因子σE和双组分系统CpxAR通过检测包膜的变化并调节基因表达来限制应激对细胞的影响,从而参与ESR途径[16, 17, 21]。这些ESR途径有一个共同的调节因子并且相互联系,导致在不同的应激条件下调节相似的基因。
(1)EnvZ/OmpR双组分系统:环境渗透压是通过EnvZ/OmpR双组分系统影响OmpF和OmpC表达的最早研究的外界压力之一[22]。EnvZ是一种膜结合的传感器激酶,OmpR是一种细胞溶质反应调节器,与porin基因的启动子区域结合。激活后,EnvZ自身磷酸化,来自EnvZ的高能磷酸基随后转移到OmpR上的保守Asp残基。磷酸化的OmpR(OmpR~P)是一种转录因子,可对ompF和ompC孔蛋白基因的表达进行不同的调节。在低渗透压下OmpR~P缺乏时,ompF基因具有高亲和力的OmpR结合位点而得以转录。然而,当OmpR~P浓度增加时,这些分子的结合增加导致ompC转录增加和ompF的抑制。此外,研究表明,高温和高渗压可通过诱导高水平的OmpR~P增加反义RNA MicF的转录[23],该RNA可以与ompF mRNA的5’端非翻译区(untranslated region,UTR))结合抑制其翻译。氧化应激和水杨酸盐的存在也会增加MicF的转录,并在转录后抑制ompF的表达。大肠埃希菌在肠道的厌氧环境下生存,然而有趣的是,有学者发现厌氧改变了OmpF和OmpC的渗透调节[24],即在厌氧状态下,即使在低渗透压的培养基中,OmpC的表达水平也相当高,而高渗透压对OmpF的抑制作用比有氧条件下更强。
(2)Cpx系统:Cpx系统包括CpxA传感器激酶和响应调节器CpxR。碱性pH、周质蛋白错误折叠和内膜(inner membrane,IM)异常(如错误折叠的转运蛋白的积累)等包膜应激可诱导辅助蛋白CpxP从CpxA解离,触发CpxA介导的CpxR磷酸化,进而改变蛋白叶酸酶和蛋白酶、呼吸复合物、IM转运体以及细胞壁生物发生酶的表达[25, 26]。Cpx介导的Porin调节发生在多个水平。在转录水平上,CpxR~P已被证明直接结合ompF和ompC启动子[27]。最近,人们发现小IM蛋白MzrA能够连接Cpx和EnvZ/OmpR[28]。在这一途径中,Cpx激活后MzrA直接与EnvZ相互作用,进而EnvZ稳定OmpR~P[28]。在感应不同信号时,Cpx和EnvZ-OmpR之间的互连允许细胞适应不同的环境压力。通过调节众多基因,Cpx已被证明会影响某些抗菌药物的耐药性[17, 25]。然而,它和其他双组分系统在临床分离株多重耐药(multiple drug resistance,MDR)中的确切作用仍缺乏文献报道。
(3)σE通路:大肠埃希菌的σE通路是第一个被鉴定的ESR。在没有诱导信号的情况下,抗sigma因子RseA(一种单程跨膜蛋白)将σE固定在内膜的细胞质侧[29]。周质蛋白RseB与RseA的周质结构域结合,并增强对σE的抑制。在细胞膜应激时,σE通过蛋白水解级联反应从RseA释放,最终导致RseA的完全降解和σE的释放以指导靶基因的转录[30]。该级联反应中的调节性膜内蛋白水解(regulated intramembrane proteolysis,RIP)蛋白酶DegS和RseP分别依次切割RseA的周质和跨膜区域[31]。与σE结合的RseA细胞质结构域(RseAcyto)随后被胞质蛋白酶(主要是ClpXP)释放和降解[32]。该蛋白水解级联是由在外膜蛋白(outermembraneproteins,OMPs)的C末端发现的保守YxF肽(x代表任何氨基酸)诱导的[33]。这种肽通常埋藏在折叠的Porin三聚体中而无法进入胞内。当Porin折叠被破坏时,YxF肽得以暴露并与DegS结合,激活DegS从而切割RseA并启动级联反应[33]。正确的Porin折叠和转运到外膜涉及一系列步骤,破坏这一途径的任何一处都可能导致Porin折叠不当和C末端的暴露。一旦σE被激活,它就会启动对其调节子基因的转录[34]。尽管σE的调节子包括影响细胞许多方面的基因,但其已知的调节子成员中有很大一部分编码OM中传递和组装LPS所需的伴侣蛋白、降解末端错误折叠Porin的蛋白酶,以及至少两个小RNA(RybB和MicA),它们靶向编码Porin的mRNA并介导其降解[35]。因此,σE通路的激活提高了细胞将蛋白质输送到外膜的能力,促进了错误折叠Porin的降解,并减少了新的Porin的合成,从而减轻了系统的负荷。这些系统中的每一个都有助于减少错误折叠的Porin,减少YxF肽诱导信号的激活,从而使DegS恢复到失活状态。
2. 抗菌药物应激反应:通过XylS-AraC家族、soxRS超氧化物反应调节子效应器SoxS、转录调节因子RamA和RamR而产生抗菌药物应激反应。
(1)XylS-AraC家族:包括抗菌药物在内的化学物质可以通过激活XylS-AraC家族的转录调节因子调节Porin的表达[36]。XylS-AraC转录家族在控制包膜通透性方面的重要作用已为人所知。这些转录调节因子包括MarA,MARAB操纵子、RamA、SoxS和Rob编码的关键转录调节因子,它们通过下调porin基因的表达和上调AcrAB等多药外排泵的表达协同减少抗菌药物的细胞内积累[37]。MarRAB在肠道细菌对外部抗菌药物的反应中起着核心作用[38]。marA可以通过与ompF启动子区域的保守marbox(MarA结合位点)结合,直接在转录水平上抑制ompF的表达,或通过激活MicF间接在转录后水平上抑制ompF的表达。在多个临床菌株中已报道了marA和marR的各种点突变或缺失突变导致临床多重耐药表型的案例[5, 38, 39]。
(2)soxRS超氧化物反应调节子效应器SoxS:SoxS与MarA具有约50%的同源性,也属于XylS-AraC转录激活因子家族[40]。在过氧化氢(H2O2)、一氧化氮(NO)或百草枯等氧化剂的存在下,SoxR从还原(非活性)状态转变为氧化(活性)状态,并激活SoxS编码基因的转录。SoxS参与了大肠埃希菌和鼠伤寒沙门菌的MDR,并可诱导MicF和多药外排转运蛋白AcrAB的转录[41]。已有报道确定了从接受喹诺酮类药物治疗的患者中分离出的大肠埃希菌和肠球菌临床菌株,以及在高浓度氟喹诺酮类药物中选择的实验室菌株中的soxR突变促进了MDR[42]。
(3)转录调节因子RamA和RamR:另外两种调节蛋白RamA(激活物)和RamR(阻遏物)已在肠杆菌属、克雷伯菌属和沙门菌属中得到鉴定,但在大肠埃希菌中缺失[43, 44]。RamA与MarA有45%的同源性。RamA通过正反馈直接增强MarA的转录,并且在这些ram基因的两个启动子中检测到保守的marbox(MarA结合位点)。在大肠埃希菌中过表达RamA可通过降低OmpF表达和促进主动外排泵的产生形成对多种抗菌药物的高水平耐药性,包括氯霉素、四环素、替加环素、氟喹诺酮类和甲氧苄啶[45]。在产气克雷伯菌中,RamA参与OM通透性的修饰和细胞内抗生素的外排[46]。RamA还能够抑制肠杆菌属和克雷伯菌属中上述介绍的porin的表达[38]。
3. 小分子调控RNA重塑Porin表达:目前已知大肠埃希菌和沙门菌等肠杆菌编码至少12种OMP调节小分子RNA(sRNA),即InvR[47]、SdsR[48]、MicA[49]、MicL[50]、MicC[51]、MicF[52]、IpeX[53]、OmrAB[54],RseX[55]、RybB[56]和CyaR[57]。这些sRNA在各种应激条件下发挥作用,并参与多种应激反应,包括σE介导的ESR。一个复杂的sRNA-OMP网络正在被发现,其中一些sRNA特异性地作用于单个OMP的mRNA(例如大肠埃希菌的MicF和MicC),而有些则控制多个OMP和非OMP的mRNA靶点(例如大肠埃希菌的MicA和RybB)。同样,同一个OMP的mRNA可能被一个以上的sRNAs靶向调控(例如OmpC的mRNA受MicC、RseX和RybB调控)。另外,这些sRNA可以提供快速反应来放大信号,或通过负反馈发挥作用。值得注意的是,所有这些sRNAs都是通过不完美的碱基配对与包含核糖体结合位点(ribosome binding site,RBS)的5’-UTR结合,从而抑制功能性翻译起始复合物的形成,并需要RNA伴侣Hfq的协助[58]。
4. Porin开-闭状态的调控:Porin的一个重要功能是经历开-闭转变,以调节其对抗菌药物等化合物的传输特性。为了描述这些转变的分子基础,Vasan等人对大肠埃希菌外膜孔蛋白OmpF进行了广泛的分子动力学(molecular dynamics,MD)模拟[4]。马尔可夫状态分析表明,内环L3的大规模运动是能量稳定的开-闭状态之间转换的基础。L3的构象由环上高度保守的酸性残基和OmpFβ桶上的碱性残基之间的氢键控制。对环路构象重要的关键残基的突变改变了开放和闭合状态之间的平衡,并调节抗生素的穿膜易位。该研究还发现有一个点突变G119D倾向于闭合状态,并已在临床耐药菌株中报道。总之,该实验分析表明,内环动力学参与了G-菌Porin的通透性和抗生素耐药性。
5. Porin与抗菌药物耐药:Porin在抗菌药物转运中的核心作用,是影响多种细菌耐药性的关键因素,当前的挑战之一是有效解释外膜Porin介导的抗菌药物渗透性的分子基础。
(1)Porin表达改变与抗菌药物耐药:近期大量文献报道了在对感染患者进行抗菌药物治疗期间收集的临床分离菌株中Porin的丢失或表达改变与抗菌药物耐药的相关性。
肠杆菌科细菌的耐药性主要是由于Porin的表达缺陷或减少,以及各种β-内酰胺酶的存在和外排泵的表达增加所致,这些机制共同降低周质中的活性抗菌药物水平[59]。在缺乏Porin的细胞中外排泵和β-内酰胺酶的效率显著提高,因为周质中抗菌药物的浓度低于酶和转运体的饱和水平[6]。据报道,存在Porin缺失的产超广谱β-内酰胺酶的肠杆菌科临床分离株对喹诺酮类等其他化合物具有耐药性[60]。最近的一项研究描述了OmpR编码基因的点突变,该蛋白是OmpC和OmpF表达的调节因子,因此可能导致碳青霉烯类抗菌药物耐药[61]。此外,在对碳青霉烯类敏感性降低的各种肠杆菌科细菌的研究中,Porin表达与碳青霉烯耐药性水平相关[59]。另外,Porin的替换(例如大通道的OmpF被狭窄的OmpC通道替换)导致的抗菌药物内流减少与在肠杆菌科分离株中观察到的耐药表型相关,即易感分离株同时表达这两种Porin,低度和中度耐药的分离株表达一个截断的Porin,而两种Porin均缺失株则表现出最高水平的耐药,导致β-内酰胺完全无法渗透[5, 6, 62]。有学者提出,这种Porin平衡(OmpF/OmpC)的改变是由Porin基因或控制包膜通透性的调节级联中的调控或突变事件引起的[39]。还有报道称,碳青霉烯类抗菌药物治疗期间,肺炎克雷伯菌中截短型Porin OmpK36的表达显著降低了对各种抗菌药物的敏感性[63]。ompK35和ompK36中的其他突变可能是导致肺炎克雷伯菌株对头孢他啶-阿维巴坦耐药[64, 65],以及对亚胺培南-瑞巴坦或美罗培南-伐博巴坦耐药的原因[66, 67]。最近,Bialek-Davenet等人[68]从一名治疗期间的患者中连续收集了3株对超广谱头孢菌素、氨基糖苷类、四环素、氯霉素和氟喹诺酮类耐药的肺炎克雷伯菌,但具有不同的碳青霉烯类药物敏感性,其中第1株表现为非碳青霉烯酶相关碳青霉烯耐药性,第2株对所有碳青霉烯类药物敏感,第3株对碳青霉烯类药物具有异质易感性。这3个分离株均为同基因株,因为它们都携带了第1个分离株中发现的碳青霉烯耐药性突变,但第2株和第3株在Pho调节子中显示出了其他的突变,这些突变诱导了Porin PhoE的表达,并恢复了对碳青霉烯类药物的敏感性[68]。
对于肺炎克雷伯菌、阴沟肠杆菌和产气克雷伯菌,已发现耐药性的发展与Porin表达水平降低或主要Porin突变缺失有关[69]。类似地,在肠链球菌中,ompC缺失也被认为是导致碳青霉烯类抗菌药物耐药的原因[70]。在临床耐药的斯氏普罗威登菌中发现了Porin OmpPst1表达缺陷或OmpPst1编码基因突变[71]。这些突变位于细胞外环,因此可能与Porin三聚体的伸缩性以及孔的活性构象有关[71, 72]。全基因组测序结合表型和生化特征的研究发现,在2名患者的抗菌药物治疗期间收集的产气荚膜杆菌分离株中出现了与Porin表达改变相关的突变[73]。
(2)Porin收缩区的突变与抗菌药物耐药:Porin和抗菌药物敏感性之间的关系似乎与Porin通道的特性有关。肠杆菌科分离株的Porin OmpC中L3环中的1个或2个氨基酸在Porin通道的收缩区域发生替换或插入突变可导致Porin突变体的产生,由此导致的孔径构象变化和位于收缩区的电荷改变大大减少了β-内酰胺类药物的易位,从而降低了细菌的敏感性[6, 9, 74]。同时,特定氨基酸残基的作用已通过多年的定点突变进行了研究,并指出了Porin通道收缩区的重要性[9]。
在产气克雷伯菌和肺炎克雷伯菌的β-内酰胺耐药分离物中检测到位于L3 loop5的PEFXGD基序中的氨基酸替换(Gly→Asp)。保守的PEFXGD基序在L3环中形成了一个转折,有助于收缩区的形成。该结构域中的Gly→Asp替换还与美罗培南-伐博巴坦耐药有关[67]。此外,一株OmpK36变体在L3环中的另外两种氨基酸位点(Asp137和Thr138)的变异显示出了厄他培南耐药性和美罗培南敏感性降低[75]。
四、结论与展望
Porin是G-菌外膜的重要组成部分,直接参与外膜的通透性,它不仅可以确保细菌代谢所需的营养物质的吸收,还可以通过其独特的结构和功能特性控制环境分子的扩散。对Porin的研究可以追溯到很多年前,但对抗菌药物易位过程的结构和机制的研究近几年才出现。Porin表达和开-闭状态的调控涉及多种调控模式,这些复杂的调控系统有效地控制了Porin的产生和通透性,进而可能控制药物的胞内浓度。Porin收缩区内通道的内部保守结构及其正负电荷的分布以及由此产生的静电场是控制小极性化合物摄入的关键因素。此外,由于Porin通道是保护细菌免受外部有毒化合物侵害的一个膜屏障中的软肋,因此在耐药的临床分离株中出现的Porin丢失的报道也就无独有偶了。另外,虽然Porin的丢失或表达下降理论上可以减少抗菌药物的摄入,提高细菌的耐药性,但Porin的缺乏对这些耐药菌株的适应性和毒力有很大的负面影响,因此,含有基因变异但仍具有功能性Porin的临床分离株可能比完全丧失Porin功能的分离株更具有生存优势,因为它们可以降低细菌的最小适应成本,同时一定程度上降低抗菌药物敏感性,这将有助于耐药性的成功传播。除此之外,以上研究的突破性进展将有助于我们设计和改良药物分子,使其在G-细菌中具有更大的渗透和积累。
参考文献
NIKAIDO H. Molecular basis of bacterial outer membrane permeability revisited [J]. Microbiol Mol Biol Rev, 2003, 67(4): 593-656.
ZGURSKAYA H I, LOPEZ C A, GNANAKARAN S. Permeability Barrier of Gram-Negative Cell Envelopes and Approaches To Bypass It [J]. ACS Infect Dis, 2015, 1(11): 512-22.
GHAI I, GHAI S. Understanding antibiotic resistance via outer membrane permeability [J]. Infect Drug Resist, 2018, 11(523-30.
VASAN A K, HALOI N, ULRICH R J, et al. Role of internal loop dynamics in antibiotic permeability of outer membrane porins [J]. Proc Natl Acad Sci U S A, 2022, 119(8):
MASI M, WINTERHALTER M, PAGES J M. Outer Membrane Porins [J]. Subcell Biochem, 2019, 92(79-123.
MASI M, REFREGIERS M, POS K M, et al. Mechanisms of envelope permeability and antibiotic influx and efflux in Gram-negative bacteria [J]. Nat Microbiol, 2017, 2(17001.
VERGALLI J, BODRENKO I V, MASI M, et al. Porins and small-molecule translocation across the outer membrane of Gram-negative bacteria [J]. Nat Rev Microbiol, 2020, 18(3): 164-76.
WEISS M S, SCHULZ G E. Structure of porin refined at 1.8 A resolution [J]. J Mol Biol, 1992, 227(2): 493-509.
ACOSTA-GUTIERREZ S, FERRARA L, PATHANIA M, et al. Getting Drugs into Gram-Negative Bacteria: Rational Rules for Permeation through General Porins [J]. ACS Infect Dis, 2018, 4(10): 1487-98.
KOJIMA S, NIKAIDO H. Permeation rates of penicillins indicate that Escherichia coli porins function principally as nonspecific channels [J]. Proc Natl Acad Sci U S A, 2013, 110(28): E2629-34.
NESTOROVICH E M, DANELON C, WINTERHALTER M, et al. Designed to penetrate: time-resolved interaction of single antibiotic molecules with bacterial pores [J]. Proc Natl Acad Sci U S A, 2002, 99(15): 9789-94.
ZIERVOGEL B K, ROUX B. The binding of antibiotics in OmpF porin [J]. Structure, 2013, 21(1): 76-87.
LOU H, CHEN M, BLACK S S, et al. Altered antibiotic transport in OmpC mutants isolated from a series of clinical strains of multi-drug resistant E. coli [J]. PLoS One, 2011, 6(10): e25825.
YE J, VAN DEN BERG B. Crystal structure of the bacterial nucleoside transporter Tsx [J]. EMBO J, 2004, 23(16): 3187-95.
PAGES J M, JAMES C E, WINTERHALTER M. The porin and the permeating antibiotic: a selective diffusion barrier in Gram-negative bacteria [J]. Nat Rev Microbiol, 2008, 6(12): 893-903.
GRABOWICZ M, SILHAVY T J. Envelope Stress Responses: An Interconnected Safety Net [J]. Trends Biochem Sci, 2017, 42(3): 232-42.
GUEST R L, RAIVIO T L. Role of the Gram-Negative Envelope Stress Response in the Presence of Antimicrobial Agents [J]. Trends Microbiol, 2016, 24(5): 377-90.
ZSCHIEDRICH C P, KEIDEL V, SZURMANT H. Molecular Mechanisms of Two-Component Signal Transduction [J]. J Mol Biol, 2016, 428(19): 3752-75.
LALOUX G, COLLET J F. Major Tom to Ground Control: How Lipoproteins Communicate Extracytoplasmic Stress to the Decision Center of the Cell [J]. J Bacteriol, 2017, 199(21):
TIWARI S, JAMAL S B, HASSAN S S, et al. Two-Component Signal Transduction Systems of Pathogenic Bacteria As Targets for Antimicrobial Therapy: An Overview [J]. Front Microbiol, 2017, 8(1878.
FROHLICH K S, GOTTESMAN S. Small Regulatory RNAs in the Enterobacterial Response to Envelope Damage and Oxidative Stress [J]. Microbiol Spectr, 2018, 6(4):
CAI S J, INOUYE M. EnvZ-OmpR interaction and osmoregulation in Escherichia coli [J]. J Biol Chem, 2002, 277(27): 24155-61.
DELIHAS N, FORST S. MicF: an antisense RNA gene involved in response of Escherichia coli to global stress factors [J]. J Mol Biol, 2001, 313(1): 1-12.
MATSUBARA M, KITAOKA S I, TAKEDA S I, et al. Tuning of the porin expression under anaerobic growth conditions by his-to-Asp cross-phosphorelay through both the EnvZ-osmosensor and ArcB-anaerosensor in Escherichia coli [J]. Genes Cells, 2000, 5(7): 555-69.
RAIVIO T L. Everything old is new again: an update on current research on the Cpx envelope stress response [J]. Biochim Biophys Acta, 2014, 1843(8): 1529-41.
DELHAYE A, COLLET J F, LALOUX G. Fine-Tuning of the Cpx Envelope Stress Response Is Required for Cell Wall Homeostasis in Escherichia coli [J]. mBio, 2016, 7(1): e00047-16.
BATCHELOR E, WALTHERS D, KENNEY L J, et al. The Escherichia coli CpxA-CpxR envelope stress response system regulates expression of the porins ompF and ompC [J]. J Bacteriol, 2005, 187(16): 5723-31.
GERKEN H, CHARLSON E S, CICIRELLI E M, et al. MzrA: a novel modulator of the EnvZ/OmpR two-component regulon [J]. Mol Microbiol, 2009, 72(6): 1408-22.
DE LAS PENAS A, ConNOLLY L, GROSS C A. SigmaE is an essential sigma factor in Escherichia coli [J]. J Bacteriol, 1997, 179(21): 6862-4.
ADES S E, ConNOLLY L E, ALBA B M, et al. The Escherichia coli sigma(E)-dependent extracytoplasmic stress response is controlled by the regulated proteolysis of an anti-sigma factor [J]. Genes Dev, 1999, 13(18): 2449-61.
ALBA B M, LEEDS J A, onUFRYK C, et al. DegS and YaeL participate sequentially in the cleavage of RseA to activate the sigma(E)-dependent extracytoplasmic stress response [J]. Genes Dev, 2002, 16(16): 2156-68.
CHABA R, GRIGOROVA I L, FLYNN J M, et al. Design principles of the proteolytic cascade governing the sigmaE-mediated envelope stress response in Escherichia coli: keys to graded, buffered, and rapid signal transduction [J]. Genes Dev, 2007, 21(1): 124-36.
WALSH N P, ALBA B M, BOSE B, et al. OMP peptide signals initiate the envelope-stress response by activating DegS protease via relief of inhibition mediated by its PDZ domain [J]. Cell, 2003, 113(1): 61-71.
RHODIUS V A, SUH W C, NonAKA G, et al. Conserved and variable functions of the sigmaE stress response in related genomes [J]. PLoS Biol, 2006, 4(1): e2.
THOMPSON K M, RHODIUS V A, GOTTESMAN S. SigmaE regulates and is regulated by a small RNA in Escherichia coli [J]. J Bacteriol, 2007, 189(11): 4243-56.
RAMANI N, HEDESHIAN M, FREUNDLICH M. micF antisense RNA has a major role in osmoregulation of OmpF in Escherichia coli [J]. J Bacteriol, 1994, 176(16): 5005-10.
DAVIN-REGLI A, BOLLA J M, JAMES C E, et al. Membrane permeability and regulation of drug "influx and efflux" in enterobacterial pathogens [J]. Curr Drug Targets, 2008, 9(9): 750-9.
DU D, WANG-KAN X, NEUBERGER A, et al. Multidrug efflux pumps: structure, function and regulation [J]. Nat Rev Microbiol, 2018, 16(9): 523-39.
DAM S, PAGES J M, MASI M. Stress responses, outer membrane permeability control and antimicrobial resistance in Enterobacteriaceae [J]. Microbiology (Reading), 2018, 164(3): 260-7.
DEMPLE B. Redox signaling and gene control in the Escherichia coli soxRS oxidative stress regulon--a review [J]. Gene, 1996, 179(1): 53-7.
MARTIN R G, GILLETTE W K, RHEE S, et al. Structural requirements for marbox function in transcriptional activation of mar/sox/rob regulon promoters in Escherichia coli: sequence, orientation and spatial relationship to the core promoter [J]. Mol Microbiol, 1999, 34(3): 431-41.
BIALEK-DAVENET S, MARCON E, LEFLON-GUIBOUT V, et al. In vitro selection of ramR and soxR mutants overexpressing efflux systems by fluoroquinolones as well as cefoxitin in Klebsiella pneumoniae [J]. Antimicrob Agents Chemother, 2011, 55(6): 2795-802.
VAN DER STRAATEN T, JANSSEN R, MEVIUS D J, et al. Salmonella gene rma (ramA) and multiple-drug-resistant Salmonella enterica serovar typhimurium [J]. Antimicrob Agents Chemother, 2004, 48(6): 2292-4.
KEENEY D, RUZIN A, BRADFORD P A. RamA, a transcriptional regulator, and AcrAB, an RND-type efflux pump, are associated with decreased susceptibility to tigecycline in Enterobacter cloacae [J]. Microb Drug Resist, 2007, 13(1): 1-6.
O'REGAN E, QUINN T, PAGES J M, et al. Multiple regulatory pathways associated with high-level ciprofloxacin and multidrug resistance in Salmonella enterica serovar enteritidis: involvement of RamA and other global regulators [J]. Antimicrob Agents Chemother, 2009, 53(3): 1080-7.
CHOLLET R, CHevalIER J, BOLLET C, et al. RamA is an alternate activator of the multidrug resistance cascade in Enterobacter aerogenes [J]. Antimicrob Agents Chemother, 2004, 48(7): 2518-23.
PFEIFFER V, SITTKA A, TOMER R, et al. A small non-coding RNA of the invasion gene island (SPI-1) represses outer membrane protein synthesis from the Salmonella core genome [J]. Mol Microbiol, 2007, 66(5): 1174-91.
FROHLICH K S, PAPENFORT K, BERGER A A, et al. A conserved RpoS-dependent small RNA controls the synthesis of major porin OmpD [J]. Nucleic Acids Res, 2012, 40(8): 3623-40.
UDEKWU K I, WAGNER E G. Sigma E controls biogenesis of the antisense RNA MicA [J]. Nucleic Acids Res, 2007, 35(4): 1279-88.
GUO M S, UPDEGROVE T B, GOGOL E B, et al. MicL, a new sigmaE-dependent sRNA, combats envelope stress by repressing synthesis of Lpp, the major outer membrane lipoprotein [J]. Genes Dev, 2014, 28(14): 1620-34.
DAM S, PAGES J M, MASI M. Dual Regulation of the Small RNA MicC and the Quiescent Porin OmpN in Response to Antibiotic Stress in Escherichia coli [J]. Antibiotics (basel), 2017, 6(4):
ANDERSEN J, FORST S A, ZHAO K, et al. The function of micF RNA. micF RNA is a major factor in the thermal regulation of OmpF protein in Escherichia coli [J]. J Biol Chem, 1989, 264(30): 17961-70.
CASTILLO-KELLER M, VUONG P, MISRA R. Novel mechanism of Escherichia coli porin regulation [J]. J Bacteriol, 2006, 188(2): 576-86.
GUILLIER M, GOTTESMAN S. Remodelling of the Escherichia coli outer membrane by two small regulatory RNAs [J]. Mol Microbiol, 2006, 59(1): 231-47.
DOUCHIN V, BOHN C, BOULOC P. Down-regulation of porins by a small RNA bypasses the essentiality of the regulated intramembrane proteolysis protease RseP in Escherichia coli [J]. J Biol Chem, 2006, 281(18): 12253-9.
WASSARMAN K M, KILEY P J. Global approaches for finding small RNA and small open reading frame functions [J]. J Bacteriol, 2010, 192(1): 26-8.
JOHANSEN J, ERIKSEN M, KALLIPOLITIS B, et al. Down-regulation of outer membrane proteins by noncoding RNAs: unraveling the cAMP-CRP- and sigmaE-dependent CyaR-ompX regulatory case [J]. J Mol Biol, 2008, 383(1): 1-9.
VALENTIN-HANSEN P, JOHANSEN J, RASMUSSEN A A. Small RNAs controlling outer membrane porins [J]. Curr Opin Microbiol, 2007, 10(2): 152-5.
DUPONT H, GAILLOT O, GOETGHELUCK A S, et al. Molecular Characterization of Carbapenem-Nonsusceptible Enterobacterial Isolates Collected during a Prospective Interregional Survey in France and Susceptibility to the Novel Ceftazidime-Avibactam and Aztreonam-Avibactam Combinations [J]. Antimicrob Agents Chemother, 2016, 60(1): 215-21.
MARTINEZ-MARTINEZ L. Extended-spectrum beta-lactamases and the permeability barrier [J]. Clin Microbiol Infect, 2008, 14 Suppl 1(82-9.
DUPONT H, CHOINIER P, ROCHE D, et al. Structural Alteration of OmpR as a Source of Ertapenem Resistance in a CTX-M-15-Producing Escherichia coli O25b:H4 Sequence Type 131 Clinical Isolate [J]. Antimicrob Agents Chemother, 2017, 61(5):
HAMZAOUI Z, OCAMPO-SOSA A, MAAMAR E, et al. An Outbreak of NDM-1-Producing Klebsiella pneumoniae, Associated with OmpK35 and OmpK36 Porin Loss in Tunisia [J]. Microb Drug Resist, 2018, 24(8): 1137-47.
FINDLAY J, HAMOUDA A, DANCER S J, et al. Rapid acquisition of decreased carbapenem susceptibility in a strain of Klebsiella pneumoniae arising during meropenem therapy [J]. Clin Microbiol Infect, 2012, 18(2): 140-6.
NELSON K, HEMARAJATA P, SUN D, et al. Resistance to Ceftazidime-Avibactam Is Due to Transposition of KPC in a Porin-Deficient Strain of Klebsiella pneumoniae with Increased Efflux Activity [J]. Antimicrob Agents Chemother, 2017, 61(10):
SHEN Z, DING B, YE M, et al. High ceftazidime hydrolysis activity and porin OmpK35 deficiency contribute to the decreased susceptibility to ceftazidime/avibactam in KPC-producing Klebsiella pneumoniae [J]. J Antimicrob Chemother, 2017, 72(7): 1930-6.
BALABANIAN G, ROSE M, MANNING N, et al. Effect of Porins and blaKPC expression on Activity of Imipenem with Relebactam in Klebsiella pneumoniae: Can Antibiotic Combinations Overcome Resistance? [J]. Microb Drug Resist, 2018, 24(7): 877-81.
SUN D, RUBIO-APARICIO D, NELSON K, et al. Meropenem-Vaborbactam Resistance Selection, Resistance Prevention, and Molecular Mechanisms in Mutants of KPC-Producing Klebsiella pneumoniae [J]. Antimicrob Agents Chemother, 2017, 61(12):
BIALEK-DAVENET S, MAYER N, VERGALLI J, et al. In-vivo loss of carbapenem resistance by extensively drug-resistant Klebsiella pneumoniae during treatment via porin expression modification [J]. Sci Rep, 2017, 7(1): 6722.
LAZARO-PERONA F, SOTILLO A, TROYANO-HERNAEZ P, et al. Genomic path to pandrug resistance in a clinical isolate of Klebsiella pneumoniae [J]. Int J Antimicrob Agents, 2018, 52(5): 713-8.
FERNANDEZ J, GUERRA B, RODICIO M R. Resistance to Carbapenems in Non-Typhoidal Salmonella enterica Serovars from Humans, Animals and Food [J]. Vet Sci, 2018, 5(2):
TRAN Q T, MAIGRE L, D'AGOSTINO T, et al. Porin flexibility in Providencia stuartii: cell-surface-exposed loops L5 and L7 are markers of Providencia porin OmpPst1 [J]. Res Microbiol, 2017, 168(8): 685-99.
EL-KHATIB M, NASRALLAH C, LOPES J, et al. Porin self-association enables cell-to-cell contact in Providencia stuartii floating communities [J]. Proc Natl Acad Sci U S A, 2018, 115(10): E2220-E8.
PHILIPPE N, MAIGRE L, SANTINI S, et al. In Vivo Evolution of Bacterial Resistance in Two Cases of Enterobacter aerogenes Infections during Treatment with Imipenem [J]. PLoS One, 2015, 10(9): e0138828.
BAJAJ H, SCORCIAPINO M A, MOYNIE L, et al. Molecular Basis of Filtering Carbapenems by Porins from beta-Lactam-resistant Clinical Strains of Escherichia coli [J]. J Biol Chem, 2016, 291(6): 2837-47.
GARCIA-FERNANDEZ A, MIRIAGOU V, PAPAGIANNITSIS C C, et al. An ertapenem-resistant extended-spectrum-beta-lactamase-producing Klebsiella pneumoniae clone carries a novel OmpK36 porin variant [J]. Antimicrob Agents Chemother, 2010, 54(10): 4178-84.




