核酸扩增检测技术不同方法特点与临床应用

段勇,医学博士,博士、硕士生导师,一级主任医师,现任昆明医科大学第一附属医院医学检验科主任,昆明医科大学第一临床学院实验诊断学教研室主任,云南省医学会检验分会主任委员。兼任国家卫生健康委临床检验标准专业委员会委员,中国医师协会检验医师分会常务委员,中华医学会检验分会第十届常务委员暨分子诊断学组组长,国家卫生健康委能力建设和继续教育检验医学专家委员会副主任委员、中国医疗保健国际交流促进会临床检验医学分会副主任委员、中国医院协会临床检验管理专业委员会常务委员等。

吴亚敏,硕士研究生毕业。现就职于昆明医科大学第一附属医院检验科,临床检验诊断学专业。
【摘要】核酸扩增检测技术具有灵敏、特异、快速、操作简便等特点,近年来广泛应用于感染性疾病诊断、遗传病的辅助诊断、病原体基因分析及耐药基因检测、个体化用药指导等方面,为医学和生命科学研究提供了强大的技术支持。近年来,新核酸扩增检测技术不断涌现,如超敏PCR技术、数字PCR技术和恒温扩增技术等,为核酸扩增检测技术向更快、更准、更简便方向发展提供了强大的技术支撑。本文就核酸扩增检测技术的进展做一简要介绍。
【关键词】核酸;核酸扩增技术;聚合酶链式反应;恒温核酸扩增
核酸是生物体不可或缺的分子,不仅储存遗传信息,编码蛋白质,而且在疾病诊断[1]、食品安全检测[2]、法医鉴定[3]等许多领域被作为重要的生物标志物。Kary Mullis等[4]在1986年发明了聚合酶链式反应(polymerase chain reaction,PCR),并借助从水生栖热菌中分离出的耐热型DNA聚合酶[5],使PCR自动化成为可能。此后,这一技术迅速发展,成为核酸检测领域最为普遍的方法之一。然而,PCR依赖于耐热的DNA聚合酶和昂贵的扩增仪,限制了其在即时检测(point-of-care testing,POCT)中的应用。为了解决上述问题,研究者们开发了恒温扩增这一新的核酸扩增技术。它与常规PCR相比,恒温核酸扩增避免了变性、退火和延伸等不同阶段的变温要求,在恒温装置中即可高效扩增,因此非常适合在资源有限的环境中使用[6]。本文主要论述和介绍了热循环扩增和恒温扩增中几种常用技术的原理及应用。
一、热循环核酸扩增技术
热循环核酸扩增经过模板DNA变性、引物与模板DNA退火、引物延伸3个步骤的不断循环,可以使待测DNA得以数百万倍的扩增。热循环核酸扩增技术发展主要经历了三个阶段:第一代聚合酶链反应(polymerase chain reaction,PCR)技术,第二代实时荧光PCR(real-time fluorescent PCR)技术,第三代数字PCR(digital PCR,dPCR)技术,表1总结了三代技术的不同特点[7]。
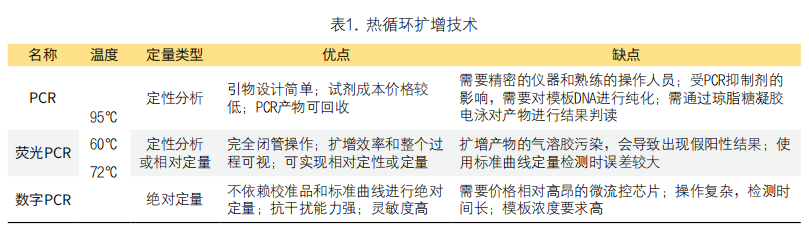
1. PCR技术:PCR是一种用于体外扩增特定双链DNA序列的方法。包括3个步骤组成的重复热循环反应:目标DNA在高温下变性,特定寡核苷酸在目标DNA上杂交,以及在耐热DNA聚合酶的作用下的延伸反应[8]。最初,PCR被用来检测导致镰状细胞性贫血HBB基因的突变,采用的是放射性标记的寡核苷酸杂交和限制性内切酶分析筛查遗传突变[9]。随后PCR技术被应用于病原微生物检测。但由于PCR产物需要应用放射性物质进行标记,然后通过琼脂糖凝胶电泳进行检测,易导致实验室污染、放射性物质会导致辐射、检测灵敏度有限、操作也比较繁琐,因此,PCR技术的临床应用受限。
2. 实时荧光PCR:实时荧光PCR是通过在PCR反应体系中加入荧光染料或荧光探针,利用荧光信号的有无或积累实现对反应过程的实时监测,可为起始目标DNA数量与扩增后扩增子的数量之间提供可靠的量化关系,借助循环阈值和标准曲线实现定性或定量检测目标DNA[10]。为了满足临床的不同需求,实时荧光PCR发展出了多种衍生技术,如熔解曲线法、扩增阻滞突变系统PCR(amplification refractory mutation system-PCR,ARMS-PCR)、多重荧光PCR等。
(1)熔解曲线法:熔解曲线法利用不同DNA序列具有不同Tm值(DNA双螺旋结构解链一半时的温度)的特征,反映了DNA双螺旋结构随温度升高的解链程度。其中,高分辨率熔解曲线法是在反应体系中加入饱和荧光染料,并在PCR反应结束后进行产物升温解链反应来获取荧光信号。通过采集和分析这种变化,记录不同目标产物的熔解曲线及熔解峰,可以用来分析已知基因突变或对未知突变进行筛查、扫描。而探针熔解曲线法则是利用含有检测位点的改良探针结合不对称PCR技术,并在扩增结束后增加熔解曲线荧光采集程序,利用探针对PCR产物的温度熔解进行实时分析,广泛应用于单核苷酸多态性的分析[11]。
(2)ARMS-PCR:又称为等位基因特异性PCR,利用PCR引物3’末端的末位碱基必须与其模板DNA互补才能有效扩增的原理,设计适当的特异性扩增引物并结合TaqMan探针,实现对基因突变位点的高特异性、高灵敏度检测。该方法实现了闭管检测,不需要进行产物的后处理,能更大程度地避免扩增产物的污染。Leelatian等[12]设计了ARMS-PCR与SYBR Green I荧光染料相结合的EGFR突变检测方法,并对扩增条件进行了优化,能够在DNA样本中检测到低至1.5个突变副本的突变,提高了检测的灵敏度。
(3)多重荧光PCR:多重荧光PCR技术是在实时荧光PCR技术的基础上,利用几种不同荧光基团的组合,结合仪器对不同通道荧光的检测能力实现对多个靶标的同时检测。荧光信号的强度与PCR扩增产物的数量成正相关,因此可以通过荧光信号的强度来定量PCR反应中的靶标序列。据不同的临床需求,基于荧光探针、探针编码、荧光染料、荧光探针熔解曲线等多重PCR技术逐步得以运用。主要应用于病原微生物检测、耐药位点的突变、单核苷酸多态性分析、甲基化分析和基因分型等领域,大幅提升了检测效率,降低了试剂成本[13]。
3. dPCR:为了解决实时荧光PCR依赖标准曲线来量化结果及其定量准确性等问题,Bert Vogelstein和Kenneth W. Kinzler在1999年首次提出了dPCR的想法[14]。通过将待测样品分成数千个甚至数百万个子样品,使每个小反应单元中理论上含有1个或者不含待测分子,然后进行检测和统计分析,不需要借助外部的校准曲线就可以对目标核酸进行绝对定量[15]。根据单个微反应单元的生成不同,dPCR可分为两类:基于腔室的数字PCR(chip-based digital PCR,cdPCR)和基于油包水液滴生成的液滴数字PCR(droplet-based digital PCR,ddPCR)。
(1)cdPCR:cdPCR通过将样品被注入由微加工技术制成的芯片中,包括滑动芯片、微阵列芯片以及集成流体电路芯片等,然后进行热循环扩增,用荧光显微镜对芯片进行成像,以确定PCR结果阳性的孔数[16]。孙等[17]报道了一种基于cdPCR实现对SARS-CoV-2病毒基因和肺癌突变基因进行高通量、高灵敏度定量检测,制备的微阵列芯片成功地量化了含有这两种基因的溶液混合物,检测极限达到10拷贝/μL。
(2)ddPCR:被不溶于水的液体(如矿物油)包裹的样品通过微流控芯片被分割成数以万计的分散液滴,形成乳状液,收集乳液后进行PCR,然后用流式细胞仪进行检测,计算PCR阳性的液滴数;或将乳化液送入芯片中,形成单层液滴,进行扩增后捕获并评价液滴的荧光图像,通过统计分析进行定量[18]。随着应用范围的不断扩大,不断开发出了基于微流控技术的ddPCR仪,可以自动产生微液滴并将反应物包裹在独立的空间内进行扩增反应,然后自动计数并显示结果[19]。目前,ddPCR主要用于细菌、病毒等病原体的检测,也可用于疾病诊断和单细胞分析包括循环肿瘤的检测、细胞和突变基因的分析。虽然存在成本高、耗时长等缺点,但ddPCR是一种高精度绝对定量的检测方法,具有广泛的应用前景。
二、恒温核酸扩增技术
恒温扩增是一种可以在恒温条件下扩增核酸的技术,不涉及变性步骤,扩增过程快。此外,恒温扩增和PCR的另一个重要区别是聚合酶的类别,PCR使用的耐热DNA聚合酶只能在单链DNA模板上延伸引物,而恒温扩增使用的聚合酶是恒温酶,扩增的模板可以是双链DNA、单链DNA或RNA。不同恒温扩增技术的建立促进了扩增产物可视化和监测反应动力学新方法的发展。目前比较常见的有环介导恒温扩增(loop-mediated isothermal amplifification,LAMP)、滚环扩增(rolling circle amplifification,RCA)、重组酶聚合酶扩增(recombinase polymerase amplifification,RPA)、它与CRISPR/Cas系统结合恒温扩增等[20]。几种常用的恒温扩增技术的特点在表2中进行了相应的总结(表2)。
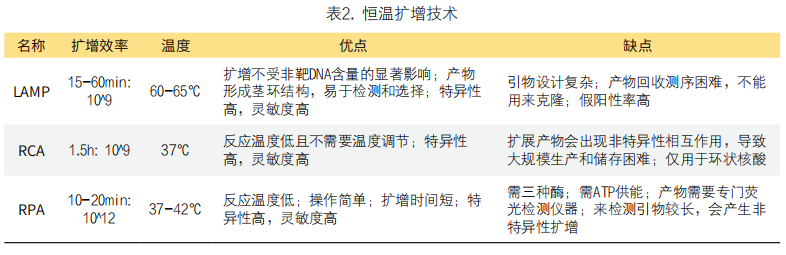
1. LAMP:自嗜热脂肪芽孢杆菌中分离出来的Bst DNA聚合酶具有较高的链置换活性,可以在等温条件下进行反应,无需DNA高温变性过程。LAMP所有阶段都在60-65℃的稳定温度下进行,不需要使用热循环仪来精确控制反应温度和时间,能在15-60分钟内产生大约10^9个拷贝数的目标DNA[21]。此外,LAMP需要四种引物:反向内引物、正向内引物、正向外引物和反向外引物。2002年Nagamine等[22]进一步优化了LAMP方法,通过添加额外的环状引物来加速反应,环状引物(正向和反向)能结合复杂哑铃结构的环状区域并促进DNA合成以加速恒温扩增。
LAMP技术主要应用于病原体检测[23]。它与RT-PCR一样,RT-LAMP也被用于病原体的RNA检测。RT-LAMP的一项重大突破是被用于新型冠状病毒SARS-CoV-2检测,成为一种高效的床旁快速检测方法[24]。有研究者在2021年报道了一种不需进行样品纯化和核酸提取的直接检测病毒的方法[25]。但LAMP技术也存在引物设计复杂;产物回收后测序困难,不能用来克隆;假阳性率高等问题。
2. RCA:Baner等[26]在1998年首次报道了RCA,利用环状模板,在DNA聚合酶的作用下生成长的线性链,与环化寡核苷酸探针相关联,在37℃、1.5小时内产生大约10^9拷贝数。RCA反应过程中有一个合成环状DNA的步骤,这些环状DNA会表现出不同的拓扑问题来阻碍DNA聚合酶的活性,而Phi29 DNA聚合酶在等温条件下具有出色的链置换能力从而解决这一问题。此外,Phi29 DNA聚合酶的3’核酸外切酶活性使RCA能够同时使用mRNA作为模板[27]。
目前RCA技术包括线性RCA、指数RCA、多引物RCA等,并已发展出锁式探针RCA和网状RCA等更加高效的技术。基于RCA设计的生物传感器由于能放大检测信号,可用于除DNA、RNA之外的其他标志物检测[28]。YOU等[29]开发了一种基于RCA和酶促信号放大的快速超灵敏ELISA(RELISA),在传统ELISA基础上,多一个RCA步骤,即可在10分钟内完成检测,使RELISA有望成为各种疾病早期诊断的有力工具。然而,由于非特异性相互作用引起的错误启动,RCA在特异性方面还面临着挑战。
3. RPA:Piepenburg等[30]在2006年开发了一种名为RPA的恒温扩增方法,具有操作简单、扩增效率高(10-20分钟达10^12)、反应温度低(37-42℃)等优点。RPA技术主要使用两种酶:重组酶T4 UvsX和枯草芽孢杆菌Pol I。在ATP和聚乙二醇存在下,重组酶T4 UvsX与RPA引物结合形成重组酶-引物复合物;然后,该复合物可以在双链DNA模板中找到同源序列并引发链置换反应;在dNTPs存在的情况下,DNA聚合酶结合到引物的3’端进行链延伸,形成新的互补链;产生的子代双链DNA又可以作为模板,实现模板上目标区域的指数扩增[31]。
目前常用的RPA方法主要包括实时RPA和RPA结合侧流试纸条等检测方法。实时RPA是将RPA与荧光探针相结合,可以快速检测目标基因,实时监测扩增过程,在耐药基因检测中发挥重要作用[32]。而RPA结合侧流试纸条检测则是将RPA扩增技术、试纸条和免疫检测技术相结合的可视化检测方法,在20分钟内即可得到检测结果,因此可以快速检测病原微生物,有利于大规模筛查[33]。然而,RPA的引物相对较长,通常为30-38 bp,可能会发生非特异性扩增[34]。
4. 恒温扩增技术CRISPR/Cas系统联用:Ishino等[34]1987年在大肠埃希菌中发现了成簇规律间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats,CRISPR)和CRISPR关联蛋白(CRISPR-associated proteins,Cas)组成的原核获得性免疫系统。目前,作为一种基因编辑工具,CRISPR/Cas系统被广泛应用于医学、农学、生物学等多个研究领域。
常用的恒温核酸扩增如LAMP、RCA、RPA等,与CRISPR/Cas系统联合应用可以增强信号,提高检测灵敏度。LAMP、CRISPR/Cas 9和比色反应相结合,实现1小时内完成对SARS-CoV-2及其变异基因的检测[36]。RT-LAMP和Cas 12a相结合的SARS-CoV-2可视化检测方法,可在45min内完成对SARSCoV-2的检测[37]。此外,RCA联合CRISPR/Cas 9平台能够特异性识别RCA扩增的长单链DNA,激活CRISPR/Cas 9对探针信号分子的切割活性,通过记录荧光信号的变化实现恒温下的高特异性检测[38]。另有研究者开发了一种RPA、CRISPR/Cas13a、横向流动试纸条生物传感器相结合的SARS-CoV-2检测方法,并设计了带有微流控芯片的荧光分析仪,可以实现高通量和快速检测[39]。
三、结论与展望
核酸扩增技术因其高敏感性和特异性在分子诊断领域得到了广泛应用。从最初的PCR到目前流行的恒温扩增技术,每一种新扩增技术都为核酸检测提供了新的思路和方法。尽管目前PCR技术应用广泛,但其高度依赖核酸样本的质量、需要耐热DNA聚合酶及热循环仪,因此,PCR难以实现在POCT场景中的应用。恒温扩增技术因具有对设备的要求低、反应温度恒定、扩增速度快、操作简便等优点,是实现POCT的理想方法,但恒温扩增技术目前还存在假阳性、核酸序列相容性及信号放大能力受限等问题。相信随着科学和技术的不断发展,核酸扩增技术尤其是恒温扩增技术将以其强大的优势逐渐成为核酸检测的主要手段。
参考文献
Gingeras TR, Richman DD, Kwoh DY et al. Methodologies for in vitro nucleic acid amplification and their applications[J]. Vet Microbiol, 1990, 24(3-4): 235-51. DOI:10.1016/0378-1135(90)90174-t.
Liu H, Wang J, Zeng H et al. RPA-Cas12a-FS: A frontline nucleic acid rapid detection system for food safety based on CRISPR-Cas12a combined with recombinase polymerase amplification[J]. Food Chem, 2021, 334: 127608. DOI: 10.1016/j.foodchem.2020.127608.
Wu J, Gao HY, Luo L et al. DNA Barcode Technology and Its Application Prospects in Forensic Medicine[J]. Fa Yi Xue Za Zhi, 2020, 36(4): 559-564. DOI: 10.12116/j.issn.1004-5619.2020.04.023.
Mullis K, Faloona F, Scharf S et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction[J]. Cold Spring Harb Symp Quant Biol, 1986, 51 (1): 263-73. DOI: 10.1101/sqb.1986.051.01.032.
Chien A, Edgar DB, Trela JM. Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus[J]. J Bacteriol, 1976, 7(3): 1550-7. DOI: 10.1128/jb.127.3.1550-1557.1976.
Boonbanjong P, Treerattrakoon K, Waiwinya W et al. Isothermal Amplification Technology for Disease Diagnosis[J]. Biosensors (basel), 2022, 12(9): 677. DOI: 10.3390/bios12090677.
Xu D, Zhang W, Li H et al. Advances in droplet digital polymerase chain reaction on microfluidic chips[J]. Lab Chip, 2023, 23(5): 1258-1278. DOI: 10.1039/d2lc00814a.
Green MR, Sambrook J. Polymerase Chain Reaction[J]. Cold Spring Harbor Protocols, 2019, 2019(6): 436-456. DOI: 10.1101/pdb.top095109.
Saiki RK, Scharf S, Faloona F et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia[J]. Science, 1985, 230(4732): 1350-4. DOI: 10.1126/science.2999980.
Arya M, Shergill IS, Williamson M et al. Basic principles of real-time quantitative PCR[J]. Expert Rev Mol Diagn, 2005, 5(2):209-19. DOI: 10.1586/14737159.5.2.209.
张亚旭, 刘紫烟. 荧光PCR扩增相关技术[J]. 中国生物化学与分子生物学报, 2021, 37(7): 890-899. DOI:10.13865/j.cnki.cjbmb.2021.01.1480.
Leelatian N, Boonchoo P, Wijitburaphat S et al. Highly sensitive EGFR mutation detection by specific amplification of mutant alleles[J]. Exp Mol Pathol, 2014, 96(1): 85-91. DOI: 10.1016/j.yexmp.2013.12.006.
钟泽澄, 王进, 张师音. 多重PCR技术研究进展[J]. 生物工程学报, 2020, 36(2): 171-179. DOI: 10.13345/j.cjb.190206.
Vogelstein B, Kinzler KW. Digital PCR[J]. Proc Natl Acad Sci USA, 1999, 96(16): 9236-41. DOI: 10.1073/pnas.96.16.9236.
Sreejith KR, Ooi CH, Jin J et al. Digital polymerase chain reaction technology - recent advances and future perspectives[J]. Lab Chip, 2018, 18(24): 3717-3732. DOI: 10.1039/c8lc00990b.
Zhu H, Zhang H, Xu Y et al. PCR past, present and future[J]. Biotechniques, 2020, 69(4): 317-325. DOI: 10.2144/btn-2020-0057.
Sun Y, Huang Y, Qi T et al. Wet-Etched Microchamber Array Digital PCR Chip for SARS-CoV-2 Virus and Ultra-Early Stage Lung Cancer Quantitative Detection[J]. ACS Omega, 2022, 7(2): 1819-1826. DOI: 10.1021/acsomega.1c05082.
Madic J, Zocevic A, Senlis V et al. Three-color crystal digital PCR[J]. Biomol Detect Quantif, 2016, 10: 34-46. DOI: 10.1016/j.bdq.2016.10.002.
Li Z, Xu X, Wang D et al. Recent advancements in nucleic acid detection with microfluidic chip for molecular diagnostics[J]. Trends Analyt Chem, 2023, 158: 116871. DOI: 10.1016/j.trac.2022.116871.
Jiang H, Li Y, Lv XF et al. Recent advances in cascade isothermal amplification techniques for ultra-sensitive nucleic acid detection[J]. Talanta, 2023, 260: 124645. DOI: 10.1016/j.talanta.2023.124645.
Soroka M, Wasowicz B, Rymaszewska A. [J]Loop-Mediated Isothermal Amplification (LAMP): The Better Sibling of PCR[J]? Cells, 2021, 10(8): 1931. DOI: 10.3390/cells10081931.
Nagamine K, Hase T, Notomi T. Accelerated reaction by loop-mediated isothermal amplification using loop primers[J]. Mol Cell Probes, 2002, 16(3): 223-9. DOI: 10.1006/mcpr.2002.0415.
Zhang X, Zhao Y, Zeng Y et al. Evolution of the Probe-based Loop-Mediated Isothermal Amplification (LAMP) Assays in Pathogen Detection[J]. Diagnostics (basel), 2023, 13(9): 1530. DOI: 10.3390/diagnostics13091530.
Park GS, Ku K, Baek SH et al. Development of Reverse Transcription Loop-Mediated Isothermal Amplification Assays Targeting Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2)[J]. J Mol Diagn, 2020, 22(6): 729-735. DOI: 10.1016/j.jmoldx.2020.03.006.
Alekseenko A, Barrett D, Pareja-Sanchez Y et al. Direct detection of SARS-CoV-2 using non-commercial RT-LAMP reagents on heat-inactivated samples[J]. Sci Rep, 2021, 11(1): 1820. DOI: 10.1038/s41598-020-80352-8.
Banér J, Nilsson M, Mendel-Hartvig M et al. Signal amplification of padlock probes by rolling circle replication[J]. Nucleic Acids Res, 1998, 26(22): 5073-5078. DOI: 10.1093/nar/26.22.5073.
Goo NI, Kim DE. Rolling circle amplification as isothermal gene amplification in molecular diagnostics[J]. Biochip J, 2016, 10(4): 262-271. DOI: 10.1007/s13206-016-0402-6.
Gao X, Li J. Research progress in rolling circle amplification in biosensing and cell imaging[J]. Sci Sin Chim, 2022, 52: 1609–1626. DOI: 10.1360/SSC-2022-0119.
You M, Peng P, Xue Z et al. A fast and ultrasensitive ELISA based on rolling circle amplification[J]. Analyst, 2021, 146(9): 2871-2877. DOI: 10.1039/d1an00355k.
Piepenburg O, Williams CH, Stemple DL et al. DNA detection using recombination proteins[J]. PLoS Biol, 2006, 4(7): e204. DOI: 10.1371/journal.pbio.0040204.
Lobato IM, O'Sullivan CK. Recombinase polymerase amplification: Basics, applications and recent advances[J]. Trends Analyt Chem, 2018, 98: 19-35. DOI: 10.1016/j.trac.2017.10.015.
Wang X, Xu LL, Zuo XY et al. Rapid detection of the New Delhi metallo-β-lactamase (NDM) gene by recombinase polymerase amplification[J]. Infect Genet Evol, 2021, 87: 104678. DOI: 10.1016/j.meegid.2020.104678.
Tan M, Liao C, Liang L et al. Recent advances in recombinase polymerase amplification: Principle, advantages, disadvantages and applications[J]. Front Cell Infect Microbiol, 2022, 12: 1019071. DOI: 10.3389/fcimb.2022.1019071.
Wang M, Liu H, Ren J et al. Enzyme-Assisted Nucleic Acid Amplification in Molecular Diagnosis: A Review[J]. Biosensors (basel), 2023, 13(2): 160. DOI: 10.3390/bios13020160.
Jansen R, Embden JD, Gaastra W et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-75. DOI: 10.1046/j.1365-2958.2002.02839.x.
Song J, Cha B, Moon J et al. Smartphone-based SARS-CoV-2 and Variants Detection System using Colorimetric DNAzyme Reaction Triggered by Loop-Mediated Isothermal Amplification (LAMP) with Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR)[J]. ACS Nano, 2022, 16(7): 11300-11314. DOI: 10.1021/acsnano.2c04840.
Wang R, Qian C, Pang Y et al. opvCRISPR: One-pot visual RT-LAMP-CRISPR platform for SARS-cov-2 detection[J]. Biosens Bioelectron, 2021, 172: 112766. DOI: 10.1016/j.bios.2020.112766.
Wang R, Zhao X, Chen X et al. Rolling Circular Amplification (RCA)-Assisted CRISPR/Cas9 Cleavage (RACE) for Highly Specific Detection of Multiple Extracellular Vesicle MicroRNAs[J]. Anal Chem, 2020, 92(2): 2176-2185. DOI: 10.1021/acs.analchem.9b04814.
Cao G, Huo D, Chen X et al. Automated, portable, and high-throughput fluorescence analyzer (APHF-analyzer) and lateral flow strip based on CRISPR/Cas13a for sensitive and visual detection of SARS-CoV-2[J]. Talanta, 2022, 248: 123594. DOI: 10.1016/j.talanta.2022.123594.




