新冠肺炎疫情前后肺炎支原体感染的流行病学

孙红妹,研究员、博士生导师。现任首都儿科研究所研究员(曾任细菌研究室主任),中国协和医科大学博士生导师。学术兼职:现担任亚洲支原体学会副理事长兼秘书长,中国微生物学会人兽共患病专业委员会委员、支原体学组组长,北京免疫学会理事,国家自然科学基金和北京市自然科学基金同行评议专家,人兽共患病学报、中华儿科杂志、JCM等多家国内外杂志编委及审稿专家。主要从事肺炎支原体研究30年,在肺炎支原体基因流行特征研究方面处于国际领先水平。发表支原体相关核心期刊和SCI学术论著70余篇;主编和副主编《支原体感染实验室诊断技术》、《支原体学》;获得国家发明专利4项,北京市科学技术奖一项。是北京市三八红旗奖章获得者,享受国务院政府特殊津贴专家。

萧丽,博士,现任亚拉巴马大学伯明翰分校医学系助理教授,支原体诊断实验室技术主任。主要研究方向包括:支原体临床诊断技术开发,支原体分子流行病学调查,支原体耐药机制及流行性研究,支原体致病机制研究等。
【摘要】肺炎支原体(Mycoplasma pneumoniae)是一种无细胞壁的呼吸道病原体,可以引起社区获得性肺炎和肺外并发症。肺炎支原体通过密切接触传播,周期性地引起地区性暴发流行。新型冠状病毒病(COVID-19)大流行阻断了肺炎支原体的全球传播。新冠疫情后新一轮肺炎支原体感染开始在我国大流行。这篇综述总结了关于肺炎支原体的发病机制、流行病特征、大环内酯类耐药性、诊断方法和临床治疗策略等方面内容,旨在协助临床医务工作者更好地了解和应对肺炎支原体的流行。
【关键词】肺炎支原体;流行病;基因型;大环内酯类耐药性;诊断治疗
肺炎支原体(Mycoplasma pneumoniae)是一种引起人类呼吸道感染的重要和常见的病原体,儿童肺炎由肺炎支原体引起的占10-30%。肺炎支原体还可以引起呼吸道以外很多器官、系统的并发症。肺炎支原体感染每2至7年引发一起地区性暴发流行。由于其独特的生物学特性,肺炎支原体感染在其流行病、临床表现以及诊断和治疗策略方面具有特殊性。新型冠状病毒(Coronavirus disease,COVID-19)流行期间采取的防控措施在一定程度上阻断了肺炎支原体在全球的传播。目前,新冠肺炎后的新一轮肺炎支原体暴发流行正在我国出现。为了帮助临床医生更好地理解、诊断和治疗肺炎支原体感染,本文论述了肺炎支原体感染的发病机制和流行病学研究的最新进展。
一、肺炎支原体的生物学特征和致病性
支原体是能够独立完成自我复制的最小生物体。由于肺炎支原体体积小、没有细胞壁,导致其很多生物学特征和致病性与其他细菌不同。支原体在加压情况下可以通过220nm的滤膜,实验室通常采用的450nm的除菌滤器不能过滤掉肺炎支原体。支原体基因组小、富含AT,降低了其代谢和生物合成能力。肺炎支原体缺乏三羧酸循环途径功能,不能进行氧化磷酸化,其所需能量是通过底物磷酸化产生的。由于细胞壁的缺失,靶向于细胞壁合成的抗生素对支原体无效。因此,肺炎支原体感染的实验室诊断对于指导临床医生正确选择抗生素至关重要。
肺炎支原体细胞呈纺锤状,具有特殊的尖端结构可使其附着在宿主细胞上[1]。肺炎支原体的基因组为816kbp,包含687个蛋白质编码基因,基因组大小不到大肠杆菌的五分之一。由于其有限的生物合成能力,肺炎支原体以寄生方式生存,依赖宿主提供氨基酸、胆固醇和叶酸等必需营养素。体外培养需要成分复杂的培养基,而且肺炎支原体生长缓慢,增殖一代需要1-6小时。肺炎支原体能够进行糖酵解,将葡萄糖代谢为乙酸和乳酸,生成三磷酸腺苷[2]。
肺炎支原体的致病机制尚不完全清楚。目前已经明确的主要机制有三个方面,包括对宿主的直接和间接损伤以及免疫逃避。直接损伤是由支原体细胞最初黏附到宿主细胞并释放有毒分子所致。细胞黏附对于肺炎支原体的定植和感染至关重要。黏附作用主要由肺炎支原体的P1黏附素和黏附辅助蛋白介导,烯醇化酶等兼职功能蛋白也参与了黏附作用[3]。肺炎支原体的黏附导致宿主细胞营养消耗、局部呼吸道上皮损伤、纤毛运动减弱和上皮细胞脱落,并引发社区获得性呼吸窘迫综合征(community-acquired respiratory distress syndrome,CARDS)毒素的释放,而该毒素参与宿主的炎症反应[4]。肺炎支原体还通过甘油-3-磷酸氧化酶(GlpO或GlpD)[5]产生过氧化氢,并通过参与细胞毒性和溶血活性的半胱氨酸脱硫酶/脱硫水解酶(HapE)产生硫化氢[6]。
这与许多其他类型的感染相似,宿主免疫反应是决定肺炎支原体感染结果的重要因素。一个完整的体液免疫系统能确保控制感染,但过度的炎症反应却又会导致间接的宿主组织损伤。肺炎支原体的细胞成分和代谢产物,如CARDS毒素、膜脂、脂蛋白和荚膜成分,可以作为促炎因子激活宿主炎症反应而产生包括干扰素γ、肿瘤坏死因子α等细胞因子,和白介素-1β(IL-1β)、IL-2、IL-4、IL-5、IL-6、IL-8、IL-10和IL-18等白细胞介素。近期研究表明,宿主非编码RNA(ncRNA),包括微小RNA(miRNA)、长非编码RNA和环状RNA可以被包装在循环体系中的细胞外囊泡中,参与肺炎支原体感染的发生与发展[7]。宿主免疫激活的程度可能决定了要么病原体被清除,要么呼吸道上皮组织加重损伤。
肺炎支原体采用几种机制进行自我保护以免受免疫清除和实现免疫逃避。一种众所周知的机制是P1黏附素的抗原变异,它是由P1基因与分散在整个基因组中的重复序列之间的同源重组介导的[8]。肺炎支原体能产生一种分泌性镁依赖性核酸酶MPN491以降解中性粒细胞胞外陷阱(neutrophil extracellular traps,NETs)[9],还能产生一种氢过氧化物酶MPN668以降解过氧化物[10]。肺炎支原体可以与补体控制蛋白因子H等人体免疫分子结合以逃避补体杀伤[11],还可以通过称为支原体免疫球蛋白结合蛋白的MPN400与免疫球蛋白IgG、IgA和IgM结合[12]。肺炎支原体可以内化并在宿主细胞中存活[13],也可以形成生物膜[14]。这些自我保护措施促进了肺炎支原体在宿主中的持久存在和扩散。
二、肺炎支原体的传播途径和易感人群
肺炎支原体从感染者的鼻、咽、喉和气管分泌物中排出,并通过飞沫或气溶胶传播。肺炎支原体在潜伏期和治疗期都具有传染性,在发病的前4-6天传染性最强,治疗3-5周后传染性逐渐消失。由于其生长缓慢,肺炎支原体感染的潜伏期为1-3周[1]。
肺炎支原体很容易在封闭或半封闭环境中的个体中流行传播,如幼儿园、学校、办公室、疗养院、军队和监狱。在家庭成员之间传播的几率也很高。免疫功能低下的个体更容易感染肺炎支原体。
三、肺炎支原体的流行特征
肺炎支原体感染易发生区域性流行传播,但全年在全球范围内都能检测到这种病原体的感染。在中国南部地区,炎热的夏季发病率会增加;而在中国北部,如北京等地区,冬季发病率会更高[15-17]。与其他病原体感染不同,肺炎支原体每2-7年(平均4年)会发生一次区域性暴发流行[18]。每次肺炎支原体暴发流行可持续1-2年,甚至更长时间。1977年至2018年,首都儿科研究所监测到1979年、1983年、1990年、1995年、2002年、2007年、2011年和2015年北京共发生的8起肺炎支原体暴发流行疫情[15]。2015年肺炎支原体的暴发流行从2015年5月持续到2017年3月。数据显示,2019年夏天新一轮疫情已经开始。其后,由于从2020年开始实施严格的新冠肺炎控制政策,这场肺炎支原体暴发流行被有效地阻断了[19]。世界其他地区也出现了这种现象。从1981年起,日本也出现了与我国类似的肺炎支原体暴发流行趋势[20]。此外,欧洲也报道了2010-2012年、2014-2015年以及2015-2017年肺炎支原体的暴发流行,且在2019年新型冠状病毒感染大流行期间,全球肺炎支原体发病率急剧下降[21]。随着新冠肺炎疫情后人们恢复正常活动,我国目前正在经历新一轮肺炎支原体流行疫情的暴发[22]。许多其他国家也在密切关注近期可能出现的肺炎支原体暴发流行。为应对此轮暴发,我们迫切需要一个有组织的区域和全球肺炎支原体感染监测项目。
四、肺炎支原体感染率
肺炎支原体的检出率因人群、检测时间和诊断方法而异。由于多数肺炎支原体感染的症状相对较轻,症状与其他病原体引起的呼吸道感染相似,以及缺乏可靠的肺炎支原体实验室诊断方法,许多感染者因没有进行肺炎支原体检测或检测漏诊而未被发现。
据报道,肺炎支原体感染的发病率为常住人口的1-3‰。肺炎支原体肺炎病例占各种病原引起的肺炎病例总数的10%-30%,流行暴发年的感染率可达到30%-50%,流行暴发期间感染高峰季节的感染率更高。据报道,肺炎支原体是导致美国2015年儿童社区获得性肺炎(CAP)住院的最常见细菌病原体,聚合酶链式反应(PCR)检出率为8%,而肺炎链球菌的检出率为4%[23]。人们普遍认为CAP是肺炎支原体感染的主要临床表现。然而,只有3%-10%的肺炎支原体呼吸道感染儿童会发展到CAP,其中不到5%的CAP病例严重到需要住院治疗。这些数据不包括在医院就诊但未接受肺炎支原体检测或未到医院就诊的肺炎支原体感染者。因此,真实的肺炎支原体感染情况远高于报道的肺炎支原体感染数据。
五、肺炎支原体感染的临床特征
大多数肺炎支原体感染患者临床症状轻微,可以自愈。肺炎支原体感染最常见的临床表现是咳嗽、发热、流涕和呕吐[24]。一些研究报告表明疾病的严重程度与肺炎支原体载量相关[25]。无症状感染在儿童中很常见[26],但有些肺炎支原体感染可以发展为持续数周或数月的严重感染。难治性肺炎支原体肺炎在我国也有频繁报道[27]。
除了引起呼吸道感染外,肺炎支原体还可引起人体多器官和系统的并发症,如胸腔积液、脑膜脑炎、心肌炎、溶血性贫血、肾小球肾炎、关节炎、皮疹、中耳炎、短暂失明和血栓形成等[3]。肺炎支原体可引发自身免疫性疾病,这是因为其具有与人类宿主相似的抗原成分刺激宿主而产生自身抗体。肺炎支原体感染可以通过引起直接局部炎症、血管栓塞或间接免疫调节等机制,在不存在肺炎的情况下直接发生独立的肺炎支原体肺外感染。因此,不仅是呼吸道感染患者需要做肺炎支原体实验室检测,对肺炎支原体可能引起的其它系统疾病也需要进行肺炎支原体实验室诊断,以便及时确诊肺炎支原体引起的肺外并发症,尤其是在肺炎支原体流行的年份和季节。
由于肺炎支原体对呼吸道上皮细胞的破坏,肺炎支原体感染通常合并和继发其他病原体的混合感染。因此,明确的细菌性或病毒性肺炎也应同时进行肺炎支原体的实验室检测,以避免由于漏诊后不正确的抗生素使用导致难治性肺炎的发生。
六、肺炎支原体的实验室诊断
常用的肺炎支原体实验室诊断检测主要包括血清学抗体检测和核酸检测。肺炎支原体培养耗时长,不适合用于指导临床肺炎支原体感染的治疗。血清学检测需要急性期和恢复期血清才能确诊。核酸检测通常快速、特异性和灵敏度高,但可能需要特定的设备和人员培训。有综述文章对这些方法进行了全面的比较 [3]。
肺炎支原体感染的核酸检测适用于人体采集的各种标本。对于呼吸道感染,常见的标本包括鼻拭子或咽拭子、气管抽吸物、肺组织、胸膜分泌物、痰液和支气管肺泡灌洗液。肺外标本包括血液、体液(如脑脊液和心包液)、皮损或任何组织[3]。下呼吸道标本是检测肺炎支原体肺炎的首选标本,与上呼吸道标本相比,它的肺炎支原体载量更高[28]。然而,由于鼻、咽拭子易于采集,且一些患者特别是儿童不能产生充足的痰液,鼻、咽拭子是最常见的肺炎支原体检测样本。另外,为确保从粘蛋白基质中有效释放病原体,在处理痰液样本时还需要采取特殊的化学和/或机械液化程序,如粘液溶解剂二硫苏糖醇等。
由于检测方法简单,在我国和世界许多地区广泛使用血清学方法检测肺炎支原体感染。在儿童首次感染的1周内,肺炎支原体特异性IgM抗体开始上升,2-3周抗体水平达到峰值。发病28天后,血清IgM抗体水平显著下降。肺炎支原体特异性IgG水平比IgM晚1-2周上升,并可在数月甚至更长时间内维持在较高水平。肺炎支原体特异性IgM抗体用于诊断近期感染仅适用于儿童,成人在反复感染肺炎支原体后一般不再产生特异性IgM抗体。一旦成年人感染肺炎支原体后特异性IgM抗体呈阳性,该抗体可能会持续阳性数月或数年。在这种情况下,特异性IgM抗体阳性不能用于诊断肺炎支原体的当前或近期感染[3]。对肺炎支原体近期感染的确诊建议是,在不同时间(最好间隔2周)采集的两份配对血清中,肺炎支原体特异性IgM或IgG抗体滴度变化≥4倍。选择商业血清学检测试剂盒时应谨慎,因为一种试剂盒若仅检测一种肺炎支原体特异性抗体可能会导致假阴性结果。肺炎支原体感染也会引起冷凝集素反应,通常在感染后第一周末出现,2-3个月后消失。冷凝集素反应是非特异性的,主要涉及IgM,如果滴度≥1:64(同时出现肺炎支原体抗体阳性)提示近期肺炎支原体感染[29]。肺炎支原体特异性抗体阳性结合冷凝集试验结果有助于诊断无症状肺炎支原体感染。
核酸检测可直接检测肺炎支原体,尽管可能受到抑制剂和模板数量的影响,该方法仍具有较高的特异性和敏感性。目前,世界各地都有许多商业和实验室开发的核酸检测方法可用于肺炎支原体的诊断。大多数核酸检测是使用DNA作为模板进行核酸扩增,靶向目标是肺炎支原体的特异基因或遗传因子(包括p1和非编码重复元件repMp1)。检测方法主要是实时PCR法,而环介导的等温扩增法可以简化实验操作。使用肺炎支原体RNA为模板的核酸检测法可以检测活体肺炎支原体[30,31]。欧洲和我国市场上已经有一些RNA检测试剂盒上市。很多商业检测是全自动化的,并能同时检测其他病原体。技术的进步极大地解放了人力和减少了实验误差,并扩大了人们获得综合诊断信息的能力。最近,二代测序技术已应用于包括肺炎支原体在内的多种呼吸道病原体的诊断,并可预测微生物耐药性[32]。16SrRNA基因测序和宏基因组学已被用于研究肺炎支原体感染者上呼吸道、下呼吸道以及肠道的微生物群变化。多项研究表明,呼吸道菌群多样性降低和肺炎支原体丰度增加与肺炎支原体感染有关,这也揭示了这些技术的在肺炎支原体诊断上的价值[33,34]。
总之,考虑到肺炎支原体的特性和治疗特殊性,DNA和/或RNA核酸检测与血清学检测相结合可以更全面地确保肺炎支原体感染诊断的准确性。在肺炎支原体感染的早期阶段,针对肺炎支原体的特异性抗体的产生和抗体滴度的增加需要时间,因此,该阶段的血清学检测可能会导致肺炎支原体感染漏诊,而核酸检测可能会提供更准确的结果。在肺炎支原体感染的恢复期,来自死亡肺炎支原体细胞的DNA以及可能发生的肺炎支原体特异性抗体的持续增加都会导致DNA和血清学检测的假阳性结果。在这一阶段,基于RNA检测肺炎支原体活体的RNA核酸检测法能更可靠地确定临床治疗的有效性[35]。
关于肺炎支原体的抗菌药物敏感性检测,2011年国际临床和实验室标准研究所发布了抗菌药物敏感性测试标准方法[36]。然而,这种方法的建立需要采用难以获得的肺炎支原体临床标本分离培养物。作为替代方案,基于PCR的对大环内酯类药物耐药性(MR)的检测方法已经被开发且已投入市场[37]。
总而言之,临床医生对肺炎支原体感染的最终诊断需要根据实验室检测结果、临床症状、体征和其他辅助检查(如胸部放射线检查等)进行综合分析判断。
七、大环内酯类药物耐药性
大环内酯类抗生素是治疗肺炎支原体感染的首选药物,尤其是对儿童。21世纪初,日本出现了对大环内酯类药物产生耐药性的肺炎支原体[38],之后在全球范围内不断检测到耐药性肺炎支原体。肺炎支原体对大环内酯类药物的耐药性与23S rRNA第V结构域2063和2064位点的点突变,以及核糖体蛋白L4和L22的突变相关。最常见的耐药突变是23S rRNA中2063位点从A到G的基因点突变(A2063G)。
肺炎支原体的大环内酯类药物耐药率在不同区域和不同时段有所不同。综合分析2000年至2020年间22个国家的数据显示,在17873株肺炎支原体中,8836株(49%)对大环内酯类药物具有耐药性[39]。亚洲的耐药率较高(63%),欧洲、北美和世界其他地区的耐药率较低(0%-8.6%)。在过去20年中,全球肺炎支原体大环内酯类耐药率从2008年前的27%增加到2012-2014年的51%,其后在2018-2020年降至23%。从地区来看,我国的耐药率一直在较高水平随时间波动,平均为81%。日本的耐药趋势变化比较明显,从2009-2011年的峰值72%下降到2018-2020年的14%。大环内酯类药物耐药率的变化与在不同地区、不同时段对大环内酯类药物的使用情况有关。在日本,大环内酯类药物的使用在2011年达到峰值,同年推荐采用托氟沙星作为治疗儿童肺炎支原体肺炎的次选药物,这与其大环内酯类药物耐药率下降密切相关。
八、肺炎支原体基因型与临床表现
根据P1基因的序列变化,肺炎支原体菌株可分为两个主要的基因型,分别对应于P1-1和P1-2基因型[40,41]。其他基因分型方法,如多位点可变串联重复序列分析(MLVA)和多位点序列分型(MLST),也可以将肺炎支原体分为不同的簇[42-44]。基因分型已被用于监测肺炎支原体流行期间菌株的传播,及研究基因型与临床表现之间的相关性。肺炎支原体P1-1和P1-2两种基因型每8-10年发生一次流行优势变化,过渡期为2-3年[45]。世界上不同的地区可能表现出不同的基因型转变速度。这种转变被认为与群体免疫的转变有关[46]。由于新冠肺炎疫情对肺炎支原体流行的阻断,人群对肺炎支原体任何基因型的群体免疫力都大大降低,这使新出现的任何肺炎支原体基因型在人群中均有可能快速传播。我国目前的肺炎支原体暴发流行就是这种案例的表现。
最近研究发现,大环内酯类药物的耐药性与肺炎支原体基因型有关。整体分析显示,P1-1型(94.8%)和MLVA 4-5-7-2型(85.3%)在大环内酯类耐药菌株中占主导地位[39]。然而这一观察结果可能只是反应了在研究期间占优势的肺炎支原体菌株对抗生素广泛暴露的情况。最近报告的数据显示,在中国、日本和韩国,从P1-1向P1-2的基因型转变伴随着总体肺炎支原体耐药率的短暂下降和P1-2型及其他分型系统中与其相应的基因型的快速增加[20,47]。本文表1总结了P1-2型或其相关基因型MLVA 3-5-6-2和MLST克隆复合物2(CC2)比例及其大环内酯类药物耐药率在新冠肺炎大流行之前和期间的变化情况(见表1)。2019年后,我国P1-2菌株的大环内酯类耐药率迅速上升至90%以上[48-52],这与之前的P1-1优势菌株耐药率相似。感染大环内酯类药物耐药的肺炎支原体可能会使临床表现更加复杂,导致发热时间延长,住院时间延长,肺外并发症增加以及严重的影像学表现等。
表1. 新冠肺炎疫情前和期间P1-2及其相应基因型菌株比例和大环内酯耐药率(MR)(中国、日本、韩国)
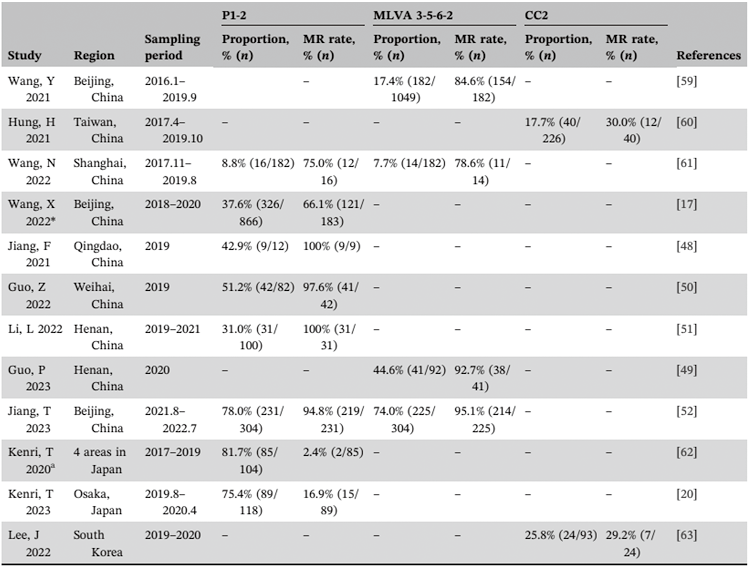
注:a仅使用了指定时间段内的部分数据
关于临床表现的严重程度与肺炎支原体基因型之间的相关性存在着相互矛盾的研究发现。最近一些研究结果显示,P1-2或MLVA 3-5-6-2基因型菌株与严重肺炎和史蒂文斯-约翰逊综合征有关[53,54]。这可能是与CARDS毒素在这些菌株中的高表达和更强的生物膜形成相关[14,55]。因此,随着当前优势菌株基因型从P1-1(或MLVA 4-5-7-2和CC1)转变为P1-2(或MLVA 3-5-6-2和CC2),应密切重视严重和难治性肺炎支原体肺炎增加的潜在可能性。
九、肺炎支原体感染的治疗
大环内酯类药物可有效治疗未产生耐药性的肺炎支原体感染。治疗大环内酯类药物耐药的肺炎支原体感染具有一定的挑战性。由于阿奇霉素具有抗炎症的特性,用其治疗一些耐药菌株感染仍然是有效的。能够替代大环内酯类药物治疗肺炎支原体感染的抗生素包括喹诺酮类药物(环丙沙星或左氧氟沙星)、四环素类药物(多西环素或米诺环素)或甘氨酰环素类-替加环素。然而,考虑到对儿童的副作用,在儿科患者中需要慎重使用喹诺酮类或四环素类药物。最近的一项研究表明,莫西沙星在治疗儿童严重难治性肺炎支原体肺炎方面具有较好的可行性和安全性[56]。日本指南建议将托氟沙星作为对大环内酯类药物治疗无效的儿科病例的次选药物[47]。2023年的中国治疗小儿肺炎支原体肺炎指南也建议托氟沙星作为替代药物,并且将16元环大环内酯吉他霉素列为首选药物之一[48]。将吉他霉素引入肺炎支原体治疗可能是基于临床经验,因为关于该药物治疗肺炎支原体感染的临床有效性和安全性尚缺乏公开信息,目前仅在一项体外研究中发现了支持性数据[49]。在对另一种16元环大环内酯麦迪霉素的体外研究中也观察到了类似结果,其对大环内酯类药物耐药的肺炎支原体菌株显示出更低的最低抑制浓度[50]。麦迪霉素对肺炎支原体耐药性的诱导能力较差,而对16元环大环内酯类药物耐药的突变菌株对14元环和15元环大环内酯类药物仍然保持敏感,这表明麦迪霉素有可能可以被用作治疗肺炎支原体感染的首选替代药物[51]。值得注意的是,考虑到肺炎支原体的高耐药突变率,在应用任何新的抗生素治疗后都有可能导致新的耐药性出现。临床上对肺炎支原体感染的治疗需要用耐药检测结果来指导治疗方案的制定。
对肺炎支原体感染的治疗,除了使用抗菌药物,还可以使用皮质类固醇等来缓解症状。2023年中国治疗小儿肺炎支原体肺炎指南还推荐使用中药治疗[57]。最近一项研究系统回顾了小儿清热咳喘联合阿奇霉素治疗儿童肺炎支原体感染的临床有效性和安全性的随机对照试验,并证实该联合用药比单独使用阿奇霉素治疗效果更好[58]。
结论:肺炎支原体独特的生理和致病特征导致了其暴发流行的特殊模式,并为其感染的诊断和治疗带来困难。目前面临的挑战是,在某些地区,特别是东亚,大环内酯类药物耐药病例的持续高流行率。为监测后新冠肺炎疫情时代的新的肺炎支原体暴发流行,建立和维护一个有效的监测系统非常重要。为控制当前和未来的肺炎支原体暴发流行,至关重要是在肺炎支原体流行年份和高发季节采取必要的预防和控制措施,以及恰当的诊断方法和治疗策略。
参考文献
Waites KB, Talkington DF. Mycoplasma pneumoniae and its role as a human pathogen. Clin Microbiol Rev. 2004;17(4):697-728. https://doi.org/10.1128/CMR.17.4.697-728.2004.
Yus E, Maier T, Michalodimitrakis K, van Noort V, Yamada T, Chen WH, et al. Impact of genome reduction on bacterial metabolism and its regulation. Science. 2009; 326(5957): 1263-8. https://doi.org/10.1126/science.1177263
Waites KB, Xiao L, Liu Y, Balish MF, Atkinson TP. Mycoplasma pneumoniae from the respiratory tract and beyond. Clin Microbiol Rev. 2017; 30(3): 747-809. https://doi.org/10. 1128/CMR.00114-16
Kannan TR, baseman JB. ADP-ribosylating and vacuolating cytotoxin of Mycoplasma pneumoniae represents unique virulence determinant among bacterial pathogens. Proc Natl Acad Sci U S A. 2006; 103(17): 6724-9. https://doi.org/10.1073/ pnas.0510644103 .
Hames C, Halbedel S, Hoppert M, Frey J, Stülke J. Glycerol metabolism is important for cytotoxicity of Mycoplasma pneumoniae. J Bacteriol. 2009; 191(3): 747-53. https://doi.org/ 10.1128/JB.01103-08 .
Großhennig S, Ischebeck T, Gibhardt J, Busse J, Feussner I, Stülke J. Hydrogen sulffde is a novel potential virulence factor of Mycoplasma pneumoniae: characterization of the unusual cysteine desulfurase/desulfhydrase HapE. Mol Microbiol. 2016; 100(1): 42-54. https://doi.org/10.1111/mmi.13300
Gan T, Yu J, He J. miRNA, lncRNA and circRNA: targeted molecules with therapeutic promises in Mycoplasma pneumoniae infection. Arch Microbiol. 2023; 205(8): 293. https:// doi.org/10.1007/s00203-023-03636-3.
Dumke R, Schurwanz N, Jacobs E. Characterisation of subtype-and variant-speciffc antigen regions of the P1 adhesin of Mycoplasma pneumoniae. Int J Med Microbiol. 2008; 298 (5-6): 483-91. https://doi.org/10.1016/j.ijmm.2007.06.002
Yamamoto T, Kida Y, Sakamoto Y, Kuwano K. Mpn491, a secreted nuclease of Mycoplasma pneumoniae, plays a critical role in evading killing by neutrophil extracellular traps. Cell Microbiol. 2017; 19(3): e12666. https://doi.org/10.1111/cmi. 12666
Chen LS, Li C, You XX, Lin YW, Wu YM. The mpn668 gene of Mycoplasma pneumoniae encodes a novel organic hydroperoxide resistance protein.Int J Med Microbiol. 2018; 308(7): 776-83. https://doi.org/10.1016/j.ijmm.2018.04.006
-Yu Y, Wang J, Han R, Wang L, Zhang L, Zhang AY, et al. Mycoplasma hyopneumoniae evades complement activation by binding to factor H via elongation factor thermo unstable (EF-Tu). Virulence. 2020; 11(1): 1059-74. https://doi.org/10. 1080/21505594.2020.1806664
-Blötz C, Singh N, Dumke R, Stülke J. Characterization of an immunoglobulin binding protein (IbpM) from Mycoplasma pneumoniae. Front Microbiol. 2020; 11: 685. https://doi.org/10. 3389/fmicb.2020.00685
Dallo SF, baseman JB. Intracellular DNA replication and long-term survival of pathogenic mycoplasmas. Microb Pathog. 2000; 29(5): 301-9. https://doi.org/10.1006/mpat.2000.0395
Simmons WL, Daubenspeck JM, Osborne JD, Balish MF, Waites KB, Dybvig K. Type 1 and type 2 strains of Mycoplasma pneumoniae form different biofflms. Microbiology. 2013; 159(Pt 4): 737-47. https://doi.org/10.1099/mic.0.064782-0
Yan C, Sun H, Zhao H. Latestsurveillance data on Mycoplasma pneumoniae infections in children, suggesting a new epidemic occurring in Beijing. J Clin Microbiol. 2016; 54(5): 1400-1. https://doi.org/10.1128/jcm.00184-16.
花盛浩, 邵雪君, 何萍, 等. 2007~2013年苏州地区住院儿童肺炎支原体感染流行特点[J]. 国际检验医学杂. 2015; 18: 2638-2639, 2642. 2015. DOI:10.3969/j.issn.1673-4130.2015.18.009
Wang X, Li M, Luo M, Luo Q, Kang L, Xie H, et al. Mycoplasma pneumoniae triggers pneumonia epidemic in autumn andwinter in Beijing: a multicentre, population-based epidemiological study between 2015 and 2020. Emerg Microb Infect. 2022; 11(1): 1508-17. https://doi.org/10.1080/22221751.2022.2078228
吴移谋,邵国青. 支原体学. 第三版,北京: 人民卫生出版社; 2022.
Cheng Y, Cheng Y, Dai S, Hou D, Ge M, Zhang Y, et al. The prevalence of Mycoplasma pneumoniae among children in Beijing before and during the COVID-19 pandemic. Front Cell Infect Microbiol. 2022; 12: 854505. https://doi.org/10. 3389/fcimb.2022.854505
Kenri T, Yamazaki T, Ohya H, Jinnai M, Oda Y, Asai S, et al. Genotyping of Mycoplasma pneumoniae strains isolated in Japan during 2019 and 2020: spread of p1 gene type 2c and 2j variant strains. Front Microbiol. 2023; 14: 1202357. https://doi. org/10.3389/fmicb.2023.1202357.
Meyer Sauteur PM, Beeton ML, Uldum SA, Bossuyt N, Vermeulen M, Loens K, et al. Mycoplasma pneumoniae detections before and during the COVID‐19 pandemic: results of a global survey, 2017 to 2021. Eur Commun Dis Bull. 2022;27(19): 2100746. https://doi.org/10.2807/1560-7917.ES.2022.27.19. 2100746.
Xu K. Pediatricians warn of mycoplasma pneumonia outbreak, with infections surging among children. In: Global times. People's daily. Beijing; 2023. https://www.globaltimes. cn/page/202310/1299756.shtml
Jain S, Williams DJ, Arnold SR, Ampofo K, Bramley AM, Reed C, et al. Community-acquired pneumonia requiring hospitalization among U.S. children. N Engl J Med. 2015; 372(9): 835-45. https://doi.org/10.1056/NEJMoa1405870
Waites KB, Ratliff A, Crabb DM, Xiao L, Qin X, Selvarangan R, et al. Macrolide-resistant Mycoplasma pneumoniae in the United States as determined from a national surveillance program. J Clin Microbiol. 2019; 57(11): e00968-1019. https:// doi.org/10.1128/JCM.00968-19.
Liu J, Zhao F, Lu J, Xu H, Liu H, Tang X, et al. High Mycoplasma pneumoniae loads and persistent long-term Mycoplasma pneumoniae DNA in lower airway associated with severity of pediatric Mycoplasma pneumoniae pneumonia. BMC Infect Dis. 2019; 19(1): 1045. https://doi.org/10.1186/s12879-019-4667‐y.
Spuesens EB, Fraaij PL,Visser EG, Hoogenboezem T, Hop WC, van Adrichem LN, et al. Carriage of Mycoplasma pneumoniae in the upper respiratory tract of symptomatic and asymptomatic children: an observational study. PLoS Med. 2013; 10(5): e1001444. https://doi.org/10.1371/journal.pmed.1001444
Tong L, Huang S, Zheng C, Zhang Y, Chen Z. Refractory Mycoplasma pneumoniae pneumonia in children: early recognition and management. J Clin Med. 2022; 11(10): 2824. https://doi.org/10.3390/jcm11102824
Loens K, van Heirstraeten L, Malhotra‐Kumar S, Goossens H, Ieven M. Optimal sampling sites and methods for detection of pathogens possibly causing community-acquired lower respiratory tract infections. J Clin Microbiol. 2009; 47(1): 21-31. https://doi.org/10.1128/JCM.02037-08
Cunha BA. The clinical diagnosis of Mycoplasma pneumoniae: the diagnostic importance of highly elevated serum cold agglutinins. Eur J Clin Microbiol Infect Dis. 2008; 27(10): 1017-9. https://doi.org/10.1007/s10096-008-0526-2
Loens K,Ieven M, Ursi D, Beck T, Overdijk M, Sillekens P, et al. Detection of Mycoplasma pneumoniae by real‐time nucleic acid sequence‐based ampliffcation. J Clin Microbiol. 2003; 41(9): 4448-50. https://doi.org/10.1128/JCM.41.9.4448-4450.2003
Li W, Fang YH, Shen HQ, Yang DH, Shu Q, Shang SQ. evaluation of a real-time method of simultaneous ampliffcation and testing in diagnosis of Mycoplasma pneumoniae infection in children with pneumonia. PLoS One. 2017; 12(5): e0177842. https://doi.org/10.1371/journal.pone.0177842
Lin R, Xing Z, Liu X, Chai Q, Xin Z, Huang M, et al. Performance of targeted next‐generation sequencing in the detection ofrespiratory pathogens and antimicrobial resistance genes for children. J Med Microbiol. 2023; 72(11). https://doi.org/10. 1099/jmm.0.001771
Wang Y, Yu X, Liu F, Tian X, Quan S, Jiao A, et al. Respiratory microbiota imbalance in children with Mycoplasma pneumoniae pneumonia. Emerg Microb Infect. 2023; 12(1): 2202272. https://doi.org/10.1080/22221751.2023.2202272
Lu Z, Dai W, Liu Y, Zhou Q, Wang H, Li D, et al. The alteration of nasopharyngeal and oropharyngeal microbiota in children with MPP and non-MPP. Genes. 2017; 8(12): 380. https://doi.org/10.3390/genes8120380
Chen J, Ji F, Yin Y, Yuan S. Time to Mycoplasma pneumoniae RNA clearance for wheezy vs. non-wheezy young children with community‐acquired pneumonia. J Trop Pediatr. 2021; 67(1): fmaa109. https://doi.org/10.1093/tropej/fmaa109
Waites K, Bade D, Bébéar C, Brown SD, Davidson MK, Duffy L, et al. Methods for antimicrobial susceptibility testing for human Mycoplasmas; Approved Guideline. Wayne: Clinical and Laboratory Standards Institute; 2011.
Leal SM, Jr, Totten AH, Xiao L, Crabb DM, Ratliff A, Duffy LB, et al. evaluation of commercial molecular diagnostic methods for detection and determination of macrolide resistance in Mycoplasma pneumoniae.JClin Microbiol. 2020; 58(6): e00242-320. https://doi.org/10.1128/JCM.00242-20
Okazaki N, Narita M, Yamada S, Izumikawa K, Umetsu M, Kenri T, et al. Characteristics of macrolide‐resistant Mycoplasma pneumoniae strains isolated from patients and induced with erythromycin in vitro. Microbiol Immunol. 2001; 45(8): 617-20. https://doi.org/10.1111/j.1348-0421.2001.tb01293.x
Wang G, Wu P, Tang R, Zhang W. Global prevalence of resistance to macrolides in Mycoplasma pneumoniae: a systematic review and meta‐analysis. J Antimicrob Chemother. 2022; 77(9): 2353-63. https://doi.org/10.1093/jac/dkac170
Xiao L, Ptacek T, Osborne JD, Crabb DM, Simmons WL, Lefkowitz EJ, et al. Comparative genome analysis of Mycoplasma pneumoniae. BMC Genom. 2015; 16(1): 610. https://doi.org/10.1186/s12864-015-1801-0
Sasaki T, Kenri T, Okazaki N, Iseki M, Yamashita R, Shintani M, et al. Epidemiological study of Mycoplasma pneumoniae infections in Japan based on PCR-restriction fragment length polymorphism of the P1 cytadhesin gene. J Clin Microbiol. 1996; 34(2): 447-9. https://doi.org/10.1128/jcm.34.2.447-449. 1996.
Dégrange S, Cazanave C, Charron A, Renaudin H, Bébéar C, Bébéar CM. Development of multiple-locus variable‐number tandem‐repeat analysis for molecular typing of Mycoplasma pneumoniae. J Clin Microbiol. 2009; 47(4): 914-23. https://doi. org/10.1128/JCM.01935-08 .
Touati A, Blouin Y, Sirand‐Pugnet P, Renaudin H, Oishi T, Vergnaud G, et al. Molecular epidemiology of Mycoplasma pneumoniae: genotyping using single nucleotide polymorphisms and SNaPshot technology. J Clin Microbiol. 2015; 53(10): 3182-94. https://doi.org/10.1128/jcm.01156-15
Sun H, Xue G, Yan C, Li S, Cao L, Yuan Y, et al. Multiple-locus variable‐number tandem-repeat analysis of Mycoplasma pneumoniae clinical specimens and proposal for amendment of MLVA nomenclature. PLoS One. 2013; 8(5): e64607. https://doi.org/10.1371/journal.pone.0064607
Kenri T, Okazaki N, Yamazaki T, Narita M, Izumikawa K, Matsuoka M, et al. Genotyping analysis of Mycoplasma pneumoniae clinical strains in Japan between 1995 and 2005: type shift phenomenon of M. pneumoniae clinical strains. J Med Microbiol. 2008; 57(Pt 4): 469–75. https://doi.org/10.1099/ jmm.0.47634-0 .
Zhang XS, Zhao H, Vynnycky E, Chalker V. Positively interacting strains that co-circulate within a network structured population induce cycling epidemics of Mycoplasma pneumoniae. Sci Rep. 2019; 9(1): 541. https://doi.org/10.1038/ s41598-018-36325-z.
Zhao F, Liu J, Shi W, Huang F, Liu L, Zhao S, et al. Antimicrobial susceptibility and genotyping of Mycoplasma pneumoniae isolates in Beijing, China, from 2014 to 2016. Antimicrob Resist Infect Control. 2019; 8(1): 18. https://doi. org/10.1186/s13756-019-0469-7 .
Jiang FC, Wang RF, Chen P, Dong LY, Wang X, Song Q, et al. Genotype and mutation patterns of macrolide resistance genes of Mycoplasma pneumoniae from children with pneumonia in Qingdao, China, in 2019. J Glob Antimicrob Resist. 2021; 27: 273-8. https://doi.org/10.1016/j.jgar.2021.10.003
Guo P, Mei S, Wang Y, Zheng X, Li L, Cheng Y. Molecular typing of Mycoplasma pneumoniae and its correlation with macrolide resistance in children in Henan of China. Indian J Med Microbiol. 2023; 46: 100435. https://doi.org/10.1016/j. ijmmb.2023.100435
Guo Z, Liu L, Gong J, Han N, He L, Wang W, et al. Molecular features and antimicrobial susceptibility of Mycoplasma pneumoniae isolates from paediatric inpatients in Weihai, China: characteristics of M. pneumoniae in Weihai. J Glob Antimicrob Resist. 2022; 28: 180-4. https://doi.org/10.1016/j. jgar.2022.01.002
Li L, Ma J, Guo P, Song X, Li M, Yu Z, et al. Molecular beacon based real-time PCR p1 gene genotyping, macrolide resistance mutation detection and clinical characteristics analysis of Mycoplasma pneumoniae infections in children. BMC Infect Dis. 2022; 22(1): 724. https://doi.org/10.1186/s12879-022-07715-6
Jiang TT, Sun L, Wang TY, Qi H, Tang H, Wang YC, et al. The clinical signiffcance of macrolide resistance in pediatric Mycoplasma pneumoniae infection during COVID-19 pandemic. Front Cell Infect Microbiol. 2023; 13: 1181402. https://doi.org/10.3389/fcimb.2023.1181402.
Yan C, Xue G, Zhao H, Feng Y, Li S, Cui J, et al. Molecular and clinical characteristics of severe Mycoplasma pneumoniae pneumonia in children. Pediatr Pulmonol. 2019; 54(7): 1012-21. https://doi.org/10.1002/ppul.24327
Watkins LKF, Olson D, Diaz MH, Lin X, Demirjian A, Benitez AJ, et al. Epidemiology and molecular characteristics of Mycoplasma pneumoniae during an outbreak of M. pneumoniae-associated stevens-Johnson syndrome. Pediatr Infect Dis J. 2017; 36(6): 564-71. https://doi.org/10.1097/INF. 0000000000001476
Techasaensiri C, Tagliabue C, Cagle M, Iranpour P, Katz K, Kannan TR, et al. Variation in colonization, ADP‐ribosylating and vacuolating cytotoxin, and pulmonary disease severity among Mycoplasma pneumoniae strains. Am J Respir Crit Care Med. 2010; 182(6): 797-804. https://doi.org/10.1164/ rccm.201001-0080OC.
He YS, Yang M, Liu G, Ji J, Qian SY. Safety study of moxiffoxacin in children with severe refractory Mycoplasma pneumoniae pneumonia. Pediatr Pulmonol. 2023;58(7):2017– 24. https://doi.org/10.1002/ppul.26426
NHCotRsRo C. Guidelines for the diagnosis and treatment of Mycoplasma pneumoniae pneumonia in children (2023 edition). Inter J Epidemiol Infect Dis. 2023; 50(5): 79-85. https://doi.org/10.3760/cma.j.cn331340-20230217-00023.
Peng Y, Chen Z, Li Y, Lu Q, Li H, Han Y, et al. Combined therapy of Xiaoer Feire Kechuan oral liquid and azithromycin for Mycoplasma pneumoniae pneumonia in children: a systematic review & meta-analysis. Phytomedicine. 2022; 96: 153899. https://doi.org/10.1016/j.phymed.2021.153899
Wang Y, Xu B, Wu X, Yin Q, Wang Y, Li J, et al. Increased macrolide resistance rate of M3562 Mycoplasma pneumoniae correlated with macrolide usage and genotype shifting. Front Cell Infect Microbiol. 2021; 11: 675466. https://doi.org/10. 3389/fcimb.2021.675466
Hung HM, Chuang CH, Chen YY, Liao WC, Li SW, Chang IY, et al. Clonal spread of macrolide‐resistant Mycoplasma pneumoniae sequence type‐3 and type-17 with recombination on non-P1 adhesin among children in Taiwan. Clin Microbiol Infect. 2021; 27(8): 1169.e1-1169.e6. https://doi.org/10.1016/j. cmi.2020.09.035
Wang N, Zhang H, Yin Y, Xu X, Xiao L, Liu Y. Antimicrobial susceptibility proffles and genetic characteristics of Mycoplasma pneumoniae in Shanghai, China, from 2017 to 2019. Infect Drug Resist. 2022; 15: 4443-52. https://doi.org/10.2147/IDR.S370126.
Kenri T, Suzuki M, Sekizuka T, Ohya H, Oda Y, Yamazaki T, et al. Periodic genotype shifts in clinically prevalent Mycoplasma pneumoniae strains in Japan. Front Cell Infect Microbiol. 2020; 10: 385. https://doi.org/10.3389/fcimb.2020.00385
Lee JK, Choi YY, Sohn YJ, Kim KM, Kim YK, Han MS, et al. Persistent high macrolide resistance rate and increase of macrolide-resistant ST14 strains among Mycoplasma pneumoniae in South Korea, 2019-2020. J Microbiol Immunol Infect. 2022; 55(5): 910-6. https://doi.org/10.1016/j.jmii.2021. 07.011.
致谢:感谢汤一苇教授参与本文撰写。




