微滴式数字PCR技术在 甲型H1N1流感病毒耐药基因H275Y突变检测中的应用

谢国良,硕士,主管技师,于浙江大学医学院附属第一医院检验科分子诊断实验室、浙江省临床体外诊断技术研究重点实验室从事病原的临床分子诊断及技术研发工作10年。专业特长:病原分子诊断新技术的研究开发;新型分子诊断技术的临床应用等。获得授权国家发明专利8项,授权美国发明专利1项,以主要参与人获2017年教育部技术发明奖二等奖,主持省厅级项目3项,参与国家科技重大专项、国家重点研发计划、国家自然基金等多个科研项目。
【摘要】目的 建立起一套可用于检测临床甲型H1N1流感病毒样本H275Y突变的微滴式数字PCR(dd-PCR)检测方法。方法 设计甲型H1N1流感病毒耐药基因H275Y突变特异性的引物及TaqMan探针,建立和优化实时荧光PCR反应体系及dd-PCR反应体系,评价其灵敏度、特异性等,并利用实验室保存的甲型H1N1流感病毒临床样本(4例H275Y基因突变标本和4例H275未突变标本)对该方法进行验证。结果 建立的甲型H1N1流感病毒耐药基因H275Y突变dd-PCR检测体系具有与qRT-PCR相当的灵敏度和特异性,能够实现对样本中耐药基因H275Y突变的精准定量检测。结论 初步建立基于dd-PCR的奥司他韦耐药基因H275Y突变的检测方法,可应用于临床样本中甲型H1N1流感病毒耐药基因H275Y突变位点的检测,有望用于流感病毒感染后的耐奥司他韦用药管理。
【关键词】微滴式数字PCR;H1N1流感病毒;H275Y突变
流行性感冒作为常见的呼吸道传染病是由流行性感冒病毒(简称流感病毒)引起的,其中甲型H1N1流感病毒是较为常见且对人健康威胁较大的一类流感病毒。临床上主要有两种抗流感病毒药物,即M2离子通道阻滞剂和神经氨酸酶抑制剂(NAIs)[1]。NAIs是WHO推荐的针对流感病毒的最常见抗病毒药物,如奥司他韦和扎那米韦等[2]。然而近年来,随着抗病毒药物的广泛应用,加之流感病毒容易发生变异,NAIs导致的耐药毒株也逐渐出现。甲型H1N1流感病毒中最常见的NAIs突变位点为H275Y,因此对流感病毒H275Y突变的检测就显得尤为重要。
数字PCR是最新一代的核酸检测和定量方法,其主要工作原理为:借助微液滴或微孔将反应体系分隔为有限数量的独立微体系;对独立微体系进行PCR扩增;检测每个微体系的扩增信号并计数;利用泊松分布公式计算总体系中的模板拷贝数[3, 4]。该检测方法具有灵敏度高、准确性强、以及对检测靶标的绝对定量等优势。为此,本研究初步建立了基于微滴式数字PCR的奥司他韦耐药基因H275Y突变位点的检测方法。
一. 材料与方法
1. 样本来源:收集浙江大学医学院附属第一医院患者的呼吸道痰液或咽拭子标本,其中包括本实验室保存的4例甲型H1N1流感病毒H275Y基因突变样本和4例甲型H1N1流感病毒H275未突变标本(均经测序确认)。其他呼吸道病毒如甲型流感病毒H3N2,乙型流感病毒,副流感病毒I-IV型,偏肺病毒,鼻病毒,呼吸道病毒,腺病毒等核酸样本均由本实验室保存。H275Y-NA和H275-NA标准品为合成的阳性质粒样品。
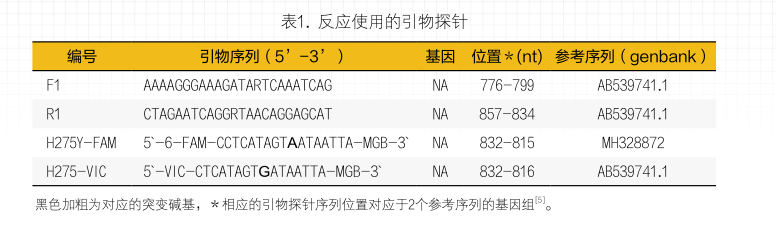
2. H275位点突变引物探针的设计:从NCBI基因库下载涵盖国内外H1N1流感病毒NA基因H275位点的多条基因序列,利用DNAman软件对其进行同源性比较,确定病毒基因组的保守区。使用Primer Express 3.0软件在其保守区设计高度特异性的引物与TaqMan探针,引物和探针序列均通过BLAST验证,具有较好特异性。根据引物设计优化原则,优化引物的Tm、GC含量等参数。引物和探针委托上海生工公司合成,反应使用的引物探针序列见表1。
3. 耐药基因H275Y突变qRT-PCR方法的建立:根据病毒RNA提取试剂盒说明书操作提取病毒的核酸样本,取5ul已抽提的待测样品核酸作为模板。反应总体积为25μl,其中qRT-PCR反应液12.5μl,酶混合液1μl,引物探针混合液(20μmol/L,包含两组特异性引物和相应的两种荧光探针)共3μl,模板5μl,加水补足至25μl。在ABI7500荧光定量PCR仪上进行检测。反应参数为:反转录50℃,30min;95℃,5min热启动;然后95℃,15s;55℃,45s;在55℃进行双重荧光检测,共进行40个循环。验证耐药基因H275Y突变的双重实时荧光qRT-PCR的特异性和敏感性等。
4. 耐药基因H275Y突变dd-PCR方法的建立:dd-PCR试剂采用北京新羿生物一步法probe RT-ddPCR试剂盒,反应总体积为30μl,其中预混液A 9μl,预混液B 3μl,引物探针混合液(20μmol/L,包含两组特异性引物和相应的两种荧光探针)共5.5μl,模板μ,加水补足至30μl。之后将反应液在样本制备仪中生成液滴后,通过PCR仪进行扩增。反应参数为:反转录50℃,15min;95℃,10min热启动,然后94℃,30s;55℃,40s,共进行40个循环。扩增结束后,通过生物芯片阅读仪进行液滴检测。
二、结果
1. H275Y-NA突变引物探针的qRT-PCR验证:优化H275Y突变的双重实时荧光qRT-PCR反应体系,并对该方法的检测性能进行验证。其中利用已知浓度的阳性质粒样品进行灵敏度实验,H275位点双靶标灵敏度均可达103copies/ml。与甲型流感病毒H3N2、H7N9,乙型流感病毒,副流感病毒I-IV型,偏肺病毒,鼻病毒,呼吸道病毒,腺病毒等阳性核酸均无非特异性扩增,显示高度特异性。用已知浓度的阳性标准品建立的标准曲线具备较好的线性,如图1所示R2分别为0.99和1。

图1. qRT-PCR检测H275位点的标准曲线
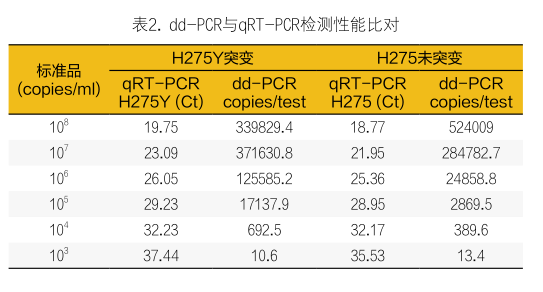
2. dd-PCR与qRT-PCR检测H275Y-NA突变引物探针的检测性能比对:将已知浓度的阳性质粒梯度稀释后作为模板,同时用qRT-PCR和dd-PCR方法进行检测性能比对,结果见表2。可见dd-PCR具备与qRT-PCR相当的检测性能,在质粒浓度在103copies/ml的时候,仍能定量检出。
3. dd-PCR检测临床样本H275Y-NA突变:对本实验室保存的4例甲型H1N1流感病毒H275Y基因突变样本和4例甲型H1N1流感病毒H275未突变标本(均经测序确认)进行dd-PCR检测,dd-PCR检测检测结果与qRT-PCR及测序结果均一致,符合率均为100%。部分检测结果如图2所示。

图2. dd-PCR检测甲型H1N1流感病毒临床样本(其中A、B为H275Y基因突变样本,C、D为H275基因未突变样本)
三、讨论
随着抗病毒药物的广泛应用,流感病毒耐药毒株也逐渐出现,病毒耐药成为当前引起重视的公共卫生问题。据报道,2009-2010年分离的甲型H1N1流感病毒几乎所有毒株对NAIs敏感[6]。2016-2017年中国大陆最常见的NAIs位点为H275Y,奥司他韦和扎那米韦耐药率为0.7%[7]。我们课题组对2017-2018年期间的甲型H1N1流感病毒测序发现,约2%(7/300)的甲型H1N1流感病毒发生H275Y-NA突变,其中重症患者比例高达14.7%(5/34)。尽管NAIs仍然是治疗和预防流感病毒感染的推荐抗病毒药物,但对流感病毒耐药基因突变的检测也越来越重要[8]。
目前,流感病毒NAIs耐药性检测方法基本上可以分为两类:表型分析和基因型分析。表型分析法是流感病毒耐药检测的“金标准”,但病毒在细胞中传代可能会导致原始临床标本中病毒耐药株的比例改变或发生新的突变,另外,该方法检测时间长且生物安全要求高。基因型分析主要通过实时荧光定量PCR或测序实现[9]。实时荧光定量PCR需要通过设置标准曲线实现定量,且受样本内抑制物质影响较大,而通过测序和后续生物信息软件分析也能获得耐药相关位点和比例[10],但检测周期较长,且成本相对较高。
数字PCR是继实时定量PCR之后新兴发展起来的一种绝对定量分析技术。与常用的实时荧光定量PCR(qRT-PCR)方法相比,其不需要标准曲线进行绝对定量是dd-PCR的最大优势之一[4]。本文初步建立了基于数字PCR的奥司他韦耐药基因H275Y-NA突变的检测方法,可以直接用于H275位点突变的载量监测,有望用于流感病毒感染后的耐奥司他韦用药指导,对流感病毒的耐药性监测也具有非常重要的意义。作者在同步建立qRT-PCR与dd-PCR方法的过程中发现,虽然二者均是基于PCR原理,但qRT-PCR优化后的条件并不一定是dd-PCR的最佳条件。研究者需深入优化引物探针比例、反应温度、模板浓度等条件,才能发挥数字PCR精准绝对定量的最大优势。另外,目前的数字PCR检测平台仍普遍存在检测过程繁琐,检测时间相对较长的劣势,相信随着技术的改进和发展,数字PCR在病原体检测这一领域会发挥更为重要的作用。
参考文献
Lampejo T. Influenza and antiviral resistance: an overview[J]. Eur J Clin Microbiol Infect Dis, 2020, 39(7): 1201-1208.
Lackenby A, Besselaar T G, Daniels R S, et al. Global update on the susceptibility of human influenza viruses to neuraminidase inhibitors and status of novel antivirals, 2016-2017[J]. Antiviral Res, 2018, 157: 38-46.
Hindson B J, Ness K D, Masquelier D A, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Anal Chem,2011, 83(22): 8604-8610.
Zhang K, Lin G, Li J. Quantitative nucleic acid amplification by digital PCR for clinical viral diagnostics[J]. Clin Chem Lab Med, 2016, 54(9): 1427-1433.
Escuret V, Cornu C, Boutitie F, et al. Oseltamivir-zanamivir bitherapy compared to oseltamivir monotherapy in the treatment of pandemic 2009 influenza A(H1N1) virus infections[J]. Antiviral Res, 2012, 96(2): 130-137.
Gubareva L V, Trujillo A A, Okomo-Adhiambo M, et al. Comprehensive assessment of 2009 pandemic influenza A (H1N1) virus drug susceptibility in vitro[J]. Antivir Ther, 2010, 15(8): 1151-1159.
Huang W, Cheng Y, Li X, et al. Neuraminidase inhibitor susceptibility profile of human influenza viruses during the 2016-2017 influenza season in Mainland China[J]. J Infect Chemother, 2018, 24(9): 729-733.
Lackenby A, Besselaar T G, Daniels R S, et al. Global update on the susceptibility of human influenza viruses to neuraminidase inhibitors and status of novel antivirals, 2016-2017[J]. Antiviral Res, 2018, 157: 38-46.
Nguyen H T, Fry A M, Gubareva L V. Neuraminidase inhibitor resistance in influenza viruses and laboratory testing methods[J]. Antivir Ther, 2012, 17(1 Pt B): 159-173.
Pichon M, Gaymard A, Josset L, et al. Characterization of oseltamivir-resistant influenza virus populations in immunosuppressed patients using digital-droplet PCR: Comparison with qPCR and next generation sequencing analysis[J]. Antiviral Res, 2017, 145: 160-167.




